肠道定植产KPC酶植生拉乌尔菌的耐药基因环境分析
葛超荣
浙江大学医学院附属第一医院,杭州310003
肠道定植产KPC酶植生拉乌尔菌的耐药基因环境分析
葛超荣
浙江大学医学院附属第一医院,杭州310003
碳青霉烯酶编码基因的传播是临床抗菌治疗面临的严重威胁之一。采用PCR、Southern杂交、测序比对等方法筛查和研究了碳青霉烯类抗菌药物的相关耐药基因。首次在国内发现了产KPC酸的植生拉乌尔菌两株。分离到的植生拉乌尔菌对被测试的19种常用抗菌药物中的16种耐药,只对多粘菌素B、粘菌素和米诺环素敏感。它们均产KPC⁃2型碳青霉烯酶,其编码基因blaKPC⁃2位于质粒上,两侧(包括blaKPC⁃2)长度为7 498 bp的DNA序列所包含的结构从上游至下游分别为Tn3⁃transposase,Tn3⁃resolvase,IS Kpn8,blaKPC⁃2和IS Kpn6⁃like元件。
植生拉乌尔菌;碳青霉烯酶;质粒
碳青霉烯类抗菌药物是目前所使用的抗菌药物中针对革兰阴性菌抗菌谱最广、抗菌活性最强的一类抗菌药物,尤其是对产超广谱β⁃内酰胺酶(extended spectrumβ⁃lactamases,ESBLs)和 β内酰胺酶(AmpC)的肠杆菌科细菌[1]。但随着该类抗菌药物在临床应用的增加,对此类抗菌药物的耐药现象也日趋增多。革兰阴性菌对碳青霉烯类抗菌药物耐药的机制主要有以下几种:①外膜孔蛋白减少或缺失;②碳青霉烯类抗菌药物作用靶位,主要为青霉素结合蛋白的改变[2];③产碳青霉烯酶,主要包括Ambler分类中A类、B类及D类的某些OXA酶[3],其中,肠杆菌科细菌耐碳青霉烯类抗菌主要由于其产生A类酶中的KPC型碳青霉烯酶[4]。
自从2001年自肺炎克雷伯菌中发现KPC酶以后[5],现已在很多种属的细菌中发现该酶[6~9],这些产KPC酶的菌株主要有肠杆菌科细菌,包括大肠埃希氏菌、克雷伯菌属细菌、肠杆菌属细菌、枸橼酸杆菌属细菌、沙门菌属细菌、沙雷氏菌属细菌以及奇异变形杆菌等[10~13],在其他细菌中也有零星报道,例如铜绿假单胞菌、恶臭假单胞菌和鲍曼不动杆菌等[14~16]。迄今只有于2009年在美国发现过2株产KPC的植生拉乌尔菌[17],本文是国内首次在植生拉乌尔菌中鉴定到KPC酶。
1 材料与方法
1.1 实验菌株
2009年8~10月期间,于浙江大学医学院附属第一医院重症监护室住院病人的粪便/直肠拭子标本中分离获得碳青霉烯类抗菌药物敏感性降低的革兰阴性临床菌株286株。标准菌株为大肠埃希氏菌Escherichia coli ATCC25922。
1.2 实验方法
1.2.1 blaKPC⁃2基因筛查 利用 blaKPC⁃2扩增引物[18]对286株临床菌株进行blaKPC-2基因PCR扩增。用煮沸法提取模板DNA(调成一定浓度的菌悬液煮沸10min,4 000 r/min离心10min,取上清-20℃保存备用)。PCR反应体系(50μL)为:模板1.0μL,10×PCR缓冲液 5.0μL,MgCl2(25 mmol/L)4.0μL,dNTPs(10 mmol/L)0.5μL,上下游引物(10μmol/L)各1.0μL,Taq酶(5 U/μL)0.3μL。PCR扩增程序为:94℃预变性5 min;94℃变性50 s,60℃退火30 s,72℃延伸50 s,共30个循环;72℃延伸10 min。扩增后用1%琼脂糖120 V电泳20 min。阳性标本的扩增产物纯化后送博赛生物公司测序,测序结果经http://blast.ncbi.nlm.nih.gov/Blast比对。
1.2.2 药物敏感性试验 用E⁃Test法测定阳性细菌对常见抗菌药物的最低抑菌浓度(minimum inhibitory concentration,MIC)。药敏判断标准主要参照CLSI 2013;头孢哌酮/舒巴坦的判断标准参照CLSI 2013头孢哌酮的折点标准[19];粘菌素和替加环素的药敏结果判断参照欧洲抗菌药物敏感性试验委员会推荐的标准(http://www.eucast.org/);多粘菌素B的判断标准参照粘菌素的药物敏感性标准。操作严格按照厂家说明书进行。
1.2.3 blaKPC⁃2基因定位 采用 Qiagen Plasmid Midi Kit(Qiagen公司,德国)抽提质粒。用Hin d III和Eco RI内切酶对提取到的质粒进行消化后判断质粒大小,酶切操作参照以上两种内切酶的使用说明书。采用Roche Dig Kit(Roche公司,美国),用blaKPC⁃2基因特异探针与质粒酶切产物进行Southern杂交,具体操作参照试剂盒说明书进行。接合和转化试验的具体操作方法参照文献[9,20]。
1.2.4 基因环境分析 用blaKPC⁃2基因引物对目的细菌基因组DNA进行基因扩增。扩增产物纯化后送博尚生物公司测序,测序结果经http://blast.ncbi.nlm.nih.gov/Blast比对,获得KPC的基因环境。
2 结果与分析
2.1 KPC阳性菌株筛选及药敏试验
通过PCR筛选及测序确认,286株碳青霉烯类抗生素敏感性下降的革兰阴性菌中有40株(占14%)携带有blaKPC⁃2基因,其中2株为植生拉乌尔菌,定名为RP1和RP2。经临床病历分析,这两株细菌均为肠道定植菌。
对分离得到的2株植生拉乌尔菌进行常见药物耐药性分析,发现这两株菌均为产ESBLs的泛耐药菌株,除对多粘菌素B、粘菌素敏感,对米诺环素中度敏感外,对其他16种被检抗菌药物均耐受。对三代、四代头孢菌素和抗菌活性最强的碳青霉烯类抗菌药物均耐受。替加环素是治疗碳青霉烯类抗菌药物耐药细菌的常用药物,这2株细菌也对此药物耐受(见表1)。
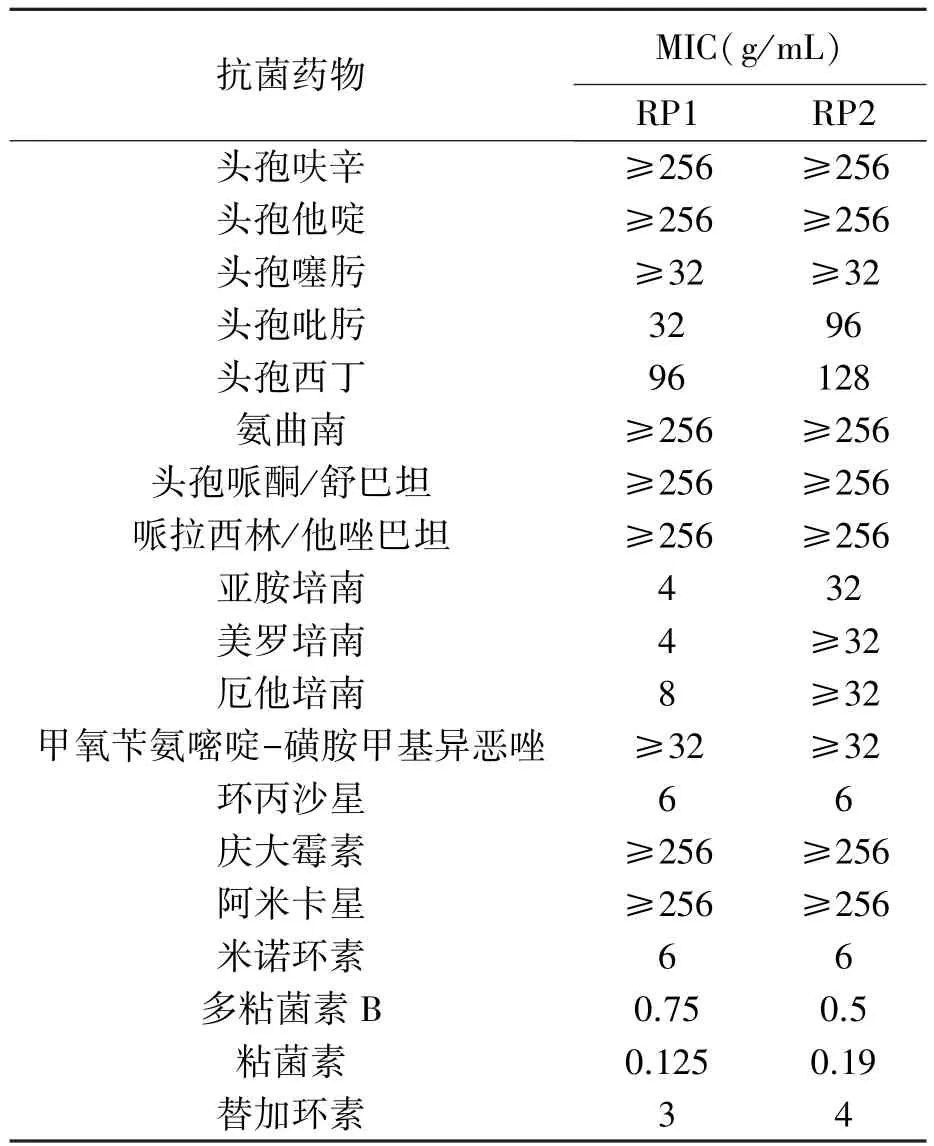
表1 产KPC植生拉乌尔菌对19种抗菌药物的MIC值Table 1 MIC values of 19 antibiotics for the KPC⁃producing Raoultella planticola.
2.2 基因定位
本文通过接合试验、转化试验及Southern杂交判断 blaKPC-2基因位于质粒还是染色体上。Southern杂交显示blaKPC⁃2基因特异探针能和提取自原始菌株和转化阳性克隆并经 Hin dⅢ和Eco RⅠ内切酶消化后的质粒条带杂交显色,表明该基因位于质粒上,结果见图1。采用接合试验得到了含blaKPC⁃2基因的接合子,转化试验也得到了含 blaKPC⁃2基因的阳性克隆,进一步证明了blaKPC⁃2基因位于质粒上。
2.3 基因环境研究
2株细菌均获得长度为7 498 bp的blaKPC⁃2基因及周围序列,其所包含的结构从上游至下游分别 为 Tn3⁃transposase、Tn3⁃resolvase、 IS Kpn8、blaKPC⁃2和 IS Kpn6⁃like元件,见图2。该结构以转座子Tn3为基础并整合了在美国、哥伦比亚及希腊等地广泛流行的被认为是blaKPC⁃2起源的转座子Tn4401的部分基因原件[21],和 Shen等[18]首次报道的在中国流行的含KPC酶编码基因的质粒pKP048中的blaKPC⁃2周围结构相一致。
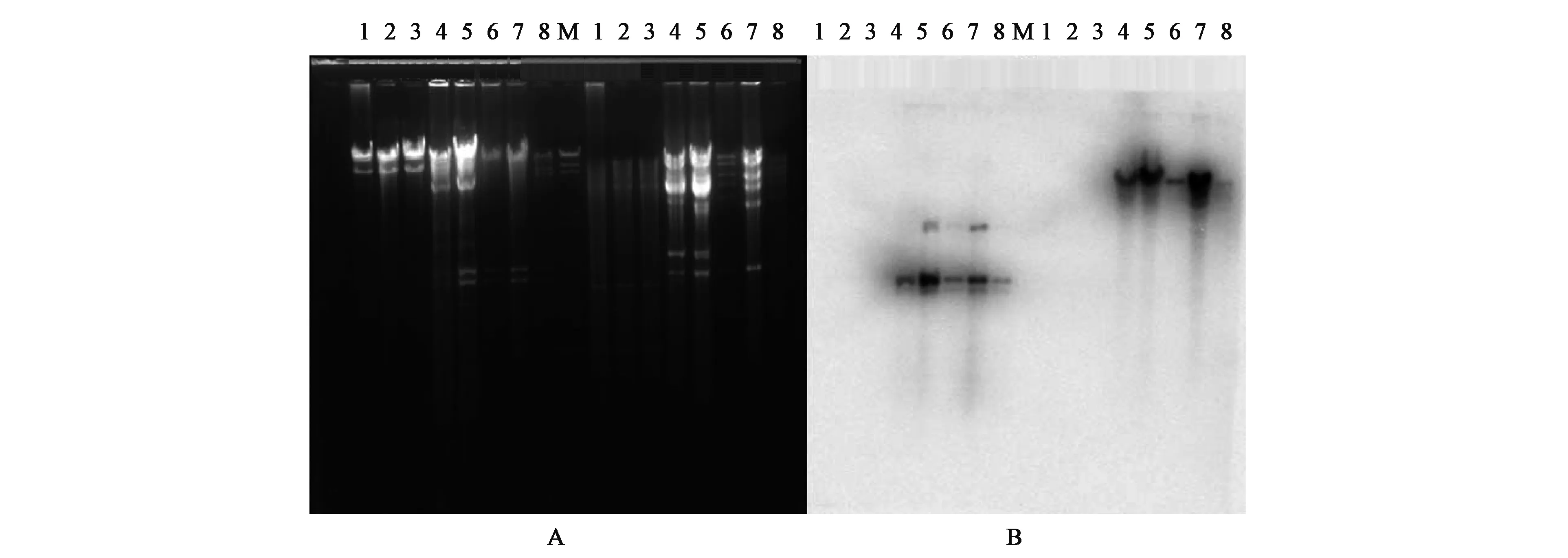
图1 blaKPC⁃2基因的定位Fig.1 Location of blaKPC⁃2gene.
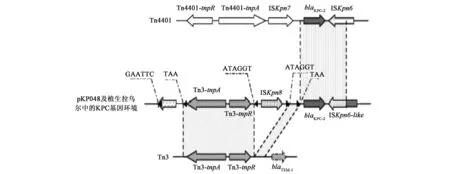
图2 携带blaKPC⁃2基因的转座子结构Fig.2 Structure of transposes harbouring blaKPC⁃2gene.
3 讨论
中国是碳青霉烯类KPC型耐药流行区,目前主要分布在浙江、上海、江苏、安徽及河南等9个省市[22]。浙江大学医学院是我国首次发现KPC酶的医院[9],因此我们对院感最易发生的ICU病房进行主动监测,留取了我院ICU病房住院病人的粪便/直肠拭子标本,筛查产KPC酶的革兰阴性菌,分离得到40株产KPC的菌株,其中产KPC的铜绿假单胞菌和植生拉乌尔菌都是首次在中国被发现[23]。拉乌尔菌属以前被认为是克雷伯菌属的成员,后来才从克雷伯菌属中独立出来。因此2株初次鉴定为克雷伯菌的阳性菌株,后来根据16S rRNA基因才被鉴定为拉乌尔菌。拉乌尔菌的致病性和感染所致的临床症状和克雷伯菌相似。
美国报道的2株植生拉乌尔菌分别分离自新泽西州和俄亥俄州。前者携带blaKPC⁃3基因,位于质粒携带的转座子Tn4401c上;后者携带blaKPC⁃2基因,位于染色体包含的转座子Tn4401b上[17]。本文发现的2株碳青霉烯类耐药的植生拉乌尔所产的KPC酶编码基因均为blaKPC⁃2,目前我国发现的所有KPC编码基因均为blaKPC⁃2。本研究结果显示这2株细菌携带的blaKPC⁃2的周围结构和中国流行的质粒pKP048所携带的blaKPC⁃2周围结构相一致。
本文分离的2株产KPC酶的植生拉乌尔菌均为多药耐受,对当前临床常用的抗菌药物均耐受,一旦感染此菌治疗非常棘手,因此监护室应对携带这2株细菌的病人进行隔离。这2株细菌均从粪便标本中分离,因此医护人员除了按院感规定进行操作外,还要注意对其大便处理程序进行严格控制以阻断此细菌的蔓延。
[1] Nordmann P,Cuzon G,Naas T.The real threat of Klebsiella pneumoniae carbapenemase⁃producing bacteria[J].Lancet Infect.Dis.,2009,9(4):228-236.
[2] Fernandez⁃Cuenca F,Martinez⁃Martinez L, Conejo M C,et al..Relationship between beta⁃lactamase production,outer membraneprotein and penicillin⁃binding protein profiles on the activity of carbapenems against clinical isolates of Acinetobacter baumannii[J].J.Antimicrob.Ch.,2003,51(3):565-574.
[3] Nordmann P,Poirel L.Emerging carbapenemases in Gram⁃negative aerobes[J].Clin.Microbiol.Infect.,2002,8(6):321-331.
[4] Deshpande L M, Rhomberg P R, Sader H S, et al..Emergence of serine carbapenemases(KPC and SME)among clinical strains of Enterobacteriaceae isolated in the United States Medical Centers:report from the MYSTIC Program(1999-2005)[J].Diagn.Micr.Infec.Dis.,2006,56(4):367-372.
[5] Yigit H,Queenan A M, Anderson G J, et al..Novel carbapenem⁃hydrolyzing beta⁃lactamase, KPC⁃1, from a carbapenem⁃resistant strain of Klebsiella pneumoniae[J].Antimicrob.Agents Ch.,2001,45(4):1151-1161.
[6] Naas T,Nordmann P,Vedel G,et al..Plasmid⁃mediated carbapenem⁃hydrolyzing beta⁃lactamase KPC in a Klebsiella pneumoniae isolate from France[J].Antimicrob.Agents Ch.,2005,49(10):4423-4424.
[7] Bratu S,Landman D,Haag R,et al.Rapid spread of carbapenem⁃resistant Klebsiella pneumoniae in New York City:a new threat to our antibiotic armamentarium[J].Arch.Inter.Med.,2005,165(12):1430-1435.
[8] Navon⁃Venezia S,Chmelnitsky I,Leavitt A,et al..Plasmid⁃mediated imipenem⁃hydrolyzing enzyme KPC⁃2 amongmultiple carbapenem⁃resistant Escherichia coli clones in Israel[J].Antimicrob.Agents Ch.,2006,50(9):3098-3101.
[9] Wei ZQ,Du X X,Yu Y S,et al..Plasmid⁃mediated KPC⁃2 in a Klebsiella pneumoniae isolate from China[J].Antimicrob.Agents Ch.,2007,51(2):763-765.
[10] Hossain A,Ferraro M J,Pino R M,et al..Plasmid⁃mediated carbapenem⁃hydrolyzing enzyme KPC⁃2 in an Enterobacter sp.[J].Antimicrob.Agents Ch.,2004,48(11):4438-4440.
[11] Yigit H,Queenan A M,Rasheed J K,et al..Carbapenem⁃resistant strain of Klebsiella oxytoca harboring carbapenem⁃hydrolyzing beta⁃lactamase KPC⁃2[J].Antimicrob.Agents Ch.,2003,47(12):3881-3889.
[12] Bratu S,Brooks S,Burney S,et al.Detection and spread of Escherichia coli possessing the plasmid⁃borne carbapenemase KPC⁃2 in Brooklyn,New York[J].Clin.Infect.Dis.,2007,44(7):972-975.
[13] Zhang R,Yang L,Cai J C,et al..High⁃level carbapenem resistance in a Citrobacter freundii clinical isolate is due to a combination of KPC⁃2 production and decreased porin expression[J].J.Med.Microbiol.,2008,57(3):332-337.
[14] Bennett JW,Herrera M L,Lewis J S,et al..KPC⁃2⁃producing Enterobacter cloacae and Pseudomonas putida coinfection in a liver transplant recipient[J].Antimicrob.Agents Ch.,2009,53(1):292-294.
[15] Villegas M V,Lolans K,Correa A,et al..First identification of Pseudomonas aeruginosa isolates producing a KPC⁃type carbapenem⁃hydrolyzing beta⁃lactamase[J].Antimicrob.Agents Ch.,2007,51(4):1553-1555.
[16] Robledo I E,Aquino E E,Sante′M I,et al..Detection of KPC in Acinetobacter spp.in Puerto Rico[J].Antimicrob.Agents Ch.,2010,54(3):1354-1357.
[17] Mariana C,Lalitagauri M D,Joseph R D.First descriptions of blaKPCin Raoultella spp. (R. planticola and R.ornithinolytica): report from the SENTRY Antimicrobial Surveillance Progra[J].J.Clin.Microbiol.,2009,47(12):4129-4130.
[18] Shen P,Wei ZQ,Yan J,et al..Novel genetic environmentof the carbapenem⁃hydrolyzing β⁃lactamase KPC⁃2 among Enterobacteriaceae in China[J].Antimicrob.Agents Ch.,2009,53(10):4333-4338.
[19] Clinical and Laboratory Standards Institute.Performance Standards for Antimicrobial Susceptibility Testing:M100⁃S23[S].Wayne,PA:CLSI(USA),2013.
[20] Wei ZQ,Chen Y G,Yu Y S,et al..Nosocomial spread of multi⁃resistant Klebsiella pneumoniae containing a plasmid encodingmultipleβ⁃lactamases[J].J.Med.Microb.,2005,54(9):885-888.
[21] Naas T,Cuzon G,Villegas MV,et al..Genetic structures at the origin of acquisition of the beta⁃lactamase bla KPC gene[J].Antimicrob.Agents Ch.,2008,52(4):1257-1263.
[22] Qi Y,Wei Z Q,Ji S J,et al.ST11,the dominant clone of KPC⁃producing Klebsiella pneumoniae in China[J].J.Antimicrob.Chemother.,2011,66(2):307-312.
[23] Ge C R,Wei ZQ,Jiang Yan,et al..Identification of KFC⁃2⁃producing Pseudomonas aeruginosa isolates in China[J].J.Antimicrob.Chemother.,2011,66(5):1184-1186.
Genetic Environment of blaKPC⁃2in the Intestinal Colonized Raoultella planticolas
GE Chao⁃rong
First Affiliated Hospital,College ofMedicine,Zhejiang University,Hangzhou 310003,China
The increasing emergence and spread of Klebsiella pneumoniae carbapenemases(KPCs)leaves fewer available therapeutic options due to their broad⁃spectrum hydrolytic activity.Two KPC⁃2⁃producing Raoultella planticola isolates were first identified and analyzed using PCR,Southern hybridization and sequencing in China.Two isolates had an identical antimicrobial susceptibility profile,blaKPC⁃2gene aswell as its flanking sequences.They were resistant to 16 antimicrobial agentswhile sensitive to polymyxin B,colistin and minocycline.The results demonstrated that the blaKPC⁃2gene was located on a 7 498 bp plasmid segment that encoded five open reading frames with the order of Tn3⁃transposase,Tn3⁃resolvase,IS Kpn8,blaKPC⁃2gene and IS Kpn6⁃like.
Raoultella planticola;carbapenemase;plasmid
10.3969/j.issn.2095⁃2341.2013.05.10
2013⁃07⁃19;接受日期:2013⁃08⁃22
国家自然科学基金项目(81101283)资助。
葛超荣,副主任技师,主要从事医学微生物研究。E⁃mail:gechaorong@126.com

