微生物源化合物合成基因簇异源表达研究进展
黄 颖 赵 晨 关 雄 张晓琳
(国家粮食局科学研究院1,北京 100037)(福建农林大学生物农药与化学生物学教育部重点实验室2,福州 350002)
微生物源化合物合成基因簇异源表达研究进展
黄 颖1,2赵 晨1关 雄2张晓琳1
(国家粮食局科学研究院1,北京 100037)
(福建农林大学生物农药与化学生物学教育部重点实验室2,福州 350002)
微生物次级代谢产物具有多种多样的生物活性,在医疗、农业和食品领域有着广阔的应用前景。异源表达策略与不断发展的DNA克隆和改造技术相结合,成为一种有效的研究手段,能够用于提高天然活性化合物的产量,获得新结构类似物,阐明沉默基因簇的功能,还能够通过表达宏基因组DNA来获得新活性物质。本文对异源表达次级代谢基因簇的研究策略和成功案例进行综述,侧重于聚酮类物质和非核糖体肽的异源表达。
异源表达 次级代谢基因簇 聚酮类物质 非核糖体肽
微生物的次级代谢产物多种多样,其中有许多天然产物具有无可比拟的生物学活性,在医疗领域和农业生产上有极大的应用价值。聚酮类化合物是由聚酮合酶(polyketide synthase,PKS)催化低级羧酸缩合而成,是次级代谢产物中最大的种类,临床上所使用的多种药物都属于这一类化合物,在农业上使用的一些农药、兽药等也属于这类化合物[1]。一些对人类极为安全并能有效杀灭靶标生物的聚酮类化合物也被应用于食品领域[2-3]。由非核糖体肽合酶(nonribosomal peptide synthetase,NRPS)合成的肽类化合物同样具有广泛的生物活性[4]。许多重要次级代谢产物的合成是由PKS和NRPS两类合成酶所催化的,被称为杂合 NRPS/PKSs[5]合成途径。这些微生物次级代谢产物的开发和利用对于人类社会意义巨大,但原始产生菌的发酵产量通常比较低,或有应用上的问题,比如基因操作困难、生长缓慢、不易培养,甚至是无法培养时,使得化合物的分离、生产和修饰改造受到限制。研究表明,天然产物的合成、调控和抗性基因都是成簇排列在微生物的基因组内,因此在合适的宿主内异源表达天然和人工的生物合成基因簇就成为获得大量天然产物及其衍生物的捷径[6]。异源表达还能够激活“沉默”的生物合成基因簇,从而达到获得新活性化合物的目的[7]。
本研究对目前异源表达天然产物生物合成基因簇的策略进行综述,侧重于聚酮合酶(PKSs)、非核糖体肽酶(NRPSs)和杂合 NRPS/PKSs合成途径。同时,为了对异源表达策略的实际应用有所帮助,对生物合成基因簇的克隆、操作和在异源宿主中的遗传方式,以及异源宿主的选择及改造进行了深入的讨论(如图1),并对如何利用异源表达开发新化合物进行综述。
1 生物合成基因簇的克隆策略
近年来,发展出了一些将小片段DNA组装成基因、生物合成基因簇乃至整个基因组的方法,例如Golden Gate组装方法[8]、不依赖于基因序列和连接反应的基因克隆方法(Sequence and Ligation Independent Cloning)[9]、Gibson等温一步法[10]等。DNA合成和组装策略的优势在于能够实现基因簇的密码子优化和调控基因的选择,并可以添加或删除酶切位点[11]。在一个简单的宿主中(例如大肠杆菌(Escherichiacoli)或黄色粘细菌 (Myxococcusxanthus))中表达人工设计并合成组装的生物合成基因簇将成为新型化合物的来源之一[12]。
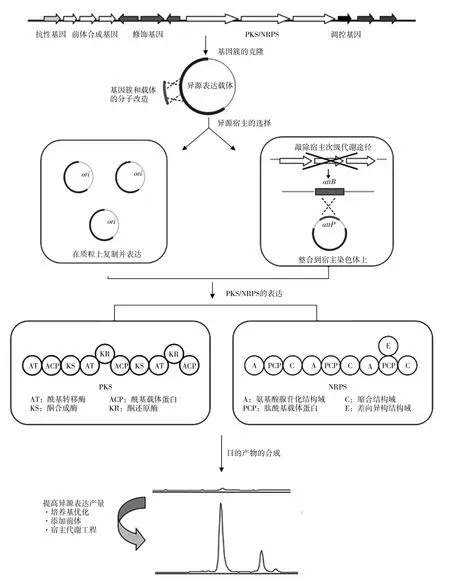
图1 异源表达生物合成基因簇的常规流程图
PKS和NRPS基因簇一般在10~120 kb之间。通过构建基因文库的手段来克隆基因簇时,一般会使用黏粒(cosmid)或福斯质粒(fosmid)来克隆30~35 kb的 DNA大片段,或者用细菌人工染色体(BAC)或P1派生人工染色体(PAC)来获得100kb以上的插入片段[13-14]。一个基因簇很有可能被分别克隆在几个载体上。利用Red/ET同源重组技术,能够在大肠杆菌中快速的完成缝合的过程[15]。而完整的RecE和RecT所介导的线性DNA之间的同源重组被称为 LLHR(linear plus linear homologous recombination),可用于直接克隆基因簇,而无需构建基因组文库[16]。首先,将基因组DNA进行酶切,以释放含有目的基因簇的DNA片段;然后构建一个线性质粒,使其两端与目的基因簇末端同源;最后在大肠杆菌内进行LLHR同源重组,从而将目的基因簇直接克隆到载体上。利用这样的方法,已从基因组中成功克隆了一系列 PKS/NRPS基因簇[17]。TAR(transformation-associated recombination)技术则利用了酿酒酵母(Saccharomycescerevisiae)中天然的高效重组能力,可以从已测序的基因组中直接克隆大片段的基因簇,但对于基因簇的进一步操作并不便捷[18]。Brady小组构建了一个S.cerevisiae/E.coli/Streptomyces穿梭BAC质粒pTARa,从柯氏柠檬酸杆菌(Citrobacterkoseri)的基因组DNA中直接克隆了56 kb的大肠杆菌毒素 colibactin基因簇[19]。LLHR和TAR方法将加速对“沉默”基因簇和环境宏基因组的潜力挖掘,从而推动新活性化合物的开发和利用[20]。
2 生物合成基因簇在异源宿主中的遗传策略
生物合成基因簇在宿主中的遗传方式分为两种,一种是在质粒上进行复制和表达,另一种则是直接整合到宿主的染色体上。通过质粒表达基因簇时,在前体供应充足的情况下,增加生物合成基因簇的拷贝数能够促进异源表达产物的合成[21]。将基因簇整合在宿主染色体上会增加基因簇的遗传稳定性,方法包括同源重组、转座和噬菌体介导的重组系统。同源重组是定点插入,可直接失活宿主次级代谢途径[22]。转座的整合效率比同源重组要高,60 kb的埃博霉素(epothilone)基因簇是目前通过转座技术插入宿主染色体的最大基因簇[23]。噬菌体所介导的重组系统则被广泛应用于以链霉菌为宿主的异源表达研究中,最近也被用于在M.xanthus中表达人工合成的 epothilone基因簇[24]。
3 异源宿主
3.1 表达PKSs途径的异源表达宿主
天蓝色链霉菌(Streptomycescoelicolor)自身产生几种聚酮类化合物,是最早用于表达聚酮类化合物的宿主。S.coelicolor有许多可以运用的载体和启动子系统,例如actI、mel、aphI、ermE、tipA、PnitA、gylP1/P2等[25-27]。6-脱氧红霉内酯 B(6-dEB)、榴菌素(granaticin)、美达霉素(medermycin)等都在天蓝色链霉菌中得以成功表达[28-29]。变铅青链霉菌(S.lividans)和S.coelicolor一样是表达聚酮类化合物的常用宿主,S.coelicolor的遗传改造方法和工具也同样适用于S.lividans[30]。
E.coli表达聚酮类化合物有很多优势,例如生长周期短(增倍时间20 min)、遗传操作简便等。但其与放线菌在生理、代谢、调控等方面差异较大,其自身的磷酸泛酰巯基乙胺基转移酶PPTase对底物有较强的选择性,无法对异源的ACP进行翻译后修饰,并且在大肠杆菌中大分子酶系的折叠存在困难。此外,E.coli中可能缺乏一些前体物质。经过一系列遗传改造,E.coliBAP1成功表达了 6dEB[31]。
来源于真核生物的聚酮类化合物可能更适合于在Sacharomycescerevisiae、黑曲霉(Aspergillusniger)、米曲霉(Aspergillusoryzae)和构巢曲霉(Aspergillus nidulans)等真核宿主中表达,以简化需要删除内含子等问题。曲霉属能够产生许多天然次级代谢产物,一旦克隆了聚酮类化合物的基因簇就可以通过原生质体融合和电转化将其转入曲霉中进行表达,可使用的启动子系统包括amyB、trpC、alcA等等[32]。目前已在曲霉属中成功地异源表达了黑色素(melanin)、角鲨抑素(squalestatin)、莫纳可林(monacolin J)等多种化合物[33-34]。
3.2 表达NRPS和杂合NRPS/PKSs合成途径的异源表达宿主
链霉菌和E.coli都可作为非核糖体肽类的异源表达宿主。目前在链霉菌中已成功表达了epothilone、达托霉素(daptomycin)、卷曲霉素(capreomycin)和噻可拉林(thiocoraline)。E.coli自身能够产生一种非核糖体肽(铁螯合载体肠菌素(siderophore enterobactin))[35],其作为异源宿主表达 NRPS的能力可能高于PKSs,目前已经成功表达一系列NRPS和杂合NRPS/PKSs合成途径,如耶尔森菌素(yersiniabactin)、棘霉素(echinomycin)和 epothilone等[36-37]。
枯草芽胞杆菌(Bacillussubtilis)拥有生长迅速(繁殖增倍时间1h)、转化容易、背景清晰的优势和天然的非核糖体肽合成能力[38],但目前缺少自我复制的质粒,仍有许多整合性质粒可供使用[39]。Bacillus subtilis中异源表达地衣芽孢杆菌(B.licheniformis)的杆菌肽bacitracin基因簇时,bacitracin的产量较原始菌株提高了50%。49kb的基因簇是分2次整合到染色体中[40],每次插入的片段比较小,这可能与大片段的插入效率较低有关,但有报道认为100kb大片段的插入是可能的[41]。
恶臭假单胞菌(Pseudomonasputida)也是NRPS合成途径的常用异源宿主,尽管是革兰氏阴性菌,却具备了产生多种次级代谢产物的能力以及与放线菌和粘细菌相近的GC含量,并且生长迅速、研究透彻,有许多可以运用的遗传工具,已被用于异源表达黏液噻唑(myxothiazol)、环肽化合物(myxochromide S)等[42]。
3.3 敲除宿主的主要次级代谢途径
不同次级代谢途径之间是相互竞争的,敲除宿主菌的主要次级代谢基因簇,能够减少对异源表达合成途径的竞争作用。阿维链霉菌(S.avermitilis)基因组左端粒区域含有许多次级代谢途径基因簇,将该区域逐步敲除,构建不产次级代谢产物的异源表达通用菌株[43],将20个次级代谢基因簇导入到所构建的通用宿主S.avermitilisSUKA17或 22中,大部分得以成功表达,少数不能够表达的异源基因簇也在更换了基因簇启动子或者调控基因的启动子之后获得了异源表达产物[44]。对S.coelicolorM145的4个主要次级代谢基因簇进行敲除,也能提高该菌株异源表达的能力[45]。生二素链霉菌(S.ambofaciens)BES2074菌株的主要次级代谢产物是螺旋霉素(spiramycin),阻断其合成途径后,异源表达脂肽类抗生素A54145,未经培养基优化,产量便达到了385 mg/L,是原始产生菌产量的 285%[46]。
4 新化合物的开发
大多数化合物的生物合成途径在现有的培养条件下是不表达的,或者表达量很低,被称为“沉默”的生物合成基因簇[12],是新型化合物的宝库。诱导沉默基因簇表达的方法主要包括培养条件的优化和调控基因的改造[12],以及进行异源表达。在纤维堆囊菌SorangiumcellulosumSo ce56的基因组中发现一个沉默的Ⅲ型PKS[47],将这个PKS在Pseudomonasputida中进行异源表达,证实了该PKS的产物是淡黄霉素flaviolin。
环境中存在大量不可培养的微生物,能够培养的微生物所占的比例不足1%[48],利用宏基因组文库技术能够深入挖掘微生物天然产物。通常的做法是构建宏基因组DNA的cosmid、fosmid或者BAC文库,并在E.coli、S.lividans或P.putida等通用菌株中进行异源表达[49-50]。近来,Brady等[51]采用基于PCR的筛选策略,在环境DNA cosmid文库中分离出38 kb的BE-54017基因簇,在白色链霉菌S.albus中成功地表达了这一抗癌物质。
异源表达天然和人工改造过的生物合成基因簇都会产生新的衍生物[52]。将来自蓝藻Mooreaproducens的新型脂肽barbamide生物合成基因簇在委内瑞拉链霉菌S.venezuelae中进行表达,产生了新的衍生物4-O-demethylbarbamide[53]。将新生霉素 novobiocin生物合成基因簇中的甲基转移酶基因novO失活后在S.coelicolor中异源表达,产生了C8位未被甲基取代的novobiocin类似物,再表达卤化酶基因clohal,产生了2个C8位被氯原子取代的novclobiocin类似物[54]。将金链菌素aureothin生物合成基因簇中参与起始单位(对硝基苯甲酰CoA)合成的基因aurG失活后在S.lividans中异源表达,并不能产生aureothin,加入非天然底物对氰基苯甲酸后,产生了一种aureothin的类似物——aureonitrile,具有更强的生物活性[55]。
5 结论与展望
微生物源活性天然产物在医疗、农业和工业领域具有极大的商业价值。天然产生菌的产量通常无法满足工业生产的需要,在原始菌株生长缓慢、不易培养、基因操作困难的情况下,异源表达其合成基因簇无疑是一条捷径。
靶标生物耐药性的增强迫使我们必须寻找新的活性化合物。对可培养微生物次级代谢产物的筛选已经难以发现新型的天然产物,因而微生物基因组中种类繁多的生物合成基因簇引起了极大的关注。利用异源表达的手段来激活“沉默”基因簇,或挖掘宏基因组,将成为发现新活性化合物的一大途径。此外,在异源宿主中改造已知的天然基因簇也是获得新化合物的重要途径,还将有助于阐明天然产物生物合成途径的分子机理,从而推动今后组合生物学的发展。
[1]Hertweck C.The biosynthetic logic of polyketide diversity[J].Angewandte Chemie International Edition,2009,48(26):4688-4716
[2]te Welscher Y M,ten Napel HH,BalaguéM M,et al.Natamycin blocks fungal growth by binding specifically to ergosterol without permeabilizing the membrane[J].Journal of Biological Chemistry,2008,283(10):6393-6401
[3]Kirst H A.The spinosyn family of insecticides:realizing the potential of natural products research[J].The Journal of Antibiotics,2010,63(3):101-111
[4]Marahiel M A,Essen L O.Nonribosomal peptide synthetases:mechanistic and structural aspects of essential domains[J].Methods in Enzymology,2009,458:337-351
[5]Fischbach M A,Walsh CT.Assembly-line enzymology for polyketide and nonribosomal peptide antibiotics:logic,machinery,and mechanisms[J].Chemical Reviews,2006,106(8):3468-3496
[6]Fortman JL,Sherman D H.Utilizing the power of microbial genetics to bridge the gap between the promise and the application of marine natural products[J].ChemBioChem,2005(6):960-978
[7]Chiang Y M,Chang S L,Oakley B R,et al.Recent advances in awakening silent biosynthetic gene clusters and linking orphan clusters to natural products in microorganisms[J].Current Opinion in Chemical Biology,2011,15(1):137-143
[8]Engler C,Kandzia R,Marillonnet S.A one pot,one step,precision cloning method with high throughput capability[J].PloSOne,2008,3(11):e3647
[9]Li M Z,Elledge SJ.Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC[J].Nature Methods,2007,4(3):251-256
[10]Gibson D G,Young L,Chuang R Y,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases[J].Nature Methods,2009,6(5):343-345
[11]Cobb R E,Zhao H.Direct cloning of large genomic sequences[J].Nature Biotechnology,2012,30(5):405-406
[12]Zotchev SB,Sekurova O N,Katz L.Genome-based bioprospecting of microbes for new therapeutics[J].Current O-pinion in Biotechnology,2012,23:941-947
[13]Alduina R,Gallo G.Artificial chromosomes to explore and to exploit biosynthetic capabilities of actinomycetes[J].BioMed Research International,2012:462049
[14]Jones A C,Gust B,Kulik A,et al.Phage p1-derived artificial chromosomes facilitate heterologous expression of the FK506 gene cluster[J].PloSone,2013,8(7):e69319
[15]Sharan SK,Thomason L C,Kuznetsov SG.Recombineering:a homologous recombination-based method of genetic engineering[J].Nature Protocols,2009,4(2):206-223
[16]Fu J,Bian X,Hu S,et al.Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J].Nature Biotechnology,2012,30(5):440-446
[17]Bian X,Huang F,Stewart F A,et al.Direct cloning,genetic engineering,and heterologous expression of the syringolin biosynthetic gene cluster in E.coli through Red/ET Recombineering[J].ChemBioChem,2012,13(13):1946-1952
[18]Kallifidas D,Brady S F.Reassembly of functionally intact environmental DNA-derived biosynthetic gene clusters[J].Methods in Enzymology,2012,517:225-239
[19]Kim JH,Feng Z,Bauer J D,et al.Cloning large natural product gene clusters from the environment:Piecing environmental DNA gene clusters back together with TAR[J].Biopolymers,2010,93(9):833-844
[20]Wolpert M,Heide L,Kammerer B,et al.Assembly and heterologous expression of the coumermycin A1 gene cluster and production of new derivatives by genetic engineering[J].Chembiochem,2008,9(4):603-612
[21]Fong R,Vroom J A,Hu Z,et al.Characterization of a large,stable,high-copy-number streptomyces plasmid that requires stability and transfer functions for heterologous polyketide overproduction[J].Applied and Environmental Microbiology,2007,73:1296-1307
[22]Perlova O,Fu J,Kuhlmann S,et al.Reconstitution of the myxothiazol biosynthetic gene cluster by Red/ET recombination and heterologous expression in Myxococcus xanthus[J].Applied and Environmental Microbiology,2006,72(12):7485-7494
[23]Fu J,Wenzel S C,Perlova O,et al.Efficient transfer of two large secondary metabolite pathway gene clusters into heterologous hosts by transposition[J].Nucleic acids research,2008,36(17):e113-e113
[24]Oßwald C,Zipf G,Schmidt G,et al.Modular construction of a functional artificial epothilone polyketide pathway[J].ACSsynthetic biology,2012,doi:10.1021/sb300080t
[25]Herai S,Hashimoto Y,Higashibata H,http://www.ncbi.nlm.nih.gov/pubmed?term=Maseda%20H%5bAuthor%5d&cauthor=true&cauthor_uid=15377796 et al.Hyperinducible expression system for streptomycetes[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(39):14031-14035
[26]Rodríguez-García A,Combes P,Pérez-Redondo R,et al.Natural and synthetic tetracycline-inducible promoters for use in the antibiotic-producing bacteriaStreptomyces[J].Nucleic Acids Research,2005,33(9):e87
[27]Dangel V,Westrich L,Smith M C,http://www.ncbi.nlm.nih.gov/pubmed?term=Heide%20L%5bAuthor%5d&cauthor=true&cauthor_uid=20127238 et al.Use of an inducible promoter for antibiotic production in a heterologous host[J].Applied Microbiology Biotechnology,2010,87(1):261-269
[28]Tang L,Shah S,Chung L,et al.Cloning and heterologous expression of the epothilone gene cluster[J].Science,2000,287(5453):640-642
[29]Eustáquio A S,Gust B,Galm U,et al.Heterologous ex-pression of novobiocin and clorobiocin biosynthetic gene clusters[J].Applied Microbiology Biotechnology,2005,71(5):2452-2459
[30]Lussier F X,Denis F,Shareck F.Adaptation of the highly productive T7 expression system toStreptomyceslividans[J].Applied and Environmental Microbiology,2010,76(3):967-970
[31]Pfeifer B A,Admiraal SJ,Gramajo H,et al.Biosynthesis of complex polyketides in a metabolically engineered strain ofE.coli[J].Science,2001,291(5509):1790-1792
[32]Lu P,Zhang A,Dennis L M,et al.A gene(pks2)encoding a putative 6-methylsalicylic acid synthase fromGlarea lozoyensis[J].Molecular Genetics and Genomics,2005,273(2):207-216
[33]Bailey A M,Cox R J,Harley K,et al.Characterisation of 3-methylorcinaldehyde synthase(MOS)in Acremoniumstrictum:first observation of a reductive release mechanism during polyketide biosynthesis[J].Chemical Communications,2007,(39):4053-4055
[34]Cox R J,Glod F,Hurley D,et al.Rapid cloning and expression of a fungal polyketide synthase gene involved in squalestatin biosynthesis[J].Chemical Communications,2004(20):2260-2261
[35]Rusnak F,Sakaitani M,Drueckhammer D,et al.Biosynthesis of theEscherichiacolisiderophore enterobactin:sequence of theentFgene,expression and purification of EntF,and analysis of covalent phosphopantetheine[J].Biochemistry,1991,30(11):2916-2927
[36]Gruenewald S,Mootz H D,Stehmeier P,et al.In vivo production of artificial nonribosomal peptide products in the heterologous hostEscherichiacoli[J].Applied and Environmental Microbiology,2004,70(6):3282-3291
[37]Watanabe K,Hotta K,Praseuth A P,et al.Total biosynthesis of antitumor nonribosomal peptides inEscherichiacoli[J].Nature Chemical Biology,2006,2(8):423-428
[38]Kleinkauf H,von Döhren H.The nonribosomal peptide biosynthetic system-on the origins of structural diversity of peptides,cyclopeptides and related compounds[J].1995,67(3):229-242
[39]Bron S,Meijer W,Holsappel S,et al.Plasmid instability and molecular cloning inBacillussubtilis[J].Research in Microbiology,1991,142(7-8):875-883
[40]Eppelmann K,Doekel S,Marahiel M A.Engineered biosynthesis of the peptide antibiotic bacitracin in the surrogate hostBacillussubtilis[J].The Journal of Biological Chemistry,2001,276(37):34824-34831
[41]Itaya M,Kaneko S.Integration of stable extracellular DNA released from Escherichia coli into the Bacillus subtilis genome vector by culture mix method[J].Nucleic acids Research,2010,38(8):2551-2557
[42]Gross F,Ring M W,Perlova O,et al.Metabolic engineering ofPseudomonasputidafor methylmalonyl-CoA biosynthesis to enable complex heterologous secondary metabolite formation[J].Chemistry&Biology,2006,13(12):1253-1264
[43]Komatsu M,Uchiyama T,■mura S,et al.Genome-minimizedStreptomyceshost for the heterologous expression of secondary metabolism[J].Proceedings of the National A-cademy of Sciences,2010,107(6):2646-2651
[44]Komatsu M,Komatsu K,Koiwai H,et al.EngineeredStreptomycesavermitilishost for heterologous expression of biosynthetic gene cluster for secondary metabolites[J].ACS synthetic biology,2013,doi:10.1021/sb3001003
[45]Gomez-Escribano JP,Bibb M J.EngineeringStreptomyces coelicolorfor heterologous expression of secondary metabolite gene clusters[J].Microbial Biotechnology,2011,4(2):207-215
[46]Alexander D C,Rock J,He X,et al.Development of a genetic system for combinatorial biosynthesis of lipopeptides in Streptomyces fradiae and heterologous expression of the A54145 biosynthesis gene cluster[J].Applied and Environmental Microbiology,2010,76(20):6877-6887
[47]Gross F,Luniak N,Perlova O,et al.Bacterial type III polyketide synthases:phylogenetic analysis and potential for the production of novel secondary metabolites by heterologous expression in pseudomonads[J].Archives of Microbiology,2006,185(1):28-38
[48]Kaeberlein T,Lewis K,Epstein S S.Isolating uncultivable microorganisms in pure culture in a simulated natural environment[J].Science,2002,296(5570):1127-1129
[49]Brady SF,Wright SA,Lee JC,et al.Pantocin B,an antibiotic fromErwiniaherbicoladiscovered by heterologous expression of cloned genes[J].Journal of the American Chemical Society,1999,121(50):11912-11913
[50]Martinez A,Kolvek S J,Yip C L T,et al.Genetically modified bacterial strains and novel bacterial artificial chromosome shuttle vectors for constructing environmental libraries and detecting heterologous natural products in multiple expression hosts[J].Applied and Environmental Microbiology,2004,70(4):2452-2463
[51]Chang F Y,Brady S F.Cloning and characterization of an environmental DNA-derived gene cluster that encodes the biosynthesis of the antitumor substance BE-54017[J].Journal of the American Chemical Society,2011,133(26):9996-9999[52]Smanski M J,Casper J,Peterson R M,et al.Expression of the platencin biosynthetic gene cluster in heterologous hosts yielding new platencin congeners[J].Journal of Natural Products,2012,75(12):2158-2167
[53]Kim E J,Lee JH,Choi H,et al.Heterologous production of 4-O-demethylbarbamide,a marine cyanobacterial natural product[J].Organic letters,2012,14(23):5824-5827
[54]Eustáquio A S,Gust B,Li SM,et al.Production of 8′-halogenated and 8′-unsubstituted novobiocin derivatives in genetically engineeredStreptomycescoelicolorstrains[J].Chemistry&Biology,2004,11(11):1561-1572
[55]Ziehl M,He J,Dahse H M,et al.Mutasynthesis of aureonitrile:an aureothin derivative with significantly improved cytostatic effect[J].Angewandte Chemie International Edition,2005,44(8):1202-1205.
Recent Developments Towards the Heterologous Expression of Microbial Compound Biosynthetic Gene Clusters
Huang Ying1,2Zhao Chen1Guan Xiong2Zhang Xiaolin1
(Academy of State Administration of Grain1,Beijing 100037)
(Key Laboratory of Biopesticide and Chemical Biology,Ministry of Education,Fujian Agriculture and Forestry University2,Fuzhou 350002)
Microbial secondary metabolites have varieties of biological activities,which have broad application prospect in the fields of pharmaceutical,agrochemical and food.Heterologous expression coupled with developing DNA clone and transformation technology has become an efficient pathway that enables the optimization of natural active compounds yields,production of new structural analogues,functional elucidation of silent gene clusters and generation of new active compounds through expressing metagenomic DNA.In this review,remarkable success stories as well as the experimental approaches will be presented with the emphasis on heterologous expression of polyketide and non-ribosomally biosynthesized peptide compounds.
heterologous expression,secondary metabolite gene cluster,polyketide,nonribosomal peptide
Q936
A
1003-0174(2015)09-0133-07
国家自然科学基金 (31300092),粮食公益性行业科研专项(201313002-3)
2014-03-10
黄颖,女,1989年出生,硕士,抗生素发酵和基因工程研究
张晓琳,女,1975年出生,研究员,微生物发酵和基因工程研究

