放牧对草地土壤微生物的影响
苟燕妮,南志标
(草地农业生态系统国家重点实验室,兰州大学草地农业科技学院,甘肃 兰州 730020)
放牧对草地土壤微生物的影响
苟燕妮,南志标*
(草地农业生态系统国家重点实验室,兰州大学草地农业科技学院,甘肃 兰州 730020)
摘要:土壤质量是草地生态系统生产力可持续发展的关键所在。健康的土壤必定拥有丰富的物种多样性、活跃的生命体、高效的内部养分循环及较好的抗干扰能力。土壤微生物作为土壤系统中各种生物进程的主要推动者,其作用不可小觑。因此,我们可以利用土壤微生物的数量、群落结构和多样性等特征监测土壤质量。本文综述了放牧对草地土壤微生物数量、生物量、群落结构和多样性的影响。指出通过了解土壤微生物与放牧行为间的关系,如何评估土壤质量,选取适宜的放牧梯度,完善草地管理模式,并介绍了土壤微生物群落结构和多样性研究方法的利弊。解决两个关键问题:1)土壤微生物的各种特征是如何响应放牧行为的。2)如何通过这些指标评价土壤质量,然后提出合理的放牧模式,改善和维护草地的生产力。
关键词:放牧;土壤;微生物生物量;微生物多样性
The impacts of grazing on the soil microorganism population of grassland
GOU Yan-Ni, NAN Zhi-Biao*
StateKeyLaboratoryofGrasslandAgro-ecosystems,CollegeofPastoralAgriculturalScienceandTechnology,LanzhouUniversity,Lanzhou730020,China
Abstract:Soil quality is crucial to sustainable development of grassland ecosystems and to their productivity. Healthy soil must have rich species diversity, active microorganism populations, an efficient internal nutrient cycle and good buffering against disturbance. Soil microorganisms are the main driver of various biological processes and are therefore very important in soil systems. Hence, soil microbial community characteristics such as population numbers, community structure and species diversity can be used as a measure of soil health. The present paper summarizes the effects of grazing on the grassland soil microbial populations, community structure and diversity. We propose that it is essential to understand the impact of grazing management on soil microorganisms. A methodology is introduced for evaluation of soil health, and selection of a suitable grazing level to improve grassland management, and the advantages and disadvantages are discussed. Two key questions are addressed; namely the responses of the soil microbial community to grazing activity, and how to use these indicators to evaluate soil quality and propose a reasonable grazing practice to sustainably improve the productivity of grassland.
Key words:grazing; soil; microbial biomass; microbial diversity
放牧是调节草地生产力最经济有效的管理方式[1-2],对草地生态系统地上和地下均有影响[3-4]。有蹄类动物通过加强土壤微生物营养富集和根际作用,提高矿物质有效利用率,最终促进植物营养能量的流动和光合作用[5]。土壤微生物作为草地地下生态系统最大的资源库,推动整个环境中物质和能量的流动和转移,对维持草地生态系统有重要的作用[6]。土壤微生物对土壤质量变化敏感,尤其是土壤微生物多样性作为土壤生物学性质的敏感指标,能够较早地指示草原生态环境变化和生态系统功能的变化[7]。研究掌握土壤微生物对放牧强度的响应在草原生态系统保护、恢复及重建中具有重要理论和实践意义。
因此,为了明确放牧与土壤微生物群落结构、营养物质循环和分解过程之间的重要关系,关于放牧对土壤微生物群落影响的研究越来越多,主要有以下几个方面:土壤微生物数量、生物量、活性、群落结构和多样性的影响。研究表明,家畜的采食、踩踏和粪尿回地都会对土壤生物区系带来影响[8-11]。而且随着放牧率的增大,营养循环加速、凋落物生物量和土壤微生物生物量均增多。但是重度放牧会消耗土壤资源,从而减少地上和地下的生物量[12-13]。随着研究的深入,学者们发现,由于不同的地理位置和季节气候下土壤微生物对放牧的响应方式不一致[14-16],因此,放牧与土壤微生物间关系复杂,而且又受环境因素的影响而呈现多变的变化趋势。所以本文对不同环境条件下放牧与土壤微生物间的关系做一综述,以期为以后的研究提供一些可参考的信息和依据。
1放牧对草地微生物数量的影响
土壤微生物是陆地生态系统中非常庞大的一个资源库。每克土壤含有几百万个微生物[17],2000~18000种细菌[18]。近年来,Gans等[17]运用现代DNA测序技术研究得出,每克土壤里约有107种微生物。土壤微生物数量是土壤微生物最基础的研究对象,有些研究者在分析土壤微生物时通常会先对微生物的数量和分布变化情况进行数据采集,现今,最为常见的计数方法为平板表面涂抹计数法。
目前,土壤微生物主要研究对象为3大类群:细菌、真菌和放线菌。不同类型草地各放牧强度下三大类群微生物的数量变化趋势由表1呈现。赵吉[19]对内蒙古冷蒿(Artemisiafrigida)小禾草草原进行4年的轮牧试验发现,土壤各微生物类群数量均随放牧率的增大而减少,重牧区的微生物总数下降约30%。尤其是自由放牧区的微生物数量显著低于放牧试验区。他们认为过度放牧不利于土壤微生物的生长,并且发现微生物数量与土壤有机碳含量显著正相关。张成霞和南志标[20]对陇东黄土高原天然草地土壤微生物和土壤理化性质相关性进行研究发现:土壤微生物总数和细菌数量随放牧强度的增大呈先减少后增多的趋势,而真菌和放线菌数量随放牧强度的增大而减少。土壤微生物总数在重牧条件下最多,这是由细菌数量最多引起的。因为,不同草地类型和利用方式下,与真菌和放线菌相比,细菌占有绝对优势,所以,土壤微生物数量主要取决于细菌数量[21]。对于细菌数量在重牧地出现最高值这一现象,张成霞和南志标[20]认为是由于牲畜的践踏使重牧地土壤变得紧实,通气性下降,从而使嫌气性细菌数量略有增加,再加上细菌适应不良环境的能力强,因而导致这种现象的发生。关于土壤微生物与土壤理化性质间相关性结果[22]表明,细菌数量与铵态氮、硝态氮及pH值呈正相关,与全氮和速效磷呈负相关。刘世贵等[22]对川西北退化草甸草地土壤微生物主要生理类群数量进行研究发现,土壤微生物总数随草地退化程度的加深而减少,而且3种退化草地中嫌气性纤维素分解细菌数量最多,这为重牧地细菌数量最多提供了参考依据。李春莉等[23]对内蒙古短花针茅(Stipabreviflora)草原研究显示:土壤细菌、真菌和放线菌数量随放牧强度变化趋势均为:重牧地<不放牧<轻牧地,表明适度的放牧率会引起土壤微生物数量增多,而不放牧和过度放牧不利于土壤微生物的生长。土壤微生物与土壤有机质和土壤全氮呈较强的正相关关系。单贵莲等[24]对内蒙古半干旱典型围封草原研究发现,围封草地与自由放牧地相比,细菌、真菌和放线菌数量无显著增加,且随围封年限的延长呈递增趋势,但当围封年限达到20年时,细菌数量显著增多,而真菌和放线菌数量有所下降。顾爱星等[25]对天山北坡温带极端干旱天然草地研究发现,退化草地的微生物数量高于未退化草地,且重度退化草地的微生物数量最多,土壤微生物数量随草地退化程度的加深呈现先增加后减少的关系。同时根据Wang和Sheng[26]关于松嫩平原农牧交错带天然草地的研究发现,土壤细菌、放线菌和真菌数量随放牧强度的增加也呈现先增多后减少的趋势,中牧地的土壤微生物数量最多。
由以上学者的研究可见,草地土壤微生物数量与放牧强度间关系呈现3种变化趋势。1)土壤微生物随放牧强度的增加或者退化程度的加深而减少,也就是放牧强度越大,草原退化越严重则土壤微生物数量越少。2)随放牧强度的增加先增多后减少的趋势,即一定的放牧率引起土壤微生物增多,且适度的放牧率微生物数量达到最大值,而过度放牧不利于土壤微生物的生长所以减少。3)随放牧强度的增加而减少,但在重度放牧时突然增多。多数研究结果呈现第二种变化趋势。根据研究者们对试验结果的分析发现,土壤微生物数量与土壤某些理化性质(比如:有机碳、全氮、pH值等)呈正相关关系。可见,一般情况下土壤微生物数量多时表示土壤质量较好,放牧率合适,草地健康状况良好(表1)。

表1 不同草地类型不同放牧强度下土壤微生物数量统计表
2放牧对土壤微生物生物量的影响
土壤微生物生物量是土壤营养元素的资源库,是植物生长所需养分的重要来源[27-28]。有研究认为与微生物个体数量相比,微生物生物量更能反映土壤中微生物的实际含量和潜在能力,所以土壤微生物生物量是反映土壤质量和性能的一项重要的指标[29-30]。在草地生态系统中,大部分的营养物质被植物吸收利用,这些营养物质又直接通过动物排泄物进入能量循环过程,导致土壤中碳、氮加速分解转化。已有少数学者研究发现,放牧地比未放牧地的有机碳和氮的含量高,且微生物生物量也较高[31-33]。但是,有些学者认为,草地放牧时存在潜在的氨挥发损失,所以有机氮含量较少[34-35]。可见土壤微生物生物量受放牧影响也较复杂。不同放牧方式下土壤微生物生物量变化见表2。
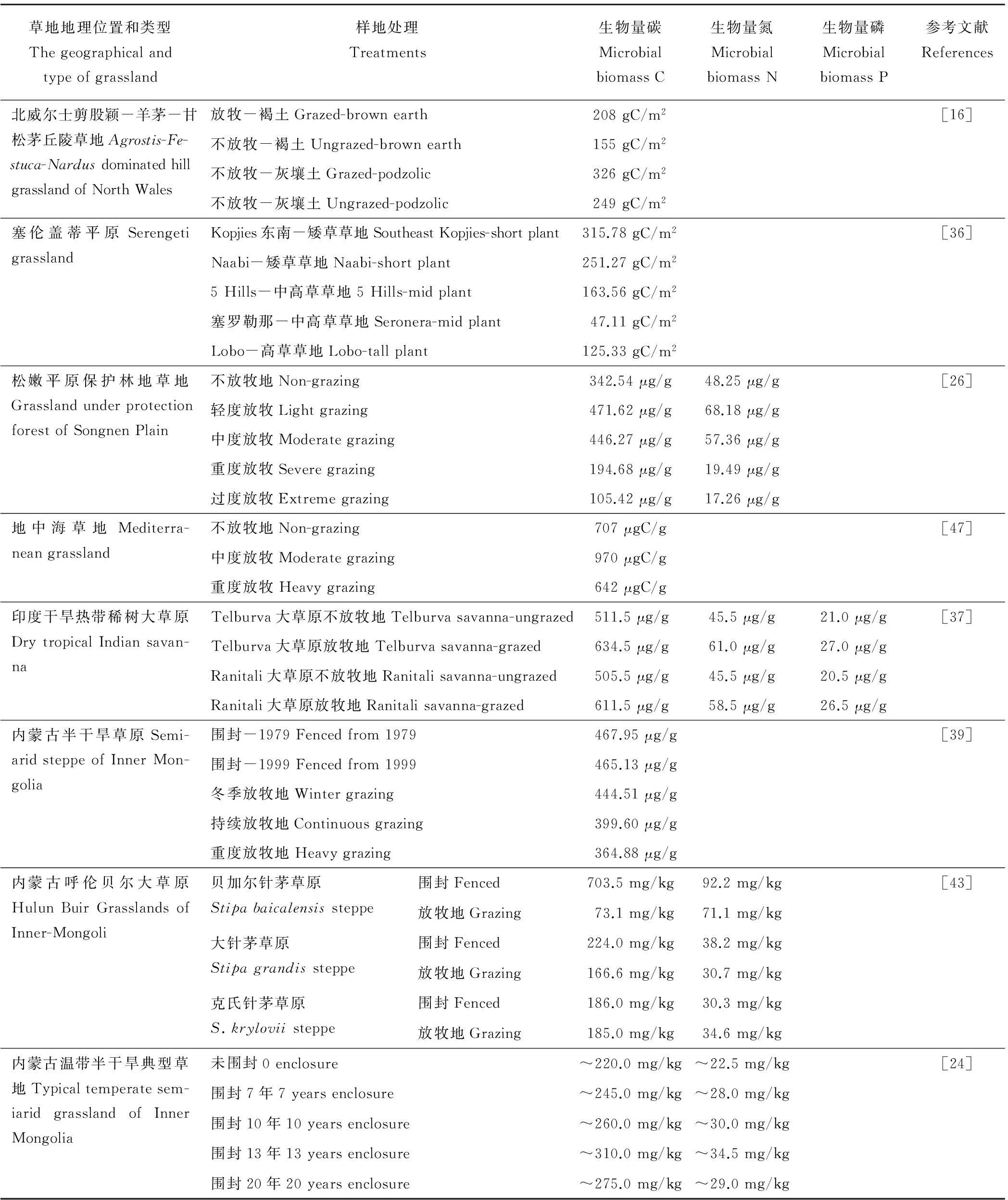
表2 不同草地类型不同放牧强度下土壤微生物生物量统计
Bardgett等[16]对两种不同类型草原[剪股颖羊茅(Agrostisfestuca)草原和甘松茅(Nardusstricta)草原]放牧地研究发现,两类草原均表现出不放牧草地的表层土壤微生物生物量显著低于放牧地,停止放牧时微生物生物量减少。Ruess和McNaughton[36]对塞伦盖蒂平原矮草草地研究发现,随着放牧强度的增大,土壤微生物生物量碳含量增加,且与土壤有机碳呈正相关关系。Singh等[37]认为放牧增加土壤有效营养物质和微生物生物量,他们对印度干热带稀树大草原研究发现,放牧增加土壤微生物生物量碳(15%~18%)、氮(14%~23%)、磷(19%~29%),且与土壤有机氮、磷呈正相关关系。Bardgett等[38]对山地草甸放牧地研究发现,放牧地的真菌生物量显著高于未放牧地,而细菌生物量无显著变化。研究者认为,这种现象可能是由于酸性土壤的草地以及家畜排泄物有益于真菌的生长,并且放牧地出现嗜粪类微生物种类,导致放牧地真菌生物量高于未放牧地。
Qi等[39]对内蒙古半干旱放牧地进行研究发现重牧梯度下土壤微生物生物量碳最低,且与土壤有机碳和土壤全氮呈正相关关系。他们认为土壤有机碳和全氮是决定土壤微生物生物量的主要影响因子,而且重牧地土壤有机碳和全氮减少是由于家畜过度采食导致植物生物量变少而引起的[40],同时裸地斑块由于风蚀作用导致土壤有机质减少[41-42],也使土壤微生物生物量减少[12]。所以,重牧梯度下土壤有机碳含量低导致土壤微生物生物量降低。赵帅[43]对呼伦贝尔针茅(Stipacapillata)草原研究,结果显示围封样地的微生物生物量碳、氮含量显著高于放牧地。李玉洁等[44]研究表明,土壤微生物生物量随休牧年限的增加而增多,休牧9年的微生物生物量碳、氮含量均最高。单贵莲等[24]研究表明,围封草地与自由放牧草地相比,微生物生物量碳、氮含量显著增加,且随围封年限的延长呈增加趋势。Northup等[13]关于热带禾草草原不同放牧梯度的研究显示,随放牧梯度的增大土壤微生物生物量减少。他们认为这是由于放牧导致地表覆盖度降低,植被凋落物生物量减少,从而减少了土壤有机物质和碳的投入,限制了微生物生长的可用能源导致的[45]。
张蕴薇等[46]关于人工草地的研究结果显示,在放牧期间中牧区的微生物生物量碳、氮均最高,而在停牧期间轻牧区和不放牧区较高;随放牧时间的延长,放牧强度越大微生物生物量碳、氮含量迅速降低。Wang和Sheng[26]研究表明土壤微生物生物量碳、氮随放牧强度的增大呈先增多后减少的趋势。以上两位学者的研究都与Li 等[47]的研究结果一致。Li等[47]在地中海高山草甸试验结果表明,中度放牧条件下的土壤呼吸速率和土壤微生物生物量最高,而重牧地的土壤微生物生物量最低。而且中牧地的土壤全氮、有机质及钠离子和钾离子显著高于重牧地。说明这些土壤理化性质有益于土壤微生物群落的生长,从而加强了土壤营养物质的循环,使生产能力最大化。所以中度放牧梯度为最适放牧率。
总结以上关于土壤微生物生物量的研究,我们可以发现土壤微生物量与放牧强度间存在3种关系(表2):1)放牧增加土壤微生物生物量,且随放牧强度的增加而增多。2)放牧减少土壤微生物生物量,且随放牧强度的增加而减少。3)土壤微生物生物量随放牧强度的增加,呈先增多后减少的趋势。首先,虽然土壤微生物量与放牧间的关系复杂,但是从以上学者们的研究中发现土壤微生物生物量与土壤有机碳、有机氮、全氮、磷等其他土壤理化性质呈正相关关系。不论在何种放牧率条件下,土壤有机碳等物质含量高时,土壤微生物生物量就高。其次,学者们研究结果不一致,其中最主要的原因是因为放牧率不明确,由于试验不同而放牧率不同,在大多数研究中并未明确放牧率,研究者们只是根据各自试验区域的情况而划分为轻度、中度、重度、过度放牧地。可见放牧率的范围很模糊,这也为定量分析造成影响。因此,关于放牧对土壤微生物量的研究,一定要结合土壤理化性质的分析,并且要以定量的放牧率为基础。
3放牧对土壤微生物多样性的影响
生物多样性、干扰行为及生态系统功能三者间的相互关系一直是国际生态学界讨论的热点,Schwartz等[48]总结前人的研究结果发现,95%的试验结果证明生物多样性与生态系统功能和过程间关系密切,但因为物种的特异性,它们之间的关系无规律。Hughes[49]指出干扰行为与生物多样性间存在复杂的关系,并且总结出几种常用的假说来解释它们之间的相互作用。在草地生态系统中,土壤是一个包含有大量微生物生活的复杂生态系统,土壤微生物多样性与土壤代谢能力紧密相关[50],对环境的影响比在理化性质方面更加重要[51]。有学者发现土壤微生物多样性指标与生态系统抵御外界干扰能力密切相关[52-53]。放牧是草地生态系统中长久以来最主要的一种干扰行为。因此,很多学者致力于放牧与土壤微生物多样性间关系的研究,以期明确它们之间的关系,选择最适放牧率,从而维持草地生态系统健康发展。随着研究方法的不断完善和出新,学者们运用不同的研究方法对各种类型草地放牧与土壤微生物多样性间的关系进行研究。
3.1 围封与自由放牧对土壤微生物多样性的影响
赵帅[43]运用PLFAs法和16S rDNA-DGGE法对呼伦贝尔针茅草原研究,结果显示放牧条件下细菌磷脂脂肪酸含量显著低于围封草地,并发现存在7个菌门:变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、芽单胞菌门(Gemmatimonadetes)、酸杆菌门(Acidobacteria)、厚壁菌门(Firmicutes)和梭杆菌门(Fusobacteria)。李玉洁等[44]运用BIOLOG法对贝加尔针茅草原研究,发现自由放牧、休牧3年和休牧6年样地间的土壤微生物群落碳源利用方式及代谢功能无差异,而休牧9年样地的微生物群落具有不同的碳源利用方式和代谢功能。李梓正等[54]采用16S rDNA-DGGE对呼伦贝尔草原研究,结果表明:羊草(Leymuschinensis)草甸草原和贝加尔针茅草甸草原的微生物丰富度和多样性在8月份最高,并且土壤微生物优势种群不受草地退化程度的影响。张海芳等[55]通过BIOLOG法对内蒙古贝加尔针茅草原研究,发现自由放牧土壤微生物丰富度、均匀度和优势度均最高,围封次之,刈割最低。周文萍等[56]采用高通量测序法对不同放牧强度下的AM真菌群落进行研究,结果显示AM真菌多样性指数和均匀度指数在长期封育小区最低,且显著低于中度放牧区,而各放牧小区间差异不显著。邹雨坤等[57]运用PLFAs法对羊草草原进行研究,发现刈割条件下的PLFA总量显著高于围封和放牧。Bardgett等[16]的研究结果为,长期围封地表层土壤的真菌丰富度和真菌/细菌PLFA值显著低于放牧地,但是细菌多样性不受放牧地的影响。
3.2 不同放牧梯度对微生物多样性的影响
Patra等[58]对不同放牧梯度下的功能微生物进行研究,发现重牧地的细菌PLFAs高于轻牧地但真菌PLFAs没有差异,而且重牧地与轻牧地间细菌群落结构差异明显,这与McCaig等[59]和Grayston等[60]的研究结果一致。Wakelin等[61]运用PCR-DGGE和TRFLP两种方法研究发现,放牧对土壤真菌和细菌群落结构有影响,而且真菌丰富度随放牧强度的增大而增多。Zhou等[62]对不同放牧梯度的细菌多样性进行研究,结果表明轻牧地和中牧地的细菌多样性显著高于未放牧地和重牧地。Howe等[63]研究认为中度放牧强度下细菌多样性显著增多。刘天增和南志标[64]运用BIOLOG法研究发现不放牧和中度放牧样地的土壤微生物多样性是最高的,而重度放牧下的土壤微生物多样性最低[65]。Yang等[6]通过高通量测序方法研究表明,微生物遗传多样性随放牧强度增大而减少。Nacke等[66]运用高通量测序法对森林和草地放牧地进行研究发现,未放牧的森林地土壤细菌多样性高于放牧地,施肥未放牧地的细菌多样性最高,然后是施肥放牧地,最低的是不施肥放牧地,可见放牧使草地细菌多样性降低。Ford等[67]对沿海地区的盐泽和沙丘两种草地类型研究,结果显示放牧对细菌PLFAs和真菌PLFAs影响不明显。
3.3 放牧对土壤原生动物多样性的影响
关于放牧对原生动物和线虫的研究发现,有些研究区域放牧会使线虫的丰富度增加2倍[39],Schönborn[68]研究发现土壤重度紧实使原生动物种类显著减少,土壤轻微紧实对土壤原生动物有抑制作用,这可能是由土壤孔隙度和土壤水分含量降低造成的。同时,Qi等[39]还发现,中度放牧下变形虫的生物多样性最高,这是因为变形虫中的优势种躯体柔软可以进入到其他原生动物进入不了的微孔中,然后获得食物和水进行生活[69]。
由各位学者们研究可知,放牧对土壤微生物多样性的影响规律不一致:第一,未放牧与放牧区间比较,一些学者研究显示未放牧区的土壤微生物多样性多于放牧区,而有学者发现长期封育的小区多样性指数低于放牧区;第二,不同放牧强度样地间比较,土壤微生物多样性与放牧干扰间的关系表现为:1) 放牧强度对土壤微生物多样性无显著影响;2) 土壤微生物多样性随放牧强度或草地退化程度的加深而增多;3) 土壤微生物多样性随放牧强度增多而减少以及中度放牧强度下土壤微生物多样性最多。这与Hughes[49]提出的关于干扰与生物多样性间关系假说中的其中几种假说一致。
4土壤微生物群落结构和多样性研究方法
土壤微生物多样性,即群落结构及功能、遗传多样性,在评估土壤质量方面越来越受重视[70]。
4.1 磷脂脂肪酸谱图法(PLFAs)
结构多样性的定义是系统内部各部分或者组成元素所占比例情况,比如可用物种、基因组、群落数量来表示[71]。如今最常采用的是PLFAs法。由于土壤微生物拥有各自特定的磷脂脂肪酸(PLFAs),因此,可以直接从土壤中提取活的微生物细胞膜中的PLFAs,通过测定PLFAs的种类和含量得到微生物群落结构组成的信息。自1990年以后,这种方法已经广泛应用于各种环境问题引起微生物群落组成变化的研究[67,72-73],使土壤微生物多样性得以更深入的研究。PLFAs法省时快速、耗费少、可重复而且能够定量表征微生物的多样性及群落结构组成变化[74]。但是PLFAs法仍有不足:首先,关于脂肪酸表征特定微生物存在3个争论点。第一,某些脂肪酸尚未确定类型,而且某些脂肪酸类型与微生物种类间对应关系不明确。比如,PLFAs cy17:0 和cy19:0通常用于表征革兰氏阴性细菌,但是Schoug 等[75]发现某些革兰氏阳性菌里也大量存在这两种PLFAs。Nichols等[76]在细菌中发现PLFAs 16:1ω5,而这种脂肪酸通常用于表征丛枝菌根真菌。第二,微生物生长环境会导致脂肪酸的类型及含量发生变化。比如,森林地土壤中,PLFAs 18:1ω9总是用于表征真菌群落,但是在农耕地土壤中含量特别少,不能用来指代真菌[77]。第三,PLFAs法鉴定微生物种类水平不能到种,而且由于古细菌特殊的极性脂质结构无法用PLFAs方法进行分析[78]。其次,PLFAs多样性并不能真实反映土壤微生物多样性。比如,细菌和真菌分别只有十几种主要的PLFAs,而土壤中的细菌和真菌种类大约有上千种。所以,通过PLFAs计算得到的多样性指数只能代表很小一部分的微生物多样性。总之,PLFAs主要用于分析微生物种群结构组成的变化。
4.2 遗传分析技术
土壤微生物的遗传多样性分析是微生物多样性的基础和本质。微生物在基因水平上的分类学多样性主要依靠DNA编码rRNA进行分析。16S rRNA由于细菌和古细菌系统发育分析,18S rRNA用于真菌系统发育分析。遗传多样性的研究方法都基于PCR进行分析的。下面主要介绍两种方法:PCR-DGGE、高通量测序法。
4.2.1变形梯度凝胶电泳(PCR-DGGE)PCR-DGGE法广泛应用于微生物指纹识别,是基于16S rDNA片段的碱基组成和二级结构变化进行分析检测的。运用特定的引物扩增16S rDNA靶序列,然后将扩增产物在含有梯度变性剂的聚丙烯酰胺凝胶中进行电泳后形成谱带,同样长度但序列不同的DNA片段滞留于凝胶的不同位置,这些条带的数量和位置来反映群落的优势种及其多样性[79]。Bloem和Breure[80]运用PCR-DGGE法对60个农场的土壤微生物多样性进行了监测,发现不同的土壤类型及利用方式的微生物群落和多样性不同。这一技术可以同时对多个样品进行比较分析,并且检测到群落中的优势群落信息,然后通过对条带切胶进行序列分析,从而鉴定微生物群落种类。而且相较其他分子技术,最突出的优点为操作方便、耗时短且可重复实验[81]。但是,同其他技术一样,PCR-DGGE法也只能检测到群落的部分多样性。因为,每克土壤可能拥有1000种不同的微生物,但是这种方法只能获得20~50个条带,得到的系统进化信息可能只有全部的1%[45],而且只能对500~1000 bp大小的基因片段进行分离。此外,PCR会导致一些细菌种类基因多拷贝或者拷贝之间的异质性问题,最终出现土壤微生物的多样性被过多估计的情况。
4.2.2454焦磷酸测序法(Roche 454)Roche 454法是第二代高通量测序技术之一,已被广泛应用于宏基因组研究。可对环境中全部微生物的基因进行测定,分析群体微生物的基因组成和多样性及丰富度。Roche 454法的技术路线为:设计、合成含454接头序列和Barcodede 的引物-PCR扩增-PCR产物定量、均一化-454测序仪测序-数据回归样品-生物信息学分析。笔者总结他人研究发现,关于微生物多样性研究的最大的难点是,无法获得土壤中全部微生物的种类信息。但是,第二代高通量测序技术解决了这一问题。尤其是Roche 454法与传统测序技术相比有以下几个优点:1) 实验操作简单、结果可重复性强、样品DNA直接进行测序、实验周期短。2) 测序序列长、通量高,获取信息量大,数据无偏向性,可以如实反映样品微生物全部信息。3) 可以同时对土壤微生物中的优势种群、稀有种群及未培养菌群进行检测,到达微生物种的分类水平。在草地不同放牧管理方式对土壤微生物影响的研究中,近几年越来越受重视。学者们已经采用Roche 454法对青藏高原[6]、老挝热带草原[65]、德国森林地和草地[66]的不同管理方式对土壤微生物细菌和真菌影响进行了深入的研究,获得全面有意义的研究结果。笔者总结Roche 454法主要不足之处为:1) 测序费用过高,不利于一般小规模实验研究。2) 所得数据结果信息量庞大,研究者必须从中仔细整理发现有价值、有意义的数据,且要运用到较多分析软件。总之,笔者认为,Roche 454法是推进环境微生物全面研究的革命性技术,研究者们应当充分应用发挥其优点,为相关研究做出贡献。
5研究展望
很多研究已经证实,土壤功能微生物在养分循环的各个过程中起着重要的作用,因此全面了解土壤微生物多样性及其相关功能对于草地可持续发展有着重要的意义。土壤微生物的数量、多样性及群落结构均是指示土壤质量和草地生产力的重要指标。适当的放牧改变土壤微生物的生活环境,有利于微生物的生长,增多微生物的多样性,改变微生物的群落结构,进而提高土壤质量,使草地生产能力最大化。
关于放牧对土壤微生物的影响以及通过土壤微生物各种特性评估土壤质量的研究越来越受重视。目前,虽然基本确定了应该针对微生物的哪几种指数进行分析,但是很难明确哪种指数最适于作为指示指标,为了解决这个问题,需要对土壤微生物随放牧行为的变化进行更加深入的研究,进而对于如何评价土壤质量做出合理的解释。此外,前文总结了放牧引起土壤微生物各种变化,但是还不清楚土壤微生物的数量和多样性受到抑制时土壤质量较好,还是土壤微生物产生功能冗余时表示土壤质量较好。基于以上两个问题,本文认为未来的相关研究应注意以下几点:
1)要从土壤功能微生物着手研究。首先,土壤是各种物质转化进程进行的场所,比如:土壤碳转化、氮转化、磷铁硫等元素的转化以及土壤酶的作用,这些物质的循环都是由土壤微生物驱动完成的。其次,土壤微生物数量庞大、体积微小、种类非常丰富。从宏观和整体上,即大的分类群方面难以把握,造成分析结果往往没有侧重点,缺少针对性,从而很难揭示土壤微生物受干扰行为影响发生变化的本质和规律。因此,加强功能微生物的研究,化整体为部分,将土壤某一物质转化进程和相对的功能微生物结合起来,对它们的相关关系加以研究,更有利于深入研究土壤微生物是如何响应干扰行为,能够提供更为有效、直接的基础数据。
2)要加强植物多样性和土壤微生物多样性相互关系的研究。首先,供试区域的植物种类及其多样性是构成整个环境生态体系的最重要的部分。放牧行为最先直接影响地被植物,进而对土壤微生物形成间接作用。其次,供试区域不同,植被种类不同,从而土壤微生物类群不同。放牧行为与植物多样性及土壤微生物多样性,三者间相互联系,互相影响。因此,注重这方面的研究,对于明确草地土壤微生物对放牧行为的响应机制有重要的意义。
3)要将土壤理化性质与土壤微生物相结合进行分析。土壤微生物不仅可以作为一个综合指标,单独用于评估土壤质量。而且作为土壤生化性质,又是土壤各种性质中的一个指数。因此,必须将它与土壤的理化性质相结合,才能全面合理的表征土壤质量。同时,根据它与土壤理化性质间的相互关系,可以明确土壤微生物的什么指数具体是怎么响应放牧行为的,与放牧强度间是呈正相关关系还是呈负相关关系抑或不相关。
4)最终建立生态模型,对草地生态系统做出合理预测和评估。在关于草地生态系统的研究中,对我国草地资源生态可持续发展性和可利用性进行评价和预测是很重要的一部分研究。我们对各种导致草地退化的干扰行为和草地恢复机制的研究,最终目的都是为了掌握草地退化过程中可预见性的问题,从而做出相应的对策,以期维护草地长久良好的发展下去。因此,土壤微生物作为土壤重要的生化性质,将它与其他环境指标结合,建立更加合理科学的生态模型是非常重要的一部分工作。
References:
[1]McNaughton S J. Grazing as an optimization process: grass-ungulate relationships in the Serengeti. American Naturalist, 1979, 113(5): 691-703.
[2]Dyer M I, Turner C L, Seastedt T R. Herbivory and its consequences. Ecological Applications, 1993, 3(3): 10-16.
[3]Frank D A, Groffman P M. Ungulate vs. landscape control of soil C and N processes in grasslands of Yellowstone National Park. Ecology, 1998, 79(7): 2229-2241.
[4]Bezemer T M, van Dam N M. Linking aboveground and belowground interactions via induced plant defenses. Trends in Ecology & Evolution, 2005, 20(11): 617-624.
[5]Hamilton III E W, Frank D A. Can plants stimulate soil microbes and their own nutrient supply? Evidence from a grazing tolerant grass. Ecology, 2001, 82(9): 2397-2402.
[6]Yang Y F, Wu L W, Lin Q Y,etal. Responses of the functional structure of soil microbial community to livestock grazing in the Tibetan alpine grassland. Global Change Biology, 2013, 19(2): 637-648.
[7]Qu T B, Zhang J F, Du W C,etal. Research progress on effects of grazing on diversity of microorganisms in grassland soil. Contemporary Eco-Agriculture, 2012, (3): 14-20.
[8]Bardgett R D, Wardle D A, Yeates G W. Linking above-ground and below-ground interactions: how plant responses to foliar herbivory influence soil organisms. Soil Biology and Biochemistry, 1998, 30(14): 1867-1878.
[9]Wardle D A, Nicholson K S. Synergistic effects of grassland plant species on soil microbial biomass and activity: implications for ecosytem-level effects of enriched plant diversity. Functional Ecology, 1996, 10: 410-416.
[10]Kohler F, Hamelin J, Gillet F,etal. Soil microbial community changes in wooded mountain pastures due to simulated effects of cattle grazing. Plant and Soil, 2005, 278(1-2): 327-340.
[11]Maharning A R, Mills A A, Adl S M. Soil community changes during secondary succession to naturalized grasslands. Applied Soil Ecology, 2009, 41(2): 137-147.
[12]Holt J. Grazing pressure and soil carbon, microbial biomass and enzyme activities in semi-arid northeastern Australia. Applied Soil Ecology, 1997, 5(2): 143-149.
[13]Northup B K, Brown J R, Holt J A. Grazing impacts on the spatial distribution of soil microbial biomass around tussock grasses in a tropical grassland. Applied Soil Ecology, 1999, 13(3): 259-270.
[14]Wang K H, McSorley R, Bohlen P,etal. Cattle grazing increases microbial biomass and alters soil nematode communities in subtropical pastures. Soil Biology and Biochemistry, 2006, 38(7): 1956-1965.
[15]Regan K M, Nunan N, Boeddinghaus R S,etal. Seasonal controls on grassland microbial biogeography: Are they governed by plants, abiotic properties or both? Soil Biology and Biochemistry, 2014, 71: 21-30.
[16]Bardgett R D, Leemans D K, Cook R,etal. Seasonality of the soil biota of grazed and ungrazed hill grasslands. Soil Biology and Biochemistry, 1997, 29(8): 1285-1294.
[17]Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in soil. Science, 2005, 309(5739): 1387-1390.
[18]Daniel R. The metagenomics of soil. Nature Reviews Microbiology, 2005, 3(6): 470-478.
[19]Zhao J. Effect of stocking reats on soil microbial number and biomass in steppe. Acta Agrestia Sinica, 1999, 7(3): 223-227.
[20]Zhang C X, Nan Z B. Changeable characteristics of three soil microbial groups under different grazing intensities in Loess Plateau. Pratacultural Science, 2011, 27(11): 131-136.
[21]Wang C T, Long R J, Wang G X,etal. Relationship between plant communities, characters, soil physical and chemical properties, and soil microbiology in alpine meadows. Acta Prataculturae Sinica, 2010, 19(6): 25-34.
[22]Liu S G, Ge S R, Long Z F. Studies on soil microorganism numbers andmicrobiota of degenerated rangelands in northwest region of Sichuan P R.China. Acta Prataculturae Sinica, 1994, 3(4): 70-76.
[23]Li C L, Zhao M L, Han G D,etal. The effect of grazing on soil micro-organism and soil nutrients and their seasonal dynamics inStipabrevifloraSteppe. Journal of Arid Land Resources and Environment, 2009, 23(4): 184-190.
[24]Shan G L, Chen G, Ning F,etal. Dynamics of soil microorganism and enzyme activity in typical steppe of restoration succession process. Acta Agrestia Sinica, 2012, 20(2): 292-297.
[25]Gu A X, Fan Y M, Wu H Q,etal. Relationship between the number of three main microorganisms and the soil environment of degraded grassland on the north slope of the Tianshan Mountains. Acta Prataculturae Sinica, 2010, 19(2): 116-123.
[26]Wang X Z, Sheng L X. Effect of grazing intensity on microorganisms quantity and microbial biomass of soil in grassland under protection forest of songnen plain. Journal of Animal and Veterinary Advances, 2012, 11(24): 4549-4552.
[27]Smith J L, Paul E A. The significance of soil microbial biomass estimations. Soil Biochemistry, 1990, 6: 357-396.
[28]Kennedy A C, Smith K L. Soil microbial diversity and the sustainability of agricultural soils. Plant and Soil, 1995, 170(1): 75-86.
[29]Chen G C. Soil microbial quantity determination method and its application in red soil. Chinese Journal of Soil Science, 1999, 30(6): 197-200.
[30]Gao Y C, Zhou W S, Chen W X,etal. Estimation for biomass and turnover of soil microorganisms. Chinese Journal of Ecology, 1993, 12(6): 6-10.
[31]Risser P G, Birney E C, Blocker H D,etal. The True Prairie Ecosystem[M]. Pennsylvania: Hutchinson Ross Publishing Company Stroudsburg, 1981.
[32]Ross D J, Tate K R. Microbial biomass in soil: effects of some experimental variables on biochemical estimations. Soil Biology and Biochemistry, 1984, 16(2): 161-167.
[33]Sarathchandra S U, Perrott K W, Upsdell M P. Microbiological and biochemical characteristics of a range of New Zealand soils under established pasture. Soil Biology and Biochemistry, 1984, 16(2): 177-183.
[34]Martin J P, Chapman H D. Volatilization of ammonia from surface-fertilized soils. Soil Science, 1951, 71(1): 25-34.
[35]Stillwell M A, Woodmansee R G. Chemical transformations of urea-nitrogen and movement of nitrogen in a shortgrass prairie soil. Soil Science Society of America Journal, 1981, 45(5): 893-898.
[36]Ruess R W, McNaughton S J. Grazing and the dynamics of nutrient and energy regulated microbial processes in the Serengeti grasslands. Oikos, 1987, 49(1): 101-110.
[37]Singh R S, Srivastava S C, Raghubanshi A S,etal. Microbial C, N and P in dry tropical savanna: effects of burning and grazing. Journal of Applied Ecology, 1991, 28(3): 869-878.
[38]Bardgett R D, Hobbs P J, Frostegård Å. Changes in soil fungal: bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biology and Fertility of Soils, 1996, 22(3): 261-264.
[39]Qi S, Zheng H, Lin Q,etal. Effects of livestock grazing intensity on soil biota in a semiarid steppe of Inner Mongolia. Plant and Soil, 2011, 340: 117-126.
[40]Gao Y Z, Giese M, Lin S,etal. Belowground net primary productivity and biomass allocation of a grassland in Inner Mongolia is affected by grazing intensity. Plant and Soil, 2008, 307(1-2): 41-50.
[41]Hoffmann C, Funk R, Wieland R,etal. Effects of grazing and topography on dust flux and deposition in the Xilingele grassland, Inner Mongolia. Journal of Arid Environments, 2008, 72(5): 792-807.
[42]Schneider K, Huisman J, Breuer L,etal. Ambiguous effects of grazing intensity on surface soil moisture: A geostatistical case study from a steppe environment in Inner Mongolia, PR China. Journal of Arid Environments, 2008, 72(7): 1305-1319.
[43]Zhao S. Effects of Grazing and Fenced on Soil Microbial Diversity in Stipa Steppes of Hulunbeier, Inner Mongolia[D]. Beijing: Chinese Academy of Agricultural Sciences, 2011.
[44]Li Y J, Li G, Song X L,etal. The influence of rest-grazing on the soil microbial community functional diversity ofStipabaicalensisSteppe. Acta Prataculturae Sinica, 2013, 22(6): 21-30.
[45]Casamayor E O, Schäfer H, Baeras L,etal. Identification of and spatio-temporal differences between microbial assemblages from two neighboring sulfurous lakes: comparison by microscopy and denaturing gradient gel electrophoresis. Applied and Environmental Microbiology, 2000, 66(2): 499-508.
[46]Zhang Y W, Han J G, Han Y W,etal. The content of soil micro-biomass carbon and nitrogen of different grazing Intensities on Pasture. Acta Agrestia Sinica, 2003, 11(4): 343-346.
[47]Li Q, Mayzlish E, Shamir I,etal. Impact of grazing on soil biota in a Mediterranean grassland. Land Degradation & Development, 2005, 16(6): 581-592.
[48]Schwartz M W, Brigham C A, Hoeksema J D,etal. Linking biodiversity to ecosystem function: implications for conservation ecology. Oecologia, 2000, 122(3): 297-305.
[49]Hughes A. Disturbance and diversity: an ecological chicken and egg problem. Nature Education Knowledge, 2012, 3(10): 48.
[50]Nannipieri P, Ascher J, Ceccherini M,etal. Microbial diversity and soil functions. European Journal of Soil Science, 2003, 54(4): 655-670.
[51]Insam H. Developments in soil microbiology since the mid 1960s. Geoderma, 2001, 100(3): 389-402.
[52]Johnsen K, Jacobsen C S, Torsvik V,etal. Pesticide effects on bacterial diversity in agricultural soils-a review. Biology and Fertility of Soils, 2001, 33(6): 443-453.
[53]Sugden A M. Diversity & ecosystem resilience. Science, 2000, 290(5490): 233-235.
[54]Li Z Z, Zhu L B, Lin Y C,etal. Seasonal variation of soil bacterial community under different degrees of degradation of Hulunbuir grassland. Acta Ecologica Sinica, 2010, 30(11): 2883-2889.
[55]Zhang H F, Li G, Song X L,etal. Functional diversity of soil microbial communities inStipabaicalensissteppe in Inner Mongolia as affected by different land use patterns. Chinese Journal of Ecology, 2012, 31(5): 1143-1149.
[56]Zhou W P, Xiang D, Hu W J,etal. Influences of long-term enclosure on the restoration of plant and AM fungal communities on grassland under different grazing intensities. Acta Ecologica Sinica, 2013, 33(11):3383-3393.
[57]Zhou Y K, Zhang J N, Yang D L,etal. Phospholipid fatty acid analysis of microbial community structure under different land use patterns in soil ecosystems ofLeymuschinensissteppes. Acta Prataculturae Sinica, 2011, 20(4): 27-33.
[58]Patra A K, Abbadie L, Clays-Josserand A,etal. Effects of grazing on microbial functional groups involved in soil N dynamics. Ecological Monographs, 2005, 75(1): 65-80.
[59]McCaig A E, Glover L A, Prosser J I. Molecular analysis of bacterial community structure and diversity in unimproved and improved upland grass pastures. Applied and Environmental Microbiology, 1999, 65(4): 1721-1730.
[60]Grayston S J, Griffith G S, Mawdsley J L,etal. Accounting for variability in soil microbial communities of temperate upland grassland ecosystems. Soil Biology and Biochemistry, 2001, 33(4): 533-551.
[61]Wakelin S A, Gregg A L, Simpson R J,etal. Pasture management clearly affects soil microbial community structure and N-cycling bacteria. Pedobiologia, 2009, 52(4): 237-251.
[62]Zhou X, Wang J, Hao Y,etal. Intermediate grazing intensities by sheep increase soil bacterial diversities in an Inner Mongolian steppe. Biology and Fertility of Soils, 2010, 46(8): 817-824.
[63]Howe H F, Brown J S, Zorn-Arnold B. A rodent plague on prairie diversity. Ecology Letters, 2002, 5(1): 30-36.
[64]Liu T Z, Nan Z B. Advances in nitrification and nitrifying bacteria in grassland soil. Pratacultural Science, 2011, 28(6): 951-958.
[65]Lienhard P, Terrat S, Chemidlin N. Pyrosequencing evidences the impact of cropping on soil bacterial and fungal diversity in Laos tropical graddland. Agronomy for Sustainable Development, 2013, 34: 525-533.
[66]Nacke H, Thürmer A, Wollherr A,etal. Pyrosequencing-based assessment of bacterial community structure along different management types in German forest and grassland soils. PloS One, 2011, 6(2): e17000.
[67]Ford H, Rousk J, Garbutt A,etal. Grazing effects on microbial community composition, growth and nutrient cycling in salt marsh and sand dune grasslands. Biology and Fertility of Soils, 2013, 49(1): 89-98.
[68]Schönborn W. Population dynamics and production biology of testate amoebae (Rhizopoda, Testacea) in raw humus of two coniferous forest soils. Archiv für Protistenkunde, 1986, 132(4): 325-342.
[69]Gao Y, Zhu W, Chen W. Structure of the protozoan community in soil and its ecological functions. Chinese Journal of Ecology, 2000, 19(1): 59.
[70]Visser S, Parkinson D. Soil biological criteria as indicators of soil quality: soil microorganisms. American Journal of Alternative Agriculture, 1992, 7(1-2): 33-37.
[71]Øvreås L. Population and community level approaches for analysing microbial diversity in natural environments. Ecology Letters, 2000, 3(3): 236-251.
[72]Clegg C D. Impact of cattle grazing and inorganic fertiliser additions to managed grasslands on the microbial community composition of soils. Applied Soil Ecology, 2006, 31(1): 73-82.
[73]Bardgett R D, Jones A C, Jones D L,etal. Soil microbial community patterns related to the history and intensity of grazing in sub-montane ecosystems. Soil Biology and Biochemistry, 2001, 33(12): 1653-1664.
[74]Frostegård Å, Tunlid A, Bååth E. use and misuse of PLFA measurements in soils. Soil Biology and Biochemistry, 2011, 43: 1621-1625.
[75]Schoug Å, Fischer J, Heipieper H J,etal. Impact of fermentation pH and temperature on freeze-drying survival and membrane lipid composition of Lactobacillus coryniformis Si3. Journal of Industrial Microbiology & Biotechnology, 2008, 35(3): 175-181.
[76]Nichols P, Stulp B K, Jones J G,etal. Comparison of fatty acid content and DNA homology of the filamentous gliding bacteria Vitreoscilla, Flexibacter, Filibacter. Archives of Microbiology, 1986, 146(1): 1-6.
[77]Olsson P A. Signature fatty acids provide tools for determination of the distribution and interactions of mycorrhizal fungi in soil. FEMS Microbiology Ecology, 1999, 29(4): 303-310.
[78]Lin X G. Soil Microbial Research Principles and Methods[M]. Beijing: High Education Press, 2010.
[79]Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[80]Bloem J, Breure A M. Microbial indicators. Trace Metals and other Contaminants in the Environment, 2003, 6: 259-282.
[81]Ma Y X, Holmstrm C, Webb J. Application of denaturing gradient gel electrophoresis (DGGE) in microbial ecology. Acta Ecologica Sinica, 2003, 23(8): 1561-1569.
参考文献:
[7]曲同宝, 张建峰, 杜玮超, 等. 放牧对草地土壤微生物多样性影响研究进展. 当代生态农业, 2012, (3): 14-20.
[19]赵吉. 不同放牧率对冷篙小禾草草原土壤微生物数量和生物量的影响. 草地学报, 1999, 7(3): 223-227.
[20]张成霞, 南志标. 不同放牧强度下陇东天然草地土壤微生物三大类群的动态特征. 草业科学, 2011, 27(11): 131-136.
[21]王长庭, 龙瑞军, 王根绪, 等. 高寒草甸群落地表植被特征与土壤理化性状, 土壤微生物之间的相关性研究. 草业学报, 2010, 19(6): 25-34.
[22]刘世贵, 葛绍荣, 龙章富. 川西北退化草地土壤微生物数量与区系研究. 草业学报, 1994, 3(4):70-76.
[23]李春莉, 赵萌莉, 韩国栋, 等. 放牧对短花针茅草原土壤微生物和土壤养分的影响及其季节动态. 干旱区资源与环境, 2009, 23(4): 184-190.
[24]单贵莲, 陈功, 宁发, 等. 典型草原恢复演替过程中土壤微生物及酶活性动态变化研究. 草地学报, 2012, 20(2): 292-297.
[25]顾爱星, 范燕敏, 武红旗, 等. 天山北坡退化草地土壤环境与微生物数量的关系. 草业学报, 2010, 19(2): 116-123.
[29]陈国潮. 土壤微生物量测定方法现状及其在红壤上的应用. 土壤通报, 1999, 30(6): 197-200.
[30]高云超, 朱文珊, 陈文新. 土壤微生物生物量周转的估算. 生态学杂志, 1993, 12(6): 6-10.
[43]赵帅. 放牧与围封对呼伦贝尔针茅草原土壤微生物多样性的影响[D]. 北京: 中国农业科学院, 2011.
[44]李玉洁, 李刚, 宋晓龙, 等. 休牧对贝加尔针茅草原土壤微生物群落功能多样性的影响. 草业学报, 2013, 22(6): 21-30.
[46]张蕴薇, 韩建国, 韩永伟, 等. 不同放牧强度下人工草地土壤微生物量碳, 氮的含量. 草地学报, 2003, 11(4): 343-346.
[54]李梓正, 朱立博, 林叶春, 等. 呼伦贝尔草原不同退化梯度土壤细菌多样性季节变化. 生态学报, 2010, 30(11): 2883-2889.
[55]张海芳, 李刚, 宋晓龙, 等. 内蒙古贝加尔针茅草原不同利用方式土壤微生物功能多样性.生态学杂志, 2012, 31(5): 1143-1149.
[56]周文萍, 向丹, 胡亚军, 等. 长期围封对不同放牧强度下草地植物和 AM 真菌群落恢复的影响. 生态学报, 2013, 33(11): 3383-3393.
[57]邹雨坤, 张静妮, 杨殿林, 等. 不同利用方式下羊草草原土壤生态系统微生物群落结构的PLFA分析. 草业学报, 2011, 20(4): 27-33.
[64]刘天增, 南志标. 草地硝化微生物与硝化作用研究进展. 草业科学, 2011, 28(6): 951-958.
[78]林先贵. 土壤微生物研究原理与方法[M]. 北京: 高等教育出版社, 2010.
[81]马悦欣, Holmstrm C, Webb J. 变性梯度凝胶电泳(DGGE)在微生物生态学中的应用. 生态学报, 2003, 23(8): 1561-1569.
http://cyxb.lzu.edu.cn
苟燕妮, 南志标. 放牧对草地土壤微生物的影响. 草业学报, 2015, 24(10): 194-205.
GOU Yan-Ni, NAN Zhi-Biao. The impacts of grazing on the soil microorganism population of grassland. Acta Prataculturae Sinica, 2015, 24(10): 194-205.
通讯作者*Corresponding author. E-mail: zhibiao@lzu.edu.cn
作者简介:苟燕妮(1987-), 女, 甘肃临洮人,在读博士。E-mail: gouyannigg@126.com
基金项目:国家重点基础研究发展计划(“973”计划)项目(编号:2014CB138702)资助。
收稿日期:2014-11-06;改回日期:2015-03-18
DOI:10.11686/cyxb2014460

