湖羊SPLUNC1基因序列的生物信息学分析
陈凯丽 孙延鸣++沈文++陈冬梅 +郭海英


摘要:以湖羊SPLUNC1基因为研究对象,利用生物信息学方法对其编码的蛋白进行理化性质、信号肽、跨膜区域、亚细胞定位、二级结构、三级结构、保守区域分析,并构建系统发育树。结果表明,湖羊SPLUNC1蛋白的分子量为26.53 ku,理论等电点为5.07。SPLUNC1蛋白N端存在信号肽,亚细胞定位在细胞外。SPLUNC1蛋白质的二级结构主要为α螺旋和无规则卷曲。该蛋白由4 股反平行的肽段形成的β折叠片和2个α螺旋组成的桶形结构域组成。系统发育树分析表明,湖羊的SPLUNC1蛋白在山羊、牛、猪、人、小鼠中,与山羊首先聚为一类,后与牛聚为一类,这与动物学分类结果一致。
关键词:湖羊;短的上腭、肺及鼻咽上皮克隆1(SPLUNC1);生物信息学分析
中图分类号: Q781文献标志码: A
文章编号:1002-1302(2016)02-0048-03
收稿日期:2015-01-27
基金项目:国家自然科学基金(编号:31460686)
作者简介:陈凯丽(1988—),女,河南南阳人,硕士,主要从事动物学研究。E-mail:kellyhenan@163.com。
通信作者:孙延鸣,博士,教授,主要从事临床兽医学研究。E-mail:sym@shzu.edu.cn。
短的上腭、肺及鼻咽上皮克隆1(short palate,lung and nasal epithelium clone 1,[WTBX][STBX]SPLUNC1[WTBZ][STBZ])因其特异表达于上腭、肺及鼻咽上皮而被命名。Weston等首次克隆了小鼠的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因[1]。SPLUNC1具有分泌物的生化特性[2],分布于细胞胞浆中[3]。人、小鼠、牛、猪[WTBX][STBX]SPLUNC1[WTBZ][STBZ]分别定位于20号染色体、2号染色体、13号染色体、17号染色体。研究表明哺乳类动物物种拥有共同的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]表达模式,即在口咽部、上呼吸道、唾液腺和气管表达,且在革兰阴性细菌引起的呼吸道感染中能起到抗菌及抗炎双重作用[4]。Liu等证明了SPLUNC1是吸道病原菌感染肺部黏膜固有免疫防御相关决定性成分[5]。Chu等证实SPLUNC1是一种新的宿主防御肺炎支原体蛋白,体内过表达的SPLUNC1能显著地抑制肺部肺炎支原体的载荷量[6]。此外,SPLUNC1在维持上呼吸道稳态方面也起了重要的作用[7]。目前,有关人和小鼠的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因功能已有相关报道,但未见有关羊的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]相关功能及其生物信息学分析的报道。在此前的研究中,笔者采用RACE技术,从湖羊的肺组织中克隆得到[WTBX][STBX]SPLUNC1[WTBZ][STBZ] cDNA序列,并在GenBank上登录(登录号:KJ749828.1)。本研究利用生物信息学方法对湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因序列进行分析,为进一步研究湖羊SPLUNC1蛋白的生物学功能奠定基础。
1材料与方法
1.1生物信息学数据库和软件
NCBI(美国生物技术研究中心):http://www.ncbi.nlm.nih.gov/blastEXPASY:http://www.us.expasy.ch(蛋白质数据库);MEGA 5.05软件;RasMol软件。
1.2湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因cDNA全长的获得
从湖羊肺组织中提取RNA,利用RACE技术分别扩增出[WTBX][STBX]SPLUNC1[WTBZ][STBZ]的3′末端和5′末端序列并送基因公司测序,用DNAMAN软件将测序结果进行拼接,获得湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ] cDNA 的全长序列。湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ] cDNA的全长序列的具体克隆步骤参照文献[8]。
1.3生物信息分析方法
利用在线分析工具ProtParam(http://web.expasy.org/protparam/)对湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因编码的蛋白质进行氨基酸理化分析。利用在线分析工具NetNGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc/)对湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因编码的蛋白质进行糖基化位点分析。利用在线分析工具WoLF PSORT(http://wolfpsort.org)、TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)和SignalP-3.0(http://www.cbs.dtu.dk/services/SignalP-3.0/)对湖羊SPLUNC1蛋白在真核细胞的亚细胞位置和信号肽进行分析。利用在线分析工具TMPred(http://www.ch.embnet.org/software/TMPRED_form.html)对湖羊SPLUNC1蛋白进行跨膜区分析。利用在线分析工具SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测湖羊SPLUNC1蛋白二级结构进行分析。利用NCBI的在线分析工具Blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastHome)对湖羊SPLUNC1蛋白的保守结构域进行预测。利用在线分析工具3D-PSSM(http//www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)和RasMol软件对湖羊SPLUNC1蛋白的三级结构进行分析。利用NCBI 的在线分析工具Blast对湖羊的SPLUNC1进行氨基酸序列比对。利用MEGA 5.05构建湖羊SPLUNC1蛋白的分子进化树。
2结果与分析
2.1湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因基本信息与理化性质分析
研究利用ExPaSy蛋白数据库中的ProtParam在线工具对湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因编码的蛋白质(GenBank登录号:AIG92771.1)进行氨基酸理化性质分析,结果表明,蛋白质分子式为C1206H2031N305O348S5,原子总数为3 895,蛋白质分子质量为26.53 ku,理论等电点为5.07;不稳定系数为 29.88,说明该蛋白为稳定蛋白;脂肪系数为152.08,总平均亲水性为0.704,说明该蛋白是疏水蛋白。湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因编码的255个氨基酸中含量最高的是Leu、Gly 和Val,各有62个、24个和24个,分别占24.3%、9.4%和9.4%,且不含有Pyl、 Trp和Sec。利用NetNGlyc 1.0 Server预测N-糖基化序列,发现湖羊的SPLUNC1序列中含有2个糖基化序列,分别是:NITA和NLTG。由图1可知,使用亮氨酸拉链公式:Leu-X6-Leu-X6-Leu-X6-Leu (X 指代任意氨基酸残基),在N末端发现了亮氨酸拉链序列。
[FK(W6][TPCKL1.tif][FK)]
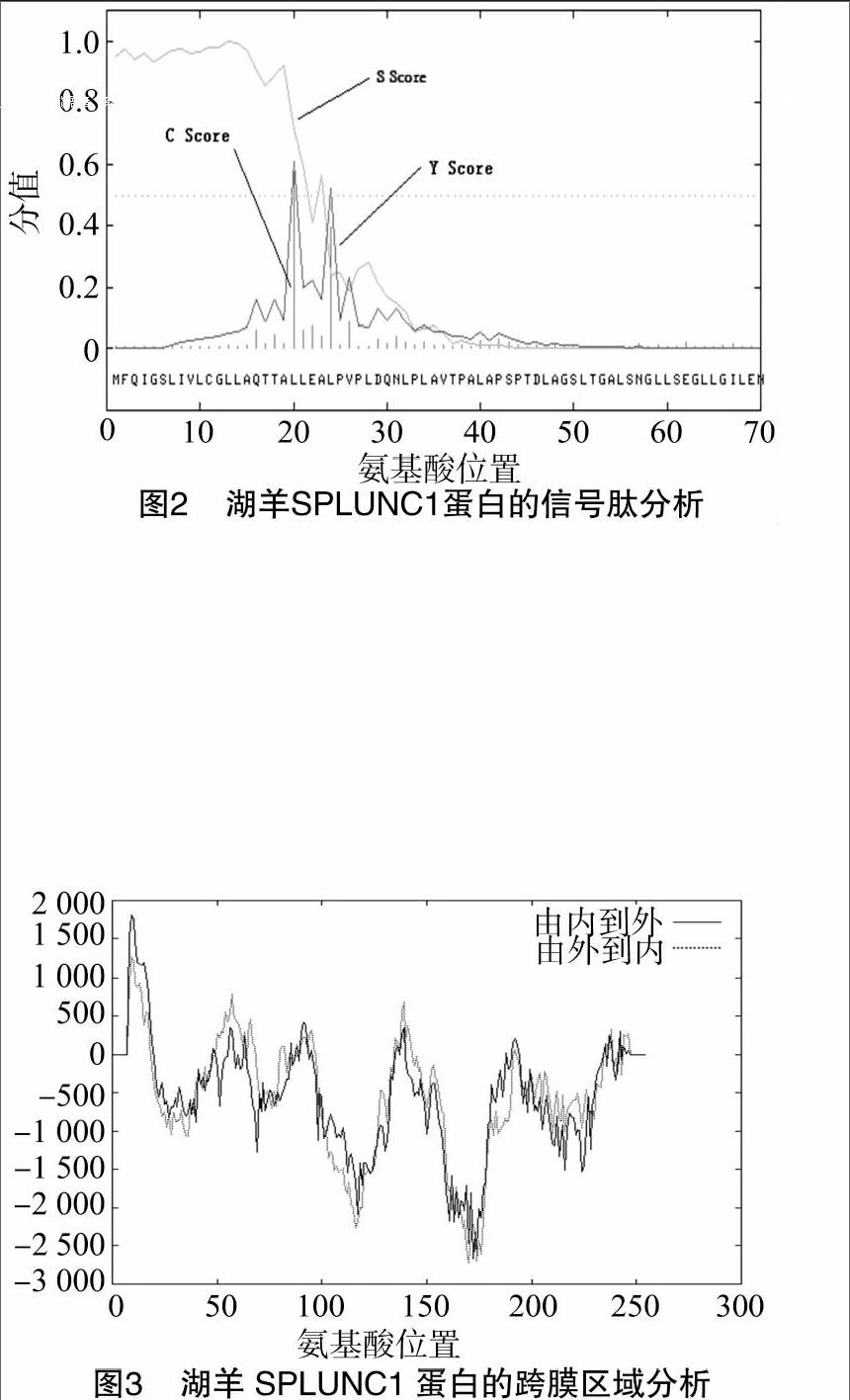
2.2湖羊SPLUNC1蛋白的信号肽、亚细胞定位及跨膜区域分析
结合Emanuelsson1等和Paul等的亚细胞定位方法对湖羊的SPLUNC1进行信号肽、亚细胞定位分析[9-10]。使用这2篇文献中提到的在线分析工具进行分析。
WoLF PSORT预测运用最邻近结点算法(k-nearest neighbor algorithm,KNN),K值为32,queryProtein details extr:28,E.R.:3 (extr即extracellular细胞外的,E.R.即内质网)。点击网页结果中的”details“查看与湖羊SPLUNC1匹配的32个蛋白中,有28个与细胞外蛋白相似,且评论大部分为分泌蛋白;有3个与内质网有关,1个与溶酶体蛋白相似。
TargetP 1.1 Server结果表明湖羊SPLUNC1具有分泌途径,且含有19个氨基酸的信号肽。
按照Emanuelsson1等的建议[9],对TargerP 1.1 Server预测的信号肽使用SignalP-3.0进行再次分析,得出的结果采用neural networks (NN)算法,湖羊SPLUNC1蛋白的信号肽切割位点为第19个氨基酸和第20个氨基酸之间(图2)。
[FK(W10][TPCKL2.tif][FK)]
利用在线TMPred工具对目标蛋白质进行跨膜区预测,结果为:有2个可能的模型值得考虑,其中一个模型是N末端在内部,有2个大的跨膜螺旋,分别位于1~21位氨基酸(由内到外),47~68位氨基酸(由外到内),总分:2 611;另一种模型是有1个大的跨膜螺旋,位于1~19位氨基酸(由外到内),总分:1 260(图3)。
[FK(W10][TPCKL3.tif][FK)]
综上所述,湖羊SPLUNC1蛋白含有1个19位氨基酸的信号肽,亚细胞定位于细胞外,有2个大的跨膜螺旋。
2.3湖羊SPLUNC1蛋白二级结构预测分析
本研究利用SOPMA工具预测该基因编码蛋白质的二级结构,结果显示参与形成螺旋(alpha helix)的氨基酸有135个、占52.94%,参与形成延伸直链(extended strand)的氨基酸有38个,占14.90%,参与形成无规则卷曲(random coil)的氨基酸有69个,占27.06%。二级结构分布见图4。
[FK(W9][TPCKL4.tif][FK)]
2.4湖羊SPLUNC1蛋白三级结构预测分析
本研究利用SWISS-MODEL蛋白质三维结构建模工具预测湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因编码蛋白质的三维结构,获得该蛋白的三维结构模型。使用SWISS-MODEL模板库匹配靶序列的进化相关结构进行Blast和HHBlits搜索,在匹配的23个模板里,模板4 kgo.1.A与湖羊SPLUNC1序列的相似最高。使用RasMol软件查看预测结果,发现该蛋白由4 股反平行的肽段形成的β折叠片和2个α螺旋组成的桶形结构域组成(图5)。
2.5湖羊SPLUNC1蛋白保守区域预测
利用NCBI的Blast比对预测湖羊SPLUNC1的结构域,由图6可见,湖羊SPLUNC1蛋白有2个结构域,其中第58个氨基酸到第233个氨基酸构成的保守结构域与LBP_BPI_CEPT结[CM(25]构域相似,第104个氨基酸到第231个氨基酸构成的保守[CM)]
[FK(W6][TPCKL5.tif][FK)]
结构域与BPI1结构域相似。
2.6湖羊SPLUNC1蛋白的系统发育树分析
使用NCBI 的Blast在线工具对湖羊SPLUNC1的氨基酸进行序列比对,结果表明,湖羊与山羊、家牛、猪、人、小鼠物种相似性为98%、92%、78%、79%、67%。利用Clustal X 1.81软件对6个物种[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因氨基酸序列进行比对,然后使
[FK(W6][TPCKL6.tif][FK)]
用MEGA 5.05的邻近法(Neighbor-Joining,NJ)基于该基因氨基酸序列的比对结果构建6个物种的系统发育树,进而反映不同物种的系统进化及亲缘关系(由图7可见,分支节点处数字为1 000次Bootstrop检验的支持百分比)。结果表明,湖羊和山羊的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因同属一个分支,亲缘关系比较密切,聚为一类,随后这2者与牛聚为一类,而后与猪聚为一类,而人和小鼠的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因单独为1支。
[FK(W9][TPCKL7.tif][FK)]
3结论与讨论
本研究通过DNAMAN、NCBI等在线工具和软件,对湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因的蛋白结构和功能进行了分析。由分析结果可知,克隆的湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因cDNA全长(GenBank登录号:KJ749828.1)为1 091 bp,含1个完整的开放阅读框(ORF)768 bp,共编码255个氨基酸。将湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因翻译的氨基酸序列与已发表文章小鼠和猪[1,11]SPLUNC1的信息进行比较,发现湖羊的SPLUNC1与小鼠在N端都有亮氨酸拉链,且排列的位置相同。小鼠的为MFLVGSLVVLCGLLAHSTAQLAGLPLPL,湖羊的为MFQIGSLIVLCGLLAQTTALLEALPVPL,猪的N端未发现,表明亮氨酸拉链在物种进化上存在差异。另外小鼠的糖基化序列是NPTD、NITA和NLTG,湖羊是NITA和NLTG,猪只有1个糖基化序列NITV。同时,在小鼠的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]序列上发现的PLPL结构域以及保守结构域蛋白激酶C(SLK)和酪蛋白激酶Ⅱ(SFVD和SGLD)在湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]上没有被发现,而猪的[WTBX][STBX]SPLUNC1[WTBZ][STBZ]序列上却发现有蛋白激酶C(SLK)序列。
最早先采用的是信号肽预测软件SignalP 4.0分析湖羊SPLUNC1蛋白,发现信号肽切割位点在23~24,而报道的小鼠和猪的信号肽切割位点在19~20之间,人的SPLUNC1 蛋白的N 端的信号肽长度为19个氨基酸[12],但是结合了亚细胞定位后,支持了信号肽切割位点为19~20。在亚细胞定位时参考了孙伟等和杜慕云等的亚细胞定位方法[13-14],对序列进行分析发现,用Signal 3.0分析有2个结果,neural networks (NN)的分析结果为信号肽切割位点为 19~20,hidden Markov models (HMM)的分析结果为信号肽切割位点为23~24。综合比较后,判断湖羊SPLUNC1的信号肽切割位点应为 19~20之间,但是仍需试验数据支持。
从近些年的研究报道中,可以看出大部分的SPLUNC1生物学研究功能主要在人和鼠上,鲜见羊SPLUNC1生物学功能的相关报道。此次研究运用生物信息学的方法对湖羊SPLUNC1蛋白的基本理化性质、信号肽、跨膜区域、二级结构等进行分析,初步预测了湖羊SPLUNC1蛋白所拥有的部分特性,以期为深入探讨其生物学功能提供理论依据。
[HS2][HT8.5H]参考文献:[HT8.SS]
[1][ZK(#]Weston W M,Leclair E E,Trzyna W,et al. Differential display identification of plunc,a novel gene expressed in embryonic palate,nasal epithelium,and adult lung[J]. The Journal of Biological Chemistry,1999,274(19):13698-13703.
[2]Campos M A,Abreu A R,Nlend M C,et al. Purification and characterization of PLUNC from human tracheobronchial secretions[J]. American Journal of Respiratory Cell and Molecular Biology,2004,30(2):184-192.
[3]肖丁良,钟礼立,梁沫,等. 肺炎支原体脂质膜蛋白诱导人支气管上皮细胞[WTBX][STBX]SPLUNC1[WTBZ][STBZ] 的表达[J]. 医学临床研究,2013,30(6):1041-1045.
[4][JP3]Di Y P,Harper R,Zhao Y,et al. Molecular cloning and characterization of spurt,a human novel gene that is retinoic acid-inducible and encodes a secretory protein specific in upper respiratory tracts[J]. The Journal of Biological Chemistry,2003,278(2):1165-1173.
[5]Liu Y,Bartlett J A,Di M E,et al. SPLUNC1/BPIFA1 contributes to pulmonary host defense against Klebsiella pneumoniae respiratory infection[J]. The American Journal of Pathology,2013,182(5):1519-1531.
[6]Chu H W,Thaikoottathil J,Rino J G,et al. Function and regulation of SPLUNC1 protein in Mycoplasma infection and allergic inflammation[J]. Journal of Immunology,2007,179(6):3995-4002.
[7]Mcgillivary G,Bakaletz L O. The multifunctional host defense peptide SPLUNC1 is critical for homeostasis of the mammalian upper airway[J]. PLoS One,2010,5(10):e13224.
[8]陈凯丽,孙延鸣,沈文,等. RACE法克隆湖羊[WTBX][STBX]SPLUNC1[WTBZ][STBZ]基因及序列分析[J]. 西北农业学报,2015,24(3):8-13.
[9][ZK(#]Emanuelsson O,Brunak S,Von Heijne G,et al. Locating proteins in the cell using TargetP,SignalP and related tools[J]. Nature Protocols,2007,2(4):953-971.
[10][ZK(#]Horton P,Park K J,Obayashi T,et al. WoLF PSORT:protein localization predictor[J]. Nucleic Acids Research,2007,35(Web Server issue):W585-W587.
[11]Larsen K,Madsen L B,Bendixen C. Porcine SPLUNC1:molecular cloning,characterization and expression analysis[J]. Biochimica et Biophysica Acta,2005,1727(3):220-226.
[12]郭小芳,陈攀,李夏雨,等. 新的天然免疫保护分子SPLUNC1的结构与功能研究进展[J]. 生物化学与生物物理进展,2012,39(10):945-951.
[13]孙伟,李达,苏锐,等. 绵羊[WTBX][STBX]YAP1[WTBZ][STBZ]基因全长cDNA克隆及生物信息学分析[J]. 中国农业科学,2013,46(8):1725-1735.
[14]杜慕云,杨仁德,李剑,等. 草菇α-淀粉酶基因的生物信息学分析[J]. 食用菌学报,2014,21(3):6-12.

