念珠菌RPB1基因片段PCR扩增条件的优化**?
吕 倩,王颜颜,刘涛华,牟丽丽,陈 燕,康颖倩
(贵州医科大学微生物学教研室,贵州贵阳 550004)
念珠菌RPB1基因片段PCR扩增条件的优化**?
吕倩,王颜颜,刘涛华,牟丽丽,陈燕,康颖倩**
(贵州医科大学微生物学教研室,贵州贵阳550004)
[摘要]目的:优化念珠菌RNA聚合酶Ⅱ大亚基(RPB1)基因片段的PCR扩增条件,筛选适宜念珠菌RPB1 的PCR扩增体系。方法:使用改良CTAB法提取念珠菌基因组DNA,分别调整念珠菌RPB1基因片段PCR扩增过程中ddH2O含量、DNA模板含量、退火温度或循环次数,比较不同反应体系的PCR扩增效果。结果:在26 μL 的PCR反应体系中,DNA模板含量2 μL、退火温度为56℃、循环40次时是念珠菌RPB1基因片段最稳定的PCR扩增反应体系,扩增得到24条皱褶念珠菌RPB1基因片段序列,上传至GenBank。结论:优化PCR扩增条件后,念珠菌RPB1基因片段的目的条带更明亮、清晰,为基因测序及深入研究念珠菌的工作奠定了基础。
[关键词]念珠菌属; RPB1基因片段; CTAB;基因扩增
**通信作者E-mail: joycekangtokyo@ qq.com
网络出版时间: 2016-02-23网络出版地址: http: / /www.cnki.net/kcms/detail/52.5012.R.20160223.2106.062.html
近年来,由于免疫抑制剂和广谱抗生素的滥用,以及艾滋病患者的增多,人群感染念珠菌呈明显上升趋势[1]。目前,除了间隔区和核糖体大亚基序列被广泛应用于念珠菌菌属的分类鉴定之外,RPB1基因片段与其他基因片段(如ITS、ACT1)相结合也广泛地应用于真菌研究中[2]。RPB1基因片段是负责编码RNA聚合酶Ⅱ最主要的功能亚基,具有单拷贝和进化速率慢的特点[3],Ku等[4-5]认为以RPB1基因作为物种之间鉴定的标准比核糖体基因有优势。本课题前期研究发现,多位点序列分型方法对皱褶念珠菌RPB1基因扩增后往往会出现目的条带不清晰、扩增特异性差等情况,不利于测序及序列分析,因此,此次研究通过探索RPB1基因片段扩增的条件,以找到最佳PCR反应体系,为今后对念珠菌RPB1基因片段的研究提供可靠的试验数据。
1 材料与方法
1.1菌种与试剂
24株经间隔区和核糖体大亚基序列鉴定过的皱褶念珠菌,由荷兰真菌多样性研究中心惠赠,见表1。CTAB缓冲液: 1 mol/L Tris-HCl (pH8.0) 50 mL + NaCl 40.91 g + 0.5 mol/L EDTA(pH8.0) 20 mL + CTAB10 g,ddH2O定容至500 mL; SEVAG(氯仿∶异丙醇= 24∶1),异丙醇,6Xloading Buffer (0.25%溴酚蓝)。TE(pH8.0) : 10 mmol/L Tris-HCl (pH8.0) +1 mmol/L EDTA(pH8.0)。PDA培养基购自日本Ltd公司,PCR Mix及DNA marker购自永博鑫生物技术公司。

表1 24株试验皱褶念珠菌种Tab.1 24 isolates of C.rugosa species included in the study
1.2提取念珠菌DNA
采用CTAB法[6]提取念珠菌基因组DNA:取菌体约60 mg于1.5 mLEP管中,加入CTAB-buffer-2X 490 μL及10%聚乙烯吡咯烷酮100 μL,Mo-Bio漩涡器充分混匀10 min,60℃水浴箱60 min,冰箱片刻使其快速降到室温,待菌落溶解后,加入SEVAG 500 μL,震荡2 min,形成乳浊液,14 000 r/ min离心10 min,收集上清液于1 mL EP管中,加入冰预冷异丙醇(体积为上清液的2/3),混匀,上下颠倒离心管,-20℃过夜培养,14 000 r/min离心10 min,弃上清,加入冰预冷70%乙醇1 mL,轻轻混匀,14 000 r/min离心2 min,弃乙醇,重复两次,室温干燥后加入1×TE 50 μL,于-20℃冰箱保存备用。
1.3调整C.pseudorugosa扩增条件
根据参考文献[7]RPB1基因片段扩增条件,对可能影响扩增效果的因素DNA模板量、ddH2O、退火温度及循环次数进行单因素调整试验。以念珠菌(CBS12857)基因组DNA为模板,以Chaves[7]扩增引物(GenBank编号EF599409) rbp1F-5'CATGTGAGCTGGTGTGTATGC和rbp1R-5' CGAGCTTGAACGTCAAATCA分别为前后引物,用PCR beads (Ready-To-Go; Amersham Pharmacia Co.Ltd.,Piscataway,NJ,USA)在26 μL反应体系中扩增,PCR反应体系含Mix 13 μL,ddH2O 10 μL,前后引物各1 μL,DNA模板浓度分别为1、1.5、2、2.5及3 μL,梯度PCR(7个不同退火温度: 53、54、55、56、57、58 及59℃) 45 s;循环次数分别为35、36、37、38、39、40次。扩增结束将产物于1.0%琼脂糖凝胶电泳(电压100 V),紫外灯下观察,并将PCR产物送测序公司测序。
2 结果
PCR产物电泳结果显示,扩增的目的条带在400 bp左右。当反应体系为Mix 13 μL、前后引物各1 μL、ddH2O 9 μL、DNA模板量2 μL、退火温度56℃、循环次数为40次时,PCR产物条带最清晰明亮,且大小完整,无明显拖带,见图1-C6;当DNA模板量为1 μL、ddH2O 10 μL时,扩增目的条带相对较淡,稍有杂带,见图1-A2;当退火温度为56℃时,虽然目的条带清晰明亮,但有明显杂带,见图1-B4,不能用于念珠菌RPB1的直接测序分析。本研究成功扩增出24株念珠菌RPB1基因片段,测序结果峰图效果理想,没有出现套峰现象。序列长度约为460 bp,GenBank登录号见表2。

表2 24株念珠菌RPB1基因扩增片段在GenBank中的菌株及序列登录号Tab.2 Strains and sequence registration number of 24 strains of Candida RPB1 gene amplification fragment in GenBank
3 讨论
本课题前期研究发现,RPB1基因在PCR扩增过程中,会出现目的条带不清晰、扩增特异性差等情况。推测RPB1基因末端的CTD结构域(C-terminal domain)在酵母菌中拷贝时,由于拷贝数量的改变引起细胞生长缺陷或死亡。因此,为保证在提取基因组DNA之前的菌株活性,选用营养条件稍高的马铃薯琼脂培养基对其进行培养,在37℃条件下活化两次。本次研究采用改良CTAB方法提取菌株DNA,通过利用冰冻试剂快速析出DNA,-20℃过夜,冰预冷2次,除去杂质,吹干后利用TE进行稀释,提高了DNA纯度。
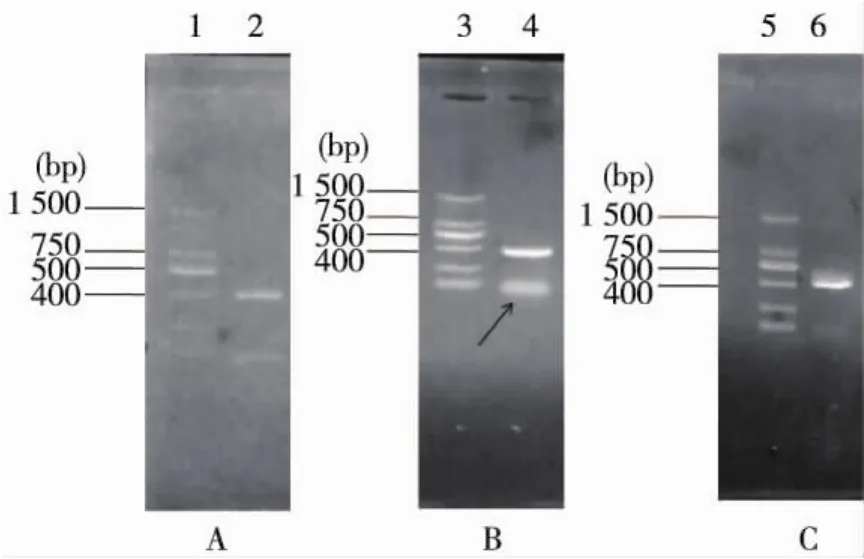
注: 1、3、5为DNA marker; 2为DNA模板量1 μL; 4为DNA模板量2 μL,退火温度55℃; 6为DNA模板量2 μL,退火温度56℃图1 念珠菌RPB1基因扩增产物凝胶电泳结果Fig.1 Result of PCR gel electrophoresis of Candida RPB1 gene
PCR反应是对皱褶念珠菌进行测序分析中的一个重要环节,它的扩增效果是反应体系和反应程序综合作用的结果[8]。本研究通过在其他因素不变的情况下,变化其中的一个因素,得到最适宜的RPB1基因片段的PCR扩增条件。首先通过改变反应体系中模板DNA的体积以找到最适的模板DNA量,试验结果表明通常加入的模板DNA体积为2μL时,扩增出的目的条带清晰。由此可见,模板DNA含量的高低会影响PCR反应的结果。模板DNA量过低,会导致PCR产物的浓度也过低,目的条带暗淡;模板DNA量过高时会导致模板与引物竞争,模板与模板之间互相结合,杂带增多。PCR反应程序中变性温度与DNA模板中G-C含量有关,G-C间由三个氢键相连,而A-T间只有两个氢键相连,相比较来说,G-C结构更加稳定。所以当模板G-C含量较高时,往往需要较高的解链温度或添加一定量的二甲基亚砜(DMSO)。试验中的引物长度为21 bp,G-C含量约为52%,不需要加入DMSO,采用常用变性温度94℃。退火温度是影响PCR特异性的较重要因素,退火温度太低,会导致目的条带不出现、不清晰或出现非目的条带;退火温度太高,扩增量会减少,有可能导致目的条带的缺失。因此适宜的退火温度能够提高扩增的特异性,在理想状态下,退火温度足够低,以保证引物同目的序列有效退火,同时还要足够高,以减少非特异性结合[9]。循环次数决定PCR扩增程度,一般选在30~40次之间,循环次数越多,非特异性产物的量亦随之增多。Mg2 +、Taq DNA酶、dNTP浓度对PCR反应体系的影响也很大。Taq DNA酶浓度过高可引起非特异性扩增,浓度过低则合成产物量减少; dNTP浓度过低会降低PCR产物的产量、影响扩增效率,浓度过高时又能与Mg2 +结合,使游离的Mg2 +浓度降低。本试验直接使用的是PCR Mix(由试剂公司将Mg2 +、Taq DNA酶、dNTP等成分混合在一起合成),降低了配制试剂过程中出现误差的几率,减少试验时间,提高实验效率。
综上,在26 μL的PCR反应体系中,DNA模板含量为2 μL、退火温度为56℃、循环40次,所得PCR产物条带最清晰明亮。在此扩增条件下,本研究成功得到了24株念珠菌的RPB1基因序列。
4 参考文献
[1]Pemán J,Zaragoza R,Salavert M.Prevention and control of nosocomial and health-care facilities associated infections caused by species of Candida and other yeasts[J].Rev Esp Quimioter,2013(4) : 298-311.
[2]Reeb V,Lutzoni F,Roux C.Contribution of RPB2 to multilocus phylogenetic studies of the euascomycetes (Pezizomycotina,fungi) with special emphasis on the lichenforming Acarosporaceae and evolution of polyspory[J].Molecular Phylogenetics and Evolution,2004(3) : 1036-1060.
[3]Murakami S.Hepatitis B virus X protein: structure,function and biology[J].Intervirology,1999(2/3) : 81-99.
[4]燕薇,沈中元,唐旭东,等.蓝叶虫微孢子虫RPB1基因片段序列的测定及其系统发育分析[J].中国农业科学,2014(23) : 4736-4744.
[5]Ku CT,Wang CY,Tsai YC,et al.Phylogenetic analysis of two putative Nosema isolates from Cruciferous Lepidopteran pests in Taiwan[J].Journal of Invertebrate Pathology,2007(1) : 71-76.
[6]吴则东,王茂芊,胡珅,等.一种适用于PCR扩增的甜菜干种子DNA快速提取方法的研究[J].中国农学通报,2013(30) : 69-72.
[7]Guilherme MC,Giselar RT,Ana Carolina BP,et al.Candida mesorugosa sp.Nov.,a novel yeast species similar to Candida rugosa isolated from a tertiary hospital in Brazil[J].Medical Mycology,2012(1) : 1-12.
[8]李立群,王小利,郑锦娟,等.影响聚合酶链式反应实验效果的基本因素[J].实验室研究与索,2011(7) :38-40.
[9]金方,陈玉如,王颜颜,等.放线菌16S rDNA片段扩增方法的优化[J].贵州医药,2014(6) : 483-486.
(2015-10-27收稿,2015-12-30修回)
中文编辑:戚璐;英文编辑:赵毅
Optimization of PCR Amplification Condition of Candida RPB1
LV Qian,WANG Yanyan,LIU Taohua,MOU Lili,CHEN Yan,KANG Yingqian
(Department of Micro-biology,Guizhou Medical University,Guiyang 550004,Guizhou,China)
[Abstract]Objective: To optimize PCR amplification condition of Candida RNA polymeraseⅡrbcl (RPB1) gene fragment,screening appropriate PCR amplification system of Candida RPB1.Methods: Adopting modified CTAB method to extract Candida genome DNA,adjusting ddH2O content of Candida RPB1 gene fragment PCR amplification process,DNA mould content,annealing or number of cycles respectively; comparing PCR amplification effect of different response system.Results: In 26 μL PCR response system,Candida RPB1 gene fragment was in the most stable PCR amplification amplification when DNA mould content was 2 μL,annealing was 56℃and number of cycles was 40; amplification yielded 24 strains of Candida rugosa RPB1 gene fragment sequence and uploaded to GenBank.Conclusion: After optimizing PCR amplification conditions,target band of Candida RPB1 gene fragment was obvious and clear,which laid the foundation of gene sequencing and in-depth research of Candida.
[Key words]Candida; RPB1 gene fragment; CTAB; gene amplification
*[基金项目]国家自然科学基金(31060006,31260029) ;贵州省社会发展科技攻关项目[黔科合SY字(2011) 3017号];贵阳市科技局社会发展与民生计划[筑科合同(201103) 16号]
[中图分类号]R34-33
[文献标识码]A
[文章编号]1000-2707(2016) 02-0135-04

