新型基因编辑技术CRISPR/Cas9系统研究现状
骞蕾阳,周志军,常岩林
(河北大学生命科学学院,河北省无脊椎动物系统学与应用重点实验室,河北保定 071002)
新型基因编辑技术CRISPR/Cas9系统研究现状
骞蕾阳,周志军,常岩林
(河北大学生命科学学院,河北省无脊椎动物系统学与应用重点实验室,河北保定071002)
摘要:规律成簇间隔短回文重复序列及其相关系统(clustered regularly interspaced short palindromic repeats/CRISPR associated,CRISPR/Cas system)是细菌和古细菌防御外来噬菌体、质粒或其他外源DNA侵染的获得性免疫系统.依据Cas蛋白种类和同源性,CRISPR/Cas系统被分为3类,其中,Ⅰ类和Ⅲ类需要多种Cas蛋白参与,而Ⅱ类系统,即CRISPR/Cas9组成简单,仅需Cas9蛋白参与即可.经过遗传工程改造后的CRISPR/Cas9已经作为一种新型的基因编辑工具被用于多种生物的基因组编辑.本文就CRISPR/Cas9系统的发现、结构组成、作用机制、研究现状、面临的困境及应用前景等几方面进行了总结.
关键词:基因编辑;CRISPR/Cas9系统;CRISPR干扰;干扰机制;基因治疗
基因编辑(gene editing)技术是指在基因组水平进行基因定点插入/缺失突变、敲除、多位点同时突变和小片段删除等精确操作的技术.目前,已报道的基因编辑技术包括锌指核酸酶(zinc-finger nucleases,ZFN)[1-2]、类转录激活因子核酸酶(transcription activator like effector nucleases,TALEN)[3-6]和新近兴起的CRISPR/Cas9[7-9],3种核酸内切酶都可以特异性识别、切割靶DNA序列,引起DNA双链断裂(DSB).其中,ZFN与TALEN复合体由多个酶亚基组成,分别执行靶DNA序列识别与内切活性.ZFN和TALEN技术都需要针对不同靶DNA序列重新构建切割工程酶,任务繁重且容易出错.CRISPR/Cas9系统是一种新兴的基因编辑技术,仅由CRISPR-derived RNA(crRNA)、反式激活crRNA(trans-activating crRNA,tracrRNA)和Cas9核酸内切酶3个元件组成[10],依靠简单的向导RNA(single-guide RNA,sgRNA)实现靶DNA序列识别,可以通过对sgRNA进行简单修改完成不同基因的靶向编辑[11-12].该系统目前已成功应用于多种生物的基因组编辑,极大地促进了功能基因研究[8]1266.本文就CRISPR/Cas9系统的发现、结构组成、作用机制、研究现状、面临的困境及应用前景等几方面进行了总结.
1CRISPR/Cas系统发现与结构组成
规律成簇间隔短回文重复序列及其相关系统(clustered regularly interspaced short palindromic repeats/CRISPR associated,CRISPR/Cas system)[13]存在于大约50%的细菌和90%的古细菌基因组中[14],是细菌和古细菌抵御外来噬菌体、质粒或其他移动元件[15]侵染的获得性免疫系统[14,16-20].CRISPR位点最早由Ishino等[21]在大肠杆菌Escherichiacoli碱性磷酸酶基因iap的侧翼序列中发现.Bult等[22]描述了产甲烷古细菌Methanocaldococcus(Methanococcus)jannaschii基因组中的18个CRISPR位点.直至Jansen等[23]对40余种原核生物基因组中的CRISPR位点进行描述后被正式命名.此后,多项研究发现CRISPR中的间隔区(spacer)与侵染细菌的病毒、质粒等外源核酸序列一致[24-26],Makarova等[27]提出CRISPR具有免疫防御作用的假说.Barrangou等[28]发现嗜热乳酸链球菌Streptococcusthermophilus的CRISPR可以通过从侵染的噬菌体基因组上获取新的间隔DNA序列,从而获得对该噬菌体的特异性免疫能力.
CRISPR由1个前导序列(Leader)、多个短而保守的重复序列区(Repeat)和多个间隔区(Spacer)组成[27,29-31](图1).Leader位于上游,没有编码活性,可能是CRISPR簇的启动子序列,具有物种特异性.不同系统的Repeats长度为23~47 bp,在同一个CRISPR排列中,Repeats是高度保守的,而且在一些近缘种间也具有一定相似性[14].Spacers是噬菌体或质粒侵入后留下的痕迹,从而赋予细胞获得对相应的噬菌体和质粒的免疫防御能力.在同一个CRISPR排列中,位于2个Repeats之间的Spacers长度相似,但序列不同[14-15,20].预测HaliangiumochraceumDSM 14365拥有最长的CRISPR序列,587个Spacers仅2个相同[15].
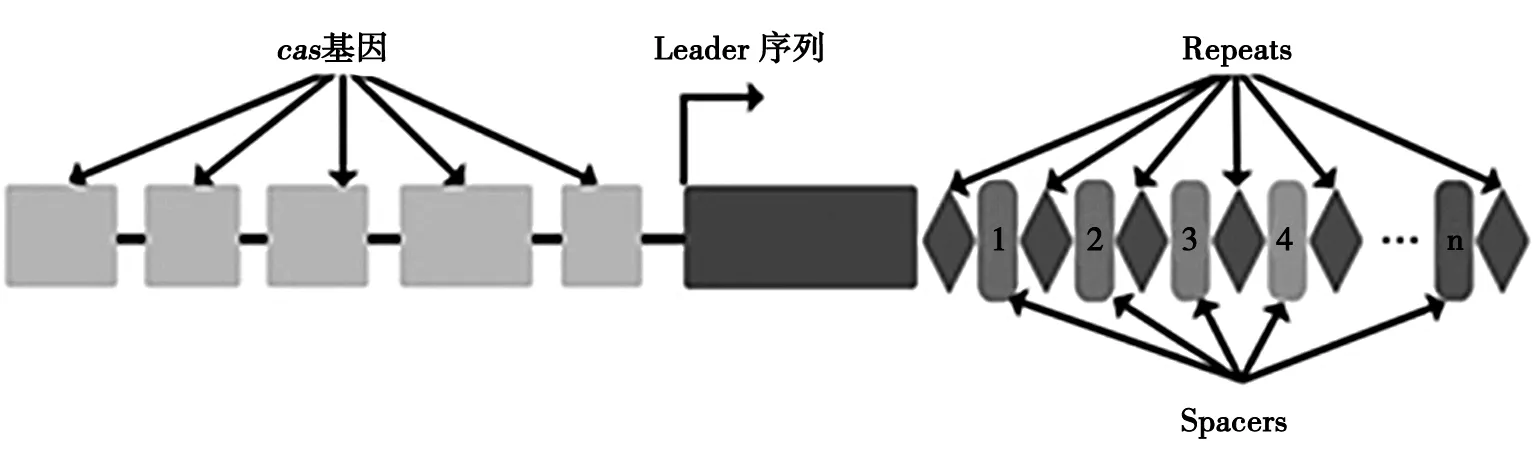
图1 细菌及古细菌中CRISPR系统的基因座组成[8]1267Fig.1 CRISPR loci in bacteria and archaea
cas基因位于CRISPR位点附近,主要编码核酸酶和DNA解旋酶等切割修饰核酸的相关的Cas蛋白[14,32].Cas是基因组剪辑和基因调控的强有力工具[19].根据CRISPR/Cas系统中Cas蛋白的种类和同源性,可将CRISPR/Cas系统区分成Ⅰ~Ⅲ 3种类型[14-15,33],Ⅰ型系统在细菌和古细菌中都有发现;Ⅱ型系统仅在细菌中发现;Ⅲ型系统主要发现于古细菌和少数细菌之中.根据cas基因的遗传信息以及所编码蛋白的结构和功能的多样性,这3种类型可分为12种亚型.它们的保守中心包括cas1到cas6 6个基因,其中cas1和cas2 是3种CRISPR/Cas系统的共有基因[14,34],而cas3、cas9和cas10 是特定的“标记”基因[32].cas1、cas2基因和Cas1蛋白对crRNA的生物合成以及靶向等与CRISPR相关的过程是不可缺少的.Cas1以序列独立的方式紧紧结合于dsDNA,Cas1和 Cas2是金属依赖性核酸内切酶,Cas1能够将dsDNA切割成短片段[32].多个CRISPR/Cas系统可共存于单个基因组,每套cas基因跟各自的CRISPR基因座功能相关[14].
2CRISPR/Cas9系统的免疫干扰机制及基因工程改造
目前,CRISPR/Cas9的研究最为深入,通过对SpyCas9 和AnaCas9 的结构分析确定了Cas9酶家族的分子构架,1个保守的结构中心包含RuvC和HNH 2个结构不同的核酶区域[33],分别负责切割目标DNA的2条链,同时也包含识别5′-NGG或5′-NAG 等与原间隔区临近基序(protospacer adjacent motif,PAM)[35-37]的结构.Cas9酶在apo状态下接收并催化不活跃的构象,DNA识别和切割也需要构象激活[38].
CRISPR/Cas9系统的作用机制分为3个阶段:间隔序列获取,crRNA表达、加工和成熟,免疫干扰[32-34].当噬菌体或质粒首次侵染细菌或古细菌时,Cas9核酸内切酶通过PAM信号识别并切割靶向DNA序列[15,19,39],酶切产生的DNA短片段被插入到前导序列和第1个重复序列之间,并伴随重复序列复制[40].当该噬菌体或质粒再次侵染细菌或古细菌时,在前导序列调控下,CRISPR被转录生成crRNA前体(pre-crRNA),Cas操纵子上游与pre-crRNA重复序列互补的tracrRNA也被同时转录合成[41],tracrRNA能够激发Cas9和双链RNA特异性RNase Ⅲ核酸酶对pre-crRNA进行加工,形成包含保守重复序列和间隔区的成熟crRNA[14,39,42].crRNA与tracrRNA通过碱基配对结合形成双链RNA[42-43],并与Cas9结合形成复合体[44].该复合体识别并结合与crRNA互补的靶DNA序列,使其双链解开形成R-loop,Cas9的HNH核酶区识别、切割与crRNA配对的靶DNA互补链;而RuvC核酶区切割处于游离状态的非互补链[19,29,44-45](图2),最终在靶DNA中PAM上游3nt 处双链断裂(double strand broken,DSB).
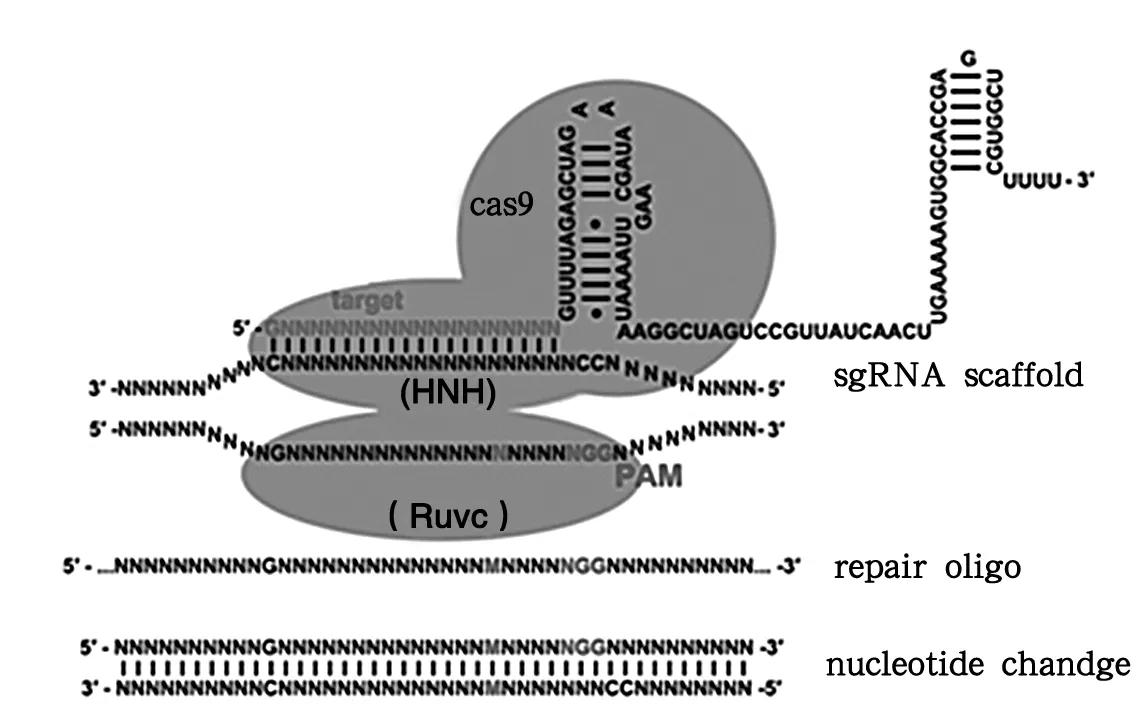
图2 CRISPR/Cas9免疫干扰示意[45]248 Fig.2 diagram of CRISPR/Cas9 immune interference
经过遗传工程改造的CRISPR/Cas9系统已作为一种新型的基因编辑工具被广泛使用[46].该系统仅包括1个Cas9蛋白和1条sgRNA[47],其中,sgRNA由tracrRNA和crRNA融合形成.Cas9 蛋白作为核酸酶切割双链DNA,而sgRNA则通过碱基互补配对决定靶序列的特异性,靶序列可以是DNA双链的任意链.基因组中的靶DNA序列与crRNA存在约20 bp的互补配对区,靶DNA序列3′末端的PAM(5′-NGG-3′)为Cas9识别位点,不能包含在sgRNA之中,切割导致靶DNA双链断裂[37,48-49].双链断裂后的靶DNA通过HR或NHEJ途径修复,以NHEJ为主,该方式较HR出错率更高.修复过程将在靶DNA序列引入插入/缺失突变[35,50-51],修复完成后,CRISPR/Cas9系统将再次作用于那些没有引入突变的靶DNA序列[11].利用载体,或通过与CPP(cell-penetrating peptide)连接/络合的方式将Cas9蛋白和sgRNA导入受体细胞,或直接将编码Cas9蛋白和sgRNA的mRNA注入受精卵等方法,快速生成靶向突变个体.CRISPR/Cas9系统在靶向编辑新的基因位点时,无需重新构建核酸内切酶,仅需根据靶基因序列对sgRNA做少许修改即可,相比于其他基因组编辑技术,靶向突变新基因位点的效率显著提高[52].CRISPR/Cas9系统不仅可以靶向编辑单个基因,而且可以组合多条sgRNA同时完成多个基因位点的靶向编辑.Cong等[53]基于在细菌/古细菌基因组中CRISPR系统基因座的间隔序列排列形式,最先使用单一CRISPR/Cas9系统同时完成了EMX1和PVALB的靶向编辑,并利用CRISPR/Cas9系统形成双链断裂完成了EMX1的大段删除.Mali等[54]对人基因组中潜在的sgRNA资源进行了估算,发现约190 kb独特的sgRNA 可以靶向约40.5%外显子.
3CRISPR/Cas9系统在介导基因敲除研究中的应用
CRISPR/Cas9系统作为一种最新涌现的基因编辑工具,由于能够完成RNA导向的DNA识别及编辑,为构建更高效的基因定点修饰技术提供了全新平台.除在细菌中被广泛使用之外,CRISPR/Cas9系统还被用于人、老鼠、斑马鱼、果蝇、酵母、线虫和农作物细胞内目标基因的删除、插入、激活或抑制.
3.1CRISPR/Cas9系统在植物基因敲除研究中的应用
CRISPR/Cas9系统已在拟南芥、烟草、水稻、玉米、高粱、小麦、甜橙和番茄等多个植物物种中得到了应用.地钱Marchantiapolymorpha被认为是研究陆生植物进化的理想模型.Sugano等[55]利用CRISPR/Cas9系统对地钱编码生长素应答因子1(ARF1)进行了定点突变.在该项研究中,地钱的U6启动子(MpU6-1pro)被用于构建针对ARF1基因设计sgRNA序列的表达载体;而经人类密码子优化后的hCas9则被35Spro或自身MpEFpro驱动过表达.利用农杆菌转化法将含有hCas9和sgRNA的表达载体导入地钱萌芽孢子.通过1-萘乙酸(NAA)和潮霉素筛选hCas9和sgRNA同时表达的转导细胞,并通过RFLP多态性分析最终证实在NAA中存活的地钱ARF1靶位点存在突变.Feng等[56]利用农杆菌侵染方法稳定转化拟南芥和水稻,除SPP sgRNA1仅5%之外,突变效率达26%~84%.Zhang等[57]将CRISPR/Cas9系统用于水稻riceoutermostcell-specificgene5(ROC5),stromalprocessingpeptidase(SPP) 和youngseedlingalbino(YSA)基因编辑.针对上述目的基因设计sgRNA,使用水稻自身的U6RNAs启动子表达sgRNA.使用的hSpCas9优化编码序列由CaMV35S启动子驱动.为提高转运效率,sgRNA和hSpCas9被亚克隆到同一表达载体中,通过农杆菌转化法导入水稻组织培养中[56].被编辑的基因能够传递到下一代,在第1代水稻中出现了所编辑的目标基因的纯合体.表明CRISPR/Cas9系统可以有效地诱导水稻中目标基因的编辑.Zhou 等[35]利用CRISPR/Cas9系统在水稻原生质体中实现115~245 kb 长染色体大片段(包含2~3 个不同的基因簇)删除.
3.2CRISPR/Cas9系统在构建人类疾病动物模型中的应用
人类疾病动物模型对病理研究及临床治疗非常重要.CRISPR/Cas9系统是继锌指核酸酶(ZFN)、ES 细胞打靶和 TALEN 等技术后,又一种可用于定点构建基因敲除大、小鼠动物的方法,且有效率高、速度快、生殖系转移能力强及简单经济的特点,在许多人类重要疾病动物模型构建的应用前景非常广阔.Li等[58]使用CRISPR/Cas9系统构建了Uhrf2基因敲除小鼠模型,Mc3R和Mc4R双基因敲除的大鼠和小鼠模型.研究结果证明通过向小鼠受精卵中注射编码CRISPR-Cas系统的mRNA比DNA在胚胎中产生定点突变的效率更高;且不受小鼠遗传品系限制,能够对基因组的大片段DNA进行删除;同时注射针对不同基因设计的sgRNA序列能够在同一只小鼠(或大鼠)中产生多个基因突变.
Yoshimi等[59]将Cas9 mRNA和靶向大鼠毛色相关的络氨酸酶基因(Tyr)的sgRNA显微注射入Wistar大鼠受精卵雄性原核,PCR扩增分析发现41.2%的Tyr位点表现出大量插入/缺失突变.将Cas9-sgRNA处理后的2细胞期胚胎转移到代孕母体中,幼鼠DNA序列分析发现Tyr位点都携带插入/缺失突变,并且Tyr位点都是杂合或嵌合式的,说明Cas9-sgRNA介导的突变能够稳定地传递到下一代.将针对Tyrc基因的sgRNA、Cas9 mRNA和80 bp的TyrC等位基因的单链寡聚脱氧核糖核酸(single stranded oligodeo xynucleotide,ssODN)同时注入F344大鼠胚胎,结果发现,7.7%的子代个体不再是白化皮毛,成为头部有颜色的非刺鼠.序列分析发现23.1%的幼鼠的Tyr位点有插入缺失突变,7.7%的幼鼠有精确的单核苷酸多态性交换.
3.3CRISPR/Cas9系统在人细胞系基因组编辑中的应用
Sakuma等[18]研究表明由Cas9和多个sgRNA 形成的复合CRISPR/Cas9系统可同时编辑多个靶基因,将经过修改同时表达Cas9核酸酶和7个sgRNA的pX330导入人HEK293T细胞.PCR分析表明:同时表达7个sgRNA的复合CRISPR/Cas9系统的诱导突变效率与表达单个sgRNA的系统相同.Ramakrishna等[60]在Cas9 的C端添加了1个半胱氨酸Cys,通过Cys游离的SH残基与转膜肽(cell-penetrating peptide,CPP)第1个氨基形成硫醚键(4-maleimidobutyryl-4G9R4L,m9R)连接形成Cas9-m9R;sgRNA和9R(包含1个Cys,3个Gly,9个Arg,4个 Leu和 1个Cys的CPP)在一定的比例下混合形成带正电的纳米粒子.通过将Cas9和sgRNA与CPP连接(或混合)促进Cas9和sgRNA跨膜转运进入人HEK293T细胞、HeLa细胞和 NCCIT细胞、皮肤纤细胞和胚胎干细胞对CCR5基因进行突变.
3.4CRISPR/Cas9系统在插入标签基因研究中的应用
Cas9对DNA 双链的切割分别由2个功能结构域执行,1个结构域突变后的Cas9成为仅能切割DNA 1条链的切口酶,通过引入修复模板,以极低的突变活性完成同源定向修复,显著降低了Cas9 切割双链引起的随机突变概率[53].Zhang等[61]利用CRISPR/Cas9系统对约氏疟原虫丝氨酸蛋白酶基因Pysera1进行编辑,构建1个包含46 bp标签DNA的质粒pYC-Pysera1,Pysera1的2个纯合区域被此DNA序列隔开.针对Cas9-sgRNA介导的可能导致突变的靶位点,设计了2个sgRNA靶向Pysera1外显子2的3′端,形成质粒pYC-sera1-sgRNA1和pYC-sera1-sgRNA2.通过电转化将质粒导入P.yoelii17XNL菌株.亲本17XNL和转染质粒的约氏疟原虫基因组DNA的PCR分析表明,在sgRNA1和sgRNA2的指导下,左右2个纯合臂在特定位点成功融合.荧光蛋白或报告基因标记技术常被用于研究蛋白质亚细胞分布和相互作用,利用CRISPR/Cas9系统能够在约氏疟原虫内源基因Py03652中插人荧光蛋白基因gfp,首先构建pYC-Py03652-gfp,在Py03652基因开放阅读框的3′端(710 bp)与3′端非编码区(779 bp)之间插入gfp基因.Py03652基因编码早期的转录膜蛋白.针对需要插入的荧光蛋白基因,设计sgRNA,构建载体pYC-Py03652-gfp-sgRNA.转染后,获得报告基因与Py03652基因3′末端融合的基因重组寄生虫.通过荧光激活细胞分选分析发现,38%的细胞存在绿色荧光蛋白的表达.
3.5CRISPR/Cas9系统在功能基因研究中的应用
基于CRISPR/Cas9系统发展而来的CRISPR干扰(CRISPRi)技术是一种新的、高度特异性的工具,该技术能够在不改变DNA序列的情况下,通过阻遏转录的延伸和RNA聚合酶的结合来实现类似RNAi的效果[62].目前,CRISPRi技术已经被成功地用于细菌、酵母、小鼠、人类等多种模式生物的功能基因转录调控研究[19,62].通过突变使Cas9的2个核酶区域失活,形成可以与靶DNA结合但没有核酶活性的缺陷Cas9(dCas9),dCas9在sgRNA的指导下与靶DNA启动子结合,特异性阻止RNA聚合酶与该启动子序列结合或作为转录终止子阻断RNA聚合酶的继续运行[63].dCas9与sgRNA复合体靶向乳糖途径的调控基因和结构基因,能够出现所需要的表型.利用该平台,在sgRNA的指导作用下,dCas9与转录抑制结合位点相互作用能够激活基因表达.通过将特定的转录激活因子和抑制因子与Cas9复合体融合,能够精确地激活或沉默基因表达.如果对CRISPR转录因子(Cas9转录效应物区域融合蛋白)和sgRNA重编程,能够对靶位点的基因表达产生预期的影响.顺着基因启动子区域,利用多个sgRNA靶向多个位点能够调节这种效果[63].在细菌细胞中,这种沉默可以逆转且具有可诱导性和高度特异性,通过在sgRNA碱基配对区域引入1个或几个错配碱基,或与基因不同区域靶向作用以调节抑制效率.多条sgRNA可以同时调控多个基因表达,也可以协同地控制单个基因以加强抑制或调整基因表达的抑制水平[64].基因治疗(gene therapy)指将外源正常基因导入靶细胞,以纠正或补偿因基因缺陷和异常引起的疾病,以达到治疗目的.Gilbert等[65]利用CRISPRi技术抑制人HeLa细胞的转铁蛋白受体(CD71)和C-X-C趋化因子受体(CXCR4)基因表达.针对靶基因编码区双链设计的10个sgRNA横跨转录起始点的上、下游500 bp,3/10的sgRNA抑制率达60%~80%,RNA 测序表明CRISPRi介导的转录抑制具有很高的靶向特异性.Feng等[66]通过电转化将绿色荧光蛋白融合表达载体U6sgRNA-Cas9-2A-GFP导入人骨肉瘤KHOS和U-2OS细胞系,结果显示CRISPRi技术可以显著抑制CDK11蛋白表达.罗斌[67]发现CRISPRi技术可以下调人肺癌细胞系A549中乳酸脱氢酶A(lactate dehydrogenase,LDHA)基因的mRNA和蛋白表达水平,抑制癌细胞增殖.
4CRISPR/Cas9系统应用的影响因素
4.1脱靶效应
CRISPR/Cas9不仅靶向编辑目标DNA位点,而且也会切割那些与靶位点序列相同(或高度相似)的其他DNA位点,使其产生突变,即脱靶效应(off-target effect)[62].脱靶效应是基于CRISPR/Cas9的基因编辑技术所面临的最主要问题.
首先,由于Cas9蛋白对sgRNA 5′端序列与靶位点间的错配不敏感,其脱靶风险较ZFN和TALEN方法更高.为了提高靶向特异性,研究人员通过点突变灭活核酸内切酶结构域(RuvC-Ⅰ)构建了1种仅切割靶DNA单条链的突变型Cas9蛋白(Cas9 D10A).为了形成靶DNA双链断裂,需要2条sgRNA同时引导Cas9 D10A识别并切割靶DNA双链的相邻位置.这种突变后的“双切口酶”产生双链断裂效率和野生型Cas9相同,但由于2个sgRNA 具有加倍的识别序列长度,因此可以极大降低脱靶突变,特异性提高50~1 500倍,显著提高了靶位点的特异性[53-54,62-63].
其次,PAM序列也可影响CRISPR/Cas9介导的基因组编辑效率和特异性.CRISPR/Cas9作用于靶DNA序列,不仅依赖sgRNA的指导,而且与入侵DNA序列中和crRNA靶序列连接的2~5 nt 的PAM序列有关[33].不同来源的Cas9蛋白识别不同的PAM序列,如:酿脓链球菌PAM为NGG,脑膜炎双球菌的PAM为N-GGNG和 NNAGAAW[62].CRISPR/Cas9在基因组编辑中的特异性与Cas9蛋白识别的PAM序列长度有关.一般而言,CRISPR/Cas9脱靶突变率随PAM序列增长而降低,其原因主要是不同长度的PAM序列在基因组中潜在的靶位点数目不同,如:PAM NGG和NAG在基因组中很可能每8 nt可以发现1个靶位点,而NGGNG和NNAGAAW则分别要每32 nt和256 nt 才有1个靶位点[62].延长PAM序列虽然能够提高CRISPR/Cas9靶向特异性,但却限制了靶DNA序列中潜在作用位点数目.在具体研究中,CRISPR/Cas9系统需要根据靶基因选择不同的PAM.参考基因组数据将有助于合适的靶位点筛选.
再次,CRISPR/Cas9用量与脱靶效应产生相关,虽然减小sgRNA用量可以降低脱靶效应,但不利于靶基因断裂.因此在高目标效应和低脱靶效应之间存在一个最优平衡点,在实际应用中需要进行优化.
最后,sgRNA在基因组中潜在互补位点多少与脱靶效应有关,每个基因都有成百上千的位点可以进行编辑,通过选择潜在互补位点少的区域设计合成sgRNA可以降低脱靶效应,提高CRISPR/Cas9特异性[62].通过优化sgRNA 设计可以提高该系统的成功率和突变效率.为了寻找到最佳的基因组序列设计sgRNA,Hsu等[68]建立了一个Web版的、可以识别几乎任何一个基因计算机模型帮助研究人员寻求最佳编辑位点.Xiao等[69]报道了一个本地版的潜在脱靶位点分析软件CasOT,该软件包可以识别任意基因组或序列中的潜在脱靶位点.
4.2sgRNA的设计合成和效率
sgRNA的指导效率是限制CRISPR/Cas9系统应用的另一个重要因素.一些人工设计合成的sgRNA指导效率低下可能与靶DNA位点所处的染色质状态及sgRNA发夹结构等因素有关[62].RNA聚合酶Ⅱ转录生成的mRNA需要经过复杂的转录后加工和修饰过程,mRNA形成后会被转运至细胞质,而Cas9/sgRNA仅仅用于核基因组DNA,目前尚没有找到使用RNA聚合酶Ⅱ生成sgRNA的方法.目前体内生成sgRNA主要依赖RNA聚合酶Ⅲ、U3snRNA和U6snRNA启动子.由于U3snRNA和U6snRNA作为管家基因,在各种组织中均有表达,无法特异性地在某种细胞和组织中产生sgRNA;而且对很多生物的U3和U6启动子都缺少研究,缺乏商业化的RNA聚合酶Ⅲ,都在一定程度上限制了基于U3和U6启动子生成sgRNA[62].Gao等[70]设计了1个可以被任何启动子转录的人造基因RGR,该基因最初转录生成1个2端具有核酶序列的sgRNA分子,经自我催化切割形成成熟的sgRNA分子,之后便可成功地诱导体外和酵母内的序列特异性切割.这样,如果选择合适的启动子,就会允许细胞和组织特异性基因组编辑.Heigwer等[71]报道的Web版sgRNA设计软件E-CRISP使用序列比对软件Bowtie2可评估脱靶效应和靶位点同源性.
4.3CRISPR/Cas9组件转移方式有限
在利用CRISPR/Cas9系统进行基因组靶DNA序列编辑时,如何将Cas9蛋白和sgRNA转运至靶细胞中至关重要.通过农杆菌转化法(agrobacterium mediated transformation)使目的基因进入植物细胞,并将其插入到植物细胞的染色体DNA上,使目的基因的遗传特性得以稳定维持和表达,是将目的基因导入植物细胞应用最广的方法[55].对于动物细胞而言,虽然可以通过显微注射技术将Cas9蛋白和sgRNA,或表达Cas9蛋白和sgRNA的mRNA注射入靶细胞,但显微注射每次只能注射1个细胞,而且不同类型的靶细胞和组织转染效率差异显著[62].Ramakrishna等[60]将Cas9和sgRNA与CPP连接导入人胚胎干细胞、皮肤成纤维细胞、HEK293T细胞、HeLa细胞和胚胎癌细胞系.Wagner等[72],Jiang等[73]通过电转化将编码sgRNA和Cas9蛋白的质粒转入靶细胞,实现了基因敲除,但电转化效率要取决于细胞所处生长期.Gilbert等[65]通过慢病毒转染将sgRNA和Cas9的表达载体导入受体细胞.但慢病毒表达时间较慢,荧光表达所需时间较长.此外,逆转录病毒载体只能感染分裂期细胞,而且容量有限,腺病毒一般不能整合到染色体上,只能进行瞬时感染.因此,在使用CRISPR/Cas9介导的基因编辑过程中仍然面临可供选择的范围有限,今后需要进一步加强新的转运工具开发以提高转运效率.
5结语和展望
CRISPR/Cas9是继ZFN和TALEN之后的第3代基因组编辑技术.这些根据DNA切割酶,即核酸酶开发的基因编辑技术针对特定靶基因订制核酸酶难度极高,而CRISPR/Cas9系统的Cas蛋白不具有特异性,只需合成1个sgRNA即可实现对靶基因的特异性编辑.尽管由于发现时间较短,在脱靶效应、sgRNA设计以及组件转运等方面存在一些应用瓶颈,但CRISPR/Cas9系统组成简单,操作方便,价格低廉,操作周期短,突变效率高,堪称基因组编辑技术的一次非常重要的技术革新[74],在基础理论研究、综合生物学和基因治疗等领域研究中具有不可估量的应用前景[75].相信通过科研人员的不懈努力,CRISPR/Cas9基因组编辑技术的效率和特异性都会进一步提高,发展成为更可靠、方便的基因编辑工具[63],应用于农作物定向育种、快速阐明基因功能、构建人类疾病动物模型和基因治疗(如血液病、肿瘤和其他遗传疾病)等多个领域,快速对整个基因组进行功能筛选,帮助人们鉴定涉及特定疾病的基因,更好地了解疾病以及开发出新的治疗方法,而CRISPR/Cas9 对诱导人多能干细胞的基因编辑和未来人类遗传疾病的治疗具有非常重要的应用前景.
参考文献:
[1] DREIER B,FULLER R P,SEGAL D J,et al.Development of zinc finger domains for recognition of the 5'-CNN-3' family DNA sequences and their use in the construction of artificial transcription factors[J].Journal of Biological Chemistry,2005,280(42):35588-35597.DOI:10.1074/jbc.M506654200.
[2] BIBIKOVA M,GOLIC M,GOLIC K G,et al.Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc finger nucleases[J].Genetics,2002,161(3):1169-1175.
[3] TESSON L,USAL C,MENORET S,et al.Knockout rats generated by embryo microinjection of TALENs[J].Nature Biotechnology,2011,29(8):695-696.DOI:10.1038/nbt.1940.
[4] 沈延,肖安,黄鹏,等.类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术[J].遗传,2013,35(4):395-409.DOI:10.3724/SP.J.1005.2013.00395.
SHEN Yan,XIAO An,HUANG Peng,et al.TALE nuclease engineering and targeted genome modification[J].Hereditas,2013,35(4):395-409.DOI:10.3724/SP.J.1005.2013.00395.
[5] 沈延,黄鹏,张博.TALEN构建与斑马鱼基因组定点突变的实验方法与流程[J].遗传,2013,35(4):533-544.DOI:10.3724/SP.J.1005.2013.00533.
SHEN Yan,HUANG Peng,ZHANG Bo.Aprotocol for TALEN construction and gene targeting in zebrafish[J].Hereditas,2013,35(4):533-544.DOI:10.3724/SP.J.1005.2013.00533.
[6] 杨翠翠,佟慧丽,马兴红,等.利用TALEN技术在牛胎儿成纤维细胞中敲除Myostatin基因[J].遗传,2014,36(7):685-690.DOI:10.3724/SP.J.1005.2014.0685.
YANG Cuicui,TONG Huili,MA Xinghong,et al.Myostatin knockout in bovine fetal fibroblasts by using TALEN[J].Hereditas,2014,36(7):685-690.DOI:10.3724/SP.J.1005.2014.0685.
[7] 殷利眷,胡斯奇,郭斐.CRISPR-Cas9基因编辑技术在病毒感染疾病治疗中的应用[J].遗传,2015,37(5):412-418.DOI:10.16288/j.yczz.14-460.
YIN Lijuan,HU Siqi,GUO Fei.The application of CRISPR-Cas9 gene editing technology in viral infection diseases[J].Hereditas,2015,37(5):412-418.DOI:10.16288/j.yczz.14-460.
[8] 李君,张毅,陈坤玲,等.CRISPR-Cas系统:RNA靶向的基因组定向编辑新技术[J].遗传,2013,35(11):1265-1273.DOI:10.3724/SP.J.1005.2013.01265.
LI Jun,ZHANG Yi,CHEN Kunling,et al.CRISPR/Cas:a novel way of RNA-guided genome editing[J].Hereditas,2013,35(11):1265-1273.DOI:10.3724/SP.J.1005.2013.01265.
[9] 王延鹏,程曦,高彩霞,等.利用基因组编辑技术创制抗白粉病小麦[J].遗传,2014,36(8):848.
[10] CHEN H F,CHOI J,BAILEY S.Cut site selection by the two nuclease domains of the Cas9 RNA-guided endonuclease[J].Journal of Biological Chemistry,2014,289(19):13284-13294.DOI:10.1074/jbc.M113.539726.
[11] SASAKI H,YOSHIDA K,HOZUMI A,et al.CRISPR/Cas9-mediated gene knockout in the ascidian Ciona intestinalis[J].Development Growth Differentiation,2014,56(7):499-510.DOI:10.1111/dgd.12149.
[12] BAKER M.Gene editing at CRISPR speed[J].Nature Biotechnology,2014,32(4):309-312.DOI:10.1038/nbt.2863.
[13] LI M H,YANG H H,ZHAO J,et al.Efficient and heritable gene targeting in tilapia by CRISPR/Cas9[J].Genetics,2014,197(2):591-599.DOI:10.1534/genetics.114.163667.
[14] NORAIS C,MOISAN A,GASPIN C,et al.Diversity of CRISPR systems in the euryarchaeal pyrococcales[J].RNA Biology,2013,10(5):659-670.DOI:10.4161/rna.23927.
[15] BISWAS A,GAGNON J N,BROUNS S J,et al.CRISPR target:bioinformatic prediction and analysis of crRNA targets[J].RNA Biology,2013,10(5):817-827.DOI:10.4161/rna.24046.
[16] SHEN B,ZHANG W S,ZHANG J,et al.Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects[J].Nature Methods,2014,11(4):399-402.DOI:10.1038/nmeth.2857.
[17] STERNBERG S H,REDDING S,JINEK M,et al.DNA interrogation by the CRISPR RNA-guided endonuclease Cas9[J].Nature,2014,507(7490):62-67.DOI:10.1038/nature13011.
[18] SAKUMA T,NISHIKAWA A,KUME S,et al.Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system[J].Scientific Reports,2014,4:5400.DOI:10.1038/srep05400.
[19] JINEK M,JIANG F G,TAYLOR D W,et al.Structures of Cas9 endonucleases reveal RNA-mediated conformational activation[J].Science,2014,343(6176):1215-1227.DOI:10.1126/science.1247997.
[20] AUER T O,BENE D F.CRISPR/Cas9 and TALEN-mediated knock-in approaches in zebrafish[J].Methods,2014,69(2):142-150.DOI:10.1016/j.ymeth.2014.03.027.
[21] ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion inEscherichiacoli,and identification of the gene product[J].Journal of Bacteriology,1987,169(12):5429-5433.
[22] BULT C J.Complete genome sequence of the methanogenic archaeon,Methanococcusjannaschii[J].Science,1996,273(5278):1058-1073.DOI:10.1126/science.273.5278.1058.
[23] JANSEN R,EMBDEN J D,GAASTRA W,et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Molecular Microbiology,2002,43(6):1565-1575.DOI:10.1007/BF02490464.
[24] MOJICA F J,DIEZ-VILLASENOR C,GARCIA-MARTINEZ J,et al.Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J].Journal of Molecular Evolution,2005,60(2):174-182.DOI:10.1007/s00239-004-0046-3.
[25] POURCEL C,SALVIGNOL G,VERGNAUD G.CRISPR elements inYersiniapestisacquire new repeats by preferential uptake of bacteriophage DNA,and provide additional tools for evolutionary studies[J].Microbiology,2005,151(Pt 3):653-663.DOI:10.1099/mic.0.27437-0.
[26] BOLOTIN A,QUINQUIS B,SOROKIN A,et al.Clustered regularly interspaced short palindrome repeats(CRISPRs) have spacers of extrachromosomal origin[J].Microbiology,2005,151(Pt 8):2551-2561.DOI:10.1099/mic.0.28048-0.
[27] MAKAROVA K S,GRISHIN N V,SHABALINA S A,et al.A putative RNA-interference-based immune system in prokaryotes:computational analysis of the predicted enzymatic machinery,functional analogies with eukaryotic RNAi,and hypothetical mechanisms of action[J].Biology Direct,2006,1(1):1-26.DOI:10.1186/1745-6150-1-7.
[28] BARRANGOU R,FREMAUX C,DEVEAU H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709-1712.DOI:10.1126/science.1138140.
[29] 李辉,施振旦.Cas9 新型基因打靶系统的研究进展[J].江苏农业学报,2013,29(4):907-911.DOI:10.3969/j.issn.1000-4440.2013.04.037.
LI Hui,SHI Zhendan.Reaserch progress of gene targeting technology of CRISPR/Cas9 system[J].Jiangsu Journal of Agricultural Science,2013,29(4):907-911.DOI:10.3969/j.issn.1000-4440.2013.04.037.
[30] JARMAN A P,GRAU Y,JAN L Y,et al.Atonal is a proneural gene that directs chordotonal organ formation in the Drosophila peripheral nervous system[J].Cell,1993,73(7):1307-1321.DOI:10.1016/0092-8674(93)90358-W.
[31] WEI Y Z,CHESNE M T,TERNS R M,et al.Sequences spanning the leader-repeat junction mediate CRISPR adaptation to phageinStreptococcusthermophilus[J].Nucleic Acids Research,2015,43(3):1749-1758.DOI:10.1093/nar/gku1407.
[32] BARRANGOU R,MARRAFFINI L A.CRISPR-Cas systems:prokaryotes upgrade to adaptive immunity[J].Molecular Cell,2014,54(2):234-244.DOI:10.1016/j.molcel.2014.03.011.
[33] CHARPENTIER E,MARRAFFINI L A.Harnessing CRISPR-Cas9 immunity for genetic engineering[J].Current Opinion in Microbiology,2014,19:114-119.DOI:10.1016/j.mib.2014.07.001.
[34] MAKAROVA K S,HAFT D H,BARRANGOU R,et al.Evolution and classification of the CRISPR-Cas systems[J].Nature Reviews Microbiology,2011,9(6):467-477.DOI:10.1038/nrmicro2577.
[35] ZHOU H B,LIU B,WEEKS D P,et al.Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice[J].Nucleic Acids Research,2014,42(17):10903-10914.DOI:10.1093/nar/gku806.
[36] KANCHISWAMY C N,SARGENT D J,VELASCO R,et al.Looking forward to genetically edited fruit crops[J].Trends in Biotechnology,2014,33(2):62-64.DOI:http://dx.doi.org/10.1016/j.tibtech.2014.07.003.
[37] BASSETT A R,LIU J L.CRISPR/Cas9 and genome editing inDrosophila[J].Journal of Genetics and Genomics,2014,41(1):7-19.DOI:10.1016/j.jgg.2013.12.004.
[38] ANDERS C,NIEWOEHNER O,DUERST A,et al.Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease[J].Nature,2014,513(7519):569-573.DOI:10.1038/nature13579.
[39] OH J-H,VAN PIJKEREN J-P.CRISPR-Cas9-assisted recombineering inLactobacillusreuteri[J].Nucleic Acids Research,2014,42(17):1-11.DOI:10.1093/nar/gku623.
[40] 刘莹,郑鹏生,张忠明.CRISPR系统结构及功能的研究[J].医学分子生物学杂志,2014,11(4):297-302.DOI:10.3870/j.issn.1672-8009.2014.04.012.
LIU Ying,ZHENG Pengsheng,ZHANG Zhongming.Structure and function of CRISPR system[J].Journal of Medical Molecular Biology,2014,11(4):297-302.DOI:10.3870/j.issn.1672-8009.2014.04.012.
[41] DELTCHEVA E,CHYLINSKI K,SHARMA C M,et al.CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J].Nature,2011,471(7340):602-607.DOI:10.1038/nature09886.
[42] MALI P,ESVELT K M,CHURCH G M.Cas9 as a versatile tool for engineering biology[J].Nature Methods,2013,10(10):957-963.DOI:10.1038/NMETH.2649.
[43] 颜雯,李海涛,向华,等.CRISPR-Cas基因组改造技术研究进展[J].广东农业科学,2014,2:149-152.
YAN Wen,LI Haitao,XIANG Hua,et al.Research progress on the genome modification technologies of CRISPR-Cas[J].Guangdong Agricultural Sciences,2014,2:149-152.
[44] JINEK M,CHYLINSKI K,FONFARA I,et al.A programmable dual-RNA-guided DNA ndonuclease in adaptive bacterial immunity[J].Science,2012,337(6096):816-821.DOI:10.1126/Science.1225829.
[45] ZHAO P,ZHANG Z,KE H M,et al.Oligonucleotide-based targeted gene editing in C.elegans via the CRISPR/Cas9 system[J].Cell Research,2014,24(2):247-250.DOI:10.1038/cr.2014.9.
[46] PORT F,CHEN H M,LEE T,et al.Optimized CRISPR/Cas tools for efficient germline and somatic genome engineering inDrosophila[J].Proceedings of the National Academy of Sciences,2014,111(29):E2967-E2976.DOI:10.1073/pnas.1405500111.
[47] DAIMON T,KIUCHI T,TAKASU Y.Recent progress in genome engineering techniques in the silkworm,Bombyxmori[J].Development,Growth & Differentiation,2014,56(1):14-25.DOI:10.1111/dgd.12096.
[48] WANG J B,QUAKE S R.RNA-guided endonuclease provides a therapeutic strategy to cure latent herpesviridae infection[J].Proceedings of the National Academy of Sciences,2014,111(36):13157-13162.DOI/10.1073/pnas.1410785111.
[49] XIE F,YE L,CHANG J C,et al.Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 andpiggyBac[J].Genome Research,2014,24(9):1526-1533.DOI:10.1101/gr.173427.114.
[50] HRUSCHA A,KRAWITZ P,RECHENBERG A,et al.Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish[J].Development,2013,140(24):4982-4987.DOI:10.1242/dev.099085.
[51] AIDA T,IMAHASHI R,TANAKA K.Translating human genetics into mouse:the impact of ultra-rapid in vivo genome editing[J].Development Growth Differentiation,2014,56(1):34-45.DOI:10.1111/dgd.12101.
[52] KABADI A M,OUSTEROUT D G,HILTON I B,et al.Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector[J].Nucleic Acids Research,2014,42(19):e147.DOI:10.1093/nar/gku749.
[53] CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339(6121):819-823.DOI:10.1126/science.1231143.
[54] MALI P,YANG L H,ESVELT K M,et al.RNA-guided human genome engineering via Cas9[J].Science,2013,339(6121):823-826.DOI:10.1126/science.1232033.
[55] SUGANO S S,SHIRAKAWA M,TAKAGI J,et al.CRISPR/Cas9-mediated targeted mutagenesis in the liverwortMarchantiapolymorphaL.[J].Plant and Cell Physiology,2014,55(3):475-481.DOI:10.1093/pcp/pcu014.
[56] FENG Z Y,ZHANG B T,DING W N,et al.Efficient genome editing in plants using a CRISPR/Cas system[J].Cell Research,2013,23(10):1229-1232.DOI:10.1038/cr.2013.114.
[57] ZHANG H,ZHANG J S,WEI P L,et al.The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation[J].Plant Biotechnology Journal,2014,12(6):797-807.DOI:10.1111/pbi.12200.
[58] LI D L,QIU Z W,SHAO Y J,et al.Heritable gene targeting in the mouse and rat using a CRISPR-Cas system[J].Nature Biotechnology,2013,31(8):681-683.DOI:10.1038/nbt.2661.
[59] YOSHIMI K,KANEKO T,VOIGT B,et al.Allele-specific genome editing and correction of disease-associated phenotypes in rats using the CRISPR-Cas platform[J].Nature Communications,2014,5:4240.DOI:10.1038/ncomms5240.
[60] RAMAKRISHNA S,KWAKU DAD A-B,BELOOR J,et al.Gene disruption by cell-penetrating peptide-mediated delivery of Cas9 protein and guide RNA[J].Genome Research,2014,24(6):1020-1027.DOI:10.1101/gr.171264.113.
[61] ZHANG C,XIAO B,JIANG Y Y,et al.Efficient editing of malaria parasite genome using the CRISPR/Cas9 system[J].mBio,2014,5(4):e01414-01414.DOI:10.1128/mBio.01414-14.
[62] ZHANG F,WEN Y,GUO X.CRISPR/Cas9 for genome editing:progress,implications and challenges[J].Human Molecular Genetics,2014,23(R1):R40-R46.DOI:10.1093/hmg/ddu125.
[63] TERNS R M,TERNS M P.CRISPR-based technologies:prokaryotic defense weapons repurposed[J].Trends in Genetics,2014,30(3):111-118.DOI:10.1016/j.tig.2014.01.003.
[64] LARSON M H,GILBERT L A,WANG X W,et al.CRISPR interference(CRISPRi) for sequence-specific control of gene expression[J].Nature Protocols,2013,8(11):2180-2196.DOI:10.1038/nprot.2013.132.
[65] GILBERT L A,LARSON M H,MORSUT L,et al.CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J].Cell,2013,154(2):442-451.DOI:http://dx.doi.org/10.1016/j.cell.2013.06.044.
[66] FENG Y,SASSI S,SHEN J K,et al.Targeting CDK11 in osteosarcoma cells using the CRISPR-Cas9 system[J].Journal of Orthopaedic Research,2015,33(2):199-207.DOI:10.1002/jor.22745.
[67] 罗斌.CRISPR/Cas9技术介导的稳定沉默LDHA表达对肺癌A549细胞增殖的影响[J].生物技术世界,2015(1):97-98.
[68] HSU P D,SCOTT D A,WEINSTEIN J A,et al.DNA targeting specificity of RNA-guided Cas9 nucleases[J].Nature Biotechnology,2013,31(9):827-832.DOI:10.1038/nbt.2647.
[69] XIAO A,CHENG Z C,KONG L,et al.CasOT:a genome-wide Cas9/gRNA off-target searching tool[J].Bioinformatics,2014,30(8):1180-1182.DOI:10.1093/bioinformatics/btt764.
[70] GAO Y B,ZHAO Y D.Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing[J].Journal of Integrative Plant Biology,2014,56(4):343-349.DOI:10.1111/jipb.12152.
[71] HEIGWER F,KERR G,BOUTROS M.E-CRISP:fast CRISPR target site identification[J].Nature Methods,2014,11(2):122-123.DOI:10.1038/nmeth.2812.
[72] WAGNER J C,PLATT R J,GOLDFLESS S J,et al.Efficient CRISPR-Cas9-mediated genome editing in Plasmodium falciparum[J].Nature Methods,2014,11(9):915-918.DOI:10.1038/NMETH.3063.
[73] JIANG W Y,BIKARD D,COX D,et al.RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J].Nature Biotechnology,2013,31(3):233-239.DOI:10.1038/nbt.2508.
[74] DE SOUZA N.Genetics:more specific CRISPR editing[J].Nature Methods,2014,11(7):712-712.DOI:10.1038/nbt.2916.
[75] GUELL M,YANG L H,CHURCH G.Genome editing assessment using CRISPR genome analyzer(CRISPR-GA)[J].Bioinformatics,2014,30(2):2968-2970.DOI:10.1093/bioinformatics/btu427.
(责任编辑:赵藏赏)
Research status of novel gene editing technology of CRISPR/Cas9 system
QIAN Leiyang,ZHOU Zhijun,CHANG Yanlin
(Key Laboratory of Invertebrate Systematics and Application of Hebei Province,College of Life Sciences,Hebei University,Baoding 071002,China)
Abstract:Clustered regularly interspaced short palindromic repeats(CRISPR) and associated proteins(Cas) comprise the CRISPR/Cas system,which confers adaptive immunity against foreign invading phages,plasmids and other mobile elements in many bacteria and most archaea.On the basis of the species and homology of the Cas protein,CRISPR/Cas systems have been classified into three distinct groups(type Ⅰ~Ⅲ).The component of type Ⅱ CRISPR/Cas9 system is the simplest one,and is well-charactered in resent study.CRISPR/Cas9 system modified by genetic engineering has proven to be an efficient gene-targeting tool in various organisms.Here,the auther review the discovery,components,molecular mechanism,applications,challenges and future prospect of CRISPR/Cas9-mediated genome editing.
Key words:gene editing;CRISPR/Cas9;CRISPRi;interference mechanism;gene therapy
DOI:10.3969/j.issn.1000-1565.2016.01.014
收稿日期:2015-07-02
基金项目:国家自然科学基金面上项目(31471985);河北省高等学校青年拔尖人才计划项目(BJ2014006)
通信作者:周志军(1980—),男,山西长治人,河北大学副教授,博士,主要从事昆虫分子系统进化研究.
中图分类号:Q789
文献标志码:A
文章编号:1000-1565(2016)01-0084-10
第一作者:骞蕾阳(1990—),女,河北武安人,河北大学在读硕士研究生. E-mail:qianleiyanghb@163.com
E-mail:zhijunzhou@163.com

