里氏木霉QM9414尿嘧啶缺陷型转化体系改进和β-葡萄糖苷酶的过量表达
钟立霞,钱远超,戴美学,钟耀华(山东师范大学生命科学学院,山东 济南 5004;山东大学生命科学学院,微生物技术国家重点实验室,山东 济南 5000)
里氏木霉QM9414尿嘧啶缺陷型转化体系改进和β-葡萄糖苷酶的过量表达
钟立霞1,2,钱远超2,戴美学1,钟耀华2
(1山东师范大学生命科学学院,山东 济南 250014;2山东大学生命科学学院,微生物技术国家重点实验室,山东 济南 250100)
摘要:里氏木霉是广泛应用于纤维素酶生产的工业真菌,但高产突变株遗传操作困难,限制了菌种改良。首先利用定点整合策略敲除里氏木霉突变株QM9414的pyr4基因,成功构建尿嘧啶营养缺陷型菌株QP4,其遗传转化效率显著提高,而且产酶能力不受影响。进一步在QP4中过量表达β-葡萄糖苷酶(BGL)基因bgl1,经大量平板显色筛选获得2株BGL活力明显增强的工程菌QPB4和QPB5,其酶活分别提高10.01倍和8.26倍。利用发酵酶液对2种不同预处理的玉米芯底物进行水解糖化实验,结果显示以酸处理玉米芯为底物时QPB4和QPB5的葡萄糖得率比QP4分别提高60.98%和52.44%,而以脱木素处理玉米芯为底物时其葡萄糖得率分别提高80.01%和86.00%。研究表明改进里氏木霉高产突变株遗传转化体系可以显著促进菌株改良,提高糖化应用效果。
关键词:纤维素酶;里氏木霉高产突变株;遗传转化;水解糖化;尿嘧啶营养缺陷型标记;构建和表达;生物技术;生物工程
2015-11-03收到初稿,2015-12-26收到修改稿。
联系人:戴美学,钟耀华。第一作者:钟立霞(1989—),女,硕士研究生。
Received date: 2015-11-03.
Foundation item: supported by the National Natural Science Foundation of China (31370135), the Agricultural Science and Technology Achievement Transformation Fund of Shandong Province (2014No.45) and the Fundamental Research Funds of Shandong University (2015CJ005).
引 言
将生物质转变为燃料乙醇是获得可再生性能源的重要途径,越来越受到世界各国的关注。而纤维素酶系是纤维素乙醇化过程中的关键酶,其生产成本过高限制了纤维乙醇的发展[1]。改良纤维素酶系,提高底物糖化效率,对降低酶生产成本、促进纤维素转化具有重要意义。目前纤维素酶生产的工业真菌包括木霉、青霉和曲霉等[2],其中里氏木霉(Trichoderma reesei)以蛋白质分泌量大而成为纤维素酶生产的主要工业菌株[3]。因此,里氏木霉的菌种改良是构建高效纤维素酶系,提高糖化效率的最有效的途径。
里氏木霉野生型菌株QM6a是第二次世界大战期间从腐烂的军人服装和帆布帐篷中分离到的一种深绿色真菌[4]。随后经过不断的紫外线、NTG、离子诱变等筛选,获得了NG14、RUT-C30和QM9414等高产突变菌株[5]。RUT-C30和QM9414是当前常用的里氏木霉突变菌株。过度的诱变在获得高纤维素酶的同时也给菌株带来了诸多负面影响,如经过多轮诱变获得的RUT-C30虽然比QM9414的胞外蛋白质和纤维素酶活都大幅度提高[5],但其生长缓慢、生孢延迟、生孢减少。而QM9414菌株生长快、产孢多、发酵成熟,是目前进行酶系改良和纤维素酶表达调控机制研究的重要模式菌株。
当前QM9414的遗传转化操作主要依赖潮霉素B(hygromycin B)等抗性筛选,但抗生素价格昂贵、筛选背景较高且不方便实现无痕遗传操作,而营养缺陷型筛选标记可以克服上述的不足,有利于构建高效遗传转化体系[6-7]。营养缺陷型筛选标记乳清酸核苷-5′-磷酸脱羧酶(Pyr4/PyrG:木霉为Pyr4,曲霉为PyrG)是尿嘧啶核苷酸合成的一个关键酶,该基因的缺失将导致尿嘧啶核苷酸合成受阻,因此缺乏该酶的营养缺陷型菌株需要添加尿嘧啶/尿苷才能生长。当pyr4基因成功转化进pyr4基因缺失菌株后,该基因的表达使受体菌能够自身合成尿嘧啶/尿苷,从而不依赖尿嘧啶/尿苷生长,起到正向筛选的作用。另一方面,5′-氟乳清酸(5′-fluorooroticacid,5′-FOA)是尿嘧啶合成前体的类似物,在pyr4的作用下会产生对细胞有毒害物质,所以野生型菌株在5′-FOA下不能生长,而变成pyr4基因缺失菌时就呈现5′-FOA抗性,从而实现反向筛选[8-9]。目前常用的里氏木霉尿嘧啶营养缺陷型菌株是TU6,该菌是由QM9414经射线诱变而来[10],尽管TU6遗传转化效率较高,但菌株生长缓慢、生孢量少,尤其是严重影响了纤维素酶的表达和分泌。因此,在QM9414中定点敲除pyr4基因,建立高效遗传转化体系,有利于通过基因工程手段实现工业菌株的纤维素酶系改良[6,9-13]。
里氏木霉分泌的纤维素酶主要包括3类:外切纤维素酶(CBH,EC3.2.1.91)、内切纤维素酶(EG,EC3.2.1.4)和β-葡萄糖苷酶(BGL,EC3.2.1.21)。在降解复杂底物时BGL能够将纤维寡糖水解为葡萄糖,从而解除了纤维寡糖对外切酶的抑制。普遍认为里氏木霉降解纤维素的瓶颈是BGL的不足[14]。向里氏木霉酶系中外源补加BGL能够显著提高水解纤维素作底物时的葡萄糖得率[15]。另外,在里氏木霉中本源和异源表达BGL1可以提高其纤维素酶系降解纤维素底物的能力[16-17],但目前纤维素酶系组分中的BGL表达水平仍然不高。因此,在里氏木霉中高水平表达BGL是构建高产纤维素酶菌株和提升纤维素酶系整体糖化效率的关键。
为了实现遗传改良里氏木霉从而提高纤维素底物的糖化能力,本研究首先利用里氏木霉QM9414构建了用于高效遗传转化的尿嘧啶营养缺陷型菌株,在此基础上过表达β-葡萄糖苷酶BGL1,通过大量筛选转化子获得高水平表达β-葡萄糖苷酶的工程菌株,并进一步利用不同预处理的玉米芯底物进行了糖化应用。
1 实验材料和方法
1.1材料
1.1.1菌株与质粒里氏木霉(Trichoderma reesei)QM9414来源于美国菌种保藏中心(ATCC26921),TU6菌株来自奥地利维也纳技术大学Christian Kubicek教授惠赠。质粒pTHB[18]、pAB4-1和pAN7-1为本实验室保存。
1.1.2培养基PDA产孢培养基(g·L−1):去皮土豆薄片200,加水煮沸30 min,8层纱布过滤,滤液添加葡萄糖20,并补足水分至1 L,自然pH,2%琼脂粉。115℃灭菌30 min。
MM培养基(g·L−1):葡萄糖20,KH2PO415,(NH4)2SO45,CaCl20.6,MgSO4·7H2O 0.6,蛋白胨2,FeSO4·7H2O 0.005,MnSO4·H2O 0.0016,ZnSO4·7H2O 0.0014,CoCl2·2H2O 0.002,调节pH 至4.5~5.5,配固体培养基需添加2%琼脂粉,菌株表型分析时将葡萄糖替换为其他碳源。115℃灭菌30 min。
里氏木霉转化培养基(g·L−1):葡萄糖 10,MgSO4·7H2O 1,KH2PO410,(NH4)2SO46,C6H5O7Na3·2H2O 3,FeSO4·7H2O 0.005,MnSO4·H2O 0.0016,ZnSO4·7H2O 0.0014,CoCl2·2H2O 0.002,调节pH 至4.5~5.5,转化培养基加入渗透压稳定剂山梨醇182.18、琼脂粉12。115℃灭菌30 min。
LB培养基(g·L−1):酵母粉5,蛋白胨10,NaCl 10,用NaOH调节pH至7.2,固体培养基添加2%琼脂粉。115℃灭菌30 min。若需要加入氨苄青霉素至终浓度100 μg·ml−1。
七叶苷培养基(g·L−1):CMC-Na 10,七叶苷3,柠檬酸铁0.5,(NH4)2SO42,MgSO4·7H2O 0.5,K2HPO41,酵母膏 1,2.5%琼脂粉。115℃灭菌30 min。
种子培养基(g·L−1):葡萄糖 10,CaCl21,MgSO4·7H2O 0.6,KH2PO45,(NH4)2SO42.5,玉米浆20。115℃灭菌30 min。
发酵培养基(g·L−1):结晶纤维20,CaCl21,MgSO4·7H2O 0.6,KH2PO45,(NH4)2SO42.5,玉米浆20。115℃灭菌30 min。
以上培养基在培养尿嘧啶缺陷型时均需添加10 mmol·L−1的尿嘧啶。
1.1.3玉米芯底物用于纤维素酶糖化效率测定的酸处理玉米芯和脱木素处理玉米芯均来自山东龙力生物科技股份有限公司,其中酸处理玉米芯的处理方式采用Schell等[19]的方法,脱木素处理玉米芯的处理方式参照Zhao等[20]的方法。酸处理玉米芯的处理方式其化学成分为:纤维素62.6%,半纤维素2.4%,木素17.7%,灰分6.8%,其他成分为10.5%;脱木素处理玉米芯的处理方式其化学成分为纤维素65.7%,半纤维素1.8%,木素3.2%,灰分5.9%,其他成分为23.4%[21]。
1.1.4酶和试剂里氏木霉原生质体制备所用的哈茨木霉裂解酶,酶活测定所用的pNPC、pNPG、CMC-Na购自SIGMA;酶活测定所用滤纸为Whaterman 1号滤纸;琼脂糖凝胶回收纯化、PCR纯化和质粒小提试剂盒均购自OMEGA;DNA聚合酶HiFiTaq polymerase、EasyTaq polymerase 购自TransGen;DNA GeneRuler 1 kb购自ThermoFisher Scientific;潮霉素B(Hygromycin B)购自Roche;尿嘧啶(Uracil)、5′-氟乳清酸(5′-FOA)购自BB;引物由北京六合华大基因科技股份有限公司合成。1.1.5仪器设备台式离心机(上海安亭科学仪器厂),高速冷冻离心机(Eppendorf),恒温摇床(上海博彩生物科技有限公司),DYY-Ⅱ电泳仪(北京君意东方电泳设备有限公司),PCR仪(Eppendorf)。
1.2方法
1.2.2里氏木霉基因组提取里氏木霉染色体的提取参照文献[22]的方法进行。
1.2.3引物设计在里氏组数据库(http://genome. jgi-psf.org/Trire2/Trire2.home.html)中,使用BLASTN程序搜索与酿酒酵母pryG的同源基因ura3(GenBank accession no. NC_001137.3)的基因序列同源性最高的序列。结果显示编号为74020的蛋白同源性最高,根据注释为乳清酸-5′-单磷酸脱羧酶,即pyr4基因序列信息,运用Primer Premier 5软件设计引物,本研究所用的引物序列见表1。
1.2.4pyr4敲除盒的构建和黑曲霉pyrG基因片段的扩增以里氏木霉QM9414基因组为模板,利用引物pyr4-UF1/pyr4-UR1和pyr4-DF1/pyr4-DR1分别扩增得到1.3 kb的pyr4基因上游同源臂和1.3 kb 的pyr4基因下游同源臂;以PAN7-1质粒为模板,利用引物hph-F1/hph-R1扩增得到2.8 kb的潮霉素B基因。然后,利用Double-joint PCR[23]将pyr4基因上下游同源臂和潮霉素B基因利用引物pyr4-UF3/pyr4-UR3融合得到5.2 kb的pyr4敲除盒。

表1 本研究所用引物Table 1 Primers used in this study
以质粒pAB4-l为模板,利用引物pyrG-S/pyrG-A扩增得到2.7 kb的黑曲霉pyrG基因片段,用于回补pyr4敲除菌株以及与质粒pTHB共转化pyr4敲除菌株,从而构建BGL1过表达菌株。
1.2.5pyr4敲除菌株、pyrG回补菌株和BGL1过表达菌株的构建里氏木霉原生质体的制备和转化实验主要参照文献[22]的方法进行。
pyr4敲除菌株的构建:将1μg pyr4敲除盒与适量里氏木霉QM9414原生质体、100 μg·ml−1潮霉素B、1.5 mg·ml−15′-FOA混合,倾倒在已凝固的转化平板上,30°C培养至转化子出现。
pyrG回补菌株的构建:1 μg pyrG基因片段与尿嘧啶营养缺陷型菌株QP4原生质体倾倒在不含有尿嘧啶的转化下层平板上,30℃培养至转化子出现。
对于初中生而言,年龄相对较小,对任何事情都具有着很大的好奇心,因此只要初中语文教师注重活跃课堂氛围,就很容易将学生带到相应的情境之中,激发他们的学习兴趣。在过去传统的初中语文教学中,教师并不重视课堂氛围,只是简单地讲授知识点,造成学生认为语文学习枯燥乏味,出现排斥心理,影响到初中语文的教学质量。因此,为了打破过去教学方法的束缚,教师应该积极使用多元化的教学方法。在讲课的过程中,教师可以面带微笑,在课堂上营造出轻松、活跃的课堂氛围,通过提问或是小故事等形式引导学生积极发言,多与学生进行互动。例如在讲述诗歌古文时,教师可以以小组为单位举行朗诵比赛,让学生们扮演诗人来抒发自己的情怀等。
BGL1过表达菌株的构建:将2 μg含有Pcbh1启动子控制表达bgl1表达盒(Pcbh1-bgl1)的pTHB质粒[18]和1 μg pyrG基因片段同时与适量尿嘧啶营养缺陷型菌株QP4原生质体倾倒在不含有尿嘧啶的转化平板上,30℃培养至转化子出现。
1.2.6PCR验证、单孢分离和菌株表型分析PCR验证:参照1.2.2节的方法提取染色体,pyr4敲除菌株的验证利用pyr4敲除盒中潮霉素B基因验证引物Yhph-F1/Yhph-R1,上游染色体锚定引物Ypyr4-UF2/Yhph-R1和下游染色体锚定引物Yhph-F2/pyr4-DR1分别扩增800 bp、3.1 kb、2.1 kb大小片段。回补菌株的验证PCR引物为YpyrG-F1/YpyrG-R1扩增600 bp左右。BGL过表达菌株的验证以Y1/Y2为引物扩增Pcbh1-bgl1表达盒中横跨启动子和编码区的1.0 kb片段。
单孢分离:将孢子悬液稀释一定倍数后,在添加有0.5%的TritonX-100、1 mg·ml−1的尿嘧啶的MM固体培养基上涂覆或划线培养,挑取单菌落进行PCR验证,将分离纯化得到的阳性转化子命名为QP4并生孢保存。回补菌株及BGL过表达菌株单孢分离时MM培养基上不需添加1 mg·ml−1的尿嘧啶。
表型验证:取1 μl新鲜孢子(或取菌丝块),在加有尿嘧啶的MM平板上30℃培养4 d。
1.2.7生长及碳源利用情况测定菌落大小测定:取1 μl新鲜孢子点至不同碳源(葡萄糖、甘油、乳糖、纤维素)MM培养基和PDA培养基上,30℃培养60 h,记录菌落形态并测量菌落直径来表征菌落的大小。
1.2.8纤维素酶活测定取新鲜孢子悬液,以106个·ml−1的接种量接种于种子培养基,30℃、200 r·min−1振荡培养36 h,以10%的接种量转接至发酵培养基,30℃,200 r·min−1振荡培养。诱导一定时间后取上清测定滤纸酶活(FPA)、内切葡聚糖酶活(EG)、外切葡聚糖酶活(CBH)、β-葡萄糖苷酶活(BGL),FPA、EG活力测定参照曲音波等[24]的方法进行,CBH、BGL活力测定参照文献[25]。
1.2.9BGL表达菌株的平板定性检测将β-葡萄糖苷酶过表达菌株在β-葡萄糖苷酶筛选培养基上30℃培养12 h、24 h,记录菌落形态并测量菌落直径大小来表征β-葡萄糖苷酶活。
1.2.10不同底物糖化分析糖化实验参照Cheng 等[26]的方法进行。反应体系为30 ml,糖化底物为5%的酸处理玉米芯、脱木素处理玉米芯,纤维素酶添加量为10 FPU·g−1底物,为了防止染菌加入0.1% NaN3,用0.1 mol·L−1pH4.8的醋酸缓冲液补齐至30 ml,50℃、150 r·min−1进行糖化,取糖化24 h、48 h糖化液,利用SBA-40C(BISAS,China)生物传感仪进行葡萄糖的测定。

图1 里氏木霉pyr4敲除菌株QP4的PCR验证图及表型差异比较Fig.1 PCR confirmation and phenotypic assay of pyr4 deletion strain
2 实验结果与讨论
2.1里氏木霉菌株的pyr4敲除
为了在里氏木霉QM9414中敲除pyr4基因,首先利用该菌基因组扩增得到均为1.3 kb的pyr4基因上下游同源臂与pAN7-1质粒扩增得到的2.8 kb潮霉素B抗性表达元件经融合PCR得到5.2 kb的pyr4敲除盒。然后以pyr4敲除盒转化里氏木霉QM9414原生质体,在100 μg·ml−1潮霉素B、1.5 mg·ml−15′-FOA双筛选压力下初步得到2株转化子,经过复筛、单孢分离,随后挑取1株遗传稳定的转化子命名为QP4,并进行PCR验证(图1)。结果显示,QP4转化子中可以扩增得到敲除盒潮霉素编码区800 bp[图1(a)左]、染色体上游锚定片段3.0 kb[图1(a)中]和染色体下游锚定片段1.8 kb左右的条带[图1(a)右],而出发菌QM9414染色体和阴性对照H2O为模板时没有扩展片段的条带,与预期结果符合。这表明pyr4敲除盒已经发生同源交换,成功插入里氏木霉QM9414染色体中,并且染色体中没有其他位点的片段整合,从而在染色体水平验证了pyr4基因被定点敲除。
为了进一步确认pyr4基因的敲除,用来自黑曲霉(Aspergillus niger)的pyr4同源基因pyrG对QP4菌株进行回补。转化后在无尿嘧啶筛选平板上得到17个转化子,经过PCR扩增pyrG基因验证均为阳性转化子,表明利用pyr4作为正向筛选基因可大大提高转化效率。随机挑取一株转化子命名为R-pyrG,经单孢分离传代后进行表型分析。图1(b)结果显示,出发菌株QM9414和回补菌株R-pyrG表型相似,在加有尿嘧啶的MM平板和不含尿嘧啶的MM平板上都生长,而敲除pyr4的QP4菌株在加有尿嘧啶的MM上平板生长,在不含尿嘧啶的MM平板上不生长。该结果说明R-pyrG能够恢复到野生型状态,QP4菌株的表型变化确实是由pyr4基因敲除造成的。因此,本研究成功构建了里氏木霉pyr4基因敲除的并可用于高效遗传转化的尿嘧啶营养缺陷型菌株QP4。
2.2不同碳源条件下QP4生长情况分析
为验证pyr4的敲除是否影响菌株的生长,本研究分析了QP4在不同碳源条件下的生长情况(图2)。与出发菌株QM9414相比,pyr4敲除菌株QP4的菌落大小、菌落颜色和菌落蔓延状态在不同碳源(葡萄糖、甘油、乳糖、纤维素和PDA)上均变化不大,而诱变的尿嘧啶营养缺陷型菌株TU6菌落明显变小且菌落颜色也有差异。其中各菌株在葡萄糖、PDA上的生长速度最快,乳糖、纤维素上次之,甘油上最慢。另外,TU6的生孢量明显低于QP4和QM9414。对菌落大小的测定结果显示,菌株QP4菌落大小与QM9414基本一致,而TU6菌落在葡萄糖、甘油、乳糖、纤维素条件下分别减小了35.8%、28.1%、15.5%和12.5%[图2(b)]。以上结果显示,不同碳源条件下pyr4敲除菌株QP4的生长并没有受到影响,可用于后续研究。
2.3摇瓶发酵条件下QP4产酶情况分析
为进一步确认pyr4敲除对产酶能力的影响,本研究对QP4在产酶发酵条件下7 d的纤维素酶活及各组分酶活进行测定。如图3所示,pyr4敲除菌株QP4的滤纸酶活(FPA)、内切葡聚糖酶活(EG)、外切葡聚糖酶活(CBH)和β-葡萄糖苷酶活(BGL)与出发菌株QM9414相比并未发生明显变化,却都显著高于诱变菌株TU6。其中,QP4的FPA和CBH分别是TU6的2.13倍和2.12倍,而EG和BGL分别是TU6的1.36倍和1.35倍。该结果表明,pyr4的敲除并未影响里氏木霉原有纤维素酶的表达和分泌,而且明显优于诱变菌株TU6。因此,QP4可用于对里氏木霉进行高产纤维素酶菌种改良的后续遗传操作。
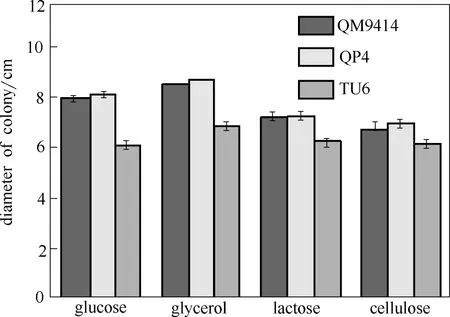
图2 里氏木霉pyr4敲除菌株QP4在不同碳源下的表型分析和生长差异比较Fig.2 Phenotypic analysis and growth comparison of QM9414, QP4 and TU6 strains on different carbon sources after 60 h (Data are represented as mean of three independent experiments. Error bars express standard deviations)
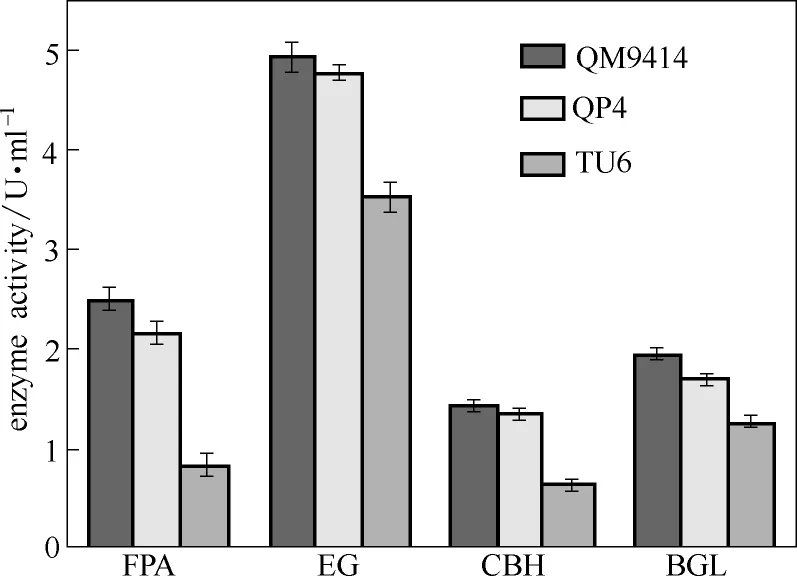
图3 里氏木霉pyr4敲除菌株纤维素酶活力分析Fig.3 Cellulase activities of T. reesei QP4, TU6 and QM9414 [Filter paper activity (FPA), endoglucanase activity (EG), cellobiohydrolase activity (CBH) and β-glucosidase activity (BGL) were measured after being induced with cellulase production medium for 7 d. Data are represented as mean ofthree independent experiments. Error bars expressstandard deviations]
2.4QP4菌株过表达β-葡萄糖苷酶改良纤维素酶系
为进一步验证QP4菌株作为高效遗传转化体系的可操作性,同时改良里氏木霉纤维素酶系组分,本研究针对里氏木霉β-葡萄糖苷酶(BGL)表达量低的问题,利用带有强启动子Pcbh1控制表达β-葡萄糖苷酶基因bgl1表达盒(Pcbh1-bgl1)的质粒pTHB[15]与来自黑曲霉的pyrG片段共转化QP4。转化平板上获得了135个转化子,经过PCR验证,123个为pyrG整合入染色体的阳性转化子,阳性转化子率达到91.1%,远高于潮霉素B抗性筛选的阳性转化子效率(63%)[12]。这表明QP4可以作为pyr4筛选标记的高效遗传转化菌株。
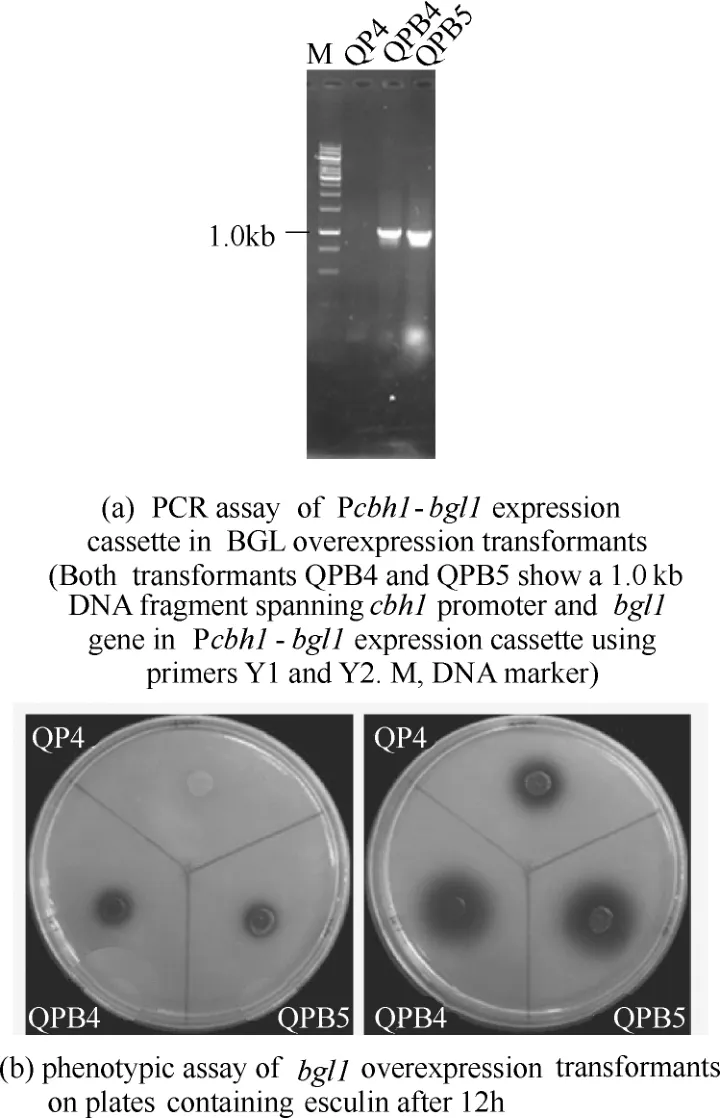
图4 里氏木霉β-葡萄糖苷酶过表达菌株的分子验证和平板BGL活力检测Fig.4 PCR analysis and detection of BGL activity on esculin substrate in bgl1 overexpression transformants
β-葡萄糖苷酶(BGL)能够将七叶苷分解为七叶素,与Fe3+螯合形成黑色的螯合物,在培养基平板上出现明显的黑色晕圈,因此利用七叶苷培养基平板可以进行高通量BGL表达菌株的筛选。本研究将获得的pyrG整合阳性转化子进行了快速七叶苷平板检测,挑选出2株BGL酶活显著增强的转化子,分别命名为QPB4和QPB5。然后进行单孢分离和PCR验证,证明Pcbh1控制表达的bgl1基因成功整合到染色体中[图4(a)]。表型分析表明,QPB4和QPB5转化子在无尿嘧啶培养基上可以生长,而QP4不能生长,说明筛选标记pyrG基因在转化子中稳定遗传。七叶苷平板测定显示,QPB4 和QPB5在12h时能够形成明显的黑色晕圈,而QP4菌株未发现明显的黑色晕圈;24 h时QPB4和QPB5的黑色晕圈的直径分别达到了1.3 cm和1.6 cm,而QP4的黑色晕圈的直径为0.7 cm[图4(b)],这说明BGL在QP4中成功实现了过表达。
2.5β-葡萄糖苷酶过表达菌株的产酶分析
为进一步确认BGL过表达菌株的纤维素酶产生情况,取摇瓶发酵培养5 d的酶液进行酶活测定。如图5所示,过表达菌株QPB4和QPB5的β-葡萄糖苷酶活力分别达到了13.65 U·ml−1和11.49 U·ml−1,比QP4(1.24 U·ml−1)分别提高了10.01倍和8.26倍。但其滤纸酶活变化有差异,QPB5的FPA为2.14 U·ml−1,比QP4提高了43.6%,而QPB4的FPA为1.29 U·ml−1,比QP4却降低了13.4%。QPB4产生这种情况的原因可能是由于其染色体中bgl1表达盒的不合理插入或cbh1启动子过表达引起转录调控因子滴定效应等导致的[27]。但测定QPB4的纤维素外切酶活和内切酶活,发现基本没有发生变化[图5(b)]。这种过表达一个纤维素酶基因而引起总纤维素酶活降低的现象在前期研究中也有报道[14]。而通过大量筛选得到的另一菌株QPB5并没有发生这种情况,BGL和FPA活力均显著提高(如上所述)。
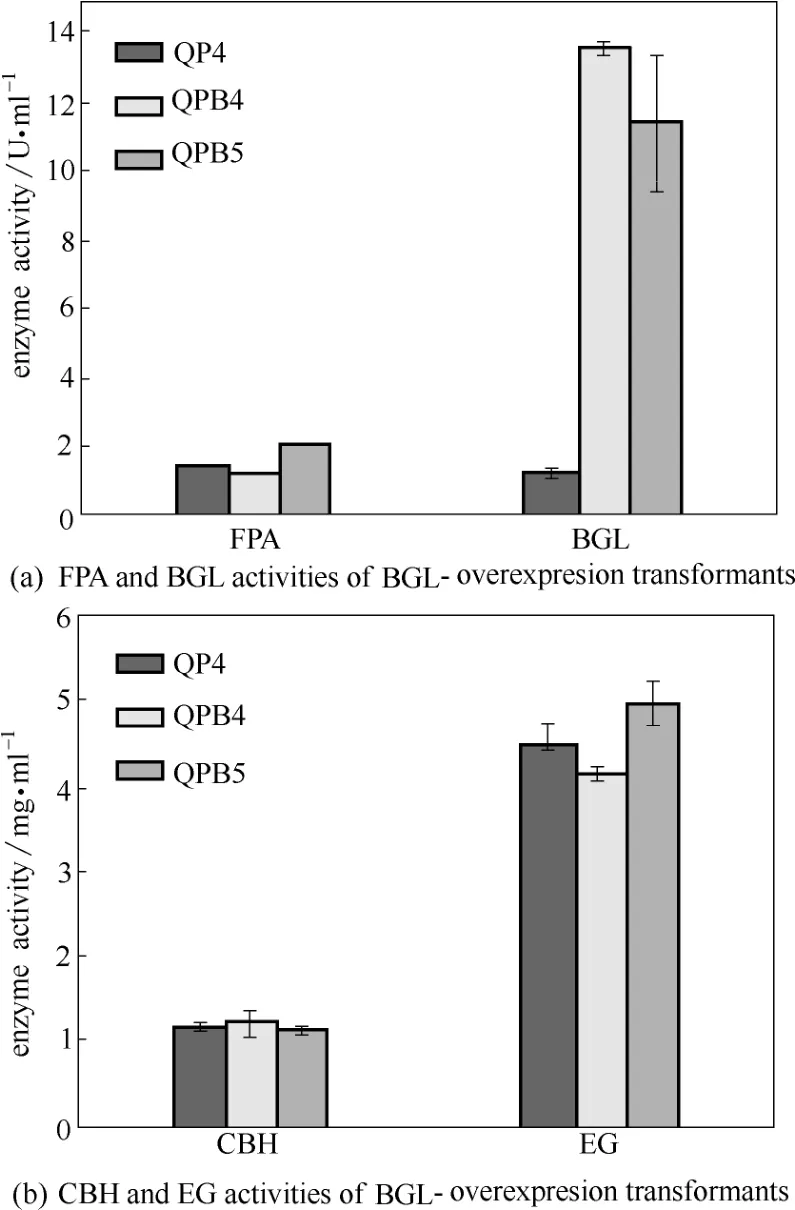
图5 里氏木霉β-葡萄糖苷酶过表达菌株纤维素酶活测定Fig.5 Enzyme activities of BGL-overexpresion transformants (FPA, BGL, CBH and EG were measured after being induced in cellulase fermentation medium for 5 d. Data are represented as mean of three independent experiments. Error bars express standard deviations)
2.6β-葡萄糖苷酶过表达菌株的糖化应用
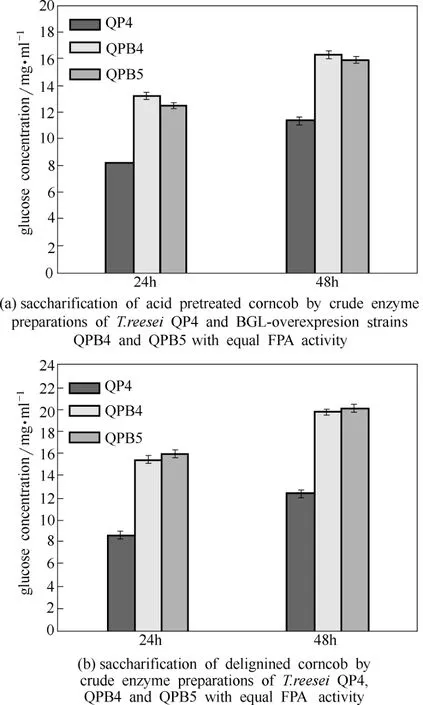
图6 过表达β-葡萄糖苷酶菌株发酵酶液对玉米芯材料的糖化应用Fig.6 Saccharification of different pretreated corncob residues by crude enzyme preparations of BGL-overexpression strains (Data are represented as mean of three independent experiments. Error bars express standard deviations)
为了检测里氏木霉高水平过表达β-葡萄糖苷酶对改良纤维素酶系的效果,本研究采用2种不同预处理的玉米芯材料作为糖化底物,测定了β-葡萄糖苷酶过表达菌株发酵酶液的糖化效率(图6)。结果显示,当以酸处理玉米芯作为底物时,糖化效率在24 h和48 h时均有大幅提升,尤其是24 h时更加明显,QPB4和QPB5发酵酶液的葡萄糖得率分别为13.20 mg·ml−1和12.50 mg·ml−1,比QP4 (8.20 mg·ml−1)分别提高了60.98%和52.44%[图6(a)]。当以脱木素处理玉米芯作为底物糖化时,过表达β-葡萄糖苷酶菌株提高糖化效率更加明显,24 h时QPB4(15.56 mg·ml−1)和QPB5(16.07 mg·ml−1)比QP4(8.64 mg·ml−1)分别提高了80.09%和86.00%[图6(b)]。QPB4和QPB5在糖化2种底物时,虽然葡萄糖得率均有大幅提升,但也出现一定差异,如QPB4糖化酸处理玉米芯底物效果比QPB5好,而QPB5糖化脱木素处理玉米芯底物效果比QPB4好,这可能与两个工程菌株酶系差异有关。总之,本研究成功实现了里氏木霉QM9414遗传操作体系的改进,并以此为基础改良纤维素酶组分,获得了糖化效率显著提高的工程菌,为下一步实际工业应用打下基础。
3 结 论
(1)通过定点敲除pyr4基因建立了里氏木霉高产突变株QM9414的尿嘧啶缺陷菌株QP4,显著提高了遗传转化效率,而且没有影响菌株生长和纤维素酶表达,成为替换尿嘧啶缺陷诱变菌株TU6进行遗传改良和纤维素酶表达机制研究的优良菌株。
(2)利用构建的尿嘧啶缺陷菌株QP4进行本源bgl1过表达,通过高效转化和筛选,仅进行一轮转化便获得了135个转化子,其中123株为遗传稳定转化子,从中进一步筛选出2株BGL活力最高的菌株,活力分别提高了10.01倍和8.26倍。这说明本研究对里氏木霉QM9414菌株的遗传转化体系改进是成功的,同时高水平过表达了β-葡萄糖苷酶。在进一步糖化应用方面,本研究获得的QPB5菌株发酵酶液将玉米芯底物水解的葡萄糖得率在24 h时就达到了16.07 mg·ml−1,比出发菌株提高了86.00%,这在本源过表达β-葡萄糖苷酶菌株提高糖化效率方面是非常显著的。
本研究借助转化体系改进和高水平过表达β-葡萄糖苷酶解决了里氏木霉高产突变株遗传操作困难和酶系组分缺陷两个限制菌种改良的因素,显著提升了里氏木霉的糖化应用能力。同时,高效遗传转化菌株的构建和高产菌株的获得也为研究里氏木霉纤维素酶高产机制以及进一步的实际生产应用打下基础。
References
[1]钟耀华, 钱远超, 任美斌, 等.丝状真菌降解转化纤维素的机制与遗传改良前景 [J]. 生物加工过程, 2014, 12 (1): 46-54. ZHONG Y H, QIAN Y C, REN M B, et al. Progress in bioconversion of cellulose by filamentous fungi and related genetic engineering strategies [J]. Chinese Journal of Bioprocess Engineering, 2014, 12 (1): 46-54
[2]CHANDELA K, CHANDRA S G, SILVA M B, et al. The realm of cellulases in biorefinery development [J]. Crit. Rev. Biotechnol., 2012, 32 (3): 187-202.
[3]LYND L R, WEIMER P J, VAN W H, et al. Microbial cellulose utilization: fundamentals and biotechnology [J]. Microbiol. Mol. Biol. Rev., 2002, 66 (3): 506-577.
[4]MANDELS M, REESE E T. Induction of cellulase in Trichoderma viride as influenced by carbon sources and metals [J]. J. Bacteriol., 1957, 73 (2): 269-278.
[5]PETERSON R, NEVALAINEN H. Trichoderma reesei RUT-C30: thirty years of strain improvement [J]. Microbiology, 2012, 158 (1): 58-68.
[6]陈永, 林良才, 肖冬光, 等. 里氏木霉RL-P37尿嘧啶营养缺陷型菌株的构建 [J].中国酿造, 2014, 33 (12): 58-62. CHEN Y, LIN L C, XIAO D G, et al. Construction of uracil-auxotrophic strain of Trichoderma reesei RL-P37 [J]. China Brewing, 2014, 33 (12): 58-62.
[7]LONG H, WANG T H, ZHANG Y K. Isolation of Trichoderma reesei pyrG negative mutant by UV mutagenesis and its application in transformation [J]. Chem. Res. Chinese Universities, 2008, 24 (5): 565-569.
[8]DU Y, XIE G, YANG C, et al. Construction of brewing-wine Aspergillus oryzae pyrG-mutant by pyrG gene deletion and its application in homology transformation [J]. Acta Biochim. Biophys. Sin. (Shanghai), 2014, 46 (6): 477-483.
[9]HIRASHIMA K, IWAKII T, TAKEGAWA K, et al. A simple and effective chromosome modification method for large-scale deletion of genome sequences and identification of essential genes in fission yeast [J]. Nucleic Acids Research, 2006, 34 (2): e11.
[10]GRUBER F, VISSER J, KUBICEK C P, et al. The development of a heterologous transformation system for the cellulolytic fungus Trichoderma reesei based on a pyrG-negative mutant strain [J]. Curr. Genet., 18 (1): 71-76.
[11]DELMAS S, LLANOS A, PARROU J L, et al. Development of an unmarked gene deletion system for the filamentous fungi Aspergillus niger and Talaromyces versatilis [J]. Appl. Environ. Microbiol., 2014, 80 (11): 3484-3487.
[12]DERNTL C, KIESENHOFER D P, MACH R L, et al. Novel strategies for genomic manipulation of Trichoderma reesei with the purpose of strain engineering [J]. Appl. Environ. Microbiol., 2015, 81 (18): 6314-6323.
[13]安莉颖, 易欣欣, 秦丽娜, 等. 里氏木霉RutC30常温常压等离子体(ARTP)诱变筛选pyr4基因缺陷型菌株及转化系统的建立 [J].菌物学报, 2014, 33 (6):1672-6472. AN L Y, YI X X, QIN L N, et al. Isolation of Trichoderma reesei RutC30 pyr4 gene mutant by atmospheric room temperature plasma (ARTP) and establishment of a transformation system [J]. Mycosystema, 2014, 33 (6): 1672-6472.
[14]SIPOS B, BENKO Z, DIENES D, et al. Characterisation of specific activities and hydrolytic properties of cell-wall-degrading enzymes produced by Trichoderma reesei Rut-C30 on different carbon sources [J]. Appl. Biochem. Biotechnol., 2010, 161 (1-8): 347-364.
[15]SALOHEIMO M, KUJA-PANULA J, YLOSMAKI E,et al.Enzymatic properties and intracellular localization of the novel Trichoderma reesei β-glucosidase BGLII(Cel1A) [J]. Appl. Environ. Microbiol., 2002, 68 (9): 4546-4553.
[16]ZHANG J W, ZHONG Y H, ZHAO X N, et al. Development of the cellulolytic fungus Trichoderma reesei strain with enhanced beta-glucosidase and filter paper activity using strong artificial cellobiohydrolase 1 promoter [J]. Bioresour. Technol., 2010, 101 (24): 9815-9818.
[17]MA L, ZHANG J, ZOU G, et al. Improvement of cellulase activity in Trichoderma reesei by heterologous expression of a beta-glucosidase gene from Penicillium decumbens [J]. Enzyme Microb. Technol., 2011, 49 (4): 366-371.
[18]LIU T, WANG T, LI X, et al. Improved heterologous gene expression in Trichodenna reesei by chhl promoter optimization [J]. Acta Biochim. Biophys. Sin. (Shanghai)., 2008, 40 (2): 158-165.
[19]SCHELL D J, FARMER J, NEWMAN M, et al. Dilute-sulfuric acid pretreatment of corn stover in pilot-scale reactor: investigation of yields, kinetics, and enzymatic digestibilities of solids [J]. Appl. Biochem. Biotechnol., 2003, 105-108: 69-85.
[20]ZHAO X, ZHANG L , LIU D . Comparative study on chemical pretreatment methods for improving enzymatic digestibility of crofton weed stem [J]. Bioresour. Technol., 2008, 99 (9): 3729-3736.
[21]LIU K, LIN X H, YUE J, et al. High concentration ethanol production from corncob residues by fed-batch strategy [J]. Bioresour. Technol., 2010, 101 (13): 4952-4958.
[22]PENTTILA M, NEVALAINEN H, RATTO M, et al. A versatile transformation system for the cellulolytic filamentous fungus Trichoderma reesei [J]. Gene, 1987, 61 (2): 155-164.
[23]YU J H, HAMARI Z, HAN K H, et al. Double-joint PCR: PCR-based molecular tool for gene manipulations in filamentous fungi [J]. Fungal Genet. Biol., 2004, 41 (11): 973-981.
[24]曲音波, 高培基, 王祖农. 青霉的纤维素酶抗降解物阻遏突变株的选育 [J]. 真菌学报, 1984, 3 (4): 238-243. QU Y B, GAO P J,WANG Z N. Screening of catabolite repressionresistant mutants of cellulase producing Penicillium spp [J]. AcfaMycol. Sin., 1984, 3 (4): 238-243.
[25]MURRAY P, ARO N, COLLINS C, et al. Expression in Trichoderma reesei and characterisation of a thermostable family 3 beta-glucosidase from the moderately thermophilic fungus Talaromyces emersonii [J]. Protein Expr. Purif., 2004, 38 (2): 248-257.
[26]CHENG Y, SONG X, QIN Y, et al. Genome shuffling improves production of cellulase by Penicillium decumbens JU-A10 [J]. J. Appl. Microbiol., 2009, 107 (6): 1837-1846.
[27]RAHMANZL, SHIDA Y, FURUKAWA T, et al. Application of Trichoderma reesei cellulase and xylanase promoters through homologous recombination for enhanced production of extracellular beta-glucosidase Ⅰ [J]. Biosci. Biotechnol. Biochem., 2009, 73 (5): 1083-1089.
Improvement of uracil auxotrophic transformation system in Trichoderma reesei QM9414 and overexpression of β-glucosidase
ZHONG Lixia1,2, QIAN Yuanchao2, DAI Meixue1, ZHONG Yaohua2
(1College of Life Sciences, Shandong Normal University, Jinan 250014, Shandong, China;2State Key Laboratory of Microbial Technology, School of Life Sciences, Shandong University, Jinan 250100, Shandong, China)
Abstract:Trichoderma reesei is an industrial filamentous fungus widely used for cellulase production. However, the difficulty in genetic modification of the high-production mutants limits the strain improvement. Firstly, the pyr4 gene was successfully deleted by target integration strategy in T. reesei QM9414 to construct a uracil auxotroph strain QP4. Compared with those in the parental strain, not only the genetic transformation efficiency was improved, but also the cellulase production was not influenced in QP4. The β-glucosidase (BGL) encoding gene bgl1 was further overexpressed in the QP4 strain. The transformants QPB4 and QPB5 with increased BGL activity were obtained by rapid screening based on the esculin plate. The BGL activities in the culture supernatants of QPB4 and QPB5 exhibited 10.01- and 8.26- fold higher than that in QP4, respectively. The crude enzyme preparations were used to saccharify two types of differently pretreated corncob residues. The glucose yields from saccharification of acid-pretreated corncob by QPB4 and QPB5 were 60.98% and 52.44% higher than that by QP4,respectively, while using the delignined corncob as substrate, the glucose yields by QPB4 and QPB5 were improved 80.01% and 86.00%, respectively. These results suggested that improvement of the genetic manipulation system in T. reesei greatly facilitated the strain improvement and increased the saccharification efficiency.
Key words:cellulases; Trichoderma reesei high-production mutant; genetic transformation; saccharification; uracil auxotroph; construction and expression; biotechnology; biological engineering
中图分类号:TQ 812
文献标志码:A
文章编号:0438—1157(2016)06—2510—09
DOI:10.11949/j.issn.0438-1157.20151654
基金项目:国家自然科学基金面上项目(31370135);山东省农业科技成果转化资金项目(2014No.45);山东大学基本科研业务费专项资金项目(2015CJ005)。
Corresponding author:DAI Meixue, daimeixue@sdnu.edu.cn; ZHONG Yaohua, zhongyaohua@sdu.edu.cn

