鹅掌楸DHHC型锌指蛋白家族基因的克隆及表达分析
徐嘉娟, 李火根
( 南京林业大学 林木遗传与生物技术省部共建教育部重点实验室, 南京 210037 )
鹅掌楸DHHC型锌指蛋白家族基因的克隆及表达分析
徐嘉娟, 李火根
( 南京林业大学 林木遗传与生物技术省部共建教育部重点实验室, 南京 210037 )
棕榈酰化修饰是一种最普遍且唯一可逆的翻译后脂质修饰方式,赋予蛋白质多样化的生理功能。DHHC(Asp-His-His-Cys)蛋白家族是一类与棕榈酰化修饰相关的蛋白,多数DHHC蛋白家族成员具有蛋白质酰基转移酶(protein S-acyltransferase,PAT)活性。该研究以鹅掌楸叶芽为材料,采用RT-PCR和RACE技术,克隆获得了3个鹅掌楸DHHC蛋白家族基因cDNA全长,命名为LcPAT7、LcPAT22、LcPAT23。序列分析结果表明:LcPAT7、LcPAT22、LcPAT23基因全长分别为1 933、2 592、2 217 bp,各包含1 332、1 839、1 662 bp的开放阅读框(Open Reading Frame,ORF),编码433、612、533个氨基酸,预测蛋白分子量分别为40.04、67.3、60.57 kDa,理论等电点为9.15、9.03、7.29。3个基因编码的蛋白均有4个跨膜区,并且都在跨膜域(transmembrane domain,TM)TM2和TM3之间存在一个DHHC蛋白家族典型的DHHC-CRD结构域。同源性分析表明:鹅掌楸LcPAT7、LcPAT22、LcPAT23编码的氨基酸序列与其他植物中预测的PAT具有较高的相似性。利用荧光定量PCR技术检测3个基因在鹅掌楸不同组织中的表达特性,发现3个基因在不同组织中均有表达,但表达量具有明显区别。同一家族基因表达模式的变化表明其功能非冗余。该研究结果将为鹅掌楸生长发育与形态建成,以及逆境响应信号传导等相关基因的调控研究提供了参考。
鹅掌楸, 棕榈酰化修饰, 蛋白质酰基转移酶, DHHC蛋白家族, 表达分析
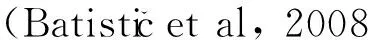
近年来,对哺乳动物棕榈酰基转移酶的研究发现,一些富含DHHC结构域的蛋白与人类某些疾病相关。如DHHC9被证明与早期智力残疾相关的X-连锁的精神迟钝有关(Mitchell et al, 2014),膀胱癌和肝母细胞瘤的发生与DHHC11密切相关(Yamamoto et al, 2007;Wu et al, 2013),DHHC17(HIP14)负责亨廷顿蛋白(Htt)的棕榈酰化,与亨廷顿舞蹈症相关(Sanders et al, 2015)。高等植物基因组中也存在数目不等的DHHC蛋白基因,例如玉米(Zeamays)中有40个,杨树(Populustrichocarpa)39个,水稻(Oryzasativa)30个,拟南芥(Arabidopsisthaliana)24个(Yuan et al, 2013)。但与哺乳动物和酵母相比,对植物PATs的研究还相对较少(Hemsley, 2013)。DHHC型锌指蛋白在植物的生长发育过程中起着非常重要的作用。例如,从拟南芥突变体中分离得到的DHHC型锌指蛋白TIPl,与酵母的Akrl和人的HIPl4有很大的相似性,它们都具有蛋白酰基转移酶的活性。TIPl在拟南芥中参与调节植物细胞的生长,对植株的生长和株型发育具有重要调控作用(Hemsley et al, 2005)。AtPAT10(At3g51390)对植株的生长发育起到重要的作用,同时参与植株对外界盐胁迫的响应过程(Zhou et al, 2013)。拟南芥DHHC型锌指蛋白基因At5g04279和水稻DHHC型锌指蛋白基因OsDHHC1与株型调控有关(Xiang et al, 2010;周波, 2011)。
木兰科(Magnoliaceae)鹅掌楸属(Liriodendron)现仅存鹅掌楸(L.chinense)和北美鹅掌楸(L.tulipifera)2个种。鹅掌楸主要分布于我国长江流域以南的亚热带中、低山区,其干形通直、树形美观、叶形奇特、花色艳丽、木材纹理通直、结构细密,材质轻软,是珍贵用材与园林观赏兼用树种(蔡伟建等, 2011;姚俊修等, 2012)。本研究以鹅掌楸叶芽为材料,根据筛选到的DHHC锌指蛋白家族基因的部分序列,采用RT-PCR和RACE技术克隆了3个鹅掌楸DHHC蛋白基因LcPAT7、LcPAT22、LcPAT23,并对其进行生物信息学分析。同时,采用实时荧光定量PCR技术检测了3个基因在鹅掌楸不同组织中的表达特性,期望为鹅掌楸生长发育、形态建成以及逆境响应信号传导等相关基因的调控研究提供参考。
1 材料与方法
1.1 植物材料
鹅掌楸(LS)采自南京林业大学下蜀实习林场的鹅掌楸属种源试验林,树龄20 a,分别取其幼嫩叶片、花芽、叶芽和盛花期的花瓣、雄蕊、雌蕊,液氮速冻后置于-80 ℃超低温冰箱保存备用。
1.2 总RNA的提取及cDNA第1链的合成
利用RNAprep Pure植物总RNA提取试剂盒(TIANGEN)分别提取上述6个不同组织样品的总RNA,用紫外分光光度计检测RNA的浓度和OD值,并取适量体积RNA用1%的琼脂糖凝胶电泳检测。
以叶芽总RNA为模板,按照RevertAid strand cDNA Synthesis Kit(Thermo Scientific)合成cDNA第1链,参照TaKaRa公司的3′-Full RACE Core Set with PrimeScript RTase和5′-Full RACE Kit试剂盒操作说明分别合成3′cDNA和5′cDNA;以6个不同组织样品总RAN为模板,均一化浓度后,按照M-MLV First Strand Kit(Invitrogen)合成用于荧光定量PCR的cDNA,存于-20 ℃备用。
1.3 基因全长cDNA的克隆
(1)以NCBI的拟南芥TIP1基因序列(NC_003076.8)为信息探针,从本实验室鹅掌楸转录组数据(Yang et al, 2014)中筛选出3个DHHC蛋白基因EST序列。利用Oligo 6软件分别设计引物P7F1、P7R1,P22F1、P22R1和P23F1、P23R1(表1),送上海捷瑞生物公司合成。以cDNA模板,进行PCR扩增。反应体系(50 μL):10 × LA PCR Buffer II(Mg2+Free)5 μL,Mgcl2(25 mol·L-1)5 μL,dNTP Mixture(2.5 mol·L-1each)8 μL,引物各2 μL,TaKaRaLATaq(5 U·μL-1)0.5 μL,cDNA 2μL,无菌双蒸水25.5 μL。扩增程序:预变性(94 ℃,3 min);变性(94 ℃,30 s),退火(30 s),延伸(72 ℃,2 min),30个循环;总延伸(72 ℃,10 min)。扩增3个基因的退火温度分别59、56、58 ℃。将得到的PCR产物经2%琼脂糖凝胶电泳检测后,切下目的条带,用胶回收试剂盒AxyPrep DNA Gel Extraction Kit(Axygen)回收,产物与pEASY-T1 Cloning Vector(TransGen Biotech)载体相连接,转化Trans1-T1感受态细胞(TransGen Biotech),进行蓝白斑筛选,挑取白斑进行菌落PCR鉴定后,将筛选到的阳性克隆菌液送往上海英骏生物技术有限公司测序,获得3个基因的部分序列。
(2)根据(1)所得3个基因测序验证的部分序列,利用Oligo 6软件按照RACE试剂盒巢式PCR的要求分别设计3个基因3′末端和5′末端各2条末端扩增引物(表1)。
3′ RACE:以3′RACE cDNA为模板,用P7-3GSP1、P22-3GSP1、P23-3GSP1分别与3′Outer primer引物进行巢式第1轮PCR扩增,反应体系同3′RACE试剂盒说明书,扩增条件:预变性(94 ℃,3 min);变性(94 ℃,30 s),退火(56 ℃,30 s),延伸(72 ℃,2 min),25个循环;总延伸(72 ℃,10 min)。以第一轮PCR的产物为模板,用P7-3GSP2、P22-3GSP2、P23-3GSP2分别与3′Inner primer引物进行巢式第2轮PCR扩增,反应体系同3′RACE试剂盒说明书。扩增条件同上,退火温度分别为58、61、62 ℃,共30个循环。克隆测序参照1.3的方法。
5′ RACE:以侧芽5′RACE cDNA为模板,以5′RACE引物P7-5GSP1、P22-5GSP1、P23-5GSP1(表1)分别与5′Outer primer引物进行巢式第一轮PCR扩增,反应体系同5′RACE试剂盒说明书,扩增条件:预变性(94 ℃,3 min);变性(94 ℃,30 s),退火(58 ℃,30 s),延伸(72 ℃,2 min),25个循环;总延伸(72 ℃,10 min)。以第一轮PCR的产物为模板,用P7-5GSP2、P22-5GSP2、P23-5GSP2(表1)分别与5′Inner primer引物进行巢式第二轮PCR扩增,反应体系同5′RACE试剂盒说明书,扩增条件同上,退火温度分别61、63、64 ℃,共30个循环。克隆测序参照1.3的方法。
1.4 基因开放阅读框(ORF)预测及扩增
将1.3中扩增得到的序列在DNAMAN 6.0软件中进行拼接获得基因全长,利用在线分析工具FGENESH,对拼接得到的基因全长序列的ORF进行预测。将预测得到的ORF编码的氨基酸序列在NCBI中进行比对以进一步确认ORF的准确性,并用Oligo 6设计引物(表1),以cDNA为模板,用2×TransStartFastPfuPCR SuperMix(TransGen Biotech)进行ORF扩增,反应体系(50 μL):2×TransStartFastPfuPCR SuperMix 25 μL,引物各2 μL,cDNA 2 μL,无菌双蒸水19 μL。反应条件:预变性(94 ℃,3 min);变性(94 ℃,30 s),退火(30 s),延伸(72 ℃,2min),30个循环;总延伸(72 ℃,10 min)。退火温度分别为60 ℃、58 ℃、58 ℃。克隆测序方法同1.3,产物用pEASY-Blunt克隆载体(TransGen Biotech)连接。

表 1 克隆用引物
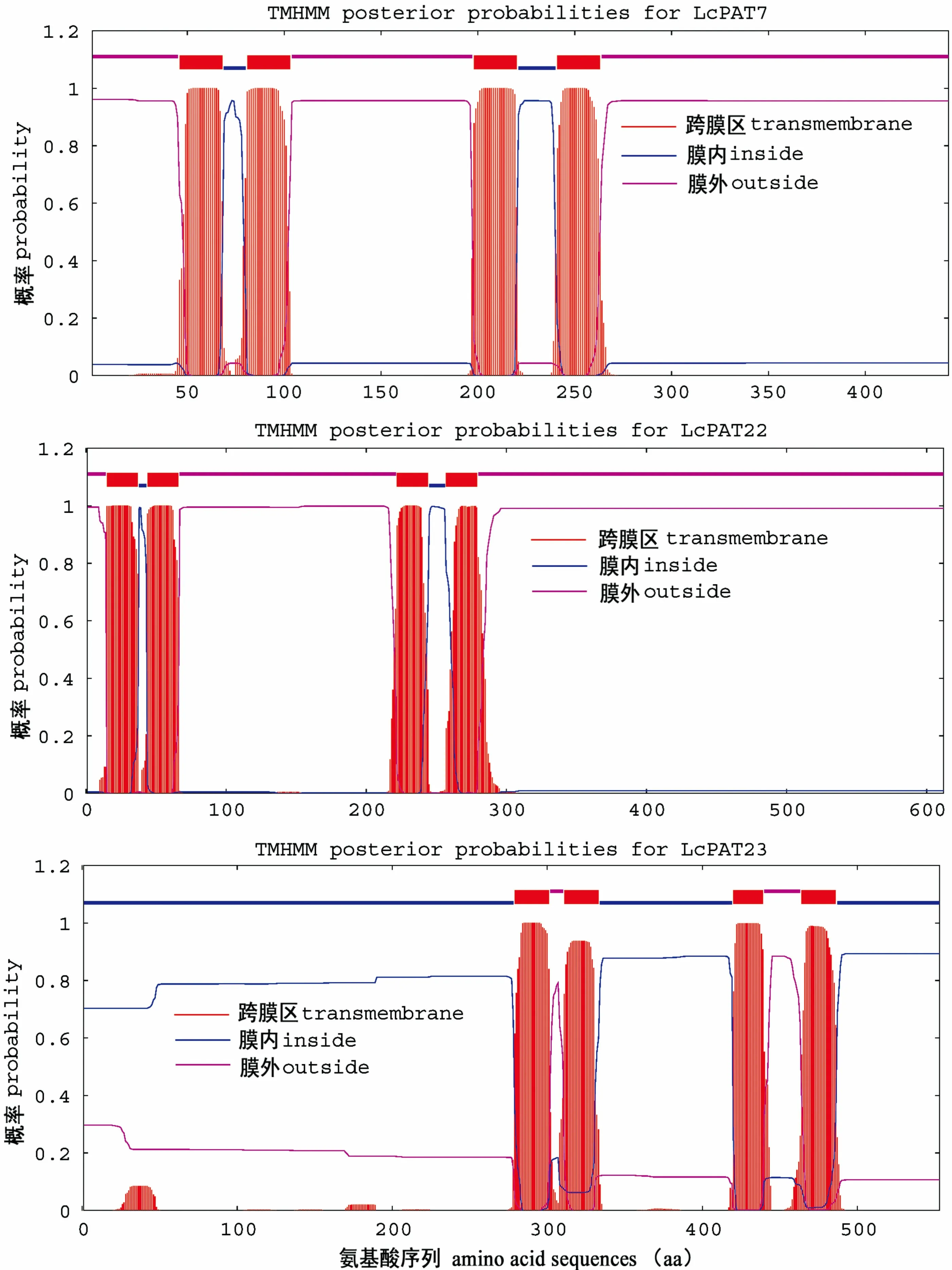
图 1 LcPAT7、LcPAT22、LcPAT23的生物信息学分析 (跨膜结构分析)Fig. 1 Bioinformatics analysis of LcPATs (Prediction of transmembrane helices in proteins)
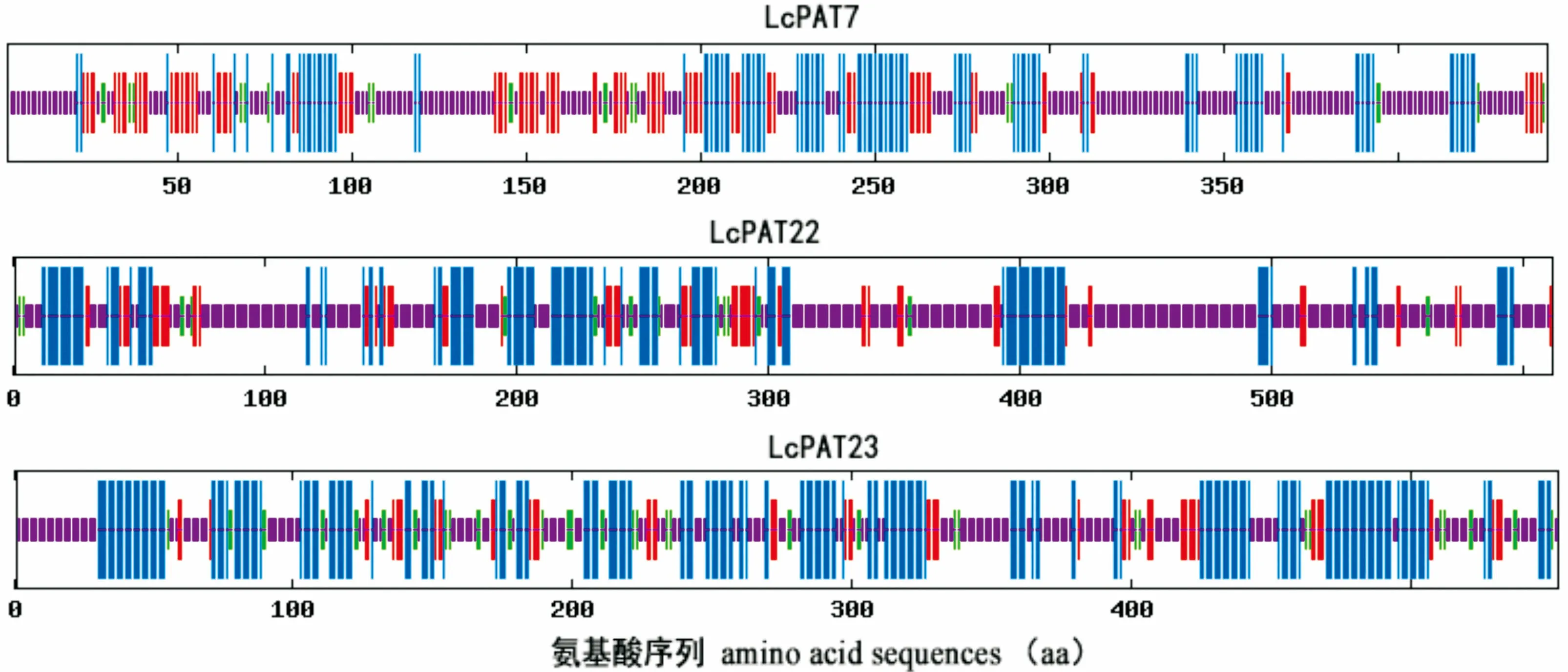
图 2 LcPAT7、LcPAT22、LcPAT23的生物信息学分析 (二级结构预测)Fig. 2 Bioinformatics analysis of LcPATs (Secondary structure prediction)
图 3 LcPAT7、LcPAT22、LcPAT23与拟南芥PAT基因家族系统进化树 At. 拟南芥; Lc. 鹅掌楸。 节点上的数值表示Bootstrap验证中基于1 000次重复该节点可信度的百分比。Fig. 3 Phylogenetic tree of LcPATs and Arabidopsis thaliana PAT family At. A. thaliana; Lc. Liriodendron chinense. The number at the nodes represents the reliability percent (%) of bootstraps values based on 1 000 replications.
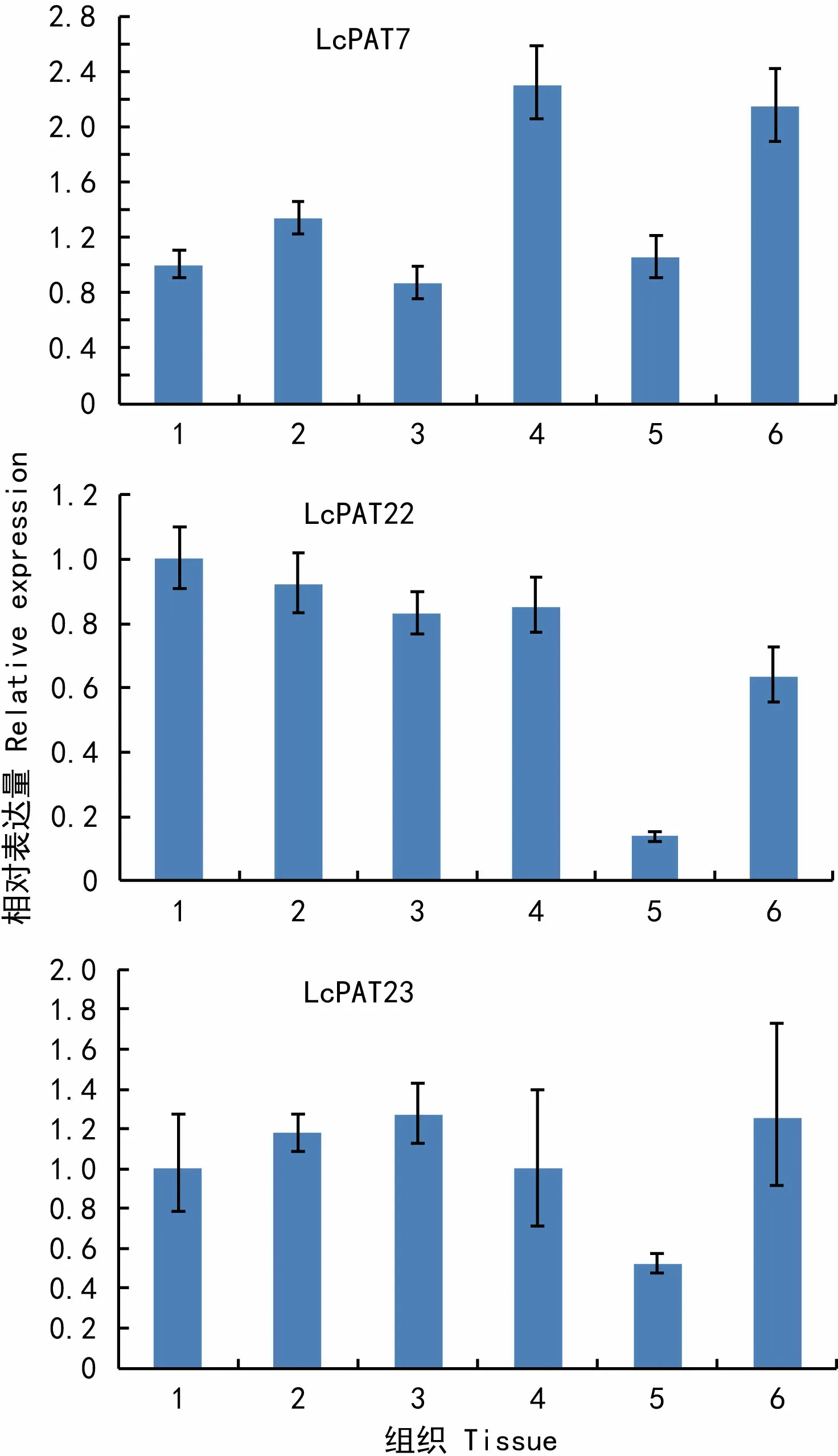
图 4 LcPAT7、LcPAT22、LcPAT23在鹅掌楸不同组织中的表达情况 1. 叶芽; 2. 叶片; 3. 花芽; 4. 花瓣; 5. 雄蕊; 6. 雌蕊。Fig. 4 Expression level of PATs in different tissues of Liriodendron chinense 1. Leaf bud; 2. Leaf; 3. Floral bud; 4. Petal; 5. Stamen; 6. Pistil.
1.5 基因的组织表达分析
利用1.2所得到的cDNA进行实时定量PCR(quantitative real-time PCR,qRT-PCR)分析,以检测目的基因在不同组织的相对表达量。采用Primer Express Software version 3.0(Applied Biosystems)软件进行qRT-PCR引物设计,以鹅掌楸肌动蛋白基因Actin为内参,所用引物见表1。用SYBRPremixExTaq(Tli RNaseH Plus)(TaKaRa)实时荧光定量PCR试剂盒进行试验,在ABI 7500 PCR仪上完成,每个反应重复3次。反应体系(20 μL):SYBRPremixExTaq(Tli RNaseH Plus)(2×)10 μL,ROX Reference Dye II(50×)0.4 μL,Forward/Reverse primer(10 μmol·L-1)0.4 μL,cDNA 2 μL,无菌双蒸水6.8 μL。反应条件:预变性(95 ℃,30 s);变性(95 ℃,5 s),退火延伸(58 ℃,34 s),40个循环。
1.6 基因生物信息学分析
利用FGENESH(http://mendel.cs.rhul.ac.uk/mendel.php?topic=fgen)软件进行ORF确定及氨基酸序列预测。用在线工具Expasy ProtParam(http://web.expasy.org/protparam/)分析蛋白的分子质量、理论等电点、氨基酸组成。通过Pfam数据库(http://pfam.xfam.org/search)推测蛋白质结构域。使用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析。利用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)程序进行蛋白序列跨膜区分析。蛋白质二级结构分析采用SOPMA(http//npsa-pbil.ibcp.fr/gi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)工具进行,利用MEGA 5.1邻接(Neighbor Joining,NJ)算法构建系统进化树。
2 结果与分析
2.1 鹅掌楸DHHC蛋白基因全长克隆与序列分析
以鹅掌楸cDNA为模板,分别以P7F、P7R,P22F、P22R和P23F、P23R为引物,扩增得到1 358、2 070、1 687 bp的片段。通过3′RACE和5′RACE技术分别获得cDNA的3′端(1 103、1 044、713 bp)和5′端(1 235、1 062、1 127 bp);采用DNAMAN 6.0软件拼接出3个DHHC蛋白基因的全长cDNA序列(1 933、2 592、2 217 bp);利用在线工具FGENESH进行ORF预测,并在NCBI蛋白质数据库中进行Blast比对。结果显示,目的序列与其他植物中预测的PAT基因高度相似,并根据与NCBI中拟南芥PAT基因的比对结果,将其分别命名为LcPAT7(1 933 bp)、LcPAT22(2 592 bp)、LcPAT23(2 217 bp)。
设计引物P7OF、P7OR,P22OF、P22OR,P23OF、P23OR进行ORF扩增,测序验证。结果表明,LcPAT7含一个1 332 bp的开放阅读框,编码443个氨基酸。其预测蛋白的分子量为49.04 kDa,理论等电点为9.15。酸性氨基酸(Asp,Glu)总数35个,碱性氨基酸(Arg,Lys)总数46个;GRAVY(grand average of hydropathicity)值为-0.021;不稳定指数为32.08,该蛋白质为稳定蛋白。LcPAT22含一个1 839 bp的开放阅读框,编码612个氨基酸,其预测蛋白的等电点和分子质量分别为9.03和67.3 kDa。酸性氨基酸(Asp,Glu)总数53个,碱性氨基酸(Arg,Lys)总数67个;GRAVY值为-0.174;不稳定指数为53.92,该蛋白质为不稳定蛋白。LcPAT23含一个1 662 bp的开放阅读框,编码553个氨基酸,其预测蛋白的等电点和分子质量分别为7.29和60.57 kDa。酸性氨基酸(Asp,Glu)总数50个,碱性氨基酸(Arg,Lys)总数50个;GRAVY值为-0.080;不稳定指数为32.83,该蛋白质为稳定蛋白。
2.2 蛋白质结构分析及系统进化树构建
以鹅掌楸叶芽cDNA为模板分离得到的3个DHHC蛋白基因,通过SignalP 4.1对3个基因进行信号肽预测。结果表明,该3个DHHC蛋白基因均不存在信号肽酶切位点,都属非分泌型蛋白。蛋白质跨膜区分析结果表明,3个基因所编码蛋白均具有4个跨膜区(图1)。Pfam数据库检索显示,LcPAT7、LcPAT22和LcPAT23均含有zf-DHHC结构域,且均位于跨膜域TM2和TM3之间,其中LcPAT23在N端有2个锚蛋白重复序列(Ank_2)。此外,3个基因所编码蛋白均具有DHHC蛋白家族典型的结构域:C-X2-C-X9-HC-X2-C-X2-C-X4-DHHC-X5-C-X4-N-X3-F-X4(C代表半胱氨酸,H代表组氨酸,X代表任意氨基酸)(Putilina et al, 1999),说明其编码的蛋白属于DHHC蛋白家族成员。
SOPMA分析表明,α螺旋(Alpha helix)和无规则卷曲(Random coil)是这3个DHHC蛋白二级结构的主要成分。LcPAT7蛋白二级结构中含有23.70%的α螺旋、20.99%的延伸链(Extended strand)、4.74%的β转角(Beta turn)和50.56%的无规则卷曲;LcPAT22蛋白二级结构中含有25.33%的α螺旋、11.11%的延伸链3.43%的β转角和60.13%的无规则卷曲;LcPAT23蛋白二级结构中含有41.05%的α螺旋、10.49%的延伸链8.32%的β转角和40.14%的无规则卷曲(图2)。ProtComp 9.0蛋白亚细胞定位分析表明,LcPAT7定位于质膜,与AtPAT7一致;LcPAT22定位于质膜;而LcPAT23定位于高尔基体,与AtPAT23和AtPAT24(TIP1)定位一致。
通过NCBI上的protein blast进行同源性搜索,发现与它们同源的序列大部分都为预测的蛋白质酰基转移酶(PAT),LcPAT7与莲(Nelumbonucifera)、葡萄(Vitisvinifera)、大豆(Glycinemax)、烟草(Solanumtuberosum)、拟南芥(A.thaliana)中预测的PAT7相似性分别为81%、77%、74%、74%、70%;LcPAT22与莲、葡萄、胡杨(Populuseuphratica)、海枣(Phoenixdactylifera)、烟草、拟南芥中预测的PAT22相似性分别为72%、70%、68%、67%、64%、61%;LcPAT23与莲、海枣、葡萄、大豆、拟南芥中预测的PAT23相似性分别为77%、75%、72%、71%、60%。
应用Clustal W软件将3个鹅掌楸DHHC蛋白家族基因与NCBI数据库下载的24个拟南芥PATs蛋白进行多重比对,再利用MEGA 5.1软件中的邻接法构建系统进化树,结果见图3。从图3可看出,LcPAT7与拟南芥中的AtPAT6和AtPAT7关系最近,LcPAT22与拟南芥中的AtPAT22关系最近,LcPAT23与拟南芥中的AtPAT23关系最近,且LcPAT23与AtPAT24(TIP1)归为同一个小的分支。这为推测LcPAT23的功能提供了一定的线索。TIP1是拟南芥中第一个进行功能研究的蛋白质棕榈酰基转移酶,研究发现TIP1突变体不仅在根毛及花粉管的生长上表现出异常,还表现出节间缩短、植株矮小、莲座叶变小、叶表皮细胞变小,且早花早衰,说明TIP1基因不但参与极性细胞的生长过程,同时还参与多种细胞的发育过程(Hemsley et al, 2005)。
2.3 DHHC基因在鹅掌楸不同组织中的表达特性
利用实时荧光定量PCR技术对3个基因在鹅掌楸不同组织中的表达模式进行分析,结果表明(图4),3个基因在鹅掌楸叶芽、花瓣、雌蕊、花芽、雄蕊、叶片中均有表达,但表达丰度存在着明显的差异。LcPAT7在花瓣中表达丰度最高,其次为雌蕊,在花芽和叶芽中的表达丰度相对较低;LcPAT22在叶芽中的相对表达丰度最高,叶片和花芽中次之,雌蕊中的表达丰度较低,而雄蕊中最低;LcPAT23在花芽和雌蕊中表达丰度较高,叶片、花瓣、花芽中表达丰度相差不大,而在雄蕊中表达丰度较低。
3 讨论
本研究首次从鹅掌楸中克隆得到3个DHHC蛋白家族基因LcPAT7、LcPAT22、LcPAT23的全长cDNA序列。其编码的蛋白均具有4个跨膜区,这与已报导的动植物DHHC蛋白跨膜域数目(4~6)一致,并且均在跨膜域TM2和TM3之间存在一个DHHC蛋白家族典型的结构域:C-X2-C-X9-HC-X2-C-X2-C-X4-DHHC-X5-C-X4-N-X3-F-X4,该结构域是动植物中S-酰基转移酶的活性位点(Putilina et al, 1999)。DHHC中的半胱氨酸(Cysteine,C)残基突变为丝氨酸(Serine,S)后,不会改变蛋白质的空间结构,但是会导致棕榈酰基转移酶丧失酶活性,表明该高度保守的半胱氨酸残基是酶活性的关键决定位点(Greaves & Chamerlain, 2011)。
高等生物的基因表达具有组织特异性和发育阶段特异性,不同基因随时间、空间选择性表达。基因的时空表达决定了生命的所有过程,如发育和分化、对逆境的反应、细胞分裂、衰老等。王倩等(2011)对拟南芥PAT基因家族中20个基因的研究表明:AtPAT7(At3g26935)在花、成熟果荚、茎中有表达,在花中表达量较高其他组织中表达量很低;AtPAT22(At1g69420)在果荚、花、幼苗、茎中有表达,在成熟果荚和花中表达量较高;AtPAT23(At2g14255)在幼苗、果荚、花、根、叶、茎中均有表达,在幼苗中表达量较高。荧光实时定量PCR分析结果表明,鹅掌楸LcPATs表达具有组织的差异性。LcPAT7、LcPAT22、LcPAT23在鹅掌楸花芽和盛花期的叶片、叶芽、花瓣、雄蕊、雌蕊中均有表达。LcPAT7在花瓣和雌蕊中表达丰度最高,功能上可能与AtPAT7具有一定的相似性,参与花瓣的扩展、心皮及胚的发育;LcPAT23在花芽和雌蕊中表达量相对较高,可能参与花发育过程;LcPAT22在叶芽中的表达量最高,其次是叶片、花芽,相对于LcPAT7、LcPAT23的表达模式,LcPAT22在叶芽、花芽中的表达丰度较其他组织高,可能参与茎端分生组织的生长调节。
ABANKWA D, GORFE AA, HANCOCK JF, 2007. Ras nanoclusters: molecular structure and assembly [J]. Semin Cell Dev Biol, 18(5): 599-607.

CAI JW, GUO X, GAO HD, et al, 2011. Research advances on cultivation ofLiriodendronplantation [J]. J Fujian For Sci Technol, 38(2): 164-170. [蔡伟建, 郭鑫, 高捍东, 等, 2011. 鹅掌楸属植物人工林培育研究进展 [J]. 福建林业科技, 38(2): 164-170.]CHAROLLAIS J, VAN DER GOOT FG, 2009. Palmitoylation of membrane proteins (Review) [J]. Mol Membr Biol, 26(1): 55-66.
CHOI Y, LEE Y, HWANG J, 2014.ArabidopsisROP9 and ROP10 GTPases differentially regulate auxin and ABA responses [J]. J Plant Biol, 57(4): 245-254.
GREAVES J, CHAMBERLAIN LH, 2011. DHHC palmitoyl transferases: substrate interactions and (patho)physiology [J]. Trends Biochem Sci, 36(5): 245-253.
GUAN XM, FIERKE CA, 2011. Understanding protein palmitoylation:biological significance and enzymology [J]. Sci Chin Chem, 54(12): 1 888-1 897.
HEMSLEY PA, KEMP AC, GRIERSON CS, 2005. The TIP GROWTH DEFECTIVE1 S-acyl transferase regulates plant cell growth inArabidopsis[J]. Plant Cell, 17(9): 2 554-2 563.
HEMSLEY PA, 2013. Assayingprotein S-acylation in plants [J]. Meth Mol Biol, 1 043: 141-146.
HUANG K, EL-HUSSEINI A, 2005. Modulation of neuronal protein trafficking and function by palmitoylation [J]. Curr Opin Neurobiol, 15(5): 527-535.
LAVY M, YALOVSKY S, 2006. Association ofArabidopsistype-II ROPs with the plasma membrane requires a conserved C-terminal sequence motif and a proximal polybasic domain [J]. Plant J, 46(6): 934-947.
LECLERCQ J, RANTY B, SANCHEZ-BALLESTA MT, et al, 2005. Molecular and biochemical characterization of LeCRK1, a ripening-associated tomato CDPK-related kinase [J]. J Exp Bot, 56(409): 25-35.
LINDER ME, DESCHENES RJ, 2007. Palmitoylation: policing protein stability and traffic [J]. Nat Rev Mol Cell Biol, 8(1): 74-84.
MITCHELL DA, HAMEL LD, REDDY KD, et al, 2014. Mutations in the X-linked intellectual disability gene, zDHHC9, alter autopalmitoylation activity by distinct mechanisms [J]. J Biol Chem, 289(26): 18 582-18 592.
MITCHELL DA, VASUDEVAN A, LINDER ME, et al, 2006. Thematic review series: lipid posttranslational modifications. Protein palmitoylation by a family of DHHC protein S-acyltransferases [J]. J Lipid Res, 47(6): 1 118-1 127.
PEDRO B, VÉRONIQUE A, LILLIAN RK, et al, 2012. Systematic functional prioritization of protein posttranslational modifications [J]. Cell, 150(2): 413-425.
PUTILINA T, WONG P, GENTLEMAN S, 1999. The DHHC domain: a new highly conserved cysteine-rich motif [J]. Mol Cell Biochem, 195(1-2): 219-226.
RESH MD, 2006. Trafficking and signaling by fatty-acylated and prenylated proteins [J]. Nat Chem Biol, 2(11): 584-590.
ROTH AF, FENG Y, CHEN L, et al. 2002. The yeast DHHC cysteine-rich domain protein Akr1p is a palmitoyl transferase [J]. J Cell Biol, 159(1): 23-28.RUAN BJ, DAI P, WANG W, et al, 2014. Progress on post-translational modification of proteins [J]. Chin J Cell Biol, 36(7): 1 027-1 037. [阮班军, 代鹏, 王伟, 等, 2014. 蛋白质翻译后修饰研究进展 [J]. 中国细胞生物学学报, 36(7): 1 027-1 037.]
SANDERS SS, HOU J, SUTTON LM, et al, 2015. Huntingtin interacting proteins 14 and 14-like are required for chorioallantoic fusion during early placental development [J]. Dev Biol, 397(2): 257-266.SCHÖNICHEN A, WEBB BA, JACOBSON MP, et al, 2013. Considering protonation as a posttranslational modification regulating protein structure and function [J]. Ann Rev Biophys, 42: 289-314.
SUN PG, MIAO HX, XU BY, et al. 2013. Proceedings in biological function of small GTPases gene ROP of plant [J]. North Hortic, (22): 188-192. [孙佩光, 苗红霞, 徐碧玉, 等. 2013. 植物小G蛋白基因ROP生物学功能研究进展 [J]. 北方园艺, (22): 188-192.]WANG Q, SUN JL, BAO L, et al, 2011. Twenty putative palmitoyl-acyl transferase genes show distinct expression pattern inArabidopsisthaliana[J]. Afr J Biotechnol, 10(52): 10 575-10 584.
WOLFF J, ZAMBITO AM, BRITTO PJ, et al, 2000. Autopalmitoylation of tubulin [J]. Protein Sci, 9(7): 1 357-1 364.
WU JF, LEE CH, CHEN HL, et al, 2013. Copy-number variations in hepatoblastoma associate with unique clinical features [J]. Hepatol Int, 7(1): 208-214.
XIANG J, LIN JZ, TANG DY, et al, 2010. A DHHC-type zinc finger protein gene regulates shoot branching inArabidopsis[J]. Afr J Biotechnol, 9(45): 7 759-7 766.
YAMAMOTO Y, CHOCHI Y, MATSUYAMA H, et al, 2007. Gain of 5p15.33 is associated with progression of bladder cancer [J]. Oncology, 72(1-2): 132-138.
YANG Y, XU M, LUO QF, et al, 2014. De novo transcriptome analysis ofLiriodendronchinensepetals and leaves by Illumina sequencing [J]. Gene, 534(2): 155-162.
YAO JX, LI HG, BIAN LM, et al, 2012. Inbreeding depression inLiriodendronrevealed by both phenotypic and genotypic characteristics [J]. J NE For Univ, 40(4): 1-5. [姚俊修, 李火根, 边黎明, 等, 2012. 鹅掌楸属树种近交衰退分析 [J]. 东北林业大学学报, 40(4): 1-5.]
YUAN XW, ZHANG SZ, SUN MH, et al, 2013. Putative DHHC-cysteine-rich domain S-acyltransferase in plants [J]. PLoS ONE, 8(10): e75985.
ZHOU B, 2011. Function analysis of the DHHC type znic finger protein geneOsDHHC1 in the plant architecture construction of rice(Oryzasativa L.) [D]. Changsha:Hunan University. [周波, 2011. DHHC型锌指蛋白基因OsDHHC1在水稻株型构建中的功能分析 [D]. 长沙:湖南大学.]
ZHOU LZ, LI S, FENG QN, et al, 2013. Protein S-ACYL Transferase10 is critical for development and salt tolerance inArabidopsis[J]. Plant Cell, 25(3): 1 093-1 107.
Gene cloning and expression analysis of DHHC protein family genes fromLiriodendronchinense
XU Jia-Juan, LI Huo-Gen
(KeyLabofForestGeneticsandBiotechnology,NanjingForestryUniversity,MinistryofEducation, Nanjing 210037, China )
Protein S-acylation is a common and unique reversible way in posttranslational lipid modification, thus confers diverse physiological functions on target proteins, such as DHHC (Asp-His-His-Cys) protein family. There was evidence that the DHHC domain was directly involved in the palmitoyl transfer reaction. In this paper, we reports gene cloning and expression analysis of DHHC protein family genes inLiriodendronchinense. Three full-length cDNA of DHHC gene were cloned fromLiriodendronleaf buds using the rapid amplification of cDNA ends (RACE) strategy, namedLcPAT7,LcPAT22 andLcPAT23 respectively. The full-length cDNAs ofLcPAT7,LcPAT22 andLcPAT23 were 1 933 bp, 2 592 bp and 2 217 bp, and contain 1 332 bp, 1 839 bp and 1 662 bp ORF, encoding 433, 612 and 533 amino acids, respectively. The predicted molecular weights of the proteins encoded byLcPAT7,LcPAT22 andLcPAT23 were 40.04, 67.3 and 60.57 kDa, respectively. The predicted isoelectric points are 9.15, 9.03 and 7.29, respectively. All the three proteins contained four putative transmembrane (TM) domain structures, and contained a typically DHHC-CRD domain between TM2 and TM3 as most known DHHC protein. Homology analysis showed that the three genes showed high similarity with predicted PAT (protein S-acyltransferase) of other plants. Tissue expression profile by Real-time PCR showed that all the three genes were expressed in various tissues, although the expression levels varied significantly in different tissues. Changes in gene expression patterns in the same gene family indicated their functionally non-redundancy. These results above will provide clues for exploring the underlying mechanism of gene regulation on growth, development, morphogenesis and signal transduction of stress response inL.chinense.
Liriodendronchinense, protein S-acylation, protein S-acyltransferase (PAT), DHHC protein family, expression analysis
10.11931/guihaia.gxzw201503026
2015-03-17
2015-04-13
国家自然科学基金(31470660,31170621);江苏省高校优势学科[Supported by the National Natural Science Foundation of China (31470660,31170621); Priority Academic Program Development of Jiangsu High Education Institutions]。
徐嘉娟(1988-),女,云南临沧人,硕士研究生,研究方向为林木分子遗传,(E-mail)rrylxjj@163.com
李火根,教授,研究方向为林木遗传育种,(E-mail)hgli@njfu.edu.cn.
Q943
A
1000-3142(2016)09-1052-09
徐嘉娟, 李火根. 鹅掌楸DHHC型锌指蛋白家族基因的克隆及表达分析 [J]. 广西植物, 2016, 36(9):1052-1060
XU JJ, LI HG. Gene cloning and expression analysis of DHHC protein family genes fromLiriodendronchinense[J]. Guihaia, 2016, 36(9):1052-1060

