基于线粒体DNA cox2基因的甜菜霜霉病的分子检测技术
李 京,张祥林,王 翀,李亚伟,张小菊
(1.新疆农业大学农学院,乌鲁木齐 830052;2.新疆出入境检验检疫局,乌鲁木齐 830063)
基于线粒体DNAcox2基因的甜菜霜霉病的分子检测技术
李 京1,2,张祥林2,王 翀2,李亚伟2,张小菊2
(1.新疆农业大学农学院,乌鲁木齐 830052;2.新疆出入境检验检疫局,乌鲁木齐 830063)
【目的】建立甜菜霜霉病菌检测技术。【方法】利用等位基因特异性PCR(AS-PCR)的原理,依据线粒体DNA(mtDNA)基因cytochrome c oxidase subunit II(cox2),运用卵菌门通用引物 (P-COX2F /P-COX2R) 扩增甜菜霜霉病菌及6种对照霜霉病菌的目的片段,测序,比对,根据等位基因特异PCR(AS-PCR)原理设计测甜菜霜霉病菌的特异性引物,并对该引物的特异性、灵敏度加以验证;分析同源性,构建系统发育树。【结果】通用引物扩增的甜菜霜霉病菌以及其他霜霉病菌片段大小约为630 bp,经比对,该霜霉病菌与Peronosporaschachtii同源性达99%,并设计出特异性引物BSP-F/BSP-R,特异性引物BSP-F/BSP-R可以检测到10-2ng/μL的模板浓度。【结论】建立的甜菜霜霉病菌快速检测技术,为口岸的进出境监测与检测提供了科学依据。
甜菜霜霉病;mtDNA;特异性引物;系统发育关系
0 引 言
【研究意义】霜霉病是较为重要的卵菌门植物病害之一,对许多农作物造成了较为严重的损失。霜霉病是危害甜菜的毁灭性病害之一,2007年被列为我国进境植物检疫性危险性有害生物。甜菜霜霉病19世纪末始于法国,后来在俄罗斯、美国等国家大面积爆发,并造成了较为严重经济损失[1]。20世纪60至90年代在我国贵州、四川以及新疆伊犁州发生此病,造成了一定程度的减产[2]。甜菜在感染霜霉病后,叶片由淡绿色变成淡黄色,且叶片变色部位背面有灰色霉层出现。大多数情况下,霉层出现在叶片背面,但当环境湿度足够的情况下,叶片正面也会有霉层出现[3]。【前人研究进展】尽管ITS区一直被看作是霜霉病最常用的分类方法[4],但由于ITS区高度保守性,且种间差异较小,不能自动鉴定它们的基因同源性[5],同时近年来对于mtDNA 检测霜霉病的报道越来越多,2000年Deborah S. S. Hudspeth等[6]根据扩增cox2 的序列成功的推断出15种霜霉菌与前毛丝壶菌的系统发育关系;2003年Hudspeth等[7]根据cox2 分析了霜霉病与白粉病间的系统发育关系;2007年Markus Göker[8]通过对多个基因(LSU、cox2 、β-Tubulin、NADH)的系统发育进行研究来分析各种霜霉病。2011年Robideau GP[9]提出在同种条件下,线粒体中细胞色素C氧化亚基1(cox1)与内转录间隔区(ITS)相对比,结果表明cox1是一种比较实用的方法。在有些情况下cox1比ITS更能区分种间水平上的差异,然而,大亚基(LSU)却很难区分一些亲缘关系较近的种;2011年Young-Joon Choi等[10]依据两部分线粒体基因cox2 、nad1的系统发育来揭示菠菜霜霉病菌(Peronosporaeffusa)的种内变异情况;2013年Anna L.Testen 等[11]根据扩增藜麦种子的ITS 与mtDNA基因cox2 的基因序列,同时分析不同品种藜麦种子间的遗传变异以及与卵菌门其他病原物的系统发育关系,显示基于cox2 的系统发育关系与ITS的分析结果大致相同,实验证明cox2 也可以作为除ITS以外的另一种分类方法;2015年Young-Joon Choi等[12]经研究提出了相比cox1,cox2对物种的鉴定成功率更高,并且,对于比较珍贵的标本采用cox2存在更高效率的PCR、测序反应[13],同时,对于一些具有历史意义的原始标本有cox2的相关序列,但不存在cox1的序列。等位基因特异性PCR(AS-PCR) 是由Newton等1989提出[14],当引物3’末端碱基与等位基因不互补时,PCR扩增将不能进行,通过单个核苷酸的多态性来达到检测效果,但在有些情况下即便3’碱基不相匹配,扩增也能进行[15-16],因此为了增加引物的特异性,人为的在3’末端第二、三的位置引入错配碱基[17-19]。2014年 Steven J. Klosterman[20]通过AS-PCR的方法成功将菠菜霜霉病菌与甜菜霜霉病菌区分开。【本研究切入点】目前对于甜菜霜霉病菌的分子检测技术相关报道较少,研究基于线粒体DNAcox2基因的甜菜霜霉病菌的分子检测技术及常见霜霉病病原系统发育分析,建立线粒甜菜霜霉病菌检测技术。【拟解决的关键问题】研究以线粒体的cox2基因为靶基因,采用AS-PCR的方法建立甜菜霜霉病菌的分子检测技术。
1 材料与方法
1.1 材 料
供试样品为甜菜霜霉病叶,均采自伊犁州尼勒克县;其他供试样品为杖藜霜霉病叶、杂配藜霜霉病叶、葡萄霜霉病叶(采自于尼勒克县、五家渠市),菠菜霜霉病叶(乌鲁木齐市三坪农场),莴苣霜霉病叶(采自于乌鲁木齐市),将实验样品进行显微镜观察与拍照。表1
表1 .供试霜霉病病原、寄主和来源
Table 1 Downy mildew pathogens,hosts and their sources of this study

序号分类地位寄主来源SequencenoClassificationstatusHostSource1Peronosporaschachtii甜菜Betavulgaris伊犁州尼勒克县2Peffuse菠菜Spinaciaoleracea乌鲁木齐市三坪农场3Pvariabilis杖藜Chenopodiumgiganteum伊犁州尼勒克县4Pfarinosafspchenopodii杂配藜Chenopodiumhybridum伊犁州尼勒克县5Plasmoparaviticola葡萄Vitisvinifera五家渠市、伊犁州尼勒克县6Plangustiterminalis苍耳Xanthiumstrumarium伊犁州尼勒克县7Bremialactucae莴苣Lactucasativa乌鲁木齐市
1.2 方 法
1.2.1 样品DNA提取
在无菌操作条件下,将甜菜霜霉病叶患病部位连带叶片一起切下,液氮充分研磨,再用试剂盒(Plant Genomic DNA Kit)提取甜菜霜霉病菌DNA,-20℃保存备用。
1.2.2 DNA扩增
采用卵菌门通用引物P-cox2F/P-COX2R[21]扩增供试样品线粒体基因cox2的序列如下:
P-COX2F :5'-GGCAAATGGGTTTTCAAGATCC-3'
P-COX2R :5'-CCATGATTAATACCACAAATTTCACTAC-3'
PCR反应体系:2×TaqPCR Green Mix 12.5 μL,10 μM上、下游引物各0.5 μL,ddH2O 9.5 μL,模板2 μL,总体积25 μL。反应程序:96℃, 4 min; 96℃, 30 s; 50℃, 30 s; 72℃,1 min共30个循环,;72℃,4 min。将PCR产物在1.0%琼脂糖凝胶,1×TAE电泳缓冲液,100 V电压条件下电泳35 min,置于凝胶成像系统中紫外照射观察并分析。
1.2.3 测序、同源性
在紫外照射仪下,用刀片切下含目的DNA的胶块,采用琼脂糖凝胶回收试剂盒胶回收目的片段,送至北京鼎国昌盛生物技术有限责任公司测序。将测序结果在NCBI网站上进行BLASTN比对,检测mtDNA的基因鉴定结果。
1.2.4 系统发育
将甜菜霜霉病菌测序得到的基因序列、其他6种对照霜霉病病原物基因序列以及Genbank数据库下载的,与所测甜菜霜霉病菌同源性较高的病原物的mtDNA基因cox2序列进行编辑,运用clustalW 软件[22-23]进行序列的整合,最后用Meg5.05软件构建系统发育树(Neighbor-joining tree)。
1.2.5 设计特异性引物与合成
将甜菜霜霉病菌及其他6种对照霜霉病菌的基因序列上传到Genbank所得到的序列号,并从Genbank数据库下载已发布与甜菜霜霉病菌同源性较高的其他mtDNA基因cox2序列,用DNAMAN8.0及GENEDOC进行比对分析选取基因cox2的保守区,根据AS-PCR的原理,对甜菜霜霉病病原物序列第279位点碱基与其他对照组及同源性较高病原物的该位置的碱基的不同,采用Primer Permier 5.0软件设计了一对引物BSP-F/BSP-R,送至生物技术公司合成。表2
表2 霜霉目种类、寄主、来源、序列号
Table 2 List of organisms,hosts,sources and sequences genbank accession number

病原物寄主来源序列号OrganismHostOrigon GenbankaccessionnoPeronosporaschachtii∗甜菜Betavulgaris本项研究KT944079Pfarinosafspbeta甜菜BetavulgarisGenbankFJ649394Pschachtii甜菜BetavulgarisGenbankKP3306161Pschachtii甜菜BetavulgarisGenbankKP3306061Peffuse∗菠菜Spinaciaoleracea本项研究KT944073Pvariabilis∗杖藜Chenopodiumgiganteum本项研究KT944074Pfarinosafspchenopodii∗杂配藜Chenopodiumhybridum本项研究KT944076Plepigoni牛漆姑草属SpergulariarubraGenbankKP3306611Pobovata牛漆姑草属SpergulaarvensisGenbankKP3306511Ppolycarpi多荚草属PolycarpondiphyllumGenbankKP3306201Prumicis酸模属RumexacetosellaGenbankKP3306341Psp2709酸模属RumexarifoliusGenbankKP3306351PspHV2657酸模属RumexacetosaGenbankKP3306301Psp1164牛漆姑草属SpergulariamarinaGenbankKP3306551Plasmoparaangustiterminalis∗苍耳Xanthiumstrumarium本项研究KT944075Plviticola∗葡萄Vitisvinifera本项研究KT944077Bremialactucae∗莴苣Lactucasativa本项研究KT944078
注:符号(*)的病原物为实验所采集到的病叶提取得到的,其中这些病原物的序列也已经上传到Genbank并获得了序列号,其余未标注的病原物信息为Genbank下载
Note: Signals (*) were used to label the organisms which were obtained from infected leaves in this study,the sequences of these organisms were submitted to genbank and obtained accession numbers,non-signals were downloaded from NCBI
1.2.6 巢式PCR检测
首先用引物P-COX2F/ P-COX2R进行第一轮扩增,然后用特异性引物BSP-F/BSP-R进行第二轮扩增(将第一轮扩增的PCR产物作为第二轮扩增的模板)。第一轮扩增程序同上,第二轮扩增程序:94℃,5 min;94℃,30 s;53℃,30 s;72℃,1 min循环38次;72℃,10 min。PCR反应体系:2×TaqPCR Green Mix 12.5 μL,10 μM上、下游引物各1 μL,ddH2O 8.5 μL,模板2 μL,总体积25 μL。
1.2.7 特异性引物灵敏度检测
将提取的甜菜霜霉病菌的模板DNA进行倍比稀释,共设置7个浓度梯度分别是:10-3ng/μL、10-2ng/ μL、10-1ng/μL、1 ng/μL、10 ng/μL、20 ng/μL、40 ng/μL,然后用特异性引物进行PCR扩增,扩增产物经电泳检测,查看该引物能检测到的最低模板浓度。
2 结果与分析
2.1 形态鉴定结果
经显微镜观察,甜菜霜霉菌(图1:A、B、C)、菠菜霜霉菌(图1D)、藜霜霉菌(图1:E、F)的孢囊梗形态较为接近,但前两者是属于主干单轴分支,后者为明显的二叉状分支,且前两者的孢囊梗比较直或者稍微有点弧度,后者的孢囊梗是整个平滑弯曲。在7种孢囊梗中,甜菜霜霉病菌与菠菜霜霉病菌的形态最为接近,但在孢囊梗分支的末端,甜菜霜霉菌孢囊梗末端锥形,不尖锐;菠菜霜霉病菌孢囊梗末端很尖锐;莴苣霜霉病孢囊梗(图1I)末端为盘状;葡萄霜霉病菌(图1H)与苍耳霜霉病菌(图1G)二者的孢囊梗单轴直角分支。因此,甜菜霜霉病菌孢囊梗为单轴二叉状分支,锐角或直角分支3~8次,末端分支(21~30 μm)× (17.5~22.5 μm)。图1
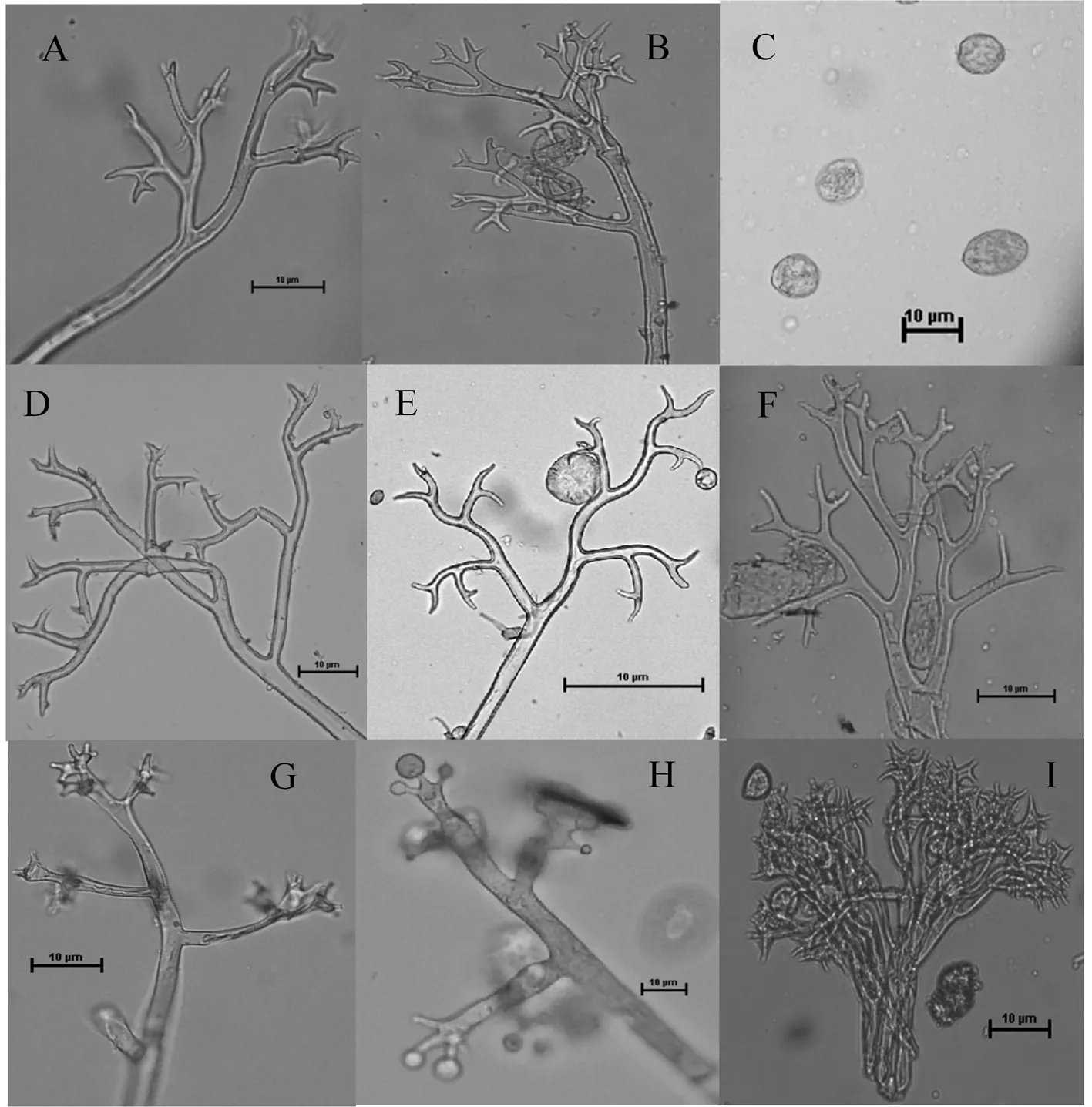
注:A~B:甜菜霜霉病菌的孢囊梗;C:甜菜霜霉病菌的孢子囊;D~I:各霜霉病菌的孢囊梗,它们的寄主分别是:菠菜、杖藜、杂配藜、苍耳、葡萄、莴苣
Note:A-B:Sporangiophores of sugar beet downy mildew pathogen;C:sporangia of sugar beet downy mildew pathogen;D-I:sporangiophores of downy mildew pathogens that the hosts of these areSpinaciaoleracea、Chenopodiumgiganteum、Chenopodiumhybridum、Xanthiumstrumarium.、Vitisvinifera、Lactucasativa
图1 甜菜霜霉病菌与其他对照样品的镜检形态
Fig.1 The morphology of sugar beet downy mildew pathogen and other controls group of oomycota through microscopy(This figure obtained from this study)
2.2 通用引物P-COX2F/ P-COX2R的扩增及测序结果
采用卵菌门通用引物P-COX2F/ P-COX2R扩增甜菜霜霉病菌及其他供试霜霉病菌(菠菜霜霉病菌、杖藜霜霉病菌、杂配藜霜霉病菌、苍耳霜霉病菌、葡萄霜霉病菌、莴苣霜霉病菌)线粒体(mtDNA)基因cox2的基因序列。PCR产物经电泳,结果显示:甜菜霜霉病叶、杖藜霜霉病叶、苍耳霜霉病叶、杂配藜霜霉病叶、葡萄霜霉病叶、菠菜霜霉病叶、莴苣霜霉病叶均能扩增出大小约为630 bp左右的DNA片段。 图2
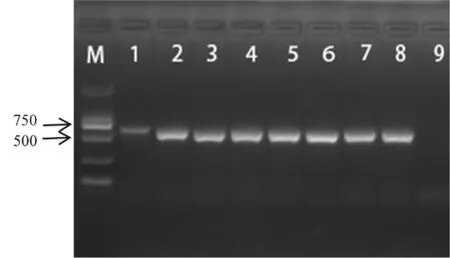
注:M:2 000 bp DNA Ladder;1~7:甜菜霜霉病菌、杖藜霜霉病菌、苍耳霜霉病菌、杂配藜霜霉病菌、葡萄霜霉病菌、菠菜霜霉病菌、莴苣霜霉病菌;8:空白对照
Note:M:2,000 bp DNA Ladder;1-2:downy mildew pathogens ofBetavulgaris;3-7:downy mildew pathogensChenopodiumgiganteum、Xanthiumstrumarium、Chenopodiumhybridum、Vitisvinifera、Spinaciaoleracea、Lactucasativa;9:Blank
图2 引物P-COX2F/ P-COX2R的扩增结果
Fig.2 The PCR amplified result by primer P-COX2F/ P-COX2R
2.3 测序及同源性
测序结果显示,甜菜霜霉病的片段大小为631 bp,将测序结果逐一在NCBI上进行BLAST比对,比对结果显示:甜菜霜霉病菌与Peronosporaschachtii同源性最高达100%,因此可认为该甜菜霜霉病菌属于P.schachtii。其他供试材料比对结果:其片段大小与甜菜霜霉病菌的片段大小相差不大,均为630 bp左右,其中菠菜霜霉病属于P.effuse;杖藜霜霉病菌属于P.variabilis;杂配藜霜霉病菌属于P.farinosaf.sp.chenopodii;苍耳霜霉病菌属于Plasmoparaangustiterminalis;葡萄霜霉病菌属于Pl.viticola;莴苣霜霉病菌属于Bremialactucae。将7种供试材料的cox2的基因序列上传至Genbank,获得7个序列号分别是:KT944079(Peronospora.schachtii);KT944073(Peronospora.Effuse);KT944074(Peronospora.Variabilis);KT944075(Plasmoparaangustiterminalis);KT944076(Peronospora.farinosaf.sp.chenopodii);KT944077(Plasmopara.viticola);KT944078(Bremialactucae)。表2,图3

图 3 引物P-COX2F/ P-COX2R测得的序列
Fig.3 Sequence of polymorphic fragment P.schachtii amplified by primers P-COX2F/ P-COX2R
2.4 系统发育
测序得到甜菜霜霉病菌(P.schachtii)mtDNA基因cox2的基因序列。根据测序得到的P.schachtii和6种对照样品的基因序列以及从Genbank数据库中下载的相关序列,运用分支进化学法构建了系统发育树,经过系统进化分析,其中甜菜霜霉病菌序列:P.schachtii(KT944079)与P.farinosaf.sp.beta(FJ649394)、P.schachtii(KP330616.1)、P.schachtii(KP330606.1)包含在了一个分支单元组内,在Neighbor-joining tree中验证可信度值93%,但这些序列与寄主为杖藜、牛漆姑草属、杂配藜、苍耳、葡萄、莴苣的遗传关系较远,同时,该系统发育树也揭示了P.schachtii与P.effuse、P.rumicis、P.sp. 2709、P.sp. HV2657(后两者的寄主为酸模属)形成了一个较大的分支,可信度值为82%。图4
2.5 常规PCR特异性引物的设计
根据Genbank数据库下载同源性较高的序列以及该试验测序获得的甜菜霜霉病菌及其他6种对照霜霉病的mtDNA基因cox2的序列,按照表2从上到下的顺序进行序列比对,(图5)其中,根据甜菜霜霉病P.schachtii(KT944079)与其他霜霉病菌第279位点(TC)等位基因的不同,但该位点与Genbank数据库下载的其他三个甜菜霜霉病基因序列相同,因此根据等位基因位点设计特异性引物BSP-F/ BSP-R,序列为(加粗碱基为SNP位点)图5:
BSP-F:5'-CAATGGATGAAGTAATTGAC -3'
BSP-R:5'-GGTTCATCTGAAAACTCTAA-3'
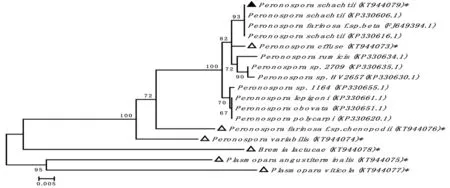
注:分支显示的数字是经过1 000 bootstap replicates得到的,且bootstap值均大于60 % ,符号(*)表示研究获得的样品,实心黑的三角形(▲)表示研究获得的的P.schachtii,对照样品用空心三角形(△)标注
Note:Numbers of the branches represent bootstap values above 60 % obtained from 1,000 bootstap replicates.Signal (*) denote sequences obtained from this study .TheP.schachtiiof this study is shown witha solid black triangle(▲),other control groups of this study is shown with hollow triangle(△)
图4 基于线粒体cox2的甜菜霜霉病菌的系统发育树
Fig.4 Phylogenetic tree of P.schachtii based on the mt DNA gene cox2
2.6 巢式PCR及特异性检测
巢式PCR电泳结果为,第一步卵菌门通用引物P-COX2F/ P-COX2R能将所有的供试材料扩增出来,片段大小均为630 bp左右。第二步用实验设计的特异性引物BSP-F/BSP-R来扩增第一步得到的PCR产物,经电泳检测,仅甜菜霜霉病菌出现条带,片段大小为103 bp。图6
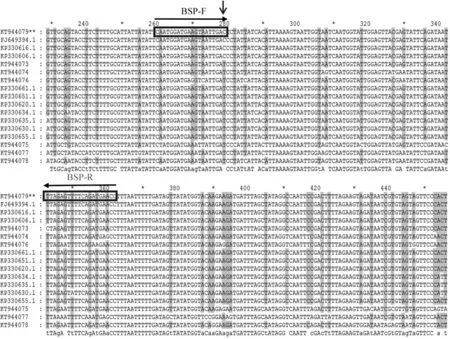
注:方框核酸显示了设计的P.schachtii特异性引物(BSP-F/ BSP-R)的具体位置(垂直向下的箭头指示的是SNP位点的位置,符号(**)标注的序列号为实验获得 )
Note:The nucletides delimited in boxes indicate locations of designed specific primers (BSP-F/ BSP-R) ofP.schachtii(the vertical arrow showed the position of the SNP ),the double asterisks (**) behind the genbank accession number denotes the P.schachtii sequence number of this study
图5 设计的特异性引物(BSP-F/ BSP-R)具体位置
Fig.5 The lacations of designed specific primers (BSP-F/ BSP-R)
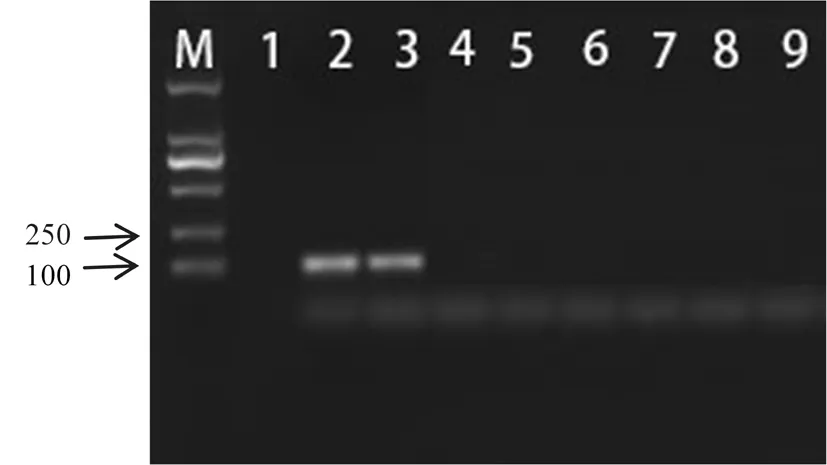
注:M:2 000 bp DNA Ladder;1:空白对照;2~3:甜菜霜霉病菌;4~9:杖藜霜霉病菌、苍耳霜霉病菌、杂配藜霜霉病菌、葡萄霜霉病菌、菠菜霜霉病菌、莴苣霜霉病菌
Note:M:2,000 bp DNA Ladder;1:Blank;2-3:downy mildew pathogens ofBetavulgaris;4-9:downy mildew pathogensChenopodiumgiganteum、Xanthiumstrumarium、Chenopodiumhybridum、Vitisvinifera、Spinaciaoleracea、Lactucasativa
图 6 特异性引物BSP-F/BSP-R扩增结果
Fig.6 The PCR amplified result by primer BSP-F/BSP-R
2.7 特异性引物灵敏度检测
将甜菜霜霉病菌的DNA进行倍比稀释,浓度分别为:10-3、10-2、10-1、1、10、20和40 ng/μL,经PCR扩增,电泳结果显示:当随着甜菜霜霉病菌模板浓度的较低,电泳条带越来越暗,当模板浓度为10-3ng/μL时电泳图上无条带出现,实验结果显示该特异性引物能检测到10-2ng/μL的模板浓度。图7

注:M:2 000 bp DNA Ladder;1-8:10-3、10-2、10-1、1 、10 、20 、40 和100 ng/μL;9:空白对照
Note:M:2,000 bp DNA Ladder;1-8:10-3ng/μL、10-2ng/μL、10-1ng/μL、1 ng/μL、10 ng/μL、20 ng/μL、40 ng/μL、100 ng/μL;9:blank
图7 特异性引物BSP-F/BSP-R灵敏度检测结果
Fig.7 The result of sensitivity detection amplified by specific primer BSP-F/BSP-R
3 讨 论
研究供试样品DNA提取方法采用了霜霉病叶直接提取法,虽然此方法在提取霜霉病菌的同时也提取了感染霜霉病叶片的DNA,但采用了常规PCR采用了巢式PCR法,该方法第一轮扩增采用了卵菌纲线粒体cox2基因通用引物,该引物只能扩增出霜霉病菌,第二轮采用甜菜霜霉病菌特异性引物,进一步将甜菜霜霉病菌扩增出来,因此经巢式PCR扩增后,产物内只能将霜霉病菌扩增出来,不用担心寄主DNA对实验结果的影响。
在实验过程中,开始仅仅根据等位基因第279位点的不同设计了一对引物,但经实验验证,该引物不仅仅扩增出了甜菜霜霉病菌,也将菠菜霜霉病菌扩增出来,因此为了设计出一对只针对甜菜霜霉病菌特异性引物根据在甜菜霜霉病菌在第279位点(TC)的不同设计了上游引物,然后根据菠菜霜霉病菌与甜菜及其他霜霉病菌第343位点(TC)的不同设计了下游引物。经实验鉴定该引物(BSP-F/ BSP-R)特异性较好,仅能将甜菜霜霉病菌扩增出来。
尽管实验设计出甜菜特异性引物BSP-F/ BSP-R,但不能证明该引物不能扩增出其他霜霉病菌。因此,实验过程中针对该引物特异性进行了进一步的验证分析,以几种常见霜霉病为例:黄瓜霜霉病(Pseudoperonosporacubensis)、向日葵霜霉病(Plasmoparahalstedii)、大豆霜霉病(Peronosporamanshurica)为例。甜菜霜霉病菌第279位点为(TC),黄瓜霜霉病菌为(TT)、大豆霜霉病菌(TT)、向日葵霜霉病菌(TC),初步可以认为该引物(BSP-F/ BSP-R)检测不到黄瓜霜霉病菌以及大豆霜霉病菌,但该引物能检测到向日葵霜霉病菌。因此,相对而言,不排除甜菜特异性引物BSP-F/ BSP-R存在特异性不高的可能性。
4 结 论
采用等位基因特异性PCR的原理,建立了快速检测甜菜霜霉病菌的常规PCR方法,并且对常见霜霉菌系统发育关系进行了分析,其中甜菜霜霉病快速分子检测技术,为快速监测甜菜上霜霉病菌的发生与预防提供了指导方法并为口岸快速检测甜菜霜霉病菌提供了技术支持。
References)
[1] 严进.甜菜霜霉病[J].植物检疫,1999,13(2):94-95.
YAN Jin. (1999). Beet downy mildew [J].JournalofPlantQuarantine, 13(2): 94-95.(in Chinese)
[2] 郭文超,杨秀荣,谢浩.新疆甜菜霜霉病的初步研究[J].新疆农业科学,1996,(3): 127-129.
GUO Wen-chao, YANG Xiu-rong, XIE Hao. (1996). The preliminary study of sugar beet downy mildew in Xinjiang [J].XinjiangAgriculturalSciences, (3):127-129. (in Chinese)
[3] Kim, J. Y., Choi, Y. J., & Shin, H. D. (2010). Downy mildew caused byPeronosporafarinosaf. sp. betae newly reported on Swiss chard in Korea.PlantPathology, 59(2):405-405.
[4] Voglmayr, H. (2008). Progress and challenges in systematics of downy mildews and white blister rusts: new insights from genes and morphology.EuropeanJournalofPlantPathology, 122(1):3-18.
[5] Choi, Y. J., Thines, M., Han, J. G., & Shin, H. D. (2011). Mitochondrial phylogeny reveals intraspecific variation in peronospora effusa, the spinach downy mildew pathogen.JournalofMicrobiology, 49(6):1,039-1,043.
[6] Hudspeth, D. S. S., Nadler, S. A., & Hudspeth, M. E. S. (2000). A COX2 molecular phylogeny of the peronosporomycetes.Mycologia, 92(4):674-684.
[7] Hudspeth, D. S. S., Stenger, D., Hudspeth, M. E. S., Hudspeth, D. S. S., Stenger, D., & Hudspeth, M. E. S. (2003). A COX2 phylogenetic hypothesis for the downy mildews and white rusts.FungalDiversity,13(4):47-57.
[8] Göker, M., Voglmayr, H., Riethmüller, A., & Oberwinkler, F. (2007). How do obligate parasites evolve A multi-gene phylogenetic analysis of downy mildews.FungalGenetics&Biology, 44(2):105-122.
[9] Robideau, G. P., De, C. O. C. K., Arthur, W., Coffey, M. D., Voglmayr, H., Brouwer, H., & Gachon, C. M. (2011). DNA barcoding of oomycetes with cytochrome c oxidase subunit I and internal transcribed spacer.Molecularecologyresources, 11(6):1,002-1,011.
[10] Choi, Y. J., Thines, M., Han, J. G., & Shin, H. D. (2011). Mitochondrial phylogeny reveals intraspecific variation in Peronospora effusa, the spinach downy mildew pathogen.JournalofMicrobiology, 49(6):1,039-1,043.
[11] Testen, A. L., Del, M. J. M., Ochoa, J. B., & Backman, P. A. (2013). Molecular detection of peronospora variabilis in quinoa seed and phylogeny of the quinoa downy mildew pathogen in South America and the United States.Phytopathology, 104(4):379-386.
[12] Choi, Y. J., Beakes, G., Glockling, S., Kruse, J., Nam, B., Nigrelli, L., ... & Voglmayr, H. (2015). Towards a universal barcode of oomycetes-a comparison of the cox1 and cox2 loci.Molecularecologyresources, 15(6):1,275-1,288.
[13] Sabine, T., & Marco, T. (2008). Amplification of cox2 (approximately 620 bp) from 2 mg of up to 129 years old herbarium specimens, comparing 19 extraction methods and 15 polymerases..PlosOne, 3(10):e3,584.
[14] Newton, C. R., Graham, A., Heptinstall, L. E., Powell, S. J., Summers, C., Kalsheker, N., ... & Markham, A. F. (1989). Analysis of any point mutation in DNA. The amplification refractory mutation system (ARMS).Nucleicacidsresearch, 17(7):2,503-2,516.
[15] Luis, U., & Wallace, R. B. (1991).Allele-specific polymerase chain reaction.Methods, 2(1):42-48.
[16] Suzuki, Y., Sekiya, T., & Hayashi, K. (1991). Allele-specific polymerase chain reaction: a method for amplification and sequence determination of a single component among a mixture of sequence variants.Analyticalbiochemistry, 192(1):82-84.
[17] 唐一通, 肖娜, 李智山, 等.单核苷酸多态性检测方法究进展[J].现代生物医学进展,2013,13(27):5 393-5 397.
TANG Yi-tong, XIAO Na, LI Zhi-shan, et al.(2013).The research progress single nucleotide polymorphism detection method [J].ProgressinModernBiomedicine,13 (27): 5,393-5,397. (in Chinese)
[18] 许家磊,王宇,后猛,等.SNP检测方法的研究进展[J].分子植物育种,2015,(2): 475-482.
XU Jia-lei, WANG Yu, HOU Meng, et al. (2015). The research progress SNP detection method [J].MolecularPlantBreeding, (2): 475-482. (in Chinese)
[19] 唐立群,肖层林,王伟平.SNP 分子标记的研究及其应用进展[J].中国农学通报 ,2012,28(12):154-158.
TANG Li-qun, XIAO Ceng-lin, WANG Wei-ping. (2012). Research and application progress of SNP Marker [J].ChineseAgriculturalScienceBulletin, 28(12): 154-158. (in Chinese)
[20] Klosterman, S. J., Amy, A., Neil, M. R., Koike, S. T., Subbarao, K. V., & Hermann, V., et al. (2014). Coupling spore traps and quantitative pcr assays for detection of the downy mildew pathogens of spinach (peronospora effusa) and beet (p. schachtii).Phytopathology, 104(12):1,349-1,359..
[21] Hudspeth, D. S. S., Nadler, S. A., & Hudspeth, M. E. S. (2000). A COX2 molecular phylogeny of the peronosporomycetes.Mycologia, 92(4):674-684.
[22] Thompson, J. D., Higgins, D. G., & Gibson, T. J. (1994). CLUSTAL W: improving the sensitiv ity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice.NucleicAcidsResVol.22:4,673-4,680.
[23] Choi, Y. J., Hong, S. B., & Shin, H. D. (2007). Re-consideration of peronospora farinosa infecting spinacia oleracea as distinct species, peronospora effusa.MycologicalResearch, 111(Pt 4):381-391.
[24] Lindqvist, H., Koponen, H., & Valkonen, J. P. (1998). Peronospora sparsa on cultivated rubus arcticus and its detection by PCR based on its sequences.PlantDisease, 82(12):1,304-1,311.
[25] Gilles, T., Phelps, K., Clarkson, J. P., & Kennedy, R. (2004). Development of milioncast, an improved model for predicting downy mildew sporulation on onions.PlantDisease, 88(7):695-702.
[26] Blanco-Meneses, M., & Ristaino, J. B. (2011). Detection and quantification of peronospora tabacina using a real-time polymerase chain reaction assay.PlantDisease, 95(6):673-682.
Fund project:Supported by The scientific research project of General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China (201310091) and Xinjiang Agriculture University research joint graduate training program ( xjaucxy-yjs-20131023)
Molecular Detecting Technology of Sugar Beet Downy Mildew Baswd on Mitochondrial DNA Genecox2
LI Jing1,2,ZHANG Xiang-lin2,WANG Chong2,LI Ya-wei2,ZHANG Xiao-ju2
(1. College of Agronomy, Xinjiang Agricultural University, Urumqi 830052, China;2.Xinjiang Entry-Exit Inspection and Quarantine Bureau, Urumqi 830063, China )
【Objective】 The purpose of this research is to establish detection technology of sugar beet downy mildew pathogen.【Method】According to cytochrome oxidase subunit II(cox2)of mitochondrial DNA (mtDNA) gene, this study used universal primers (P-COX2F/P-COX2R) of oomycota to amplify the target fragments of sugar beet downy mildew pathogen and other six control groups, sequencing, blastn, then designed specific primers to detect beet downy mildew pathogen based on the principle of allele specific PCR (AS-PCR), verify its specificity and sensitivity, then analyzed their homology and constructed phylogenetic tree, and finally infected healthy sugar beets using inoculum ofP.Schachtiiwhich was to verify whether theP.Schachtiiwas pathogenic.【Result】The experimental results showed that the fragments of sugar beet downy mildew pathogen and others downy mildew pathogens were about 630 bp using oomycota universal primers, the homology of sugar beet downy mildew pathogen of this study were 99% with those downloaded from Genbank and designed a specific primer BSP-F/BSP-R which just amplified beet downy mildew. This primer could detect 10-2ng/μL of template DNA.【Conclusion】A rapid detection ofPeronosporaschachtiihas been extablished, which provided an important scientific basis for monitoring and detecting for the port entry and exit.
sugar beet downy mildew; mtDNA; specific primer; phylogenetic
10.6048/j.issn.1001-4330.2016.05.011
2016-01-08
国家质检总局科研项目(201310091);新疆农业大学产学研联合培养研究生项目(xjaucxy-yjs-20131023)
李京(1990-),女,山东人,硕士研究生,研究方向为植物检疫病害,(E-mail)13579981032@163.com
张祥林(1964-),男,新疆人,研究员,研究方向为分子植物病理学,(E-mail)XL6479@163.com
S435.121.42
A
1001-4330(2016)05-0857-09

