花生Clp家族成员的筛选、聚类和盐胁迫响应分析
郑春花+孔祥远+陈冠旭+隋炯明+乔利仙+王晶珊+赵春梅


摘 要:Clp蛋白酶是一种ATP依赖的丝氨酸型蛋白酶,广泛存在于原核和真核生物中,在生物抗逆胁迫中发挥重要作用。为分析花生中Clp基因的情况,我们构建了花生叶片转录组数据库,通过生物信息学手段鉴定出28个Clp基因,分别位于花生A组野生种的9条染色体上;聚类分析表明,花生的28个Clp基因分别聚到已报道的ClpB、C、D、X、P、R和S 7个亚类中。利用花生耐盐突变体(S2)和对照(S4)构建了盐胁迫处理前后各时间段的表达谱数据,进行Clp基因盐胁迫表达分析,结果表明16个Clp基因在S2和(或)S4中受盐胁迫诱导表达。该研究为花生Clp基因的功能研究与利用奠定基础。
关键词:花生;Clp基因;RNA测序;盐胁迫
中图分类号:S565.203.53文献标识号:A文章编号:1001-4942(2016)12-0001-05
Abstract Clp is a kind of ATP-dependent protease widespreading in prokaryote and eukaryote, which plays an important role in resisting biotic and abiotic stresses in plants. To improve the understanding of Clp genes in peanut,28 Clp genes were screened by one transcriptome library constructed with peanut leaves. These 28 Clp genes distributed on 9 chromosomes respectively of A genome of wild peanut. Clustering analysis showed that these 28 Clp genes were assorted to ClpB, C, D, X, P,R, S groups reported previously. To analyze the response to salinity stress of 28 Clp genes,digital gene expression profiles were constructed using a mutant with higher salinity resistance (S2) and its control (S4) before and after treated with 250 mmol/L NaCl. The results showed that 16 Clp genes were responsive to salinity stress in S2 and/or S4. This study provided bases for functional researches and application of Clp genes in peanut.
Keywords Peanut (Arachis hypogaea L.);Clp gene;RNA sequencing;Salinity stress
Clp蛋白酶是一种ATP依赖的丝氨酸型蛋白酶,属于AAA+类ATP酶超级家族。Clp家族基因在植物中广泛存在,有些成员在受到非生物胁迫时可被诱导表达。蓝藻在强光、低温、UV-B等外界胁迫下的生长状况与Clp蛋白酶密切相关[1]。拟南芥和玉米胞质ClpB基因能够恢复其突变体的耐热性[2-4]。超表达拟南芥胞质ClpB基因后,拟南芥生长速度加快,而且能够耐45℃的高温胁迫[5]。反义抑制番茄的叶绿体类ClpB基因后,转基因植株对高温更为敏感,表明叶绿体类ClpB基因可能是植物耐热过程中的重要蛋白[6]。拟南芥ClpA类似物在水分胁迫下被诱导表达,在植株衰老时表达量有所增加[7]。但Clp基因与植物耐盐性关系的报道非常少。
花生是重要的油料和经济作物,培育高产耐盐花生品种不仅可以丰富育种资源,同时对花生耐盐的分子机理研究也具有重要作用。然而花生缺乏耐盐种质资源[8],利用常规杂交方法难以选育出耐盐品种。因此挖掘花生的耐盐基因并进行功能研究是花生抗逆分子育种的重要任务之一。
目前,关于花生中Clp基因的研究相对较少。纪鸿飞等[9]利用花生品种鲁花14号种子不同发育时期混合cDNA文库,筛选到1个AhClpP蛋白酶基因,但未报道该Clp基因是否受胁迫诱导。为了探究Clp基因在花生中是否受盐胁迫诱导表达,本研究从转录组整体水平上分析花生Clp基因的染色体定位和分类,并利用构建好的表达谱寻找盐胁迫响应的Clp基因,为进一步研究它们的功能以及利用它们改良花生抗逆性提供参考。
1 材料与方法
1.1 试验材料
前期我们以平阳霉素为诱变剂进行了花生离体诱变,经筛选获得一批稳定遗传的突变体[10],其中1个突变体(S2)在0.7% NaCl溶液中的发芽率可达到50%以上,而对照花育20(S4)在0.7% NaCl溶液中的发芽率只有6.7%,且S2比S4具有更高的SOD和POD值。本研究即以花生耐盐突变体(S2)和花育20(S4)为材料。
1.2 花生叶片转录组数据库构建
用250 mmol/L NaCl处理耐盐突变体S2和对照S4,在0、6、12、24、48 h取叶片分别提取总RNA,S2的5个样品分别命名为S2_0、S2_6、S2_12、S2_24和S2_48;S4的5个样品分别命名为S4_0、S4_6、S4_12、S4_24和S4_48,每个样品包含2个生物学重复。将所有样品RNA混合后构建转录组文库,送交北京诺和致源公司进行双向测序,组装处理后获得非冗余Unigene序列,在NCBI数据库注册(注册号:SRR311451)。用NR、NT、SwissProt、PFAM、KOG、GO、KEGG数据库进行基因功能注释。
1.3 盐胁迫处理前后花生叶片表达谱数据库构建
对上述样品采用Solexa技术分别进行数字化表达谱测序,构建花生叶片盐胁迫处理前后的表达谱数据库(NCBI注册号:SRR3210665和SRR3210666),并以上述转录组数据库为参考,对整理去杂后的表达谱数据库Unigene序列进行分析。
1.4 Clp家族基因的筛选、结构分析和染色体定位
根据上述7大数据库的基因功能预测搜索Clp基因。利用SMART在线工具进一步分析结构域,去除无典型结构域的基因。蛋白的分子量和理论等电点利用http://www.expasy.org进行分析。从花生全基因组数据(http://www.peanutbase.org)下载A组野生种基因组序列,进行基因序列比对,取相似度最高的序列作为目标基因,并根据数据库的预测和比对结果进行外显子-内含子结构分析。根据与基因组比对后基因位置的显示结果,标示每个基因在染色体上的位置,得到各个基因在染色体上的分布状况。
1.5 系统发育树的构建
通过Clustal X程序对花生和拟南芥Clp蛋白进行多序列联配分析,序列联配结果使用MEGA4.0程序构树。采用邻接法生成Clp基因的系统进化树,bootstrap值设置为1 000。
1.6 Clp基因的盐胁迫响应分析
应用DESeq R package (1.10.1)对S2或S4样品中某一基因在胁迫处理0、6、12、24、48 h的任意2个时间段间的表达量进行比较,如果达到显著上调或下调(应用Benjamini和Hochberg的方法调整P值,调整后的P<0.05)水平,则认为该基因对盐胁迫有响应。
2 结果与分析
2.1 花生Clp基因家族的鉴定
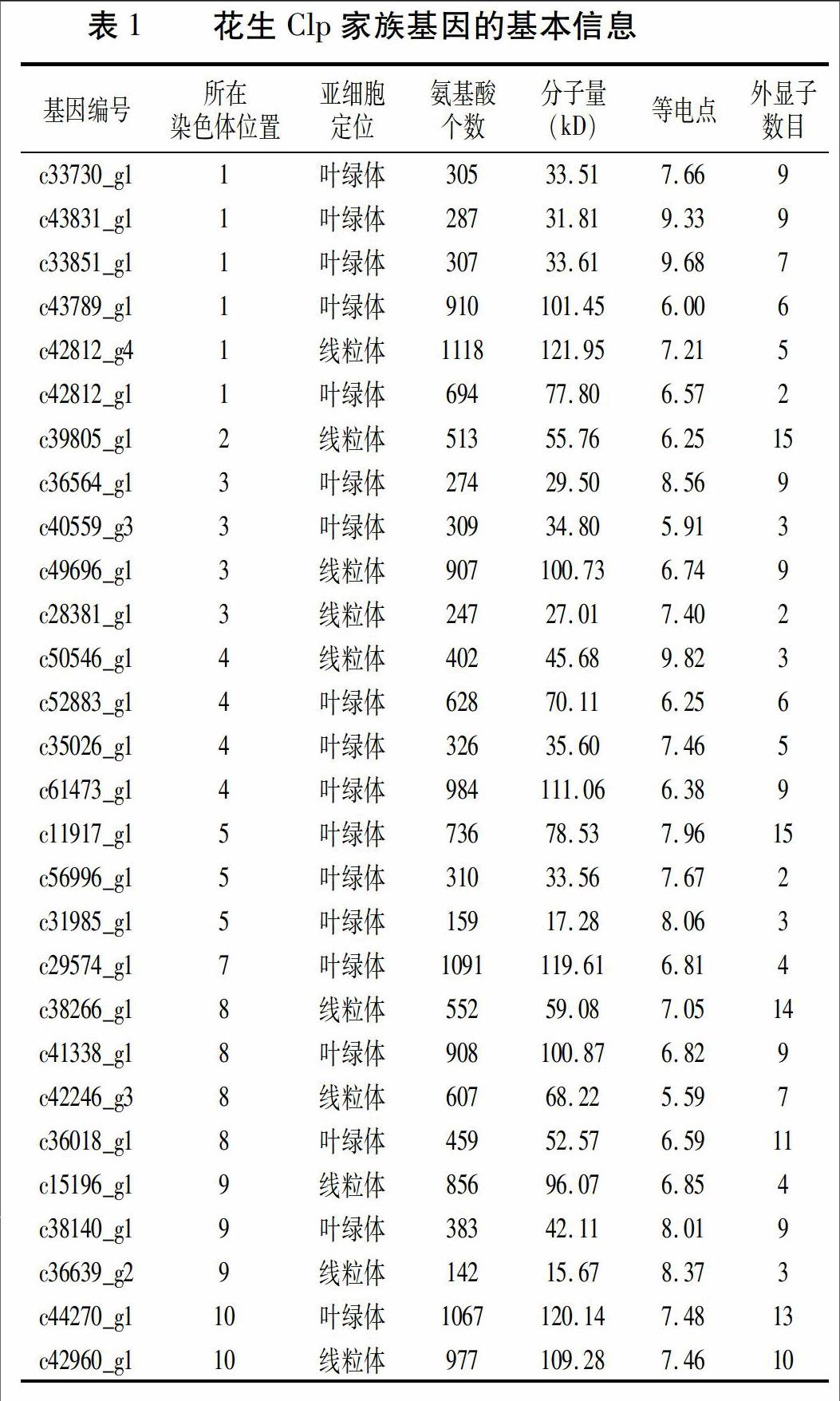
根据7大数据库的基因功能预测结果,我们从花生叶片转录组数据库中筛选出多个候选Clp基因,将其与公布的花生A组野生种基因组序列进行比对,去除比对到同一位置的Clp基因,最终得到28个花生Clp基因。根据比对的基因位置显示结果得到28个Clp基因在花生A组野生种染色体上的位置信息,这些基因分布在9条染色体上,第1号染色体分布的成员数量最多,有6个;其次为第3号、4号和8号染色体,各有4个Clp基因;而第6号染色体上不含有Clp基因;第4号染色体在63 kb范围内有3个紧密连锁的Clp基因(c52883_g1、c50546_g1和c61473_g1)。花生Clp氨基酸残基个数在不同成员间差别很大,长度范围为142~1 118个残基,其等电点范围从5.59到9.82,基因编码产物全部定位在叶绿体或线粒体上。基因结构分析显示,外显子个数从2个到15个不等(表1)。
2.2 花生Clp家族基因的聚类分析
拟南芥至少有26个Clp基因,可分为ClpB、C、D、X、P、R和S 7个亚类[11]。根据Clp蛋白家族成员长度差异、含不同特征的结构域等特点,对花生28个Clp蛋白与拟南芥的Clp蛋白进行多重序列比对和系统进化分析。结果表明,花生的28个Clp蛋白都能聚到ClpB、C、D、X、P、R和S 7个亚类中,这7个亚类分别包含6、2、1、4、5、3个和7个花生Clp蛋白。在拟南芥中ClpP亚类有6个成员,其中ClpP1由质体编码,其它由核基因编码,我们在花生中筛选到5个ClpP蛋白,分别与拟南芥的ClpP2、3、4、5、6聚到一类。
2.3 花生Clp家族基因的盐胁迫响应分析
我们利用盐胁迫处理前后S2和S4的数字表达谱,对28个Clp基因在胁迫处理前后任何2个时间段间的表达量进行比较,根据调整后的P值,确定其是否受盐胁迫诱导表达,结果列于表2和图2。可以看出,在S2中,与胁迫处理前相比,胁迫处理6 h后,差异表达的基因最多,有2个上调,2个下调;而在S4中,与胁迫处理前相比,胁迫处理12 h后,差异表达的基因最多,有2个上调,4个下调。
进一步分析发现,6个基因(c43789_g1、c36018_g1、c31985_g1、c29574_g1、c42812_g4、c38266_g1)在S2和S4中均受盐胁迫诱导表达;2个基因(c35026_g1、c56996_g1)只在S2中受盐胁迫诱导表达;8个基因(c42246_g3、c42960_g1、c61473_g1、c41338_g1、c44270_g1、c49696_g1、c42812_g1、c40559_g3)只在S4中受盐胁迫诱导表达。其中,c42246_g3、c61473_g1、c36018_g1、c42960_g3、c43789_g1这5个基因在耐盐突变体S2和对照S4中表达模式完全一致,都为上调-下调-上调-上调;而c49696_g1、c44270_g1、c42812_g1、c31985_g1、c40559_g2、c56996_g1这6个基因在S2和S4中表现出截然不同的表达模式。
各基因FPKM值差异很大, S2中FPKM值的变化范围为0.33~267.41, 而S4中FPKM值的变化范围为0.43~285.92(图2)。与胁迫处理前相比,盐胁迫处理48 h后,c43789_g1在S2和S4中的表达量均显著上调,log2(fold change)分别达到3.84和5.46;盐胁迫处理12 h时与6 h相比,c36018_g1在S2和S4中的表达量均显著下调,log2(fold change)分别达到-2.71和-3.44,而随着盐胁迫处理时间的增加,该基因的表达量迅速增加,48 h与12 h相比,该基因的log2(fold change)分别达到2.58和3.85。c31985_g1 和c38266_g1这2个基因的表达量在多个时间段间的表达量差异都达到显著水平。盐胁迫处理6 h时,c42246_g3在S2和S4间表达量差异达到显著水平,推测该基因可能与这两个材料的耐盐性差异有一定联系(结果另文发表)。
结合聚类分析结果,上述16个受盐胁迫诱导的Clp基因涉及到除ClpR亚类外的其余6个亚类,其中ClpC(2/2)和ClpD(1/1)2个亚类中的基因均受盐胁迫诱导表达;其次为ClpS(5/7)和ClpB(4/6)2个亚类,绝大多数成员受盐胁迫诱导表达;而ClpP(2/5)和ClpX(2/4)只有个别成员受盐胁迫诱导表达。
3 讨论与结论
植物在生长过程中,不可避免地会受到各种逆境胁迫条件的影响。在长期的进化过程中, 植物自身已经形成了多种抵抗胁迫的机制,例如:当植物细胞经历生化代谢或受到胁迫后, 会产生一些不可逆损伤的蛋白或多肽,这些蛋白丧失了其天然构象或活性,而生物体内的蛋白酶可以维持它们的稳定、促进其再折叠或者降解。因此,蛋白酶在调节蛋白代谢过程及清除损伤蛋白过程中起关键作用[12]。多数细胞内的蛋白水解作用通过依赖于能量的蛋白酶来完成,Clp蛋白酶就是其中之一。
为了分析花生中Clp家族成员的情况,我们利用构建的花生叶片转录组数据库筛出28个Clp蛋白酶基因,它们位于花生A组野生种的9条染色体上,不同染色体上的Clp基因数目差异很大。根据已有报道,Clp蛋白可分为两大类:ClpP蛋白家族和Hsp100/Clp分子伴侣家族。前者组成Clp蛋白酶的催化亚基,后者组成Clp蛋白酶的调节亚基(成员包括 ClpA、B、C、D、M、N、X 及Y等)[13],ClpP通常与分子伴侣组合成为Clp复合物,共同行使蛋白酶的功能。在拟南芥中已经发现与ClpP 具有相似蛋白序列的基因ClpR,但它们缺乏ClpP所具有的蛋白酶活性中心。此外在拟南芥中还发现了不属于任何已知Clp基因家族的ClpS蛋白[14]。我们对花生的28个Clp基因进行了聚类分析,结果表明这28个Clp基因分别聚到已报道的ClpB、C、D、X、P、R和S 7个亚类中,与拟南芥的Clp基因分类结果完全一样。
我们还利用花生耐盐突变体(S2)和对照(S4)构建了盐胁迫处理前后各时间段的表达谱数据,进行基因盐胁迫表达分析,结果表明16个Clp基因受盐胁迫诱导表达,这16个基因涉及除R亚类外的其它6个亚类。ClpS和ClpB亚类中受盐胁迫诱导表达的成员最多;同一亚家族内的基因表达模式并不完全相同,例如ClpP和ClpX只有个别成员受盐胁迫诱导表达,暗示它们可能在进化过程中发生了功能分化。该研究可为花生Clp蛋白酶基因的耐盐功能研究与利用提供重要参考。
参 考 文 献:
[1] 周婷,刁红丽,陈晓国. 蓝藻Clp、Deg、FtsH蛋白酶的研究进展[J]. 湖北农业科学,2010,49(3):710-714.
[2] Hong S W,Vierling E. Mutants of Arabidopsis thaliana defective in the acquisition of tolerence to high temperature stress [J]. Proc. Natl. Acad. Sci. USA,2000,97(8):4392-4397.
[3] Hong S W,Vierling E. Hsp101 is necessary for heat tolerance but dispensable for development and germination in the absence of stress [J]. The Plant Journal,2001,27(1):25-35.
[4] Nieto-Sotelo J,Martínez L M,Ponce G,et al. Maize HSP101 plays important roles in both induced and basal thermotolerance and primary root growth [J]. The Plant Cell,2002,14(7):1621-1633.
[5] Queitsch C,Hong S W,Vierling E,et al. Heat shock protein 101 plays a crucial role in thermotolerance in Arabidopsis[J]. The Plant Cell,2000,12(4):479-492.
[6] Yang J Y,Sun Y,Sun A Q,et al. The involvement of chloroplast HSP100/ClpB in the acquired thermotolerance in tomato [J]. Plant Molecular Biology,2006,62(3):385-395.
[7] Nakashima K, Kiyosue T, Yamaguchi-Shinozaki K,et al. A nuclear gene,erd1, encoding a chloroplast-targeted Clp protease regulatory subunit homolog is not only induced by water stress but also developmentally up-regulated during senescence in Arabidopsis thaliana[J]. The Plant Journal,1997,12:851-861.
[8] 禹山林. 中国花生品种及其系谱[M]. 上海:上海科学技术出版社,2008.
[9] 纪鸿飞, 彭振英, 马敬, 等. 花生Clp蛋白酶基因(AhClpP)的克隆与序列分析 [J]. 华北农学报,2010,25(S2):5-8.
[10]Sui J,Wang Y,Wang P,et al. Generation of peanut drought tolerant plants by pingyangmycin-mediated in vitro mutagenesis and hydroxyproline- resistance screening [J]. PLoS ONE,2015,10 (3):e0119240.
[11]Peltier J B,Ytterberg J,Liberles D A,et al. Identification of a 350-kDa ClpP protease complex with 10 different Clp isoforms in chloroplasts of Arabidopsis thaliana[J]. The Journal of Biological Chemistry,2001,276(19):16318-16327.
[12]Beers E P, Woffenden B J, Zhao C S. Plant proteolytic enzymes: possible roles during programmed cell death[J]. Plant Molecular Biology,2000,44 (3):399-415.
[13]Schirmer E C, Glover J R, Singer M A,et al. HSP100/Clp proteins: a common mechanism explains diverse functions[J]. Trends. Biochem. Sci.,1996,21 (8):289-296.
[14]Peltier J B,Ripoll D R,Friso G,et al. Clp protease complexes from photosynthetic and non-photosynthetic plastids and mitochondria of plants,their predicted three- dimensional structures,and functional implications [J]. The Journal of Biological Chemistry,2004,279(6):4768-4778.

