番红花种球中虎眼万年青花叶病毒的分子鉴定与序列分析
廖富荣,林武镇, 2,陈细红,,陈青,陈红运,黄蓬英,方志鹏,吴媛,沈建国,林石明
番红花种球中虎眼万年青花叶病毒的分子鉴定与序列分析
廖富荣1,林武镇1, 2,陈细红1,3,陈青1,陈红运1,黄蓬英1,方志鹏1,吴媛1,沈建国3,林石明1
(1厦门出入境检验检疫局检验检疫技术中心,福建厦门361026;2生物农药与化学生物学教育部重点实验室,福州350002;3福建出入境检验检疫局检验检疫技术中心,福州350003)
对一批荷兰进境番红花种球()进行病毒鉴定,以防止危险性病毒传入危害。根据已报道的马铃薯Y病毒科()通用引物(Sprimer和M4T),采用RT-PCR方法对一批番红花种球样品进行检测,取其中一个RT-PCR产物进行克隆测序,利用GenBank上的基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)和MEGA 6中的基于对数期望的多重序列比较(Multiple Sequence Comparison by Log-Expectation,MUSCLE)进行序列比对分析,并根据外壳蛋白基因(coat protein,)序列利用最大似然法(maximum likelihood method)进行系统发育分析。RT-PCR检测结果显示该批样品扩增到一条约1.7 kb的预期大小片段,说明该批番红花种球样品中存在科病毒。测序获得长度为1 666 bp的序列(命名为2677-NL),含有部分核内含体b基因(nuclear inclusion b,)、完整的和3′端非编码区(3′-UTR),其核苷酸序列与虎眼万年青花叶病毒(,OrMV)的SW3.3分离物具有最高序列一致性(99.6%),与OrMV参考基因组序列(Reference genomic sequences,RefSeq;NC_019409)的序列一致性为99.1%。该分离物的与6个OrMV印度分离物(JQ686722、JQ686720、JN692498、JN692497、JN692496、JF682235)的核苷酸和氨基酸序列一致性分别为71.5%—77.3%和65.7%—79.7%,与其他32个OrMV分离物的核苷酸和氨基酸序列一致性分别为77.9%—99.5%和82.9%—99.2%。基于进行的系统发育分析表明,OrMV可分为两个类群,而2677-NL与5个澳大利亚分离物(JQ807995、JQ807996、NC_019409、AF185964、AF185965)相聚成簇,表明其在系统发育关系上与OrMV澳大利亚分离物的亲缘关系最近。根据国际病毒分类委员会(ICTV)关于科病毒种类的分类标准,2677-NL为OrMV的一个分离物,证实该批番红花种球受到OrMV的侵染。这是世界上首次在番红花中发现OrMV的报道。
虎眼万年青花叶病毒;番红花;RT-PCR;新寄主
0 引言
【研究意义】番红花()又名藏红花、西红花,为鸢尾科(Iridaceae)番红花属()多年生球茎草本植物,是一种名贵药材和香料[1]。在番红花的种植过程中,易受到各种病虫的危害,包括各种植物病毒[2]。对于番红花这种以无性繁殖为主的植物来说,一方面,植物病毒可以通过繁殖体从上一生长季传播到下一生长季,成为重要的初侵染源;另一方面,通过农事操作、介体传播等方式,植物病毒可以从发病植株传播给健康植株。因此,一旦用于繁殖的番红花携带植物病毒,即使带毒率非常小,经过多年的繁殖栽培,也能够造成植物病毒在番红花植株中普遍存在,且容易造成多种病毒种类复合侵染现象,导致种球变小、品种退化,严重影响番红花的产量和质量。近年来,随着中国从国外引进优良的番红花品种越来越频繁,各种有害生物传入境内的风险也越来越高。因此,加强口岸检疫,可以有效防止各种有害生物的入侵危害。【前人研究进展】目前,已报道的侵染番红花的病毒至少有10种,分属于7个病毒属,包括马铃薯Y病毒属()的菜豆黄花叶病毒(,BYMV)、鸢尾重型花叶病毒(,ISMV)、鸢尾轻型花叶病毒(,IMMV)和芜菁花叶病毒(,TuMV);马铃薯X病毒属()的水仙花叶病毒(,NMV);线虫传多面体病毒属()的南芥菜花叶病毒(,ArMV);烟草脆裂病毒属()的烟草脆裂病毒(,TRV);黄瓜花叶病毒属()的黄瓜花叶病毒(,CMV);坏死病毒属()的烟草坏死病毒(,TNV);番茄斑萎病毒属()的番茄斑萎病毒(,TSWV)[3-8]。其中,ArMV和TSWV是中国严格禁止进境的检疫性有害生物。在中国番红花中已报道的病毒种类有3种,即BYMV[9]、IMMV[10]和TuMV[11],均属属病毒。电子显微镜观察病毒粒体形态、细胞病理学观察、鉴别寄主鉴定、DAS-ELISA检测、RT-PCR检测及序列测定分析是检测鉴定番红花病毒的主要方法[4-11]。虎眼万年青花叶病毒(,OrMV)又称天鹅绒嵌纹病毒、翅柱兰Y病毒(Pterostylis virus Y,PtVY),属于马铃薯Y病毒科()、属的成员[12-13]。病毒粒体线状,长720—760 nm,宽12 nm[14]。可通过蚜虫非持久性传播[14]、汁液传播[15]。其基因组为一条正ssRNA,长9 442、9 445或9 475 nts,含有一个大的开放阅读框,编码一个341.5 kD的聚合蛋白,具有典型的属病毒的基因组结构特征[16-17]。OrMV最早在美国报道,目前在荷兰[18]、南非[14]、法国的留尼汪岛[19]、以色列[20]、印度[21-22]、韩国[23]、日本[15,24]、新西兰[25]和澳大利亚[16,26]等地均有报道发生。在中国台湾地区曾在进境的虎眼万年青中检出该病毒[27],而在中国大陆地区则在英国等地引进种植的围裙水仙和喇叭水仙上发现该病毒[28]。自然情况下,OrMV可侵染鸢尾科(Iridaceae)的唐菖蒲属()[22]、雄黄兰属()(KF493898)、鸢尾属()[14,21];百合科(Lillaceae)的虎眼万年青属()[14,24,29];兰科(Orchidaceae)的香荚兰属()[19]、双尾兰属()[16]、绒唇兰属()[26]、翅柱兰属()[26]、铠兰属()[26]、鸟兰属()[26];天门冬科(Asparagaceae)的非洲莲香属()[14,29]、石蒜科(Amaryllidaceae)的水仙属()[28,30]、六出花科(Alstroemeriaceae)的六出花属()[18]等植物。【本研究切入点】基于PCR技术的分子检测方法,已越来越多应用于口岸植物病毒的快速检测鉴定中。其中,根据基因组序列的保守区域设计简并引物所建立的病毒属或病毒科的通用PCR或RT-PCR方法,是快速筛查检测同一类病毒的一种高效、灵敏的检测方法,结合序列分析可在短期内实现同一类病毒(如某个病毒属)种类的检测与鉴定[31-33]。【拟解决的关键问题】利用科通用RT-PCR方法[34]对一批旅客携带的番红花种球进行检测,以证实该批番红花种球携带OrMV,防止该病毒入境危害。
1 材料与方法
试验于2016年在厦门出入境检验检疫局检验检疫技术中心植物检疫实验室完成。
1.1 番红花种球
一袋番红花种球,共计25粒,从来自香港航班的旅客行李中截获,包装袋上显示来自荷兰(Aviflora Trading B.V.)。
1.2 样品制备及核酸提取
混合样品:把种球分为两组(12粒、13粒),每粒球茎切取约0.05 g的薄片,用植物汁液提取仪(MEKU Erich Pollähne Gmbh)研磨,然后用3.0 mL样品提取缓冲液冲洗收集。经8 000 r/min离心后,取250 μL的上清液,根据操作说明用TRIzol方法(Invitrogen公司)提取总RNA。
单粒种球:切取约0.1 g的球茎薄片,用研钵研磨后用TRIzol方法提取总RNA。
1.3 RT-PCR检测
取4 μL的总RNA和2 μL Oligo(dT)18引物(10 μmol·L-1),于65℃水浴中处理10 min,然后迅速冰浴5 min。继续加入5×M-MLV buffer 4 μL、dNTP Mixture(10 mmol·L-1;宝生物工程(大连)有限公司)1 μL、M-MLV反转录酶(200 U·μL-1;Promega公司)1 μL、RNase inhibitor(40 U·μL-1;宝生物工程(大连)有限公司)1.0 μL,无菌水补足至20 μL。37℃水浴处理1 h,70℃水浴10 min,冰上冷却,即得到作为PCR模板的cDNA。
采用科通用引物Sprimer:(5′-GGNAAYAAYAGYGGNCARCC-3′,N=A,T,G,C;Y=T,C;R=A,G)和M4T(5′-GTTTTCCCAGTC ACGAC(T)15-3′)进行PCR扩增[34]。PCR反应体系(50 µL):10×EasyTaq Buffer(plus Mg2+)5 µL,引物Sprimer和M4T各2 µL(10 µmol·L-1),cDNA模板6 µL,dNTP(2.5 mmol·L-1)4 µL,EasyDNA聚合酶0.5 µL(5 U·μL-1;北京全式金生物技术有限公司),双蒸水补足至50 µL。PCR反应程序:95℃预变性5 min;94℃变性30 s,47℃退火1 min,72℃延伸2 min,共35个循环;72℃延伸7 min,结束后4℃保存。取5 µL扩增产物在1.5%的琼脂糖凝胶上进行电泳,然后在凝胶成像仪上观察、拍照。
1.4 序列分析
PCR产物经回收后,进行克隆测序,由宝生物工程(大连)有限公司完成。序列拼接后,用基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)、MEGA等软件进行序列分析和系统发育分析。其中,用MEGA 6中的最大似然法(maximum likelihood,ML)构建系统发育树[35],自举检验(bootstrap)1 000次,并以石蒜花叶病毒(,ValMV,EF114726)为外群;用BLAST和基于对数期望的多重序列比较(Multiple Sequence Comparison by Log-Expectation,MUSCLE)进行序列比对分析。用于比较分析的虎眼万年青花叶病毒(OrMV)其他分离物序列下载自GenBank,其寄主、国家或地区等信息见表1。
2 结果
2.1 RT-PCR扩增
利用科的通用RT-PCR方法,在两组混合样品中均扩增到约1.7 kb的条带,且未见其他条带产生(图1-A)。对其中14粒种球进行单独检测,结果显示14个种球样品中8个种球样品明显扩增到约1.7 kb的预期条带,5号、11号样品也出现预期大小条带,但条带比较弱(图1-B)。该结果初步表明,该批番红花种球携带有科病毒,且种球带毒率在57 %以上。
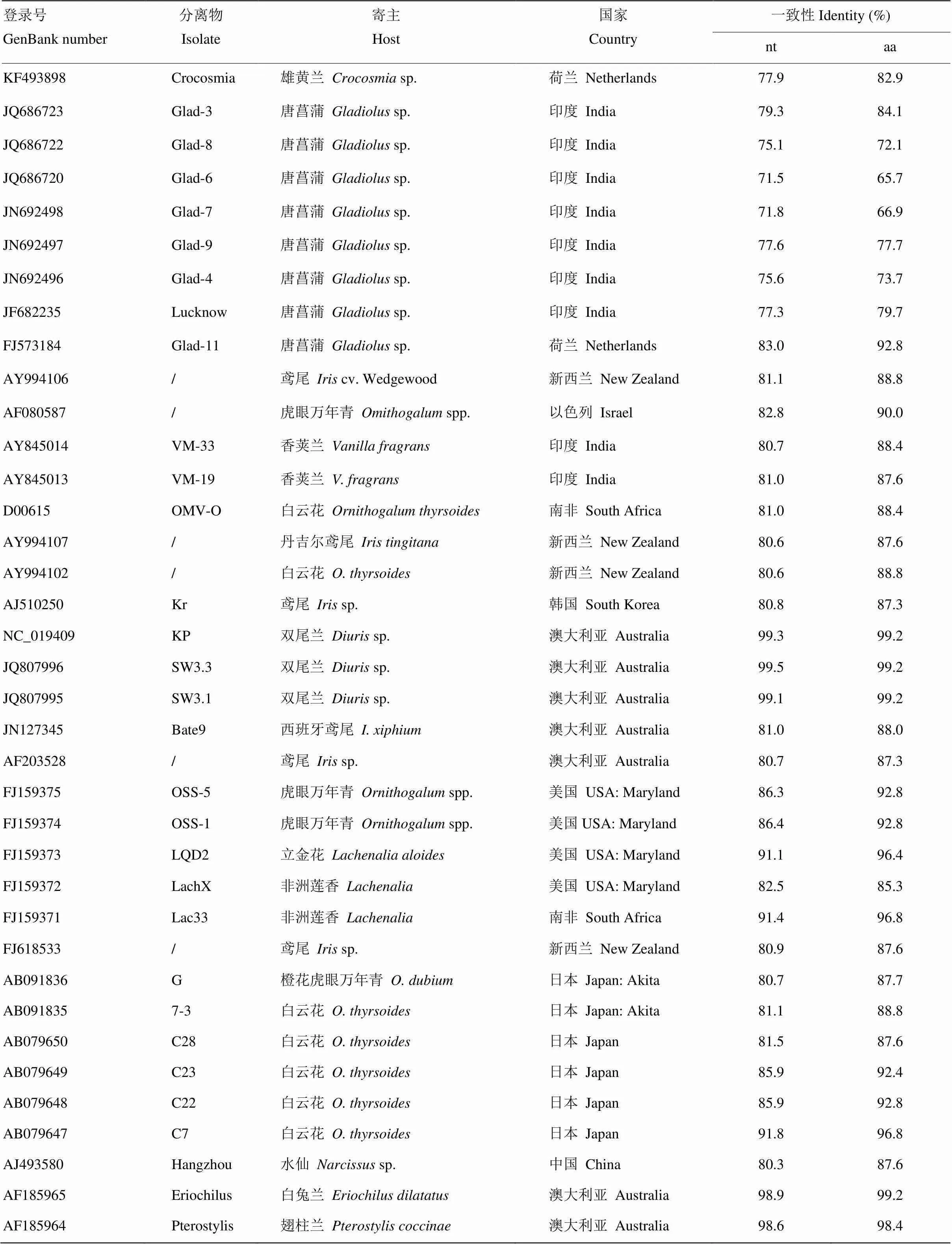
表1 2677-NL分离物与其他虎眼万年青花叶病毒分离物的外壳蛋白基因核苷酸(nt)和氨基酸(aa)序列一致性
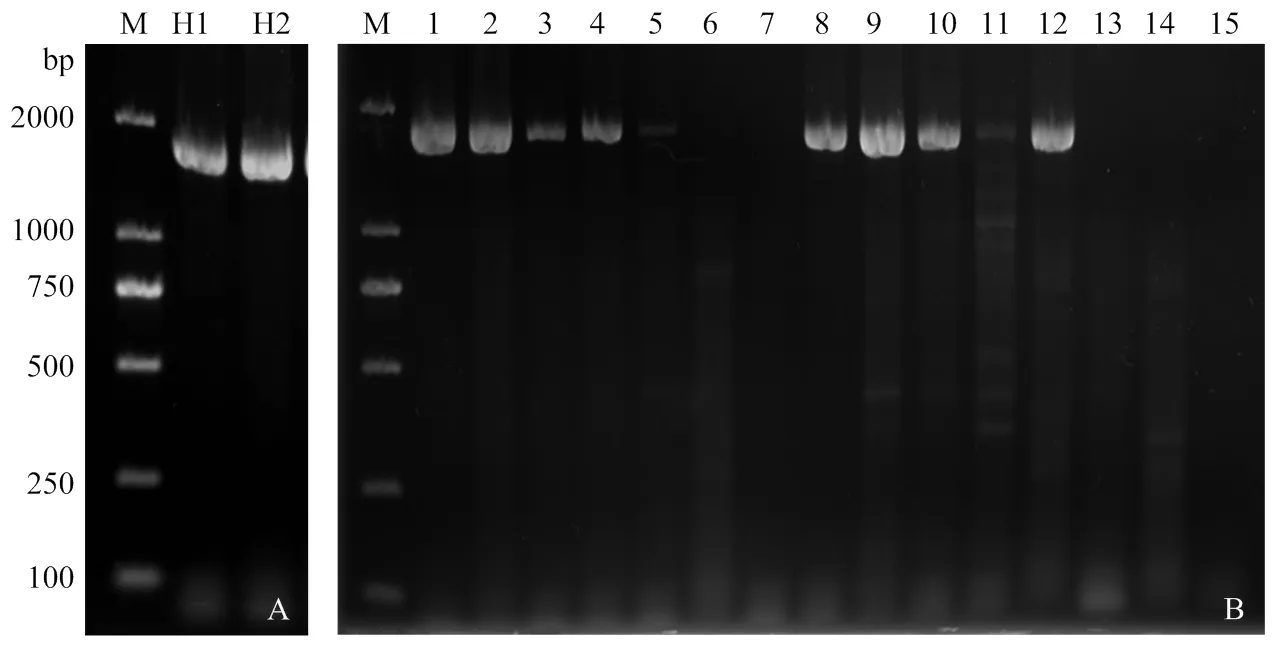
M:TaKaRa DL2000 DNA Marker;H1、H2:混合种球样品mixed corm sample;1—14:单粒种球样品single corm sample;15:空白对照Blank control
2.2 序列测定与分析
随机取其中一个混合样品的RT-PCR产物进行克隆测序,共测定1 666 nts的序列(不包括22 nts的A尾),GenBank登录号为KJ194467,命名为2677-NL。该分离物与OrMV SW3.3分离物(JQ807996)的序列一致性最高,为99.6%;与NCBI中OrMV参考基因组序列(Reference genomic sequences,RefSeq;NC_019409)的序列一致性为99.1%。序列分析显示,该序列含有部分聚合蛋白(polyprotein)基因和3′-端非编码区(3′-UTR)。其中,1—636 nts为部分核内含体b基因(nuclear inclusion b,)的3′端序列,637—1 395 nts为完整的外壳蛋白基因(coat protein,)序列。
基因序列分析显示,2677-NL与其他OrMV分离物的核苷酸和氨基酸序列一致性分别为71.5%— 99.5%和65.7%—99.2%(表1)。其中,2677-NL的核苷酸序列与Glad-4(JN692496)、Glad-6(JQ686720)、Glad-8(JQ686722)和Glad-7(JN692498)4个分离物的序列一致性<76%;与Glad-4(JN692496)、Glad-6(JQ686720)、Glad-7(JN692498)、Glad-8(JQ686722)、Glad-9(JN692497)和Lucknow(JF682235)6个分离物的氨基酸序列一致性<80%。2677-NL与其他32个OrMV分离物的核苷酸和氨基酸序列一致性分别为77.9%—99.5%和82.9%—99.2%,分别>76%和80%。国际病毒分类委员会(International Committee on Taxonomy of Viruses,ICTV)关于科病毒种类区分标准之一是:的核苷酸和氨基酸序列一致性分别<76%和80%。因此,根据ICTV分类标准,2677-NL属于OrMV的一个分离物。
2.3 系统发育分析
根据构建的系统发育分析表明,来自不同地区、不同寄主的OrMV分离物主要分归为2个大类群(I和II)。虽然这2个类群并没有完全根据寄主或地域进行分类群(如澳大利亚分离物的其中2个分离物和另外5个分离物在分属于2个不同的大类群中),但在小分支中,还是具有一定的寄主和地域相关性。如来自澳大利亚的5个分离物(JQ807995、JQ807996、NC_019409、AF185964、AF185965)处于同一个小分支中,与地域相关;而来自日本的2个分离物(AB079648、AB079649)和来自美国的2个分离物(FJ159374、FJ159375)也处于同一小分支中,寄主均为虎眼万年青属,与寄主相关;来自印度的7个分离物(JQ686720、JQ686722、JQ686723、JF682235、JN692496、JN692497和JN692498)也处于同一个小分支中,与寄主和地域相关。本研究的2677-NL分离物属于I群中,与来自荷兰的Crocosmia分离物(KF493898)处于不同类群中,与其中5个澳大利亚分离物(JQ807995、JQ807996、NC_019409、AF185964、AF185965)处于同一分支,显示出最近的亲缘关系(图2)。这5个澳大利亚分离物寄主并不相同,有侵染双尾兰属的SW3.1(JQ807995)、SW3.3(JQ807996)、KP(NC_019409)分离物,侵染白兔兰的Eriochilus分离物(AF185965),侵染翅柱兰的Pterostylis分离物(AF185964),但均属兰科。系统发育分析结果进一步证实2677-NL属于OrMV的一个分离物。
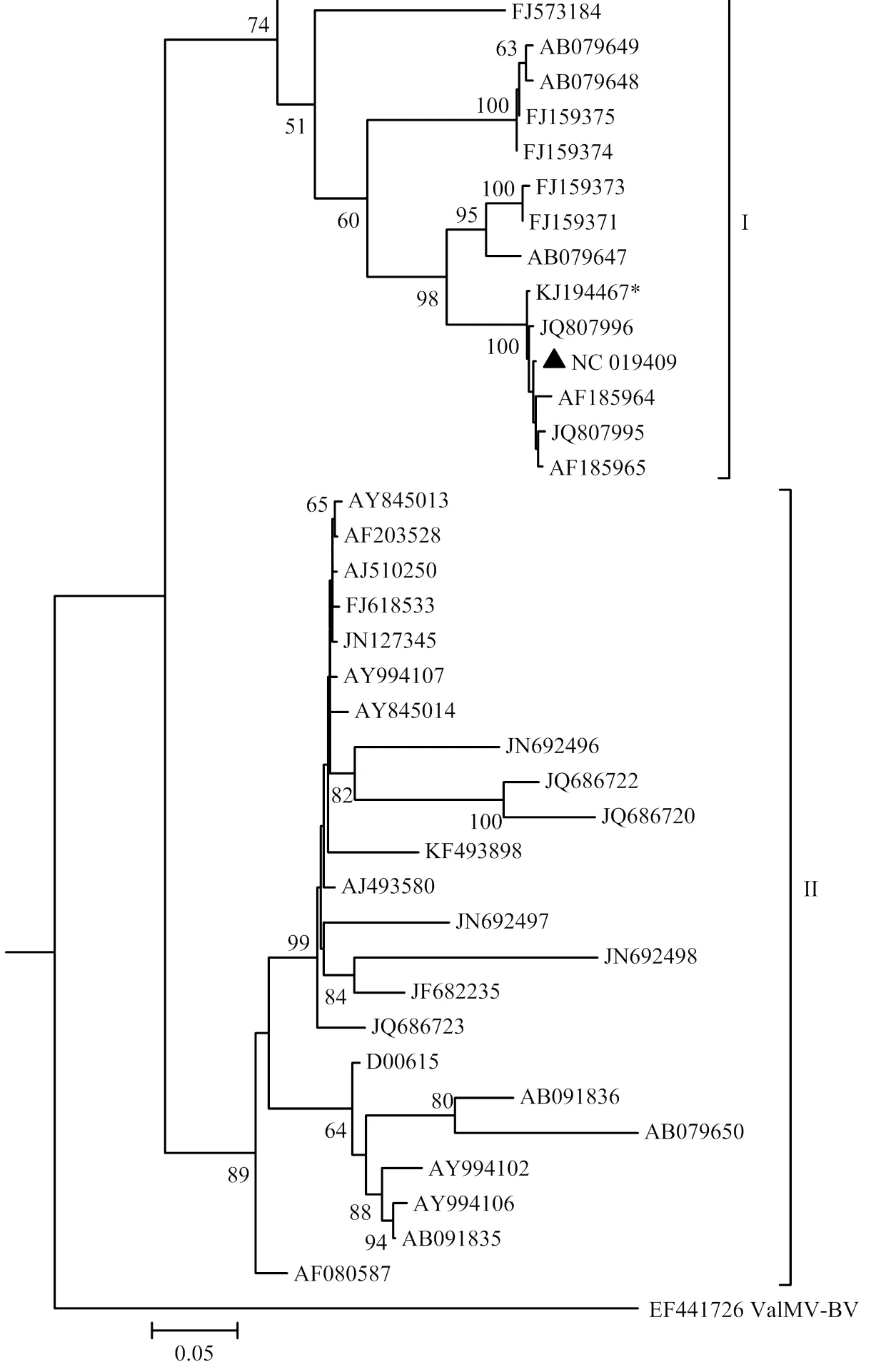
*为本研究的2677-NL分离物;▲NCBI中OrMV的参考基因组序列。节点上的数值为1 000次检验后的自举检验值(≥50) * was the 2677-NL isolate used in this study. ▲ wasNCBI reference genomic sequence of OrMV. The numbers on nodes indicated bootstrap values (≥50) after 1 000 replicates
3 讨论
ICTV第9次分类报告关于科病毒种类区分标准之一为CP蛋白序列一致性约<80%;或整个基因组的核苷酸序列一致性<76%[12,36]。本研究所测定的2677-NL部分3′末端序列与OrMV分离物最高序列一致性达99.6%,与OrMV的RefSeq(NC_019409)的序列一致性为99.1%,其与其中32个分离物(包括OrMV的RefSeq)的核苷酸和氨基酸序列一致性分别>76%和80%。因此,根据ICTV分类标准,2677-NL分离物应属于OrMV的一个分离物。这是首次在番红花中发现OrMV的报道,是侵染番红花的又一种属病毒。
在已知的OrMV分离物中,2677-NL与其中4个分离物的核苷酸序列一致性<77%,与6个分离物的氨基酸序列一致性<80%。其中,2677-NL与Lucknow分离物(JF682235)[21]的核苷酸序列一致性>76%(77.3%),但氨基酸序列一致性略<80%(79.7%),根据ICTV分类标准,应为同一病毒的不同分离物。2677-NL与Glad-9(JN692497)分离物的核苷酸序列一致性>76%但氨基酸序列明显<80%(77.7%),与Glad-4(JN692496)、Glad-6(JQ686720)、Glad-7(JN692498)、Glad-8(JQ686722)分离物的核苷酸和氨基酸序列一致性分别<76%和80%,无法通过基因序列确定为同一病毒种类。ICTV第10次分类报告中,除了明确作为科病毒种类区分标准外,还确定了整个开放阅读框(open reading frame,ORF)以及各个编码区作为种类区分的标准[37]。因此,对于这些分离物,应该进一步比较分析整个ORF或不同的编码区序列,以确认这些分离物是否为同一病毒种类。
BYMV、ISMV、IMMV和TuMV是番红花中比较常见的病毒种类,均属于属,中国的番红花已报道除ISMV外的其他3种病毒[9-11]。在本研究中,利用基于简并引物的通用检测方法并结合序列分析,发现的病毒并不是在番红花中已报道的4种属病毒,而是一种在番红花中还没有报道的OrMV。因此,利用通用RT-PCR检测方法不仅可以用来快速检测已报道的番红花病毒,而且还可以用来快速检测未报道的同一病毒属的其他病毒。该类方法通常只要一对引物、一次RT-PCR就可以检测不同种类的病毒,是特异性RT-PCR方法不能够实现的,是该类检测方法的一大优势。该类方法不仅可以减轻实验操作强度、缩短检测周期,而且可以快速检测鉴定寄主中未报道的病毒种类、甚至新的病毒种类。
另一方面,基于简并引物的通用RT-PCR方法,可以快速得知一个样品中是否受到多种病毒的复合侵染。在本试验中,如果不仅有OrMV,而且还有BYMV、IMMV、ISMV、TuMV中的一种或多种病毒的复合侵染,当PCR产物直接测序时,就会出现叠峰、套峰现象。然后进一步把PCR产物进行克隆,并挑取多个克隆子进行测序,就可以得到不同种类的病毒序列。因此,利用通用RT-PCR方法检测时,可以利用PCR产物直接测序以快速辨别是否为单一侵染或多种病毒的复合侵染。因此,进行PCR产物克隆测序时,建议至少单独测5个或以上的克隆子,以辨别是否有多种病毒的复合侵染。
本研究所使用的科病毒通用RT-PCR检测方法,已经广泛应用于该科病毒的检测。在原文献中,先使用M4T引物作为反转录合成cDNA的起始引物,而后用Sprimer和M4引物作为PCR扩增引物[34]。在本研究中,反转录时使用Oligo(dT)18引物代替M4T引物,而PCR时用M4T引物代替M4引物。Oligo(dT)18引物是反转录时常用的起始引物,在各实验室或反转录试剂盒中基本都有配置。因此,在科病毒通用RT-PCR检测中,省去M4引物,操作更为便捷。
本研究测定了2677-NL分离物的基因组3′端部分序列,含有部分、完整的和3′-UTR序列,与OrMV的SW3.3分离物具有最高的序列一致性(99.6%)。根据进行的系统发育关系分析表明,在2个大类群中,没有明显的地域、寄主相关性,但在各小分支中,具有一定的寄主、地域相关性。从包装信息来看,本次截获的番红花种球来自荷兰。2677-NL分离物与来自荷兰的Crocosmia分离物(KF493898)分属于2个类群中,显示出比较远的亲缘关系。这可能是由于当前的种子苗木贸易非常频繁,OrMV随着种子苗木的贸易在世界范围内快速扩散传播。
侵染番红花的病毒,不仅有常见的BYMV、CMV、NMV等病毒,而且还有中国严格禁止进境的检疫性病毒ArMV和TSWV[3-8]。在此之前中国口岸部门在进境的番红花种球中还未发现植物病毒,该批番红花种球中发现OrMV,是口岸部门首次在进境番红花种球中发现植物病毒。因此,口岸检验检疫部门不仅应该加强邮寄的包裹、旅客随身携带的行李检疫,而且应该加强对大规模引进种植的番红花种球的病毒检疫,以防止各种检疫性病毒的传入危害。此外,OrMV不仅寄主范围比较广泛,而且地理分布也越来越广[18-30]。应该进一步对OrMV进行风险分析、危害等方面的研究,并采取相应检疫措施,以避免该病毒对中国的农业生产和环境生态产生新的威胁。
4 结论
根据ICTV分类标准,2677-NL属于OrMV的一个分离物。该批来自荷兰的番红花种球携带有OrMV,这是OrMV首次侵染番红花的报道,是番红花上发现的又一种属病毒。
[1] 兰卫, 朱金芳, 耿直. 新疆西红花的种植技术. 新疆医科大学学报, 2010, 33(8): 874-875.
Lan W, Zhu J F, Geng Z. Planting techniques of Xinjang saffron., 2010, 33(8): 874-875. (in Chinese)
[2] Ahrazem O, Rubio-Moraga A, Castillo-López R, Mozos A T, Gómez-Gómez L.pathogens and defence responses//Husaini A M.., 2010, 4(Special Issue 2): 81-90.
[3] Caiola M G, Faoro F. Latent virus infections inand., 2011, 50(2): 175-182.
[4] Miglino R, Jodlowska A, Van Schadewijk A R. First report ofinfectingspp. cultivars in the Netherlands., 2005, 89(3): 342.
[5] Corona F M O, Lebas B S M, Elliott D R, Tang J Z, Alexander B J R. New host records and new host family range forin New Zealand., 2007, 2(1): 127-130.
[6] Ehrig F, Kegler H, Fuchs E. Detection of a virus hitherto unknown inhybrids., 1997, 30(5): 453-455.
[7] Samuitienė M, Navalinskienė M, Jackevičienė E.on ornamental plants., 2008, 54(4): 264-268.
[8] Samuitienė M, Navalinskienė M. Occurrence ofon ornamental plants in Lithuania., 2008, 95(3): 135-143.
[9] 谢礼, 吕明芳, 董峰丽, 饶君凤, 毛碧增, 洪健. 藏红花病毒病原的分子鉴定. 中草药, 2013, 44(8): 1033-1036.
Xie L, Lü M F, Dong F L, Rao J F, Mao B Z, Hong J. Molecular identification of viral pathogens in., 2013, 44(8): 1033-1036. (in Chinese)
[10] 赵培洁, 许彩霞, 徐正, 高其昌. 由栽培番红花中检出鸢尾轻花叶病毒. 中药材, 1990, 13(5): 4-6.
Zhao P J, Xu C X, Xu Z, Gao Q C. Identification ofin the cultivated., 1990, 13(5): 4-6. (in Chinese)
[11] 陈集双, 李德葆. 侵染番红花的芜菁花叶病毒. 植物病理学报, 1995, 25(4): 361-365.
Chen J S, Li D B. Ainfecting., 1995, 25(4): 361-365. (in Chinese)
[12] Burger J T, Brand R J, Rybicki E P. The molecular cloning and nucleotide sequencing of the 3′-terminal region of., 1990, 71(11): 2527-2534.
[13] Adams M J, Antoniw J F, Fauquet C M. Molecular criteria for genus and species discrimination within the family., 2005, 150: 459-479.
[14] Burger J T, Von Wechmar M B. Purification and some properties of South African isolates of., 1989, 79(4): 385-391.
[15] 松本勤, 山本英樹, 高橋春實, 神田啓臣, 勝田茂満, 相馬玲子, 藤晋一, 井上正保. オーニソガラムモザイクウイルスによるオーニソガラムモザイク病の発生と宿主範囲を異にする分離株の存在. 日本植物病理学会報, 2007, 73(2): 102-105.
Matsumoto T, Yamamoto H, Takahashi H, Kanda H, Katsuta S, Soma R, Fuji S, Inoue M. The first report on the occurrence ofin Japan and the presence of isolates differing in pathogenicity to dicot plants., 2007, 73(2): 102-105. (in Japanese)
[16] Wylie S J, Li H, Dixon K W, Richards H, Jones M G K. Exotic and indigenous viruses infect wild populations and captive collections oftemperate terrestrial orchids (species) in Australia., 2013, 171: 22-32.
[17] Cho S Y, Lim S, Kim H, Yi S I, Moon J S. Complete genome sequence ofmosaic virus infectingspp. in South Korea., 2016, 4(4): e00816-16.
[18] Bouwen I, van der Vlugt R A A. Natural infection ofwith., 2000, 84(2): 202.
[19] Grisoni M, Moles M, Farreyrol K, Rassaby L, Davis R, Pearson M. Identification of potyviruses infecting vanilla by direct sequencing of a short RT-PCR amplicon., 2006, 55(4): 523-529.
[20] Zeidan M, Cohen J, Watad A, Gera A. Improved purification and molecular properties ofin Israel., 1998, 133(2): 167-176.
[21] Chandel V, Kulshrestha S, Hallan V, Zaidi A A. Natural infection ofonfrom India., 2006, 55: 284.
[22] Kaur C, Raj S K, Snehi S K, Goel A K, Roy R K. Natural occurrence ofnewly reported on gladiolus in India., 2011, 24: 2.
[23] Yoon H I, Ryu K H. Molecular identification and sequence analysis of coat protein gene ofisolated fromplant., 2002, 18(5): 251-258.
[24] Fuji S, Inoue M, Yamamoto H, Furuya H, Naito H, Matsumoto T. Nucleotide sequences of the coat protein gene of potyviruses infecting., 2003, 148: 613-621.
[25] Wei T, Pearson M N, Cohen D. First report ofand, 2006, 55(6): 820.
[26] Gibbs A, Mackenzie A, Blanchfield A, Cross P, Wilson C R, Kitajima E, Nightingale M, Clements M. Viruses of orchids in Australia: their identification, biology and control., 2000, 65(3): 10-21.
[27] 陈金枝,张幼莉,张清安,蔡惠婷.于进口之天鹅绒发生严重嵌纹之病株中发现由天鹅绒嵌纹病毒及复合感染之分子生物学证据. 植物病理学会刊, 2000,9(4): 184-185.
Chen J Z, Zhang Y L, Zhang Q A, Cai H T. Molecular evidences for the dual infection ofandin an importedplant with severe mosaic symptom., 2009, 9(4): 184-185. (in Chinese)
[28] 郑红英, 鲁宇文, 沈嘉乐, 林林, 陈炯,陈剑平. 侵染水仙的虎眼万年青花叶病毒外壳蛋白的原核表达、抗血清制备及应用. 浙江农业学报, 2009, 21(3): 198-201.
Zheng H Y, Lu Y W, Shen J L, Lin L, Chen J, Chen J P. Prokaryotic expression of coat protein gene ofand its use in the antiserum preparation., 2009, 21(3): 198-201.(in Chinese)
[29] Burger J T, von Wechmar M B. Rapid diagnosis ofandviruses in propagation stock., 1988, 234: 31-38.
[30] Chen J, Chen J P, Langeveld S A, Derks A F L M, Adams M J. Molecular characterization of carla- and potyviruses fromin China., 2003, 151(1): 26-29.
[31] James D, Varga A, Pallas V, Candresse T. Strategies for simultaneous detection of multiple plant viruses., 2006, 28(1): 16-29.
[32] 李鑫, 陈雅诗, 朱连, 张寅寅, 黑多尔, 廖富荣. 进境荷兰百合中车前草花叶病毒的分子鉴定及序列分析. 植物病理学报, 2017, 47(2): 197-202.
Li X, Chen Y S, Zhu L, Zhang Y Y, Hei D E, Liao F R. Molecular identification and sequence analysis ofinfrom Netherlands., 2017, 47(2): 197-202. (in Chinese)
[33] 叶志红, 廖富荣, 郭木金, 方志鹏, 陈青, 陈红运, 林石明, 林毅. 同时检测豇豆花叶病毒属与蚕豆病毒属病毒的通用RT-PCR检测方法. 中国农业科学, 2015, 48(8): 1527-1537.
Ye Z H, Liao F R, Guo M J, Fang Z P, Chen Q, Chen H Y, Lin S M, Lin Y. A universal RT-PCR method for the simultaneous detection of the viruses in generaand., 2015, 48(8): 1527-1537. (in Chinese)
[34] Chen J, Chen J, Adams M J. A universal PCR primer to detect members of theand its use to examine the taxonomic status of several members of the family., 2001, 146(4): 757-766.
[35] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular evolutionary genetics analysis version 6.0., 2013, 30(12): 2725-2729.
[36] Adams M J, Zerbini F M, French R, Rabenstein F, Stenger D C, Valkonen J P T. Family//King A M, Adams M J, Carstens E B, Lefkowitz E J.. London, UK: Elsevier Academic Press, 2012: 1069-1089.
[37] Wylie S J, Adams M, Chalam C, Kreuze J, López-Moya J J, Ohshima K, Praveen S, Rabenstein F, Stenger D, Wang A, Zerbini F M, ICTV Report Consortium. ICTV virus taxonomy profile:., 2017, 98(3): 352-354.
(责任编辑 岳梅)
Molecular identification and sequence analysis ofin saffron () corms
LIAO FuRong1, LIN WuZhen1,2, CHEN XiHong1,3, CHEN Qing1, CHEN HongYun1, HUANG PengYing1, FANG ZhiPeng1, WU Yuan1, SHEN JianGuo3, LIN ShiMing1
(1Inspection and Quarantine Technology Center, Xiamen Entry-Exit Inspection and Quarantine Bureau, Xiamen 361026, Fujian;2Key Laboratory of Biopesticide and Chemical Biology, Ministry of Education, Fuzhou 350002;3Inspection and Quarantine Technology Center, Fujian Entry-Exit Inspection and Quarantine Bureau, Fuzhou 350003)
The objective of this study is to identify viruses in saffron () corms imported from the Netherlands and to prevent the entry of dangerous virus into China.Using theuniversal primers (Sprimer and M4T), the saffron corms samples were detected by reverse transcription polymerase chain reaction (RT-PCR) assay. After cloning and sequencing of the RT-PCR products, the Basic Local Alignment Search Tool (BLAST) and Multiple Sequence Comparison by Log-Expectation (MUSCLE) methods were used for multiple sequence alignment. The maximum likelihood method was used to construct the phylogenetic tree according to the coat protein () nucleotide sequences.The about 1.7 kb RT-PCR products were amplified from the samples, indicating a member ofvirus was present in the samples of saffron corms. A sequence of 1 666 bp segments was obtained from a positive sample (designated 2677-NL) and it contained a portion of nuclear inclusion b gene (), the completeand 3′ untranslated region (3′-UTR). This isolate shared the highest nucleotide sequence identity (99.6%) with the SW3.3 isolate of(OrMV) and shared 99.1% nucleotide sequence identity with the Reference genomic sequences (RefSeq) of OrMV (GenBank accession number: NC_019409). Of those, theof the 2677-NL isolate shared 71.5%-77.3% and 65.7%-79.7% nucleotide and amino acid identities, respectively, with those of six OrMV isolates from India (GenBank accession number: JQ686722, JQ686720, JN692498, JN692497, JN692496 and JF682235) whereas 77.9%-99.5% and 82.9%-99.2%, respectively, with the other 32 OrMV isolates. Phylogenetic analysis based on thegenes showed that the OrMV could be divided into two groups, in which 2677-NL was clustered together with five OrMV isolates from Australia (GenBank accession number: JQ807995, JQ807996, NC_019409, AF185964 and AF185965), suggesting these OrMV were highly homologous in the phylogenetic relationship.According to the species demarcation criteria of the familyon the International Committee on Taxonomy (ICTV), the 2677-NL was an isolate of OrMV, and it showed that the saffron corms sample was infected by OrMV. this is the first report on OrMV infectingin the world.
;; RT-PCR; new host
2017-05-02;接受日期:2017-06-25
福建省自然科学基金(2015J01148)、国家质量监督检验检疫总局科技计划(2015IK190)、厦门市科技计划(3502Z20154080)
廖富荣,Tel:0592-6806943;E-mail:lfr005@163.com。通信作者林石明,Tel:0592-6803996;E-mail:smlink@163.com

