植物基因家族全基因组分析平台构建及在Dof中的应用
王培培 刘长宁


摘要:指出了基因家族是植物基因组的重要组成部分,在植物整个生长发育过程中扮演着重要角色,参与各种生物、非生物胁迫的应答反应,而且在植物环境适应性进化过程中可发挥重要作用。植物基因家族分析方法多样,可选择软件较多,参数设置繁琐,分析流程缺少规范性。为拉低学科差异的鸿沟与大数据分析的繁琐性,构建了一个植物基因家族全基因组鉴定与分析平台,以小桐子Dof转录因子基因家族全基因组分析为例进行了平台测试,可为植物基因家族的研究提供一个流程化参考。
关键词:基因家族;分析平台;全基因组分析
中图分类号:S432.2+3
文献标识码:A
文章编号:1674-9944(2018)07-0001-05
1 研究概况
由于存在物种特异性扩张,高等生物基因组中有丰富的多基因家族及超基因家族(superfamilies),20世纪70年代开始,人们逐渐对这种在生物个体和群体中产生遗传变异并与基因组冗余有微妙关系的基因家族的研究产生了兴趣[1]。基因家族成员以簇状或无规律形式分布于基因组的不同位置并在植物生長发育过程中发挥重要作用。转录因子(transcription factor,TF)是基因表达的转录调控因子,调控复杂的DNA到RNA的时空特异性表达,转录因子基因家族是植物中最大、作用最广泛的基因家族之一[2]。由转录因子组成的转录起始前复合物可以偶联靶基因启动子中的顺式作用元件并启动基因转录。例如,转录因子通过招募启动子共激活因子、一般转录因子、染色质重塑因子等蛋白因子构成转录起始前复合物并激活RNA聚合酶,促进RNA转录链的起始及延伸[3]。
伴随着二代测序的广泛应用及生物信息学的快速发展,解析大数据密码所隐含的生物学现象是生命科学研究者要解决的首要问题.基于这样的契机,流程化分析平台应运而生。一些不断更新的分子生物学数据库作为基因组序列的仓储,为在全基因组水平研究转录因子基因家族提供了便利,常用的比较全面的大型数据库比如NCBI、Ensembl和DDBJ等是转录因子研究的重要数据来源。常用的转录因子数据库包括PlantTFDB4.0( http: //planttfdb.cbi.pku.edu.cn/)和PlnTFDB 3.0( http://plntfdb.bio.unipotsdam.de/v3.0/)等,为植物转录因子的研究提供了丰富的开源数据。
目前,有关植物基因家族全基因组分析的研究大部分都集中在一些重要的转录因子上,比如bZIP、MADS- box、SBP - box、WRKY、AP2/ERP、NAC等等,而对Dof( DNA- binding with one zinc finger)锌指蛋白的报道相对较少,它是植物特有的一类转录因子基因家族,属于锌指蛋白超家族( zine finger super - family)。Dof在多种代谢途径及植物生长发育过程中发挥着重要作用,包括C、N代谢、光响应、种子发育和萌发等[4]。首个Dof转录因子蛋白被发现于玉米中(ZmDofl),在玉米糊粉层形成过程中有重要功能朝。近年来,相继从拟南芥[6]、水稻[7]、大麦[8]、小麦[9]、大豆[10]、蓖麻[11]等物种中鉴定出Dof基因,并对其功能进行了深入地研究,不断证实了Dof基因的功能重要性。
各种模式生物基因组测序工作完成之后,基因家族成为功能基因组学的研究热点之一。基因家族全基因组分析的专业人士可以根据数据特征及分析目的自主选择分析策略。但是,对于非专业的基因家族研究者而言,目前缺少一个流程化的分析平台。基于分子生物学和生物信息学研究背景的复杂性,搭建一个基因家族分析平台有其重要的现实意义。目前关于基因家族分析有很多值得借鉴的软件工具与操作方法,但仍然存在很多值得完善的地方。例如,分析方法多样化,可选择软件较多,参数设置繁复,分析流程复杂等。研究参考已有的分析方法,利用现有的硬件设施选择合适的分析工具初步搭建了植物基因家族全基因组分析平台,为植物基因家族流程化、规范化分析提供参考。
2 平台搭建
输入并整合分析蛋白质数据、核酸数据、转录组数据,结合基因家族全基因组鉴定模块与挖掘分析模块,完成流程化基因家族全基因组鉴定与分析。
2.1 测试数据来源
平台应用测试所涉及到的物种基因组信息是 从以下数据仓储网站下载:小桐子最完整版基因组数据(Assembly JatCur1.0)来源于NCBI( https://WWW.ncbi. nlm. nih. gov/);拟南芥Dof基因家族蛋白质序列与核酸序列数据来源:TAIR 9.0(http://www. arabi-dopsis. org/);蓖麻Dof基因家族蛋白质序列与核酸序列数据来源:PlantTFDB database( http://planttfdb.cbi. pku. edu.cn/);小桐子Dof基因表达量数据收集于:SRA数据库(https://www. ncbi. nlm. nih. gov/)。
2.2 平台流程
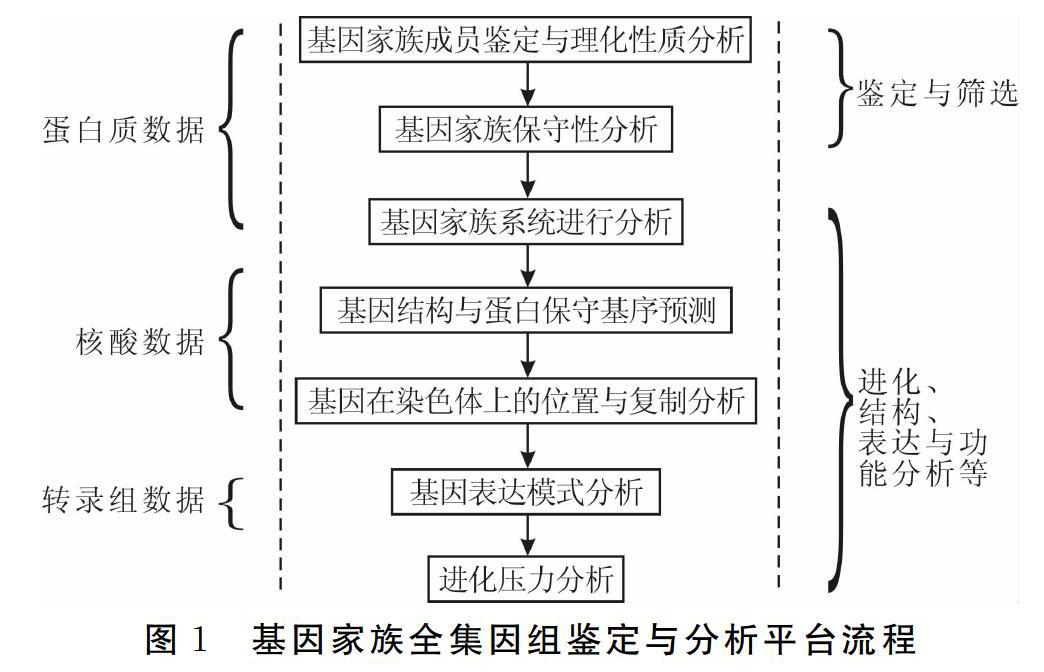
平台分析流程主要包括以下几个步骤:基因家族成员鉴定与理化性质分析;基因家族保守性分析;基因家族系统进化分析;基因结构与蛋白保守基序分析;基因在染色体上的位置与复制分析;基因表达模式分析;进化压力分析(图1)。分析流程中参考已有的分析策略结合现有的硬件设备,选择了一系列较为高效便捷的软件工具(图2)。
2.3 结构模块
平台没计主要包括三个模块,分别为可视化模块、逻辑操作模块和数据服务模块。其中可视化用户服务界面主要通过snakemake实现;逻辑操作模块主要包含各个分析模块及其涉及到的应用软件T具;数据服务模块主要是指基因家族分析使用的公共数据库中的基因组数据(图2)。
3 方法步骤
3.1 基因家族成员鉴定
为筛选出某物种基因家族所有成员,结合Blastp和hmmsearch两种程序对全基因组数据进行全面搜索。首先,利用待鉴定物种的全基因组蛋白质数据,构建本地Blast数据库,以模式植物拟南芥转录因子蛋白质作为query序列执行本地blastp程序(e- value设置为le-10)。其次,以Pfam蛋白结构域模型作为hm-mquery序列,以物种全基因组数据作为HMM数据库,执行本地hmmsearch程序。两部分筛选结果取交集,删除冗余,所得候选序列利用SMART及NCBI- CDD工具进行蛋白质结构预测[12,13],删除不含目标转录因子特定功能结构域的序列,同时剔除不含完整读码框的序列。利用ExPASy Proteomics Server(http://ex-pasy. org/)工具对所有目标转录因子蛋白氨基酸序列进行分子量、蛋白质长度以及等电点等理化性质预测分析[14]。
3.2 蛋白质保守性分析
为可视化分析目标转录因子蛋白的保守性,首先,使用DNAMAN软件来提取目标基因家族蛋白质的保守区域,结合smart验证目标蛋白是否含有该家族特定的保守的功能domain[15];其次,通过ClustalW软件对目标基因家族成员进行多序列联配比对分析,鉴定出高度保守的蛋白质结构域.找到标志性功能位点,并用同种颜色标示保守的氨基酸[16]。
3.3 系统演化分析
首先,利用guidance2 工具对蛋白质序列进行多序列联配比对分析,设置梯度参数获得信任值比较高的columns[17]。其次,使用提取得到的蛋白质序列,结合MEGA6 [18]软件采用邻接法(Neighbor - Joining NJ)生成目标基因家族的系统演化树,替换模式选用“poissonmodel”,校验参数Bootstrap设置为1000。最后,使用在线的进化树美化软件EvolView等软件对系统演化树进行二次编辑修饰[19]。
3.4 基因家族蛋白结构和功能基序的预测
每个成员基因核酸序列与核酸序列对应的CDS序列,提交到Gene Structure Display Server( GSDS2.0:http://gsds2. cbi. pku. edu. cn/)[20]软件分析基因结构组成模式,包括内含子、外显子分布模式和数量特征等;利用在线工具MEME (multiple expectation maximiza-tion for motif elicitation)[21]对转录因子蛋白的功能mo-tif进行预测分析,长度参数设定为5~150,预测数量设置为10。
3.5 染色体定位和基因复制分析
结合物种基因组注释GFF3文件,提取目标基因在染色體上的位置信息,将所有目标基因定位在该物种的染色体上,通过MapInspect[22]绘图软件绘制目标基因家族基因组染色体定位图。利用McScanX[23]软件判定基因发生片段复制,软件执行物种all-against-allblastp文件和包含基因位置信息的GFF3文件.计算该物种基因组中的共线性区段(e-value=le-10).发生于同一个共线性区段的目的基因对被认为是发生片段复制的基因对;另外,基因发生串联复制事件的判定条件为:①两个基因序列相匹配部分的长度大于较长序列的80%;②两个基因序列相匹配部分的相似性应大于80%;③紧密相连的基因中,只参与一次复制事件。结合基因在染色体上的位置,两个基因应位于同一条染色体上[24]。
3.6 选择压力分析
每一对发生复制的基因,根据其CDS序列,利用DnaSP[25]软件计算复制基因对的非同义替换率(Ka)和同义替换率(Ks)以及Ka/Ks值.分析发生复制事件的基因所受到的环境选择压力。①若Ka/Ks>1,正选择压力;②若Ka/Ks=1,受到中性选择或自然选择压力;③若Ka/Ks<1,存在纯化选择作用。
3.7 基因家族的表达模式分析
基因的差异表达模式分析是基因功能研究的重要方法,为了进一步探究目标转录因子基因功能,收集基因在不同环境条件下(例如激素处理、盐胁迫、干旱胁迫等)以及不同组织器官中的表达量数据(RNA - seq数据、表达芯片数据等),基因表达数据进行标准化处理之后,利用R、Heml[26]等软件绘制表达谱热图。
4 测试与应用
鉴于植物基因家族分析缺少流程化、规范化平台,构建了一个初级的基因家族全基因组鉴定与分析的平台,依据其具体流程步骤,以小桐子Dof基因家族的全基因组鉴定与分析为例,对该平台的方法进行实践测试。小桐子是大戟科重要的多年生木本植物,因其种子含油量较高及花发育过程的特殊性是大戟科研究的一个潜在的模式植物。平台测试分析过程中,对小桐子Dof基因进行全基因组筛选与鉴定,并对鉴定到的基因进行理化性质、保守性、基因进化、基因结构及功能mo-tif、染色体定位、表达谱、选择压力等进行系统性地研究分析,为小桐子Dof基因后续功能研究与开发利用提供理论基础(表1)。
一共鉴定出了24个Dof基因,共编码33条Dof蛋白,均属于大分子蛋白,其理化性质预测结果列表如表2。
5 总结
越来越多的植物全基因组测序完成,对基因家族的研究也越来越普遍,本研究初步搭建了一个植物基因组分析平台。主要成果包括:整理了基因家族分析的比较常见的分析方法,归纳出了主要分析流程;设计了分析平台主要结构模块主要包括可视化层一逻辑操作层一数据服务层等主要框架。利用小桐子Dof基因的流程化分析对该植物基因家族分析平台进行了测试应用,验证了平台的可行性。随着高通量测序的大量产出和发布,本课题的研究工作能为从事植物基因家族分析的工作者提供参考,辅助其完成不同目的的基因家族分析。此外,本课题的工作对相关生物信息学平台的设计与构建也有一定的参考价值。
6 展望
目前,由于時间的限制、技术的不成熟等原因,该分析平台仍然存在很多值得完善的地方。比如:本研究平台初步建立,经验不足.有待继续完善;自动化程度较低,后续snakemake可视化分析需要完善;本平台可以为基因家族功能性分析及其分析平台设计提供一定的参考。
参考文献:
[1]Hool, Campbellj, Elglns. The organization, expression, and evo-lution of antibody genes and other multigene families [J]. Annualreview of genetics, 1975, 9(1): 305~53.
[2] Van Lijsebettensm, Gasser K D. Transcript elongation factors:shaping transcriptomes after transcript initiation [J]. Trends inplant science, 2014, 19(11) : 717~26.
[3] Hahn S, Young E T. Transcriptional regulation in Saccharomy-ces cerevisiae: transcription factor regulation and function, mech-anisms of initiation, and roles of activators and coactivators [J].Genetics, 2011, 189( 3) : 705.
[4] Lijavetzky D, Carbonero P, Vicente- Carbajosa J. Genome-wide comparative phylogenetic analysis of the rice and ArabidopsisDof gene families [J]. BMC evolutionary biology, 2003, 3
[5] YaNngisawa S, Sheen J. Involvement of maize Dof zinc fingerproteins in tissue - specific and light - regulated gene expression[J]. The Plant Cell, 1998, 10(1): 75~89.
[6] Riechmann J L, Heard J, Martin G, et al. Arabidopsis transcrip-tion factors: genome - wide comparative analysis among eu-karyotes [J]. Science, 2000, 290(5499) : 2105.
[7] Lihavetaky D, Carbonero P, Vicente- Catbajosa J. Genome-wide comparative phylogenetic analysis of the rice and ArabidopsisDof gene families [J]. BMC evolutionary biology, 2003, 3
[8] Moreno-Risueno M , Mart Nez M, Vicente- Carbajosa J, et al.The family of DOF transcription factors : from green unicellular al-gae to vascular plants [J]. Molecular Genetics and Genomics,2007, 277(4) : 379.
[9] Chen ay, Guo xj, Chen zx, et al. Genome- wide characterizationof developmental stage- and tissue- specific transcription factorsin wheat [J]. BMC genomics, 2015, 16(1) : 125.
[10] Wang H W, Zhang B, Hao Y J, et al. The soybean Dof - typetranscription factor genes, GmDof4 and GmDofll, enhance lipidcontent in the seeds of transgenic Arabidopsis plants [J]. ThePlant Journal, 2007, 52(4) : 716.
[11] Jin Z, Chandrasekaran U, Liu A. Genome- wide analysis of theDof transcription factors in castor bean ( Ricinus communis L. )[J]. Genes &- genomics, 2014, 36(4): 527.
[12] Xu Q, Dunbrack R L. Assignment of protein sequences to exist-ing domain and family classification systems: Pfam and the PDB[J]. Bioinformatics, 2012, 28(21) : 2763.
[13] Marchler- Bauer A, Zhenh C, Chitsaz F, et al. CDD: con-served domains and protein three- dimensional structure [J].Nucleic acids research, 2012(11).
[14] Artimo P, Jonnalagekka M, Arnold K, et al. ExPASy: SIBbioinformatics resource portal [J]. Nucleic acids research,2012, 40(W1) : 597~603.
[15] Woffelman C. DNAMAN for windows, version 5.2. 10: LynonBiosoft [J]. Institute of Molecular Plant Sciences, Netherlands:Leiden University, 2004(4).
[16] Fukami- Kobayashl K, Salto N. How to make good use ofCLUSTALW [J]. Tanpakushitsu kakusan koso Protein, nucleicacid, enzyme, 2002, 47(9): 1237.
[17] Sela I, Ashkenazy H, Katoh K, et al. GUIDANCE2: accuratedetection of unreliable alignment regions accounting for the un-certainty of multiple parameters [J]. Nucleic Acids Research,2015, 43(W1).
[18] Tamura K, Stecher G, Peterson D, et al. MEGA6: molecularevolutionary genetics analysis version 6.0 [J]. Molecular biolo-gy and evolution, 2013(8).
[19] Zhang H, Gao S, Lercher M J, et al. EvolView, an online toolfor visualizing, annotating and managing phylogenetic trees [J].Nucleic acids research, 2012, 40(W1) : 569.
[20] Hu B, Jin J, Guo AY, et al. GSDS 2.0: an upgraded gene fea-ture visualization server[J]. Bioinformatics, 2014, 31(8) : 1296.
[21] Bailey T L, Johnson J, Grant C E, et al. The MEME suite [J].Nucleic acids research, 2015, 43( Wl) : 39~49.
[22] He H, Dong Q, Shao Y, et al. Genome- wide survey and char-acterization of the WRKY gene family in Populus trichocarpa[J]. Plant Cell Reports, 2012, 31(7) : 1199.
[23] Wang Y, Tang H, Debarry J D, et al. MCScanX: a toolkit fordetection and evolutionary analysis of gene synteny and col-linearity [J]. Nucleic acids research, 2012, 40(7) : 49.
[24] Jin J, Tian F, Yang D-C, et al. PlantTFDB 4. 0: toward acentral hub for transcription factors and regulatory interactionsin plants [J]. Nucleic acids research, 2016(4).
[25] Librako P, Rozas J. DnaSP v5: a software for comprehensive a-nalysis of DNA polymorphism data [J]. Bioinformatics, 2009,25 (11) : 1451.
[26] Deng W, Wang Y, Liu Z, et al. Heml: a toolkit for illustratingheatmaps [J]. PloS one, 2014, 9(11).

