花生出仁率和株高的QTL定位分析
陈伟刚 郭建斌 徐志军 喻博伦 邱西克 黄 莉 宋延滨陈玉宁 周小静 罗怀勇 刘 念 任小平 姜慧芳
花生出仁率和株高的QTL定位分析
陈伟刚**郭建斌**徐志军 喻博伦 邱西克 黄 莉 宋延滨陈玉宁 周小静 罗怀勇 刘 念 任小平 姜慧芳*
中国农业科学院油料作物研究所 / 农业部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
花生出仁率、株高等性状都对产量有重要影响, 鉴定出仁率和株高相关的主效QTL, 分析QTL的加性、上位性及其与环境的互作效应以及出仁率与株高之间的遗传关系, 有助于加快花生分子育种研究进程。本研究以远杂9102×徐州68-4构建的RIL群体为材料, 在4个环境中调查出仁率和株高等表型性状, 相关性分析结果表明, 4个环境中, 出仁率与株高均存在极显著负相关。利用前期构建的高密度遗传图谱, 通过QTLNetwork 2.0软件对出仁率和株高进行QTL定位分析, 检测到13个具加性效应的出仁率QTL, 8个具加性效应的株高QTL, 其中, 2个与出仁率相关的主效QTL (和)和1个与株高相关的主效QTL ()至少能在3个环境下被重复检测到。还检测到11对上位性QTL, 包括出仁率6对和株高5对, 与环境之间均存在互作效应。比较QTL在连锁群上的位置发现, 在A09染色体Ad91I24–AGGS2492区间同时检测到稳定的出仁率主效QTL ()和株高主效QTL ()。通过条件QTL排除该位点株高的效应后, 出仁率加性效应贡献率从14.37%下降到5.50%, 表明和为同一位点, 同时控制株高和出仁率。
花生; 出仁率; 株高; QTL
花生是重要的油料作物和经济作物, 籽仁产量由荚果产量和出仁率组成, 是花生最重要的经济性状。花生的经济产量不仅与出仁率相关, 还与株高相关。通过分析花生品种资源的数据信息《中国花生品种资源目录》, 发现植株偏高( > 60 cm)的材料, 其出仁率偏低, 由此推测, 花生的出仁率与株高可能存在一定的相关性。因此, 分析花生的出仁率与株高之间的相互关系, 对于定位和克隆相关基因、进行功能分析、了解产量性状的遗传网络具有十分重要的意义。
在花生产量相关性状的研究中, 大部分集中在果重、荚果大小和种子大小等性状[1-6], 对出仁率的相关研究较少。Huang等[7]利用F2群体定位到3个与出仁率相关的QTL, 贡献率为2.00%~11.78%; Chen等[3]检测到3个与出仁率相关的QTL, 贡献率为5.6%~10.9%。在株高方面的研究中, 成良强等[8]利用F2群体在2个环境下共检测到9个与株高相关的QTL; Li等[9]检测到11个与株高相关的QTL; Huang等[10]通过RIL群体定位到2个主效QTL, 并且在3个环境中重复检测到。在上述为数不多的QTL研究中, 对出仁率和株高都是独立进行的, 这2个性状的QTL之间是否存在相关性, 未见报道。而研究它们之间的关系, 对于花生的分子育种设计和亲本选配具有重要意义。蔡岩等[11]利用远杂9102和徐州68-4杂交衍生的RIL群体构建的包含365个SSR标记的连锁图, 结合2013年和2014年的出仁率调查结果, 用WinQTLCart 2.5软件共检测到22个与出仁率相关的QTL, 其中贡献率大于10%的有4个。在此基础上, 本实验室对该图谱进行了加密, 获得了1个包含830个SSR位点的高密度遗传连锁图[5]。本研究以该图谱为基础, 同时增加了2015年和2016年的表型数据, 对出仁率和株高进行了QTL定位, 以期分析出仁率与株高的关系, 发掘出在不同环境中稳定存在的主效QTL, 为分子标记辅助育种改良出仁率和株高性状奠定基础。
1 材料与方法
1.1 试验材料
以远杂9102为母本、徐州68-4为父本进行杂交, 采用单粒传法构建包含195个家系的重组自交系群体(RIL), 其中远杂9102株高较矮, 出仁率高, 徐州68-4株高中等, 出仁率低。
1.2 出仁率与株高的测定
于2013¾2016年在武汉中国农业科学院油料作物研究所试验田连续种植RIL群体的F5~F8代和亲本材料, 每份材料1行, 行长2.50 m, 行距0.30 m, 完全随机区组设计, 2次重复, 常规田间管理。收获时按家系测量株高, 晒干后, 选取每个家系成熟饱满干荚果, 剥壳后的仁重占荚果重量的百分率即为出仁率。采用Microsoft Excel软件统计与计算数据。采用SPSS软件进行方差分析。
1.3 QTL定位
利用RIL群体195个F5家系构建的包含20个连锁群、830个位点、总长度为1386.19 cM的遗传图谱[5]。结合表型数据, 采用QTLNetwork 2.0[12]软件对出仁率和株高进行QTL定位分析。按软件设置的默认参数, 以=0.05为统计检测阀值, 即当标记的值小于统计检测阈值时, 则认为该标记处存在1个与性状相关的QTL; 最后将检测到的所有QTL以及它们之间的上位性互作整合到一个全QTL模型中, 用基于Gibbs抽样的Bayesian方法估计遗传效应。用QGA Station 1.0[13]软件排除株高对出仁率的影响, 用QTLNetwork 2.0对出仁率进行条件QTL分析。以“q+性状的英文名称首字母+所在连锁群”命名QTL, 若同一连锁群上出现2个或以上QTL时, 则QTL后面加“.”和数字来区分。
2 结果与分析
2.1 RIL群体出仁率和株高的变异
从表1可以看出, 两亲本在出仁率和株高性状上均表现出显著差异, 4年结果一致。在RIL群体中, 2个性状的变异均超出了亲本间的差异, 最大值和最小值均具有超亲优势。通过在4个环境下的调查, 可以看出RIL群体的出仁率和株高性状均变异范围广且呈连续分布现象, 接近正态分布(图1和图2), 具数量性状的典型特征。出仁率的表型变异系数为3.88%~4.84% (表1), 广义遗传力为0.958 (表2); 株高的表型变异系数为12.03%~20.48% (表1), 广义遗传力为0.886 (表2)。相关性分析表明, 4个环境下出仁率和株高均存在极显著负相关, 相关性系数为0.388~0.532。
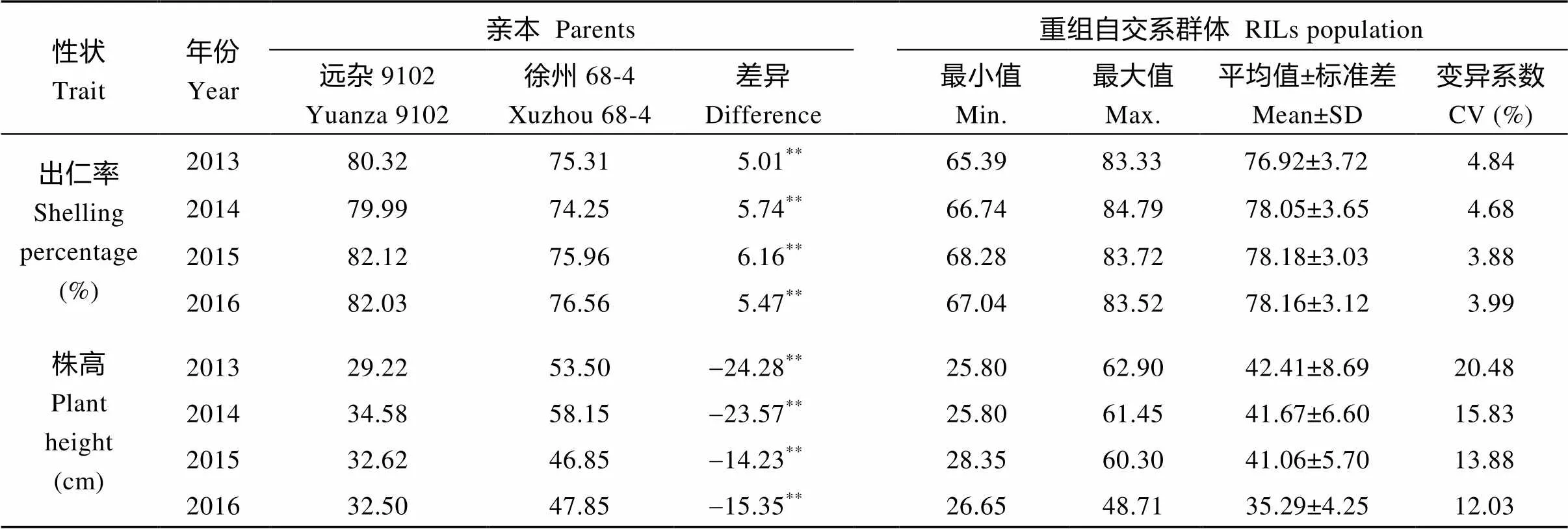
表1 亲本及RIL群体出仁率和株高
**表示两亲本在0.01概率水平上差异显著。
**Significant difference between parents at the 0.01 probability level.

图1 RIL群体出仁率性状在4个环境中的分布
虚线箭头为远杂9102, 实线箭头为徐州68-4。
Dotted arrow: Yuanza 9102; solid arrow: Xuzhou 68-4.
利用SPSS软件对RIL群体株高和出仁率性状4年间进行方差分析表明(表2), 除出仁率性状在基因型和环境间的互作效应差异不显著外, 株高和出仁率在个体间的差异、环境间差异以及株高性状在基因型和环境间的互作效应均表现出极显著差异, 说明群体内性状变异丰富, 环境对株高的影响较大。
2.2 出仁率相关QTL
结合本实验室前期通过该群体构建的遗传连锁图[5]和4年出仁率的表型值, 利用QTLNetwork 2.0软件在单一环境下及多环境下联合进行QTL定位分析, 共检测到13个与出仁率相关的加性QTL, 分布在A05、A09、B03、B01、B04、B05和B10染色体上(图3), 加性效应贡献率为1.47%~16.86%, 其中、和加性效应贡献率大于10%, 为主效QTL。3个QTL (、和)在3个以上环境中被重复检测到。6个QTL (、、、和)加性效应为正, 增效等位基因来自徐州68-4; 其余QTL加性效应为负, 增效等位基因来自远杂9102 (表3)。
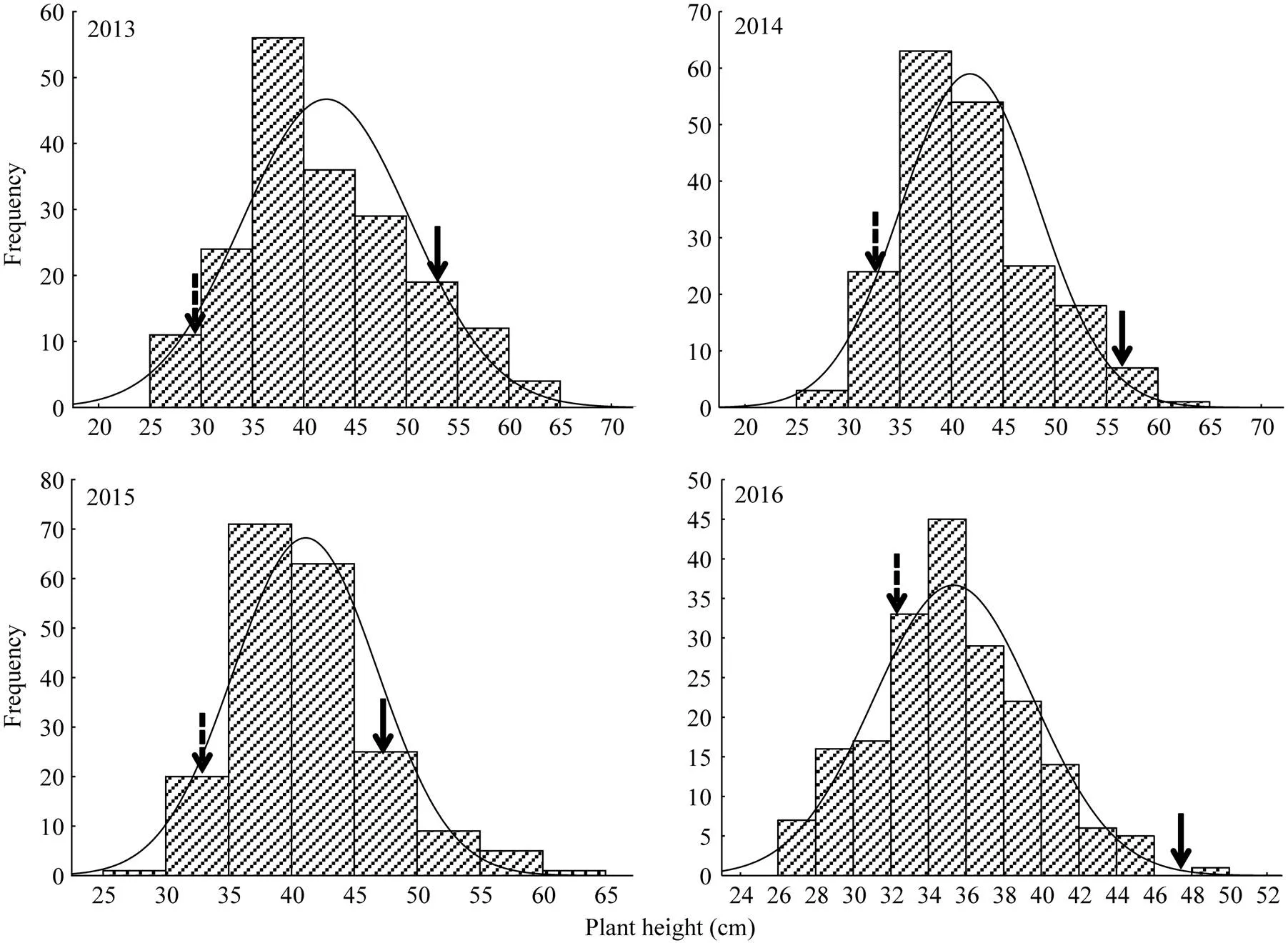
图2 RIL群体株高性状在4个环境中的分布
虚线箭头为远杂9102, 实线箭头为徐州68-4。
Dotted arrow: Yuanza 9102; solid arrow: Xuzhou 68-4.

表2 RIL群体出仁率和株高的方差分析
从表4可以看出, 在4个环境下联合(即ME环境)分析QTL与环境的互作效应,与环境的互作效应值为0, 说明该QTL不受环境的影响, 其余QTL均与环境间存在互作, 互作贡献率小于加性效应贡献率, 为0.02%~0.21%, 说明基因对出仁率的影响比环境的影响大。对于出仁率性状共检测到6对上位性QTL, 2对上位性效应值为负, 其他上位性效应为正, 贡献率为0.45%~3.72%。2对(/和/)互作QTL的上位性效应值分别为-0.6479和-0.4537, 表现出重组型大于亲本型, 与环境之间也存在互作效应, 互作贡献率为0.01%和0.02%。4对互作QTL (/、/、/和/)的上位性效应值分别为0.6695、0.2599、0.5093和0.5220, 表现出亲本型大于重组型, 贡献率分别为2.33%、0.45%、1.44%和1.56%, 同时也检测到与环境之间存在互作效应, 互作贡献率很低, 分别为0.08%、0.01%、0.05%和0.05%。

图3 部分加性QTL及上位性QTL在连锁群上的分布

表3 株高和出仁率性状的加性QTL及遗传效应
E1: 武汉2013; E2: 武汉2014; E3: 武汉2015; E4: 武汉2016; ME: 多环境。
E1: Wuhan 2013; E2: Wuhan 2014; E3: Wuhan 2015; E4: Wuhan 2016; ME: multi-environments.
2.3 株高相关QTL
利用QTLNetwork 2.0软件在单一环境下及多环境下联合进行QTL定位分析, 共检测到8个与株高相关的加性QTL, 分布在A05、A09、B03、B04和B08染色体上, 加性效应贡献率为0.51%~21.87%, 其中、、和的加性效应贡献率分别为14.19% (E3)、21.16% (E1)、12.15%和10.30%, 是主效QTL。加性效应值为负, 说明亲本远杂9102在上携带着增加株高的等位基因; 其余QTL的加性效应值均为正, 增效等位基因来源于株高较高的亲本徐州68-4 (表3)。在ME环境下检测到的5个QTL均与环境之间存在互作, 互作贡献率为0.31%~1.98%。其中的环境互作贡献率大于加性效应贡献率, 其余QTL的环境互作贡献率均小于加性效应贡献率, 说明基因对株高的影响比环境对其影响更显著。检测到5对上位性QTL, 3对互作QTL (/、/和/), 上位性效应值分别为-2.3216、-0.7828和-7.7112, 贡献率分别为3.13%、0.04%和0.13%, 上位性效应值均为负, 表现出重组型大于亲本型, 同时这3对QTL均检测到与环境之间存在互作效应, 环境互作贡献率为0.25%、0.05%和1.11%。2对(/和/)互作QTL上位性效应值为0.5921和0.6076, 亲本型大于重组型, 贡献率为1.06%和0.46%, 同时也检测到与环境之间存在互作效应, 环境互作贡献率分别为0.13%和0.18% (表4)。
2.4 出仁率QTL与株高QTL之间的关系
为了分析株高和出仁率的遗传关系, 使用条件表型值(出仁率|株高)进行条件QTL分析。从表5可以看出, 在ME环境下, 检测到8个与出仁率相关的QTL。以株高为给定条件, 检测到6个与出仁率相关的QTL, 其中、、和在条件和非条件中都检测到, 加性贡献率(1.26%~6.90%)比其相应的非条件QTL加性效应贡献率(4.09%~14.37%)变化大, 说明这4个QTL为部分通过株高变化进而影响出仁率的QTL, 并且同时作用于株高和出仁率2个性状;和是排除株高的效应下检测到的QTL, 说明这2个QTL受株高性状的影响, 由于其效应值较小, 排除株高性状的效应后, 才能检测到效应值较小的QTL。另外, 有4个QTL (、、和)没有被检测到, 说明这些QTL是通过影响株高的变化进而影响出仁率的。比较QTL在连锁群上的位置, 在A09染色体Ad91I24–AGGS2492区间存在控制出仁率相关的和控制株高相关的, 排除该位点株高性状的效应后, 出仁率QTL仍能被检测到, 而且加性效应贡献率从14.37%下降到5.5% (图3), 说明和为同一位点, 同时作用于株高和出仁率性状。
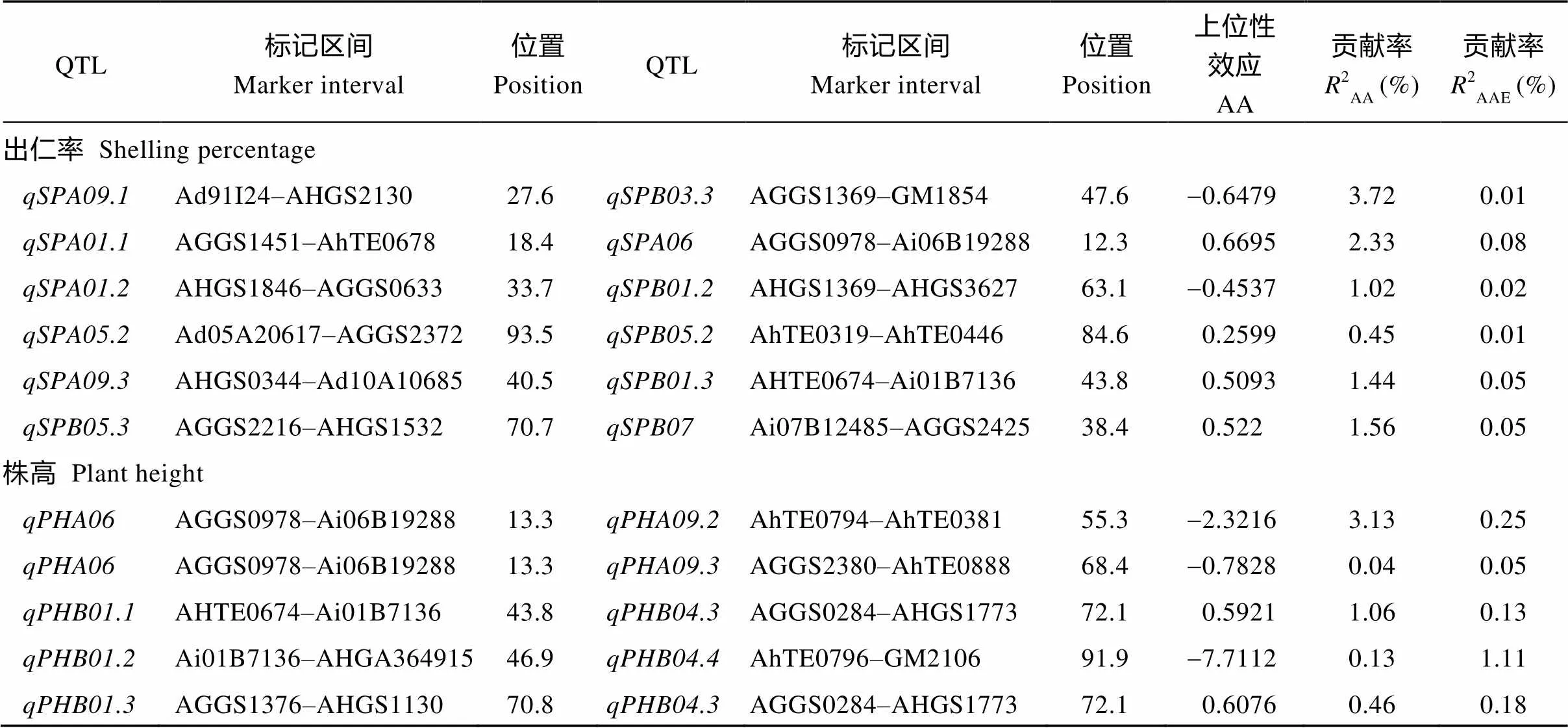
表4 RIL群体株高和出仁率性状的上位性QTL与环境互作效应

表5 出仁率的条件QTL效应
SP: shelling percentage; PH: plant height.

图4 A09染色体上同一区间内出仁率和株高QTL及LOD值
3 讨论
本研究选用的2个亲本材料(远杂9102和徐州68-4)在株高和出仁率性状方面差异显著, 远杂9102出仁率大于徐州68-4, 而株高小于徐州68-4。对杂交构建的RIL群体F5~F8代材料的出仁率和株高性状表型值分析表明, 出仁率和株高的变异均呈连续性分布, 都具有正向和负向超亲优势, 属典型的数量性状。株高变异幅度大, 并且受环境影响较大。本研究中, 出仁率和株高存在极显著负相关, 这与Faye等[6]、Huang等[7]的研究结果一致。Faye等[6]的研究中, 出仁率和株高性状间存在极显著负相关, 相关系数为–0.250; Huang等[7]报道出仁率与主茎高间也存在极显著负相关, 相关系数为0.462。

图5 重复检测到的QTL (3个或3个以上环境)在染色体上的分布
复杂性状的基因表达存在上位性作用, 单纯的定位单个加性QTL不能全面地反映出QTL 对性状调节的真实效应。因为大部分性状是由多个基因共同调节控制的, 并且其表达也受环境条件的影响。本研究表明, 对于出仁率和株高都能检测到上位性效应, 因此, 除了主效应外, 上位性也是数量性状重要的遗传基础。定位的同一性状QTL分布在不同染色体上, 且QTL 之间存在上位性互作效应和环境互作效应, 环境互作效应的存在也解释了数量性状易受环境影响的现象。本研究共检测到13个与出仁率相关的QTL, 分布在A05、A09、B03、B01、B04、B05和B10染色体上,控制出仁率的加性QTL 之间发生上位性效应, 共同调节出仁率, 与环境之间的互作贡献率很低, 仅为0.01%。
单个基因多效性或多个基因紧密连锁在一起共同控制某些性状是性状相关的遗传基础。本研究中, 与出仁率相关的和与株高相关的处于同一置信区间, 这2个QTL可能为同一QTL或紧密连锁(图3)。条件QTL分析则可以排除株高对出仁率的影响, 将控制出仁率的QTL解析为单独控制出仁率的基因、通过调控株高而影响出仁率的基因或者既控制株高又影响出仁率的基因。本研究排除该位点株高性状的效应后, 出仁率加性效应贡献率从14.37%下降到5.50%, 说明该位点同时作用于株高和出仁率性状, 为“一因多效”的QTL。因此, 条件QTL分析方法有助于更加深入地了解相互独立但又密切相关的性状间的遗传关系。
本研究共检测到21个加性QTL, 其中检测到8个与株高相关的QTL, 检测到13个与出仁率相关的QTLs, 每个性状都检测出正的和负的加性QTL, 这些QTL解释了群体中超亲分离现象。和对出仁率的贡献率较大, 可分别解释表型变异的10.06% (ME)和14.37% (ME), 增效等位基因来自于远杂9102。对株高的遗传贡献率最大, 可解释11.17%的表型变异, 其增效等位基因来自于徐州68-4。这3个QTL为主效QTL, 并且在多个环境中被检测到(图4), 为出仁率和株高的精细定位及分子标记辅助育种奠定了良好基础。对株高QTL的研究, 前人已有报道, 在染色体A03、A04、A06、A09、A10、B01、B03、B04、B06和B07上均检测到与株高相关的QTL, 除此之外, 本研究在A05染色体上也检测到与株高相关的QTL。Huang等[10]利用RIL群体在3个环境中共检测到18个QTLs, 解释4.85%~20.52%的表型变异, 其中位于A09染色体上的QTL与本研究检测到的都与标记AHGS2130连锁, 由此可看出, 本研究检测到的QTL是可靠的; Li等[7]利用RIL在6个环境中共检测到11个QTL, 解释表型变异的6.26%~ 22.53%; 刘华等[15]的研究中, 在2个环境中检测到10个QTL, 可解释表型变异的5.81%~18.00%, 这些QTL由于标记各异以及连锁群和染色体没有对应, 很难比较。在出仁率方面, 本研究检测到的13个QTL, 分布在A05、A09、B03、B04、B05和B10染色体上。与已报道的QTL相比较,与蔡岩等[11]检测到的一致, 均与标记AhTE0446连锁; 上位性QTL中的和蔡岩等[11]检测到的和Huang等[7]检测到的一致, 都与标记AHGS0344连锁, 说明该QTL是可靠的。周小静等[16]利用SNP标记构建的遗传图谱对出仁率进行了QTL定位, 由于其所用标记不同, 很难与本研究的标记比较。此外, 本研究还检测到了新的与出仁率和株高相关的QTL, 如在3个环境中重复检测到的出仁率QTL和, 在2个环境中重复检测到的株高QTL和等。本研究在7个连锁群上检测到影响出仁率的QTL, 揭示了该性状遗传基础的复杂性。QTL加性效应有正有负, 说明2个亲本均存在提高出仁率的等位基因, 通过QTL的聚合就能产生比双亲出仁率更高的家系。
4 结论
获得2个稳定与出仁率相关的主效QTL (和)和1个与株高相关的主效QTL(); 在A09染色体Ad91I24–AGGS2492区间同时存在出仁率和株高相关的稳定主效QTL。
[1] 李振动, 李新平, 黄莉, 任小平, 陈玉宁, 周小静, 廖伯寿, 姜慧芳. 栽培种花生荚果大小相关性状QTL定位. 作物学报, 2015, 41: 1313–1323 Li Z D, Li X P, Huang L, Ren X P, Chen Y N, Zhou X J, Liao B S, Jiang H F. Mapping of QTLs for pod size related traits in cultivated peanut (L.)., 2015, 41: 1313–1323 (in Chinese with English abstract)
[2] Chen W, Jiao Y, Cheng L, Huang L, Liao B, Tang M, Ren X, Zhou X, Chen Y, Jiang H. Quantitative trait locus analysis for pod- and kernel-related traits in the cultivated peanut (L.)., 2016, 17: 25
[3] Chen Y, Ren X, Zheng Y, Zhou X, Huang L, Yan L, Jiao Y, Chen W, Huang S, Wan L, Liao B, Huai D, Wei W, Jiang H. Genetic mapping of yield traits using RIL population derived from Fuchuan Dahuasheng and ICG6375 of peanut (L.)., 2017, 37: 17
[4] Luo H, Ren X, Li Z, Xu Z, Li X, Huang L, Zhou X, Chen Y, Chen W, Lei Y, Liao B, Pandey M, Varsheny R, Guo B, Jiang X, Liu F, Jiang H. Co-localization of major quantitative trait loci for pod size and weight to a 3.7 cM interval on chromosome A05 in cultivated peanut (L.)., 2017, 18: 58
[5] Luo H, Guo J, Ren X, Chen W, Huang L, Zhou X , Chen Y, Liu N, Xiong F, Lei Y, Liao B, Jiang H. Chromosomes A07 and A05 associated with stable and major QTLs for pod weight and size in cultivated peanut (L.)., 2017, 131: 267–282
[6] Faye I, Pandey M K, Hamidou F, Rothore A, Ndoye O, Vadez V, Varshney R K. Identification of quantitative trait loci for yield and yield related traits in groundnut (L.) under different water regimes in Niger and Senegal., 2015, 206: 631–647
[7] Huang L, He H, Chen W, Ren X, Chen Y, Zhou X, Xia Y, Wang X, Jiang X, Liao B, Jiang H. Quantitative trait locus analysis of agronomic and quality-related traits in cultivated peanut (L.)., 2015, 128: 1103–1115
[8] 成良强, 唐梅, 任小平, 黄莉, 陈伟刚, 李振动, 周小静, 陈玉宁, 廖伯寿, 姜慧芳. 栽培种花生遗传图谱的构建及主茎高和总分枝数QTL分析. 作物学报, 2015, 41: 979–987 Cheng L Q, Tang M, Ren X P, Huang L, Chen W G, Li Z D, Zhou X J, Chen Y N, Liao B S, Jiang H F. Construction of genetic map and QTL analysis for main stem height and total branch number in peanut (L.)., 2015, 41: 979–987 (in Chinese with English abstract)
[9] Li Y, Li L, Zhang X, Zhang K, Ma D, Liu J, Wang X, Liu F, Wan Y. QTL mapping and marker analysis of main stem height and the first lateral branch length in peanut (L.)., 2017, 213: 57
[10] Huang L, Ren X, Wu B, Li X, Chen W, Zhou X, Chen Y, Pandey M, Jiao Y, Luo H, Lei Y, Varsheny R, Liao B, Jiang H. Development and deployment of a high-density linkage map identified quantitative trait loci for plant height in peanut (L.)., 2016, 6: 39478
[11] 蔡岩, 徐志军, 李振动, 李新平, 郭建斌, 任小平, 黄莉, 陈伟刚, 陈玉宁, 周小静, 罗怀勇, 姜慧芳. 花生出仁率QTL分析及其与荚果大小的相关性. 作物学报, 2017, 43: 701–707 Cai Y, Xu Z J, Li Z D, Li X P, Guo J B, Ren X P, Huang L, Chen W G, Chen Y N, Zhou X J, Luo H Y, Jiang H F. Quantitative trait locus analysis for shelling percentage and correlation between shelling percentage and pod size related traits in., 2017, 43: 701–707 (in Chinese with English abstract)
[12] Yang J, Hu C, Hu H, Yu R, Xia Z, Ye X, Zhu J. QTLNetwork: mapping and visualizing genetic architecture of complex traits in experimental populations., 2008, 24: 721–723
[13] Zhu J. Analysis of conditional genetic effects and variance components in developmental genetics., 1995, 141: 1633
[14] Wang D, Zhu J, Li Z, Paterson A. Mapping QTLs with epistatic effects and QTL × environment interactions by mixed linear model approaches., 1999, 99: 1255–1264
[15] 刘华, 张新友, 韩锁义, 严玫, 徐静, 董文召, 孙子淇. 花生主茎高、侧枝长的遗传分析及QTL检测. 中国油料作物学报, 2013, 35: 508–514 Liu H, Zhang X Y, Han S Y, Yan M, Xu J, Dong W Z, Sun Z Q. Inheritance analysis and QTL mapping of main stem height and lateral branch length in peanut (L.)., 2013, 35: 508–514 (in Chinese with English abstract)
[16] 周小静, 董洋, 张芳, 任小平, 陈玉宁, 黄莉, 陈伟刚, 廖伯寿, 雷永, 晏立英, 罗怀勇, 姜慧芳. 利用SNP标记高密度遗传图谱进行花生出仁率QTL定位. 中国油料作物学报, 2016, 38: 750–756 Zhou X J, Dong Y, Zhang F, Ren X P, Chen Y N, Huang L, Chen W G, Liao B S, Luo H Y, Jiang H F. QTL mapping of shelling percentage using SNP-based high density genetic map in cultivated peanut., 2016, 38: 750–756 (in Chinese with English abstract)
QTL Mapping for Shelling Percentage and Plant Height in Cultivated Peanut (L.)
CHEN Wei-Gang**, GUO Jian-Bin**, XU Zhi-Jun, YU Bo-Lun, QIU Xi-Ke, HUANG Li, SONG Yan-Bin, CHEN Yu-Ning, ZHOU Xiao-Jing, LUO Huai-Yong, LIU Nian, REN Xiao-Ping, and JIANG Hui-Fang*
Oil Crops Research Institute, China Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture, Wuhan 430062, Hubei, China
Peanut yield is greatly influenced by shelling percentage (SP) and plant height (PH). Marker-assisted selection for shelling percentage and plant height improvement can be facilitated by detecting additive and epistatic QTLs, understanding their interactions with environment, as well as the genetic relationship between SP and PH. In a four-year experiment, the variations of SP and PH were evaluated with the recombinant inbred line population derived from the cross between Yuanza 9102 and Xuzhou 68-4. QTLs associated with SP and PH were analyzed using a high density linkage map and QTLNetwork 2.0 software. The results showed that SP and PH were negatively correlated (< 0.01). Thirteen and eight additive QTLs were identified for SP and PH, respectively. Major lociandfor SP and major locusfor PH were repeatedly detected in three or four years. Six pairs of epistatic QTLs for SP and five pairs of epistatic QTLs for PH were identified, and they all had interaction effects with environment.andwere located in the same interval (Ad91I24–AGGS2492) on chromosome A09. The additive effect of this locus for SP variation decreased from 14.37% to 5.50% after eliminating the PH effect, suggesting the pleiotropism offor SP andfor PH.
cultivated peanut; shelling percentage; plant height; QTL
10.3724/SP.J.1006.2018.01142
姜慧芳, E-mail: peanutlab@oilcrops.cn, Tel: 027-86711550
**同等贡献(Contributed equally to this work)
陈伟刚, E-mail:wgchen2015@163.com; 郭建斌, E-mail: guojianbin1990@163.com
2017-12-20;
2018-03-26;
2018-04-20.
本研究由国家自然科学基金项目(31471534, 31571713), 农业部农作物种质资源保护项目(2017NWB033), 国家农作物种质资源共享服务平台(NICGR2017-36)和国家现代农业产业技术体系建设专项(CARS-13-花生种质资源评价)资助。
This study was supported by the National Natural Science Foundation of China (31471534, 31571713), the Crop Germplasm Resources Protection Project (2017NWB033), the Plant Germplasm Resources Sharing Platform (NICGR2017-36), and the China Agriculture Research System (CARS-13-Germplasm Resource Evaluation for Peanut).
URL:http://kns.cnki.net/kcms/detail/11.1809.s.20180419.1321.002.html

