疱疹病毒的示踪技术:看到了什么?还能看到什么?
王亚林,仇华吉,孙元
疱疹病毒的示踪技术:看到了什么?还能看到什么?
王亚林,仇华吉,孙元
中国农业科学院哈尔滨兽医研究所 兽医生物技术国家重点实验室,黑龙江 哈尔滨 150069

王亚林, 仇华吉, 孙元. 疱疹病毒的示踪技术:看到了什么?还能看到什么?生物工程学报, 2018, 34(11): 1721–1733.Wang YL, Qiu HJ, Sun Y. Tracking of herpesviruses: what have been seen and will be seen? Chin J Biotech, 2018, 34(11): 1721–1733.
病毒感染宿主细胞是一个非常复杂的过程,涉及病毒与多种宿主成分的相互作用。目前通过可视化病毒示踪技术可对病毒复制周期的各个过程进行实时、原位观察。疱疹病毒是一类有囊膜的DNA病毒,在自然界中广泛存在,对人类和动物健康构成严重威胁。文中综述了病毒示踪技术在疱疹病毒研究中的应用。这些研究为认识疱疹病毒的感染、复制机制以及病毒与宿主相互作用的过程开拓了新的视野。但该技术还不是十分完善,包括标记物最佳插入位点的选择、无法示踪病毒生命周期的全过程等。相信随着相关技术的发展与进步,会实现对疱疹病毒复制周期更加详细地追踪,从而更为详尽地揭示其复制机制。
病毒示踪技术,疱疹病毒,荧光蛋白,复制,潜伏感染,轴突转运
病毒是在普通光学显微镜下不可见的依赖活细胞寄生的微生物。病毒广泛存在,可以感染自然界已知的所有生命体。许多病毒能造成人类的严重疾病,如近年来的SARS-CoV、H5N1亚型高致病性禽流感病毒、H1N1甲型流感病毒等曾大规模流行,造成全球恐慌。因此,人类必须加强病毒学研究,寻找攻克病毒病的方法,这就必须深入探究病毒感染宿主的详细机制。传统的研究方法无法直观地研究病毒在活细胞内的感染行为,但是在给病毒标记上荧光标签之后,就能使病毒的感染过程可视化,即病毒示踪技术。
示踪技术是利用放射性或非放射性的标记物在体内或体外跟踪其行径、转变和代谢等过程的技术。而病毒示踪技术,即利用标记物标记病毒,从而追踪其在细胞或宿主体内的复制、扩散等生命活动的示踪技术。而这一技术更是得益于绿色荧光蛋白 (Green fluorescent protein,GFP) 的发现和应用。随着基因工程技术的发展和其他荧光蛋白的发现与改造,科学家们研发了多种新型的病毒示踪策略,为生物医学研究作出了巨大贡献。
病毒示踪技术实现了病毒感染的可视化,即“看得见”的病毒感染。这使得人类对病毒感染的微观世界有了更加直观的认识。病毒感染宿主细胞是一个非常复杂的过程,包含黏附、入侵、复制、包装以及释放等多个步骤,而且还涉及病毒与细胞内多种组分的复杂相互作用。传统的技术无法直观地研究活细胞中的病毒感染过程。而病毒示踪技术能够实时、原位地研究病毒在活细胞内的感染行为[1-2]。
1 疱疹病毒的示踪技术
疱疹病毒是一类有囊膜的DNA病毒,分为α疱疹病毒、β疱疹病毒、γ疱疹病毒以及其他疱疹病毒。其中有代表性的α疱疹病毒包括能够感染人类的单纯疱疹病毒 (Herpes simplex virus type 1/2,HSV-1/2)、水痘带状疱疹病毒 (Varicella-zoster virus,VZV) 以及感染动物的伪狂犬病病毒 (Pseudorabies virus,PRV)、鸡马立克氏病病毒 (Marek’s disease virus,MDV) 等。β疱疹病毒主要有人类的巨细胞病毒 (Human cytomegalovirus,HCMV)[3]。病毒示踪技术很早就已经应用于疱疹病毒的研究当中,随着示踪技术的发展和高敏感度荧光蛋白的衍生,多种荧光蛋白已经用于标记疱疹病毒,从而研究疱疹病毒的复制周期及其与宿主细胞的相互作用。
病毒示踪剂的标记方法繁多,主要包括基因工程技术标记、化学反应法标记以及细胞结构标记等[1,4-5]。相应的病毒示踪剂包括荧光蛋白、肽/蛋白标签、有机染料、量子点以及荧光素酶等,这些标记方法和示踪剂同样适用于疱疹病毒的示踪。
自20世纪60年代初发现GFP以来,荧光蛋白在生命科学和医学研究中获得了广泛应用。研究者利用基因工程技术构建了一系列能够表达荧光蛋白的生物有机体,包括病毒、细菌、酵母、寄生虫以及转基因动植物等,然后通过光学成像技术可以直接观察到从微观到宏观各个层次上丰富多彩的生命现象[6]。肽/蛋白标签多为人工改造的多肽或酶,其本身不具有荧光特性,但可通过与荧光标记的特异性识别分子或底物之间的化学反应实现对肽/蛋白标签目的蛋白的标记[7-8]。这一类的病毒示踪剂有HaloTag蛋白标签以及利用脊髓灰质炎病毒等肠道病毒非结构蛋白2C构建的一系列Flag-2C肽标签等[2,9]。有机荧光染料大多是含有共轭双键、苯环的小分子化合物,具有分子量小、种类繁多等特点,现已广泛应用于荧光成像领域。如利用Cy5染料标记腺病毒衣壳,阐明了其感染机制[10]。DiO是一种脂质染料,可标记病毒包膜,通过观测荧光强度的变化研究病毒的感染过程[11]。量子点 (Quantum dot,QD) 是一种新型纳米材料,具有荧光强度高、稳定性好等优点,现已广泛用于活细胞标记、免疫标记及活体成像等研究领域。Li等通过共价结合将量子点嵌入人类免疫缺陷病毒1型 (Human immunodeficiency virus type 1,HIV-1) 的衣壳内,利用单颗粒示踪技术揭示了HIV-1是通过内吞的方式入侵巨噬细胞的[1]。荧光素酶是自然界中能够产生生物荧光的酶的总称,通过与相应底物发生氧化反应而产生生物冷光[12],包括最常用的萤火虫荧光素酶 (Firefly luciferase)、海肾荧光素酶 (luciferase)、荧光素酶以及NanoLuc荧光素酶等[12-15]。利用荧光素酶标记的病毒感染动物或细胞后,可通过注射或加入底物的方式实现对病毒感染的体内外示踪,已在病毒感染的相关研究中获得了广泛应用[16-18]。图1展示了荧光标记物在示踪技术中的应用。
基因工程标记法是疱疹病毒示踪技术中常用的标记方法之一,具有稳定性高、定位准确、可缩短曝光时间等优势。利用荧光蛋白标记疱疹病毒时,即使标记物和病毒蛋白的详细结构是已知的,我们也无法预测其融合蛋白的精确结构。因此,构建一株功能完善的表达荧光蛋白的重组病毒仍然是一个充满不确定性、需要反复试验的过程。疱疹病毒在结构上具有一定的共性,这使得荧光蛋白在疱疹病毒基因组中的插入位点具有普适性。例如UL35基因是荧光蛋白的常用插入位点,曾有文献报道在HSV-1、PRV的该位点插入荧光蛋白的实例[4,23]。图2展示了本研究团队在PRV不同位点插入红色和绿色荧光蛋白的重组病毒感染小鼠背根神经节 (Dorsal root ganglions,DRGs) 中的神经元和猪肾上皮细胞 (PK-15) 后荧光蛋白的表达情况。表1列出了一些目前报道的疱疹病毒中荧光蛋白的插入位点。
2 病毒示踪技术在疱疹病毒研究中的应用
可视化病毒示踪技术因其方便、快速并能实时定位病毒感染等优点而获得了广泛的应用,在疱疹病毒的研究中也不例外。此外,α疱疹病毒具有嗜神经性和在神经细胞中传导的特性。因此,可视化病毒示踪技术也促进了神经解剖学和病毒神经传导方面的研究。

图1 不同的荧光标记物在生物示踪技术中的应用 (改编自参考文献[19–22])

图2 绿色或红色荧光蛋白标记的PRV感染神经元和PK-15细胞后的荧光蛋白表达情况
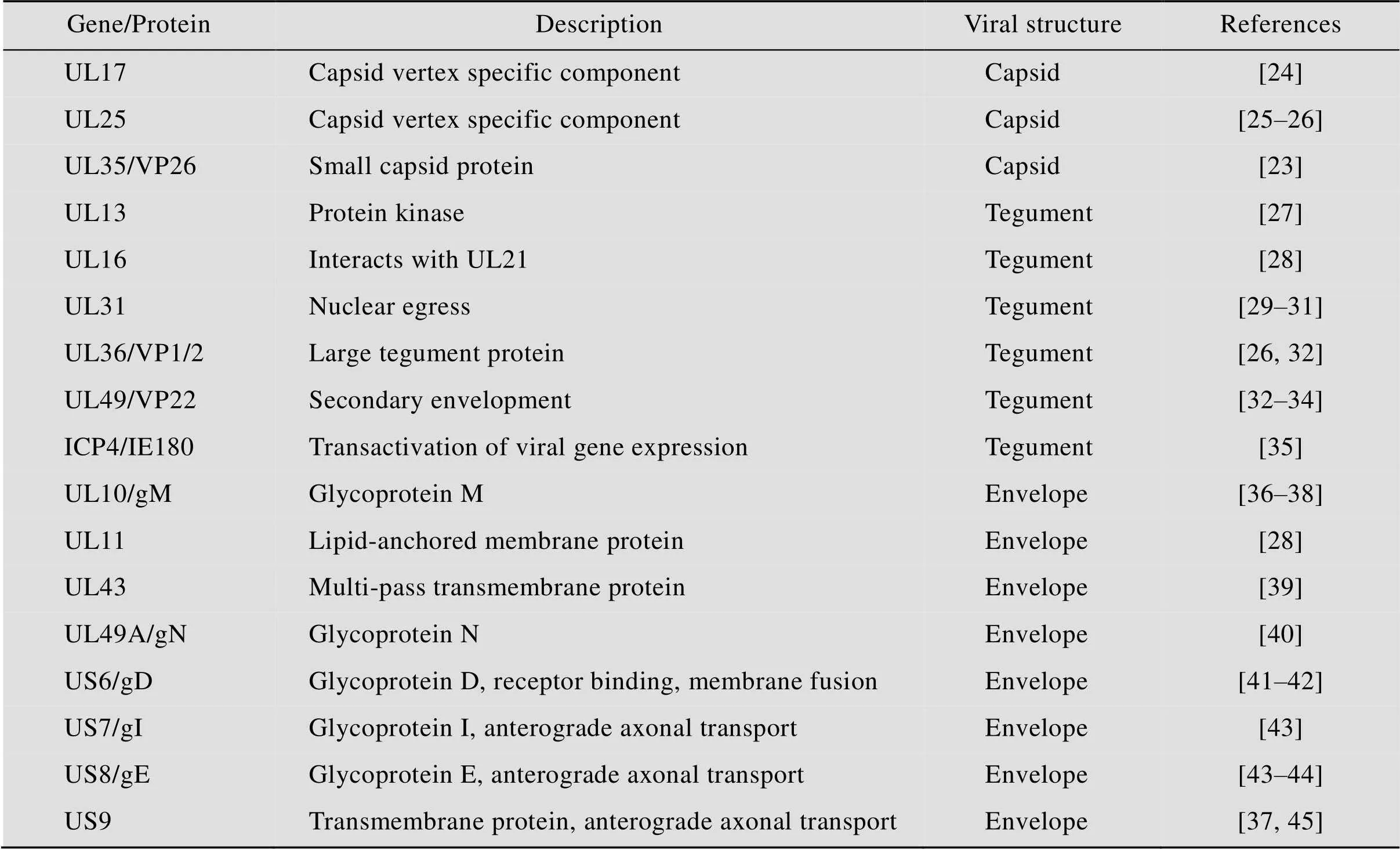
表1 荧光蛋白与疱疹病毒结构蛋白融合表达的实例 (改编自参考文献[6])
2.1 在病毒入侵、复制和释放相关研究中的应用
Liu等利用转座的原理将HaloTag蛋白标签成功插入到PRV基因组中,使HaloTag与VP26融合表达,产生重组病毒rPRV-HT。病毒感染细胞之后,可以通过不同的HaloTag配体使重组病毒产生不同颜色的荧光,从而区别亲本病毒和子代病毒,实现对病毒黏附、衣壳入侵以及子代病毒释放的可视化。结合单颗粒示踪技术,最终揭示了PRV生命周期的机制,获得了病毒入侵和复制过程的全局图像数据[2]。
疱疹病毒在宿主细胞的核内复制,最终形成相似的病毒粒子结构。病毒基因组由核孔入核,首先表达IE蛋白 (Immediate-early proteins),从而调控E蛋白 (Early proteins) 的表达,最终起始病毒基因组在复制区室 (Replication compartments,RCs) 的复制[46]。通过荧光蛋白标记可以实现病毒复制组装中间体形成过程的可视化,进而揭示病毒复制和组装的详细过程[47]。在病毒入侵的每个细胞中,仅有限数量的基因组获得表达,而且只有这些基因组进行复制并包装成病毒粒子[48]。Kobiler等利用分别与PRV VP26融合表达红色荧光蛋白 (Red fluorescent protein,RFP)、增强型绿色荧光蛋白 (Enhanced green fluorescent protein,EGFP)、蓝绿色荧光蛋白 (Cyan fluorescent protein,CFP) 的3株重组病毒研究发现,每个RC起源于单个病毒基因组,且每个细胞中仅有少于7个的病毒基因组可以进行复制[48]。
新合成病毒粒子的转运和释放是一个高度动态的过程,涉及病毒组分与宿主膜转运系统的协同作用。衣壳和病毒基因组在细胞核内组装之后,通过出芽方式穿过内外核膜离开细胞核[49]。分泌途径中产生的病毒膜蛋白被转运至二次包膜 (Secondary envelopment) 部位,目前认为这种包膜是转运高尔基体膜和/或内体膜[50-51]。病毒颗粒通过出芽的方式获得其囊膜,在细胞内囊泡中产生有囊膜的病毒粒子。之后这种含病毒粒子的转运囊泡被运输至细胞质膜,病毒粒子通过胞吐方式离开感染的细胞[36]。关于病毒释放过程的这些描述虽然被广泛接受,但涉及具体机制的相关研究还不够充分。Hogue等利用pH敏感型的GFP与病毒gM蛋白融合表达的重组PRV毒株,通过单颗粒示踪技术实现了对病毒释放过程的可视化[36]。
Scherer等利用一株能够稳定表达PRV mNG-VP26融合蛋白的PK-15细胞系和能够表达VP26-RFP融合蛋白的重组PRV构建了一个双荧光报告系统,利用这个系统构建的重组病毒命名为PRV180G,该重组病毒的衣壳能同时表达绿色和红色荧光蛋白。重组病毒在神经元细胞中感染并复制之后,所有的PRV180G子代病毒都仅含有mRFP-VP26标记的衣壳。Scherer等利用PRV180G来分析外周神经元轴突中PRV衣壳的转运动力学发现,在病毒入侵时,胞浆动力蛋白 (Dynein) 和驱动蛋白 (Kinesin) 都可介导衣壳的双向转运,而且转运活动比较活跃,而在子代病毒释放时,衣壳的转运仅由驱动蛋白介导[5]。
2.2 在病毒潜伏感染研究中的应用
疱疹病毒能在其宿主的特定细胞内建立并维持长期的潜伏感染[52]。潜伏感染的特征是仅表达有限数量的转录本——潜伏相关转录本 (Latency- associated transcripts,LATs),且不会合成子代病毒[53-55]。潜伏感染的建立、维持和再激活需要多种病毒和细胞蛋白的协同作用。
Koyuncu等利用表达单体红色荧光蛋白的PRV对神经元的感染模式进行了研究。利用经元三室神培养系统将神经元胞体和轴突在分隔的不同区室中培养。以不同感染复数 (Multiplicity of infection,MOI) 的重组病毒感染轴突区室时发现,当MOI低于0.1时,传导至神经核的病毒基因组是“静止”的,即建立了潜伏感染,而且这些“静止”的基因组可以被复制缺陷型的PRV再激活。最终结果表明感染剂量的不同和感染环境的多样性对于神经元中的感染状态 (复制或潜伏感染) 具有决定性的影响[56]。
α疱疹病毒潜伏感染时,病毒基因组仅限于表达LATs。HSV-1的全长LAT长约8.3 kb,可被剪接为稳定的1.5 kb和2 kb的内含子,以及6.3 kb的LAT外显子,6.3 kb的外显子最终被加工成一系列microRNAs[57]。已经证实LATs和相关的microRNAs能够在体外限制HSV-1 IE基因 (Immediate-early gene) 的表达[58-59],并能限制小鼠模型中病毒复制相关基因的转录[60-61]。Michael等利用一系列表达GFP和萤火虫荧光素酶 (Firefly luciferase) 的重组HSV-1毒株,结合体内和体外试验证明,LATs可以抑制病毒复制相关基因的表达,并能抑制神经元中潜伏感染的病毒基因组的再激活[57]。
HCMV在人群中的感染率极高,可达90%以上,并可对免疫缺陷患者造成严重的后果[62]。目前已在不同类型细胞上建立了HCMV潜伏感染模型,包括造血干细胞、粒细胞、巨噬细胞等,但神经细胞潜伏感染模型鲜有报道[63-64]。Cheng等利用一株表达GFP的HCMV作为指示病毒,成功在恶性胶质瘤细胞T98G细胞系中建立了潜伏感染模型,为研究HCMV引起的神经退行性后遗症奠定了基础[65]。
2.3 在病毒神经传导研究中的应用
很多病毒可以感染神经细胞,但只有少数病毒可以跨突触传播:如水泡性口炎病毒 (Vesicular stomatitis virus,VSV)、狂犬病病毒 (Rabies virus,RV) 以及α疱疹病毒。神经传导需要病毒入侵神经元 (第一级神经元) 并进行复制,然后将包装的病毒基因组在突触连接部位或其附近转运到第二级神经元进行再次复制。这种自我扩增的特性使得第一级、第二级和第三级神经元都能被标记[66]。图3是疱疹病毒在神经轴突中逆行传导后建立潜伏感染的示意图[56]。
PRV感染神经元需要病毒粒子沿着长距离轴突的双向运输[3,67]。病毒入侵外周神经元的轴突末端,并逆行运输至神经元胞体而建立潜伏感染。潜伏状态的病毒被再激活时,新合成的病毒粒子沿着轴突顺行传导至外周[3]。PRV高度保守的US9蛋白是病毒在体内外顺行传导所必需的[34]。在通过轴突运输之前,PRV核衣壳与内膜蛋白形成的病毒粒子前体通过二次包膜过程纳入由转运高尔基体衍生出的囊泡中。这一过程产生的病毒颗粒同时含有病毒和宿主的膜蛋白[3]。Taylor等利用US9-GFP融合表达的重组PRV感染大鼠颈上神经节中的原代神经细胞,通过活细胞的实时成像证实,US9-GFP能够与在轴突中顺行传导的病毒颗粒共转运[37]。若缺失US9,病毒颗粒可以在神经元胞体中组装,但不能在轴突中运输[68]。综合这些研究结果,说明US9是利用一些未知的病毒或宿主蛋白介导神经元内病毒粒子转运的。Kramer等利用质谱法鉴定了与US9-GFP相互作用的蛋白,发现KIF1A能与US9-GFP相互作用。KIF1A是宿主的一种微管依赖性驱动蛋白 (Kinesin),在轴突其他物质的转运中具有重要的作用[69-70]。KIF1A与病毒颗粒共转运,且对KIF1A的显性位点失活突变后可干扰病毒颗粒的顺行传导[45]。这些研究结果表明,PRV能够有效利用宿主神经元内轴突的运输机制在宿主体内进行传播。

图3 α疱疹病毒扩散到外周神经系统并建立潜伏感染的示意图 (改编自参考文献[56])
2.4 在神经通路示踪研究中的应用
疱疹病毒在神经通路示踪方面的应用主要包括探究外周神经系统与中枢神经系统间的联系通路和CNS内局部环路两方面。此外,疱疹病毒作为神经通路示踪剂的优势是其能够在神经环路中复制,因此可以放大示踪信号,增加示踪的灵敏度。PRV通常作为逆向的神经示踪剂揭示传入神经通路[71]。Krout等利用GFP与β半乳糖苷酶 (β-gal) 标记的PRV对中枢神经的神经元的功能进行了研究。将GFP-PRV注射入星状神经节,将β-gal-PRV注射入初级运动皮层,通过鉴定双标记的神经元,揭示了中枢神经元可能支配的神经通路[72]。而HSV-1通常作为顺行的神经环路示踪剂揭示传出神经网络[73-74]。Zeng等利用细菌人工染色体 (BAC) 技术构建了能够同时表达4个GFP分子的HSV-1 H129株,高荧光强度使其能够有效标记神经元并对其详细形态学实现可视化,包括对树突、棘突和轴突纤维的可视化。将基因敲除后,可实现对单个突触的示踪,通过对神经环路细节的可视化以及对神经网络的标记,有助于提升我们对中枢神经系统内神经网络的认识,也为研究神经退行性脑疾病提供了参考和借鉴意义[75]。
2.5 PRV报告病毒在示踪技术中的系统应用
国际著名疱疹病毒学家Enquist的团队在近20年的研究中,以PRV为模型,构建了一系列表达不同荧光蛋白的重组报告病毒,并且利用这些报告病毒对PRV感染宿主的过程进行了示踪,进而揭示了病毒的复制、细胞内转运以及释放等过程的详细机制,同时阐明了α疱疹病毒潜伏感染、在神经轴突中传导的相关机制以及宿主神经通路的结构[45,56,67,76]。他们在病毒基因组的不同位点 (gM、gG、US9、UL35等) 插入不同的荧光蛋白 (GFP、YFP、RFP、mCherry、mNeonGreen等) 构建了一系列示踪病毒,为人类疱疹病毒示踪的相关研究提供了重要参考[4,36,77]。
Enquist等以PRV Bartha株为骨架,利用Cre-Lox重组系统获得的PRV-267和PRV-263,在感染同一个神经元细胞时,由于发生不同的重组而产生不同的报告子表型,从而产生表达不同荧光的病毒,利用这两株病毒对大鼠脑干髓质区神经回路进行了分析[76]。他们也分别以PRV Becker和PRV 180 (RFP与病毒小衣壳蛋白VP26融合表达) 为骨架,在囊膜蛋白gM之后插入pH敏感型的绿色荧光蛋白pHluorin,获得重组病毒PRV 486和PRV 483,利用这一系列报告病毒并基于相关研究基础,Enquist等建立了α疱疹病毒在细胞内转运以及由胞吐作用介导的释放的完整模型[36],并阐明了病毒颗粒、L-颗粒 (仅含有衣壳、外膜蛋白或囊膜蛋白,无病毒核酸) 以及糖蛋白的分泌机制[78]。
2.6 双荧光标记在疱疹病毒示踪技术中的应用
与单独的荧光蛋白标记相比,双色荧光蛋白标记在研究病毒感染机制以及病毒与宿主相互作用中更具优势。可以通过同时标记病毒衣壳、外膜或囊膜实现对病毒粒子的双色标记[79];也可通过标记病毒粒子,利用荧光补充型细胞系实现双色标记[5];此外,还可以利用示踪剂与不同配体的相互作用实现双色甚至多色标记[2]。
Enquist等利用双色标记的PRV实现了对疱疹病毒在轴突内转运动态的追踪,阐明了不同的运输蛋白对亲代和子代病毒粒子的转运机制[5]。Kerstin等分别利用EGFP和mCherry标记了HCMV的内膜蛋白pp150和囊膜蛋白gM,实现了对HCMV感染的双色示踪,揭示了其入侵机制[79]。
图4系统展示了病毒示踪技术在疱疹病毒相关研究中的应用。

图4 病毒示踪技术在疱疹病毒研究中的应用 (改编自参考文献[2,56])
3 疱疹病毒示踪技术的发展前景
自病毒示踪技术应用到疱疹病毒的研究中以来,一个个难题得以攻克。然而,关于病毒生命周期、病毒与宿主相互作用等方面依然存在很多未阐明的机制。随着科学技术的发展、更加灵敏的新的荧光蛋白的合成、高效监测仪器的发明,相信我们未来依然会利用其解决更多的科学问题。
例如,Kobiler等利用表达RFP、EGFP、CFP三种荧光蛋白的重组PRV研究发现,疱疹病毒的RC起源于单个病毒基因组,且每个细胞中仅有极少数的病毒基因组可以启动复制[48]。但他们同时也发现部分RC来源于2个病毒基因组,将这种RC称为“双基因组RC”。对于这一现象有3种解释:一是发生了病毒的重组;二是这种“双基因组RC”事实上来源于单个基因组,只是在一个基因组复制的过程中,邻近的基因组与其复制不同步,从而只检测到一个RC;第三种可能性是“双基因组RC”可能是独立形成的两个RC的重叠[80]。因此,假设利用病毒示踪技术对单个基因组进行标记,然后持续监测到其形成RC,这样的实时活细胞可视化模型可以解决这一问题,从而对RC的形成过程进行可视化的监测。
PRV囊膜蛋白US9通过募集驱动蛋白KIF1A参与顺行神经传导[45]。但是,KIF1A与US9相互作用还需要其他未知病毒蛋白的协助,如果可以将多种病毒蛋白、宿主成分利用不同的荧光标记物同时标记,就可以将这一过程进行更加细致的可视化,从而揭示PRV在轴突传导过程的详细机制,不仅如此,这样的研究方法同样适用于其他α疱疹病毒的神经传导研究。
病毒学家也已经开始利用超高分辨率显微镜进行相关的研究[49,81-82],这非常有助于对疱疹病毒内膜层网状蛋白的观测以及对病毒相关的胞内结构的鉴定,如组装中间体 (Assemblons) 、RC等。目前已利用超高分辨率技术测量了PRV颗粒中结构蛋白的不对称分布以及HSV-1颗粒中内膜蛋白的径向分布[83-84]。未来需要对超高分辨率技术的速度和灵敏度进行优化,以实现对病毒复制周期的超高分辨率的观测。
4 结语与展望
可视化病毒示踪技术应用于疱疹病毒的相关研究,成功地揭示了疱疹病毒入侵、复制、胞内转运、组装、释放以及潜伏感染等生命活动的详细机制,也促进了神经解剖学和病毒神经传导方面的研究。随着病毒示踪技术所需的荧光标记物、标记策略和监测仪器的不断发展和完善,可视化的病毒示踪技术将更加方便、快捷、经济、有效地用于疱疹病毒的相关研究。
目前的示踪技术尚不完善,虽然可以实现对于多种病毒蛋白的可视化标记,但对病毒核酸的标记仍旧是一大难题;荧光原位杂交技术虽然可以实现对DNA或RNA的原位标记,特别是近年来报道的RNAscope技术[85],但该技术需要对组织进行固定和染色,即无法实现对病毒核酸在宿主细胞内一系列生命活动的实时追踪,期待未来在病毒核酸标记方面能有所突破;将荧光蛋白插入病毒基因组的基因工程技术具有稳定性好、荧光蛋白表达效率高等优点,但也存在使病毒感染能力减弱的问题,此外还存在荧光蛋白在细胞内形成多聚体的报道,对病毒的生命活动具有不良的影响[23]。因此,探索更加适宜插入荧光蛋白的位点或更加优良的标记方法依然是待解决的技术难题。有机染料和肽类标记物虽然对病毒的感染能力无削弱,但是存在结合不牢的缺点,跟荧光蛋白相比标记能力差。未来需要探寻分子量更小的荧光蛋白以及结合性更强、更特异的染料和肽类标记物。
利用示踪剂实现对疱疹病毒单个生命过程的研究有很多,但是对两个或多个相关生命过程的同步示踪还鲜有报道,这可能也是未来需要突破的难点。
此外,今后需要研发更加经济实惠、荧光强度高、对病毒活性影响小的标记物;仍需不断探索更加简便、普适性的标记策略;仍需不断研发更加灵敏的监测仪器。如何使病毒示踪技术与生物技术手段更加高效地结合应用,突破各领域之间的界限,一直是研究者亟待解决的难题。
[1] Li Q, Li W, Yin W, et al. Single-particle tracking of human immunodeficiency virus type 1 productive entry into human primary macrophages. ACS Nano, 2017, 11(4): 3890–3903.
[2] Liu AA, Zhang ZF, Sun EZ, et al. Simultaneous visualization of parental and progeny viruses by a capsid-specific HaloTag labeling strategy. ACS Nano, 2016, 10(1): 1147–1155.
[3] Smith G. Herpesvirus transport to the nervous system and back again. Annu Rev Microbiol, 2012, 66(1): 153–176.
[4] Hogue IB, Jean J, Esteves AD, et al. A functional carboxy-terminal fluorescent protein fusion to pseudorabies virus small capsid protein VP26. J Virol, 2017, 92(1): e01193–17, doi: 10.1128/jvi.01193–17.
[5] Scherer J, Yaffe ZA, Vershinin M, et al. Dual-color herpesvirus capsids discriminate inoculum from progeny and reveal axonal transport dynamics. J Virol, 2016, 90(21): 9997–10006, doi: 10.1128/jvi.01122–16.
[6] Hogue IB, Bosse JB, Engel EA, et al. Fluorescent protein approaches in alpha herpesvirus research. Viruses, 2015, 7(11): 5933–5961.
[7] Wombacher R, Cornish VW. Chemical tags: applications in live cell fluorescence imaging. J Biophotonics, 2011, 4(6): 391–402.
[8] Tsien RY. Building and breeding molecules to spy on cells and tumors. FEBS Lett, 2005, 579(4): 927–932.
[9] Li Q, Zheng ZH, Liu Y, et al. 2C proteins of enteroviruses suppress IKKβ phosphorylation by recruiting protein phosphatase. J Virol, 2016, 90(10): 5141–5151.
[10] Seisenberger G, Ried MU, Endreß T, et al. Real-time single-molecule imaging of the infection pathway of an adeno-associated virus. Science, 2001, 294(5548): 1929–1932.
[11] Chen C, Zhuang XW. Epsin 1 is a cargo-specific adaptor for the clathrin-mediated endocytosis of the influenza virus. Proc Natl Acad Sci USA, 2008, 105(33): 11790–11795.
[12] Luker GD, Bardill JP, Prior JL, et al. Noninvasive bioluminescence imaging of herpes simplex virus type 1 infection and therapy in living mice. J Virol, 2002, 76(23): 12149–12161.
[13] Pan WQ, Dong ZY, Li F, et al. Visualizing influenza virus infection in living mice. Nat Commun, 2013, 4: 2369.
[14] Karlsson EA, Meliopoulos VA, Savage C, et al. Visualizing real-time influenza virus infection, transmission and protection in ferrets. Nat Commun, 2015, 6: 6378.
[15] England CG, Ehlerding EB, Cai WB. NanoLuc: a small luciferase is brightening up the field of bioluminescence. Bioconjug Chem, 2016, 27(5): 1175–1187.
[16] Cook SH, Griffin DE. Luciferase imaging of a neurotropic viral infection in intact animals. J Virol, 2003, 77(9): 5333–5338.
[17] Mondotte JA, Lozach PY, Amara A, et al. Essential role of dengue virus envelope protein N glycosylation at asparagine-67 during viral propagation. J Virol, 2007, 81(13): 7136–7148.
[18] Schoggins JW, Dorner M, Feulner M, et al. Dengue reporter viruses reveal viral dynamics in interferon receptor-deficient mice and sensitivity to interferon effectors. Proc Natl Acad Sci USA, 2012, 109(36): 14610–14615.
[19] Finke S, Brzózka K, Conzelmann KK. Tracking fluorescence-labeled rabies virus: enhanced green fluorescent protein-tagged phosphoprotein P supports virus gene expression and formation of infectious particles. J Virol, 2004, 78(22): 12333–12343.
[20] Raina K, Noblin DJ, Serebrenik YV, et al. Targeted protein destabilization reveals an estrogen-mediated ER stress response. Nat Chem Biol, 2014, 10(11): 957–962.
[21] Torregrosa-Hetland CJ, Villanueva J, Giner D, et al. The F-actin cortical network is a major factor influencing the organization of the secretory machinery in chromaffin cells. J Cell Sci, 2011, 124(5): 727–734.
[22] Karakoti AS, Shukla R, Shanker R, et al. Surface functionalization of quantum dots for biological applications. Adv Colloid Interface Sci, 2015, 215: 28–45.
[23] Nagel CH, Döhner K, Binz A, et al. Improper tagging of the non-essential small capsid protein VP26 impairs nuclear capsid egress of herpes simplex virus. PLoS ONE, 2012, 7(8): e44177.
[24] Toropova K, Huffman JB, Homa FL, et al. The herpes simplex virus 1 UL17 protein is the second constituent of the capsid vertex-specific component required for DNA packaging and retention. J Virol, 2011, 85(15): 7513–7522.
[25] Cockrell SK, Sanchez ME, Erazo A, et al. Role of the UL25 protein in herpes simplex virus DNA encapsidation. J Virol, 2009, 83(1): 47–57.
[26] Bohannon KP, Sollars PJ, Pickard GE, et al. Fusion of a fluorescent protein to the pUL25 minor capsid protein of pseudorabies virus allows live-cell capsid imaging with negligible impact on infection. J Gen Virol, 2012, 93(Pt 1): 124–129.
[27] Coller KE, Smith GA. Two viral kinases are required for sustained long distance axon transport of a neuroinvasive herpesvirus. Traffic, 2008, 9(9): 1458–1470.
[28] Loomis JS, Courtney RJ, Wills JW. Binding partners for the UL11 tegument protein of herpes simplex virus type 1. J Virol, 2003, 77(21): 11417–11424.
[29] Lorenz M, Vollmer B, Unsay JD, et al. A single herpesvirus protein can mediate vesicle formation in the nuclear envelope. J Biol Chem, 2015, 290(11): 6962–6974.
[30] Guan Y, Guo L, Yang EX, et al. HSV-1 nucleocapsid egress mediated by UL31 in association with UL34 is impeded by cellular transmembrane protein 140. Virology, 2014, 464–465: 1–10.
[31] Leelawong M, Guo DS, Smith GA. A physical link between the pseudorabies virus capsid and the nuclear egress complex. J Virol, 2011, 85(22): 11675–11684.
[32] Antinone SE, Smith GA. Retrograde axon transport of herpes simplex virus and pseudorabies virus: a live-cell comparative analysis. J Virol, 2010, 84(3): 1504–1512.
[33] Xue XD, Huang JH, Wang HS. The study of the intercellular trafficking of the fusion proteins of herpes simplex virus protein VP22. PLoS ONE, 2014, 9(6): e100840.
[34] Del Rio T, Ch’ng TH, Flood EA, et al. Heterogeneity of a fluorescent tegument component in single pseudorabies virus virions and enveloped axonal assemblies. J Virol, 2005, 79(7): 3903–3919.
[35] Dukhovny A, Sloutskin A, Markus A, et al. Varicella-zoster virus infects human embryonic stem cell-derived neurons and neurospheres but not pluripotent embryonic stem cells or early progenitors. J Virol, 2012, 86(6): 3211–3218.
[36] Hogue IB, Bosse JB, Hu JR, et al. Cellular mechanisms of alpha herpesvirus egress: live cell fluorescence microscopy of pseudorabies virus exocytosis. PLoS Pathog, 2014, 10(12): e1004535.
[37] Taylor MP, Kramer T, Lyman MG, et al. Visualization of an alphaherpesvirus membrane protein that is essential for anterograde axonal spread of infection in neurons. mBio, 2012, 3(2): e00063–12.
[38] Crump CM, Bruun B, Bell S, et al. Alphaherpesvirus glycoprotein M causes the relocalization of plasma membrane proteins. J Gen Virol, 2004, 85(12): 3517–3527.
[39] Huang T, Ma GG, Osterrieder N. Equine herpesvirus 1 multiply inserted transmembrane protein pUL43 cooperates with pUL56 in downregulation of cell surface major histocompatibility complex class I. J Virol, 2015, 89(12): 6251–6263.
[40] El Kasmi I, Lippé R. Herpes simplex virus 1 gN partners with gM to modulate the viral fusion machinery. J Virol, 2015, 89(4): 2313–2323.
[41] Milne RSB, Nicola AV, Whitbeck JC, et al. Glycoprotein D receptor-dependent, low-pH-independent endocytic entry of herpes simplex virus type 1. J Virol, 2005, 79(11): 6655–6663.
[42] Antinone SE, Smith GA. Two modes of herpesvirus trafficking in neurons: membrane acquisition directs motion. J Virol, 2006, 80(22): 11235–11240.
[43] Tirabassi RS, Enquist LW. Role of the pseudorabies virus gI cytoplasmic domain in neuroinvasion, virulence, and posttranslational N-linked glycosylation. J Virol, 2000, 74(8): 3505–3516.
[44] Kratchmarov R, Kramer T, Greco TM, et al. Glycoproteins gE and gI are required for efficient KIF1A-dependent anterograde axonal transport of alphaherpesvirus particles in neurons. J Virol, 2013, 87(17): 9431–9440.
[45] Kramer T, Greco TM, Taylor MP, et al. Kinesin-3 mediates axonal sorting and directional transport of alphaherpesvirus particles in neurons. Cell Host Microbe, 2012, 12(6): 806–814.
[46] Quinlan MP, Chen LB, Knipe DM. The intranuclear location of a herpes simplex virus DNA-binding protein is determined by the status of viral DNA replication. Cell, 1984, 36(4): 857–868.
[47] de Oliveira AP, Glauser DL, Laimbacher AS, et al. Live visualization of herpes simplex virus type 1 compartment dynamics. J Virol, 2008, 82(10): 4974–4990.
[48] Kobiler O, Brodersen P, Taylor MP, et al. Herpesvirus replication compartments originate with single incoming viral genomes. mBio, 2011, 2(6): e00278–11.
[49] Müller B, Heilemann M. Shedding new light on viruses: super-resolution microscopy for studying human immunodeficiency virus. Trends Microbiol, 2013, 21(10): 522–533.
[50] Hollinshead M, Johns HL, Sayers CL, et al. Endocytic tubules regulated by Rab GTPases 5 and 11 are used for envelopment of herpes simplex virus. EMBO J, 2012, 31(21): 4204–4220.
[51] Johns HL, Gonzalez-Lopez C, Sayers CL, et al. Rab6 dependent post-Golgi trafficking of HSV1 envelope proteins to sites of virus envelopment. Traffic, 2014, 15(2): 157–178.
[52] Speck SH, Ganem D. Viral latency and its regulation: lessons from the γ-herpesviruses. Cell Host Microbe, 2010, 8(1): 100–115.
[53] Buehler J, Zeltzer S, Reitsma J, et al. Opposing regulation of the EGF receptor: a molecular switch controlling cytomegalovirus latency and replication. PLoS Pathog, 2016, 12(5): e1005655.
[54] Mahjoub N, Dhorne-Pollet S, Fuchs W, et al. A 2.5-kilobase deletion containing a cluster of nine microRNAs in the latency-associated-transcript locus of the pseudorabies virus affects the host response of porcine trigeminal ganglia during established latency. J Virol, 2015, 89(1): 428–442.
[55] Roizman B, Whitley RJ. An inquiry into the molecular basis of HSV latency and reactivation. Annu Rev Microbiol, 2013, 67(67): 355–374.
[56] Koyuncu OO, Song R, Greco TM, et al. The number of alphaherpesvirus particles infecting axons and the axonal protein repertoire determines the outcome of neuronal infection. mBio, 2015, 6(2): e00276–15.
[57] Nicoll MP, Hann W, Shivkumar M, et al. The HSV-1 latency-associated transcript functions to repress latent phase lytic gene expression and suppress virus reactivation from latently infected neurons. PLoS Pathog, 2016, 12(4): e1005539.
[58] Umbach JL, Kramer MF, Jurak I, et al. MicroRNAs expressed by herpes simplex virus 1 during latent infection regulate viral mRNAs. Nature, 2008, 454(7205): 780–783.
[59] Shen WW, Sa e Silva M, Jaber T, et al. Two small RNAs encoded within the first 1.5 kilobases of the herpes simplex virus type 1 latency-associated transcript can inhibit productive infection and cooperate to inhibit apoptosis. J Virol, 2009, 83(18): 9131–9139.
[60] Garber DA, Schaffer PA, Knipe DM. A LAT-associated function reduces productive-cycle gene expression during acute infection of murine sensory neurons with herpes simplex virus type 1. J Virol, 1997, 71(8): 5885–5893.
[61] Chen SH, Kramer MF, Schaffer PA, et al. A viral function represses accumulation of transcripts from productive-cycle genes in mouse ganglia latently infected with herpes simplex virus. J Virol, 1997, 71(8): 5878–5884.
[62] Ljungman P, Griffiths P, Paya C. Definitions of cytomegalovirus infection and disease in transplant recipients. Clin Infect Dis, 2002, 34(8): 1094–1097.
[63] Hahn G, Jores R, Mocarski ES. Cytomegalovirus remains latent in a common precursor of dendritic and myeloid cells. Proc Natl Acad Sci USA, 1998, 95(7): 3937–3942.
[64] Kondo K, Kaneshima H, Mocarski ES. Human cytomegalovirus latent infection of granulocyte-macrophage progenitors. Proc Natl Acad Sci USA, 1994, 91(25): 11879–11883.
[65] Cheng S, Jiang X, Yang B, et al. Infected T98G glioblastoma cells support human cytomegalovirus reactivation from latency. Virology, 2017, 510: 205–215.
[66] Pomeranz LE, Reynolds AE, Hengartner CJ. Molecular biology of pseudorabies virus: impact on neurovirology and veterinary medicine. Microbiol Mol Biol Rev, 2005, 69(3): 462–500.
[67] Smith GA, Gross SP, Enquist LW. Herpesviruses use bidirectional fast-axonal transport to spread in sensory neurons. Proc Natl Acad Sci USA, 2001, 98(6): 3466–3470.
[68] Lyman MG, Feierbach B, Curanovic D, et al. Pseudorabies virus Us9 directs axonal sorting of viral capsids. J Virol, 2007, 81(20): 11363–11371.
[69] Klopfenstein DR, Tomishige M, Stuurman N, et al. Role of phosphatidylinositol (4,5) bisphosphate organization in membrane transport by the Unc104 kinesin motor. Cell, 2002, 109(3): 347–358.
[70] Lo KY, Kuzmin A, Unger SM, et al. KIF1A is the primary anterograde motor protein required for the axonal transport of dense-core vesicles in cultured hippocampal neurons. Neurosci Lett, 2011, 491(3): 168–173.
[71] Nassi JJ, Cepko CL, Born RT, et al. Neuroanatomy goes viral! Front Neuroanat, 2015, 9: 80.
[72] Krout KE, Mettenleiter TC, Loewy AD. Single CNS neurons link both central motor and cardiosympathetic systems: a double-virus tracing study. Neuroscience, 2003, 118(3): 853–866.
[73] Wojaczynski GJ, Engel EA, Steren KE, et al. The neuroinvasive profiles of H129 (herpes simplex virus type 1) recombinants with putative anterograde-only transneuronal spread properties. Brain Struct Funct, 2015, 220(3): 1395–1420.
[74] McGovern AE, Driessen AK, Simmons DG, et al. Distinct brainstem and forebrain circuits receiving tracheal sensory neuron inputs revealed using a novel conditional anterograde transsynaptic viral tracing system. J Neurosci, 2015, 35(18): 7041–7055.
[75] Zeng WB, Jiang HF, Gang YD, et al. Anterograde monosynaptic transneuronal tracers derived from herpes simplex virus 1 strain H129. Mol Neurodegener, 2017, 12(1): 38.
[76] Card JP, Kobiler O, Ludmir EB, et al. A dual infection pseudorabies virus conditional reporter approach to identify projections to collateralized neurons in complex neural circuits. PLoS ONE, 2011, 6(6): e21141.
[77] Taylor MP, Kratchmarov R, Enquist LW. Live cell imaging of alphaherpes virus anterograde transport and spread. J Vis Exp, 2013, (78): 50723, doi: 10.3791/50723.
[78] Hogue IB, Scherer J, Enquist LW. Exocytosis of alphaherpesvirus virions, light particles, and glycoproteins uses constitutive secretory mechanisms. mBio, 2016, 7(3): e00820–16.
[79] Sampaio KL, Jahn G, Sinzger C. Applications for a dual fluorescent human cytomegalovirus in the analysis of viral entry//Bailer S, Lieber D, eds. Virus-Host Interactions. Totowa, NJ: Humana Press, 2013, 1064: 201–209.
[80] Taylor TJ, McNamee EE, Day C, et al. Herpes simplex virus replication compartments can form by coalescence of smaller compartments. Virology, 2003, 309(2): 232–247.
[81] Grove J. Super-resolution microscopy: a virus’ eye view of the cell. Viruses, 2014, 6(3): 1365–1378.
[82] Colberg-Poley AM, Patterson GH, Salka K, et al. Superresolution imaging of viral protein trafficking. Med Microbiol Immunol, 2015, 204(3): 449–460.
[83] Bohannon KP, Jun YG, Gross SP, et al. Differential protein partitioning within the herpesvirus tegument and envelope underlies a complex and variable virion architecture. Proc Natl Acad Sci USA, 2013, 110(17): E1613–E1620.
[84] Laine RF, Albecka A, van de Linde S, et al. Structural analysis of herpes simplex virus by optical super-resolution imaging. Nat Commun, 2015, 6(3): 5980.
[85] Wang F, Flanagan J, Su N, et al. RNAscope: a novelRNA analysis platform for formalin-fixed, paraffin-embedded tissues. J Mol Diagn, 2012, 14(1): 22–29.
Tracking of herpesviruses: what have been seen and will be seen?
Yalin Wang, Huaji Qiu, and Yuan Sun
State Key Laboratory of Veterinary Biotechnology, Harbin Veterinary Research Institute, Chinese Academy of Agricultural Sciences, Harbin 150069, Heilongjiang, China
Viral infection of cells is a highly intricate process that involves the complex virus-cell interactions. Recently, virologists can monitor the virus life cycle at the primary infection site in real-time using various virus tracking techniques. Herpesviruses, a class of large enveloped DNA viruses, are important pathogens threatening the health of humans and animals. This review discussed the applications of different virus tracking techniques in herpesvirus studies, to provide new insights into virus-cell interactions and replication mechanisms of herpesviruses. Though the techniques have widely been exploited, some issues need to be addressed, such as the selection of the optimal site to insert reporters and the inability to track the whole process of the virus life cycle. With the updated tracking techniques, hopefully, more complex replication mechanisms of herpesviruses will be revealed in detail.
virus tracking techniques, herpesvirus, fluorescent protein, replication, latent infection, transport in axon
February 8, 2018;
June 11, 2018
National Natural Science Foundation of China (No. 31570149).
Huaji Qiu. Tel/Fax: +86-451-51051708; E-mail: huajiqiu@caas.cn
Yuan Sun. Tel/Fax: +86-451-51051709; E-mail: sunyuan@caas.cn
10.13345/j.cjb.180057
国家自然科学基金(No. 31570149) 资助。
2018-07-06
http://kns.cnki.net/kcms/detail/11.1998.Q.20180704.1636.001.html
(本文责编 陈宏宇)

