湖羊转录因子CTCF基因序列分析及其对NR5A1基因转录活性的调控
李隐侠 郭潇潇 张俊 孟春花 钱勇 仲跻峰 曹少先

摘要:转录因子CTCF在动物生长发育过程中发挥重要的调控作用,但其在绵羊中的序列特征、组织器官表达及功能目前尚不清楚。本研究以湖羊CTCF基因为研究对象,采用PCR方法克隆获得其编码区全序列,发现其序列全长2 187 bp,编码727个氨基酸残基,含有11个连续的锌指结构域。组织器官表达谱分析发现CTCF基因在湖羊各个组织器官中广泛表达,在子宫中表达量相对较低,在胃中表达量相对较高。JASPAR在线软件预测发现核受体NR5A1基因内含子(翻译起始位点ATG前393 bp片段)含有3个CTCF结合位点,双荧光素酶试验结果显示,CTCF结合位点突变后NR5A1基因转录活性显著或极显著下降,表明CTCF可能通过调控NR5A1基因转录参与调控湖羊繁殖性能。
关键词:转录因子CTCF;表达特征;NR5A1 基因;转录活性
中图分类号:Q786文献标识码:A文章编号:1000-4440(2020)06-1482-07
Abstract:Transcription factor CTCF plays an important regulatory role in growth and development of animals, but its sequence characteristics, tissues and organs expression and function in sheep are still unclear. In this study, CTCF gene of Hu sheep was used as research object, the full coding sequence of CTCF was cloned by RT-PCR. The result showed that the full-length sequence of CTCF was 2 187 bp, encoding 727 amino acids and containing 11 consecutive zinc finger domains. CTCF gene was widely expressed in various tissues and organs of Hu sheep, with a relatively low expression in uterus and a relatively high expression in stomach. Predication results showed that the intron (the 393 bp fragment before translation initiation site ATG) of nuclear receptor NR5A1 gene contained three CTCF binding sites by the JASPAR online software. The results of dual luciferase assay showed that the transcription activity of NR5A1 gene decreased significantly or extrenely significantly when the CTCF binding sites were mutated, indicating that CTCF participated in the regulation of reproductive performance of Hu sheep by regulating NR5A1 gene transcription.
Key words:transcription factor CTCF;expression characteristics;NR5A1 gene;transcription activity
CTCF(CCCTC binding factor)是一个重要的转录因子,由一个N末端结构域,一个包含11个C2H2锌指中心结构域和一个C末端结构域组成[1]。哺乳动物基因组中大约含有30 000~70 000个CTCF结合位点[2],调控多种功能,包括微调基因表达、X染色体失活、印记和三维染色质组织等[3-4]。体内、外研究发现,CTCF结合位点完全破坏或缺失的纯合子在整个胚胎期都是致死的[5],且在特定细胞如卵母细胞[6]、淋巴細胞[7]、神经元[8]中特异性敲除CTCF后导致器官特异性功能失效、异常的增强子-启动子相互作用和转录失调。小鼠雄性生殖细胞中CTCF失活导致精子发生异常和不育[9]。说明CTCF在动物生长发育过程中具有非常重要的作用。
CTCF作为转录因子发挥重要的调控作用。作为转录抑制因子负调控c-myc和溶菌酶基因转录[10-12],作为转录激活因子与Cohesin蛋白参与调控人UGT1基因簇的转录[13],驱动端粒重复编码RNA的转录进而促进端粒DNA复制的完成[14]。
孤儿核受体NR5A1基因参与调控湖羊的繁殖性能[15-17],JASPAR在线软件预测发现,在NR5A1基因内含子(翻译起始位点“ATG”前393 bp内)含有3个CTCF结合位点,但CTCF是否参与调控湖羊NR5A1基因转录尚未知道。本研究以湖羊为研究对象,扩增卵巢组织中CTCF基因编码区序列,进行序列特征分析,同时鉴定其在湖羊各组织中的表达模式,最后构建CTCF结合位点野生型/突变型NR5A1序列的荧光素酶报告载体,研究CTCF对NR5A1活性的影响,解析CTCF对湖羊NR5A1基因的调控作用。
1材料与方法
1.1试验动物
试验湖羊母羊(来自西来原生态农业有限公司)屠宰后,立即采集各个组织器官样(包括心、肝、脾、肺、肾、子宫、卵巢、大肠、小肠和肌肉等)于液氮中保存备用。采用传统的酚-氯仿方法提取DNA,用RNA提取试剂盒(百迈客生物科技有限公司产品)提取RNA,并使用HiScript II Q RT SuperMix(诺唯赞生物科技股份有限公司产品)完成cDNA第一链的合成。
1.2引物的合成和扩增
用Primer Premier 5.0 软件先设计2对CTCF基因(序列号:XM_027978089.1)特异性引物P-CTCF1和P-CTCF2,用于扩增湖羊CTCF编码区;再设计P-CTCF3引物,以β-actin为内参(表1)进行湖羊CTCF基因组织表达谱的鉴定。引物由南京擎科生物有限公司合成,引物序列的相关信息见表1。
以湖羊卵巢组织为模板,扩增湖羊CTCF基因编码区序列; 以湖羊各组织逆转录的cDNA为模板,以β-actin为内参,扩增不同组织中CTCF基因及对应β-actin,分析湖羊CTCF基因组织表达谱。RT-PCR反应体系为20 μl,含模板DNA或者cDNA 60 ng、1.5 TM2X High-Fidelity Master Mix (南京擎科生物科技有限公司产品) 10 μl、引物(100 μmol/L)1 μl,加灭菌双蒸水至20 μl。PCR扩增程序:98 ℃预变性2 min;98 ℃变性10 s,53~58 ℃(表1)退火10 s,72 ℃延伸2 min ,35个循环;最后72 ℃延伸5 min。
1.3序列分析和转录因子结合位点预测
克隆测序获得的序列在NCBI 的ORF Finder(http://www.ncbi.nlm. nih.gov/projects/gorf/)中进行开放阅读框预测,并用DNAMAN6.0软件进行编码区序列翻译。用SMART(http://smart.embl-heidelberg.de/)软件预测CTCF蛋白功能域。用JASPAR在线预测数据库(http://jaspar.bin.ku.dk)预测转录因子结合位点。
1.4荧光素酶载体构建
以湖羊DNA为模板扩增NR5A1基因自翻译起始位点ATG前393 bp至ATG后85 bp的片段(片段长481 bp),连接到pMD-19T载体上,由南京擎科生物科技有限公司测序鉴定后获得pMD-19T-NR5A1载体。用内切酶Nhe I 和Hind III 双酶切荧光素酶报告载体(pGL3-basic)和pMD19T-NR5A1,目的片段切胶回收后T4连接酶连接过夜,转化DHα感受态细胞,测序鉴定后获得pGL3-NR5A1载体。
1.5点突变载体构建
以获得的pMD19T-NR5A1质粒为模板,根据TaKaRa MutanBEST Kit 引物设计原理设计3对5′端邻接、3′端方向相反的引物用以导入变异点,同时使用高保真DNA聚合酶(Pyrobest DNA Polymerase)进行PCR 扩增、连接和质粒DNA转化,构建3个NR5A1基因CTCF结合位点突变型pMD19T-NR5A1-TU1、pMD19T-NR5A1-TU2和pMD19T-NR5A1-TU3载体,用内切酶Nhe I 和Hind III 分别双酶切荧光素酶报告载体(pGL3-basic)和pMD19T-NR5A1-TU1、pMD19T-NR5A1-TU2和pMD19T-NR5A1-TU3载体,目的片段切胶回收后T4连接酶连接过夜,转化DHα感受态细胞,测序鉴定后获得pGL3-NR5A1-TU1、pGL3-NR5A1-TU2和pGL3-NR5A1-TU3载体,引物见表1。
1.6细胞转染和荧光素酶活性测定
293T细胞传代到含有10%胎牛血清[赛默飞世尔科技(中国)有限公司产品]、100 U/L氨苄/四环素[赛默飞世尔科技(中国)有限公司产品] 的DMEM(Dulbeccos modified Eagles media) 培养基的12孔板中,在5% CO2 的37 ℃培养箱中培养到细胞汇合度85%~90% 时,使用Lipofectamine 3000[赛默飞世尔科技(中国)有限公司产品]分别转染空载pGL3-basic、NR5A1野生型载体pGL3-NR5A1和3个CTCF结合位点突变型NR5A1载体pGL3-NR5A1-TU1、pGL3-NR5A1-TU2、pGL3-NR5A1-TU3于细胞中,转染后48 h 搜集细胞进行荧光素酶活性测定。
1.7数据分析
试验结果用平均数 ± 标准差表示,用SPSS16.0软件中t检验进行独立样本统计分析,用One-way中的ANOVA进行多重比较分析。
2结果与分析
2.1湖羊CTCF基因编码区序列扩增
以湖羊卵巢組织cDNA为模板,2对特异性引物扩增湖羊CTCF编码区序列,1%琼脂糖凝胶电泳检测发现2对引物在湖羊中均有特异性扩增(图1),切胶回收测序后进行序列比对和拼接,得到湖羊CTCF基因的整个编码区CDS序列。
2.2湖羊CTCF基因序列特征分析
测序获得的CTCF序列用在线软件(ORF Finder )预测发现,完整的开放阅读框长度为2 187 bp,编码727 个氨基酸残基。SMART在线预测发现湖羊CTCF编码的蛋白质与其他哺乳动物一样,含有11个连续的锌指结构域,11个锌指结构域在编码区的起始位点见表2,说明CTCF基因在哺乳动物中高度保守。
2.3CTCF基因在湖羊组织器官中的表达
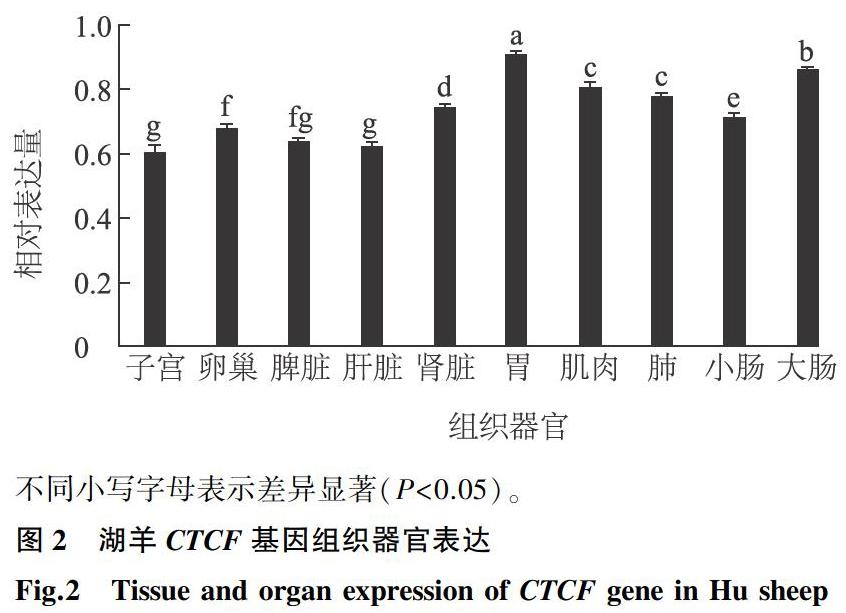
RT-PCR方法检测CTCF基因在湖羊子宫、卵巢、脾脏、肝脏、肾脏、胃、肌肉、肺脏、小肠和大肠等10个组织器官中的表达模式,结果发现CTCF基因在检测的10个湖羊组织器官中广泛表达 (图2),在子宫中表达量相对较低,胃中表达量相对最高。统计分析发现子宫、卵巢、脾脏和肝脏中CTCF表达量显著低于肾脏、胃、肌肉、肠等组织(P<0.05)(图2)。
2.4转录因子CTCF调控湖羊NR5A1基因活性
2.4.1湖羊NR5A1基因翻译起始位点ATG前393 bp序列扩增以湖羊DNA为模板,扩增湖羊NR5A1基因翻译起始位点ATG前393 bp至ATG后85 bp(共计481 bp)的片段,测序后比对分析发现其与引物源序列高度一致。JASPAR软件预测发现在翻译起始位点ATG前的393 bp片段内存在3个转录因子CTCF结合位点(图3)。
2.4.2CTCF 调控湖羊NR5A1基因转录活性构建湖羊NR5A1基因ATG前393 bp至ATG后85 bp的片段野生型荧光素酶报告载体pGL3-NR5A1和3个突变型荧光素酶报告载体pGL3-NR5A1 TU1、pGL3-NR5A1 TU2、pGL3-NR5A1 TU3 (图4A)。将4个荧光素酶报告载体转染293T细胞,双荧光素酶检测试剂盒检测荧光素酶活性,结果(图4 B)发现第一个CTCF转录因子结合位点突变后荧光活性下降,但是与野生型相比差异不显著;第二个CTCF转录因子结合位点突变后,荧光活性与野生型相比显著下降(P=0.042);第三个CTCF转录因子结合位点突变后,荧光活性与野生型相比极显著下降(P=0.001)。说明转录因子CTCF参与调控湖羊NR5A1基因的转录活性。
3讨论
转录因子CTCF在调控动物生长发育过程中发挥重要作用[18-19]。CTCF通过抑制S100A1上调RYR2,从而抑制心肌细胞的内质网应激和凋亡[20],CTCF缺失胚胎无法植入[21-22],CTCF在人类胚胎发生过程中的3D染色质结构建立中具有关键作用,CTCF缺失导致小鼠早期大脑发育中PUMA(受P53上调表达的凋亡调控基因)上调和随后大量凋亡[23]。
研究发现CTCF可调控脊椎动物大脑、心血管、四肢和肌肉发育等[24],而本研究组织器官表达谱显示CTCF基因在湖羊各个组织器官中广泛表达,是一种组成型表达基因,提示可能在湖羊各个组织中均发挥重要的调控作用,与前人的研究结果一致[24-25]。
人[26]、牛[27]、猪[28]、小鼠[29]和斑马鱼[30]等的CTCF基因均已被克隆,本研究首次克隆获得湖羊CTCF 编码区全序列,比对发现CTCF编码的蛋白质含有连续的11个锌指结构域,与其他哺乳动物一致。CTCF转录因子一般通过其第3到第7结构域结合DNA的15 bp 核心基序调控靶基因表达[31],但CTCF中的11个锌指结构域在不同物种中参与调控靶基因的功能也时有不同,转录因子CTCF调控靶基因c-myc基因时,第2至第7锌指结构域对于鸡c-myc 基因V位点的结合是必需的,而第3至第7锌指结构域对特异性结合人c-myc 基因A位点是必须的[32],说明转录因子CTCF通过不同锌指结构域与不同DNA靶序列结合参与调控多种生物学功能[33-35],是一个多价转录因子。
进一步研究发现CTCF主要与基因间区域和内含子相关联,像增强子一样作为调控元件调控基因表达[36]。CTCF通过与FMR1基因启动子区、外显子区和内含子2相结合参与调控FMR1基因转录调控[37],通过与BCL6基因第一内含子结合参与调控基因转录和表观遗传学修饰[38],CTCF与巨细胞病毒(CMV)MIE基因的内含子A结合并具有抑制MIE基因表达和病毒复制的功能[34]。NR5A1是核受体NR5A家族重要成员之一,与哺乳动物雌性生殖、卵泡发育、类固醇生成等关系密切[39-40],是维持雌性卵巢功能和繁殖力必需的因子[41-43]。在湖羊中发现NR5A1基因在卵巢组织中高表达,是调控湖羊产羔数的重要候选基因之一[15-17],5′RACE 方法鉴定其转录起始位点与翻译起始位点(ATG)间有一个长度为1 424的内含子存在[16],可能对NR5A1的转录发挥调控作用。本研究发现的NR5A1基因ATG前393 bp片段位于此內含子中, 此393 bp片段存在3个CTCF转录因子结合位点,荧光素酶活性分析结果显示CTCF转录因子参与调控湖羊NR5A1基因的转录活性,该结果初步说明转录因子CTCF可能通过调控NR5A1基因转录进而参与调控湖羊繁殖性能。
参考文献:
[1]FILIPPOVA G N. Genetics and epigenetics of the multifunctional protein CTCF[J]. Curr Top Dev Biol, 2008, 80:337-360.
[2]ARZATE-MEJA R G, RECILLAS-TARGA F, CORCES V G. Developing in 3D: the role of CTCF in cell differentiation[J]. Development,2018, 145(6):137729.
[3]FEDORIW A M, STEIN P, SVOBODA P, et al. Transgenic RNAi reveals essential function for CTCF in H19 gene imprinting[J]. Science, 2004, 303(5655): 238-240.
[4]MERKENSCHLAGER M, NORA E P. CTCF and cohesin in genome folding and transcriptional gene regulation[J]. Annu Rev Genomics Hum Genet, 2016, 17: 17-43.
[5]CARMONA-ALDANA F, ZAMPEDRI C, SUASTE-OLMOS F, et al. CTCF knockout reveals an essential role for this protein during the zebrafish development[J]. Mech Dev, 2018, 154: 51-59.
[6]WAN L B, PAN H, HANNENHALLI S, et al. Maternal depletion of CTCF reveals multiple functions during oocyte and preimplantation embryo development[J]. Development,2008, 135(16): 2729-2738.
[7]RIBEIRO D E ALMEIDA C, STADHOUDERS R, DE BRUIJN M J W, et al. The DNA-binding protein CTCF limits proximal Vκ recombination and restricts κ enhancer interactions to the immunoglobulin κ light chain locus[J]. Immunity,2011, 35(4): 501-513.
[8]HIRAYAMA T, TARUSAWA E, YOSHIMURA Y, et al. CTCF is required for neural development and stochastic expression of clustered Pcdh genes in neurons[J]. Cell Rep,2012, 2(2): 345-357.
[9]HERNNDEZ-HERNNDEZ A, LILIENTHAL I, FUKUDA N, et al. CTCF contributes in a critical way to spermatogenesis and male fertility[J]. Sci Rep,2016, 6: 28355.
[10]LOBANENKOV V V, NICOLAS R H, ADLER V V, et al. A novel sequence-specific DNA binding protein which interacts with three regularly spaced direct repeats of the CCCTC-motif in the 5′-flanking sequence of the chicken c-myc gene[J]. Oncogene,1990, 5: 1743-1753.
[11]BANIAHMAD A, STEINER C, KHNE A C, et al. Modular structure of a chicken lysozyme silencer: involvement of an unusual thyroid hormone receptor binding site[J]. Cell,1990, 61: 505-514.
[12]OUBOUSSAD L, KREUZ S, LEFEVRE P F. CTCF depletion alters chromatin structure and transcription of myeloid-specific factors[J]. J Mol Cell Biol,2013, 5: 308-322.
[13]郑晓飞,黄海燕,吴强. 染色质构架蛋白CTCF调控UGT1基因簇的表达[J]. 遗传, 2019,41(6):509-523.
[14]BEISHLINE K, VLADIMIROVA O, TUTTON S, et al. CTCF driven TERRA transcription facilitates completion of telomere DNA replication[J]. Nat Commun,2017, 8(1): 2114.
[15]李隱侠,张俊,钱勇,等. 湖羊NR5A1基因全序列克隆和表达特征分析[J]. 江苏农业学报, 2019, 35(1): 114-121.
[16]LI Y, ZHANG J, QIAN Y, et al. Mutation -388 C>G of NR5A1 gene affects litter size and promoter activity in sheep[J]. Anim Reprod Sci,2018, 196: 19-27.
[17]李隐侠,张俊,钱勇,等. 湖羊NR5A1基因SNPs筛选及其与产羔数的关联分析[J]. 江苏农业学报,2017, 33(1): 124-132.
[18]SEITAN VC, KRANGEL MS, MERKENSCHLAGER M. Cohesin, CTCF and lymphocyte antigen receptor locus rearrangement[J]. Trends Immunol,2012, 33: 153-159.
[19]OHLSSON R, BARTKUHN M, RENKAWITZ R. CTCF shapes chromatin by multiple mechanisms: the impact of 20 years of CTCF research on understanding the workings of chromatin[J]. Chromosoma,2010, 119: 351-360.
[20]ZENG Z, HUANG N, ZHANG Y, et al. CTCF inhibits endoplasmic reticulum stress and apoptosis in cardiomyocytes by upregulating RYR2 via inhibiting S100A1[J]. Life Sci,2020, 242: 117158.
[21]CHEN X, KE Y, WU K, et al. Key role for CTCF in establishing chromatin structure in human embryos[J]. Nature,2019, 576(7786): 306-310.
[22]MOORE J M, RABAIA N A, SMITH L E, et al. Loss of maternal CTCF is associated with peri-implantation lethality of Ctcf null embryos[J]. PLoS One,2012, 7: e34915.
[23]WATSON LA, WANG X, ELBERT A, et al. Dual effect of CTCF loss on neuroprogenitor differentiation and survival[J]. J Neurosci,2014, 34: 2860-2870.
[24]ARZATE-MEJíA R G, RECILLAS-TARGA F, CORCES V G. Developing in 3D: the role of CTCF in cell differentiation[J]. Developmet,2018, 145(6): 137729.
[25]PHILLIPS J E, CORCES V G. CTCF: master weaver of the genome[J]. Cell,2009, 137(7): 1194-1211.
[26]VOSTROV A A, QUITSCHKE W W. The zinc finger protein CTCF binds to the APB beta domain of the amyloid beta-protein precursor promoter. Evidence for a role in transcriptional activation[J]. J Biol Chem, 1997, 272(52): 33353-33359.
[27]蘇节,朱鹏,刘庆友,等. 水牛转录抑制因子CTCF基因克隆分析及不同组织中的表达研究[J]. 中国畜牧兽医, 2013, 40(3): 1-6.
[28]UENISHI H, EGUCHI T, SUZUKI K, et al. PEDE (pig EST data explorer): construction of a database for ESTs derived from porcine full-length cDNA libraries[J]. Nucleic Acids Res, 2004, 32 (Suppl): D484-D488.
[29]LUTZ M, BURKE LJ, BARRETO G, et al. Transcription repression by the insulator protein CTCF involves histone deacetylases[J]. Nucl Acids Res, 2000, 28(8): 1707-1713.
[30]PUGACHEVA E M, KWON Y W, HUKRIEDE N A, et al. Cloning and characterization of zebrafish CTCF: developmental expression patterns, regulation of the promoter region, and evolutionary aspects of gene organization[J]. Gene,2006, 375: 26-36.
[31]HASHIMOTO H, WANG D, HORTON J R, et al. Structural basis for the versatile and methylation-dependent binding of CTCF to DNA[J]. Mol Cell, 2017, 66: 711-720.
[32]FILIPPOVA G N, FAGERLIE S, KLENOVA E M, et al. An exceptionally conserved transcriptional repressor, CTCF, employs different combinations of zinc fingers to bind diverged promoter sequences of avian and mammalian c-myc oncogenes[J]. Mol Cell Biol,1996, 16(6): 2802-2813.
[33]KUZMIN I, GEIL L, GIBSON L, et al. Transcriptional regulator CTCF controls human interleukin 1 receptor-associated kinase 2 promoter[J]. J Mol Biol,2005, 346 (2): 411-422.
[34]AWAD T A, BIGLER J, ULMER J E, et al. Negative transcriptional regulation mediated by thyroid hormone response element 144 requires binding of the multivalent factor CTCF to a novel target DNA sequence[J]. J Biol Chem,1999, 274 (38): 27092-27098.
[35]TORRANO V, CHERNUKHIN I, DOCQUIER F, et al. CTCF regulates growth and erythroid differentiation of human myeloid leukemia cells[J]. J Biol Chem,2005, 280 (30): 28152-28161.
[36]RUIZ-VELASCO M, KUMAR M, LAI M C, et al. CTCF-mediated chromatin loops between promoter and gene body regulate alternative splicing across individuals[J]. Cell Syst,2017, 5(6): 628-637.
[37]LANNI S, GORACCI M, BORRELLI L, et al. Role of CTCF protein in regulating FMR1 locus transcription[J]. PLoS Genet,2013, 9(7): e1003601.
[38]BATLLE-LPEZ A, CORTIGUERA M G, ROSA-GARRIDO M, et al. Novel CTCF binding at a site in exon1A of BCL6 is associated with active histone marks and a transcriptionally active locus[J]. Oncogene,2015, 34(2): 246-256.
[39]MARTíNEZ F P, CRUZ R, LU F, et al. CTCF binding to the first intron of the major immediate early (MIE) gene of human cytomegalovirus (HCMV) negatively regulates MIE gene expression and HCMV replication[J]. J Virol,2014, 88(13): 7389-7401.
[40]KIM KW, LI S, ZHAO H, et al. CNS-specific ablation of steroidogenic factor 1 results in impaired female reproductive function[J]. Mol Endocrinol,2010, 24(6): 1240-1250.
[41]YIN M, L M, YAO G, et al. Transactivation of microRNA-383 by steroidogenic factor-1 promotes estradiol release from mouse ovarian granulosa cells by targeting RBMS1[J]. Mol Endocrinol,2012, 26(7): 1129-1143.
[42]JEYASURIA P, IKEDA Y, JAMIN S P, et al. Cell-specific knockout of steroidogenic factor 1 reveals its essential roles in gonadal function[J]. Mol Endocrinol,2004, 18(7): 1610-1619.
[43]BUAAS F W, GARDINER J R, CLAYTON S, et al. In vivo evidence for the crucial role of SF1 in steroid-producing cells of the testis, ovary and adrenal gland[J]. Development,2012, 139(24): 4561-4570.
(責任编辑:陈海霞)

