ACE2在不同物种间的同源性分析及其对2019-nCoV宿主范围的启示
李彦君,陈娇珊,赵 锦,闻子钰,孙彩军
新型冠状病毒肺炎(COVID-19)疫情暴发以来,在全球迅速蔓延,已造成超过410万人感染。长期以来冠状病毒家族成员与人类和动物疾病密切相关,可感染包括猪、牛、马、骆驼、猫、狗、蝙蝠、兔子、雪貂、水貂、蛇、啮齿动物、鸟类、和其他野生动物[1]。冠状病毒科按其遗传特性分为4个属,其中α和β属仅能感染哺乳动物,而γ和δ属可感染禽类和哺乳动物等[1]。2019-nCoV 属于冠状病毒科β冠状病毒属[2],近期的基因组学分析表明2019-nCoV 是一种自然进化的产物[3]。由于近期陆续报道了几起动物的核酸检测阳性事件[4-5],2019-nCoV 由人向动物传播的风险也引起了人们的关注。尽管尚缺乏确凿的证据,但从理论上说这些动物与人类处于同一水平面对病毒,也有被感染的可能性[6]。基于2019-nCoV 已经在人群广泛流行,人类感染者是否能通过呼吸道等途径再传染给人类密切接触的动物,造成该疾病成为长期存在的人兽共患病,这将是一个值得关注的科学问题。
人类血管紧张素转化酶2(angiotensin-converting enzyme 2,ACE2)包含805个氨基酸,由N端肽酶结构域(peptidase domain,PD)和C端凝集素样结构域(collectrin-like domain,CLD)组成[7-8]。病毒表面S蛋白的受体结合域(receptor-binding domain, RBD)利用其外部区域与受体ACE2的PD结构域(19-615残基)结合,介导病毒进入宿主细胞[8-9]。同时ACE2在新冠肺炎的发病中也可通过对肾素-血管紧张素系统产生负性调节,从而保护肺免受2019-nCoV所致的损伤[8,10]。因此,ACE2在COVID-19的传播和疾病的病理中起关键作用。本文系统分析了不同物种ACE2蛋白的同源性,并讨论了动物感染模型和潜在的易感动物,这将对实现人类-动物-生态的同一健康理念(One Health)以及预测预警重大疫情的流行和演变至关重要。
1 材料与方法
1.1研究对象 人类和30种与人类活动相对关系密切的动物ACE2全长氨基酸序列(1-805)。124种脊椎动物的ACE2-PD氨基酸序列(19-615残基)。人类ACE2中与SARS-CoV接口的18个氨基酸:24Q、27T、31K、34H、37E、38D、41Y、42Q、45L、79L、82M、83Y、90N、325M、329F、330W、353L、354G[11],人类ACE2中与2019-nCoV接口的8个氨基酸为24Q、30D、34H、41Y、42Q、82M、353L、357G[12]。
1.2数据来源与选择 基于NCBI网站的Protein数据库(https://www.ncbi.nlm.nih.gov/refseq/),以“ACE2”为关键词,筛选条件设置为:SPECIES 选择“animals”,SOURCE DATABASES选择“RefSeq”,下载不同物种ACE2氨基酸序列。124种脊椎动物中包含哺乳类75种、鸟类23种,鱼类18种 爬行类7种和两栖类1种。根据经验哺乳类动物与人类的同源性较高,进一步将其分为灵长目16种、奇/偶蹄目11种、食肉目23种、啮齿目11种、翼手目7种、兔形目2种和其他类别8种。通过查阅文献获取人类ACE2中与SARS-CoV和2019-nCoV接口的20个氨基酸[11-12]。
1.3软件和网站 氨基酸序列同源性比对使用NCBI网站的在线程序BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)。采用MEGA 7软件中的ClustalW 进行序列对齐,截取与人ACE2-PD 结构域(19-615位残基)的对应序列。系统发育树的构建使用MEGA 7软件,采用邻接法(NJ,Neighbor-joining),参数设置为 Bootstrap 1000个循环。使用在线程序iTOL(http://itol.embl.de/)修改树枝和字体的颜色。
2 结 果
2.1不同物种间ACE2氨基酸全长序列对比 与人类活动相对关系密切的30种动物ACE2全长氨基酸序列系统发育分析表明(图1),相比于鱼类、鸟类和爬行类动物,哺乳类动物间的ACE2同源性更高。21种哺乳类动物可主要分为2个分支,刺猬单独为一类,人类、非人灵长类、食肉类、啮齿类等动物为第二类。与人类并列的2个小类分别为:猫科动物、犬科动物、穿山甲构成第一小类;奇蹄与偶蹄类动物和蝙蝠构成第二小类。BLAST氨基酸序列对比显示,与鸟类、鱼类、两栖动物和爬行动物相比,人类ACE2全长氨基酸序列与哺乳动物更相似,高达80%以上,其中大猩猩99%,猕猴97%。
2.2不同物种间ACE2-PD氨基酸序列对比 进一步扩大不同种类动物样本的序列数据,纳入了124种脊椎动物的ACE2氨基酸序列,分析与2019-nCoV病毒S蛋白结合的ACE2肽酶结构域(ACE2-PD)序列的同源性。结果如图2,PD结构域序列在不同物种间的同源性分析结果大致与图1中的ACE2全序列分析一致。值得注意的是,与ACE2全长序列分析相比较,图2中猫科动物和反刍动物与人的识别率有所增加,而蝙蝠有所下降。
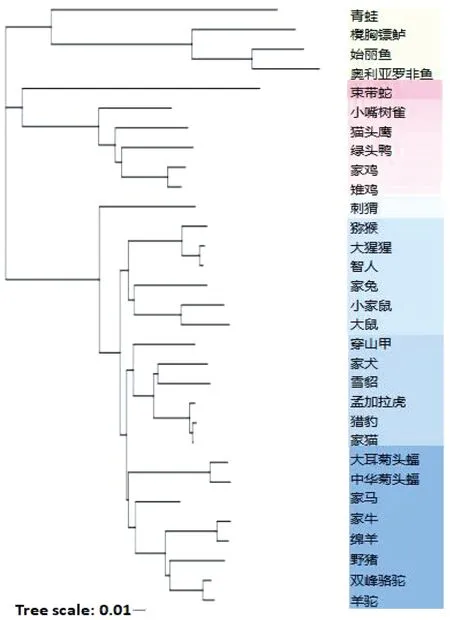
图1 AEC2氨基酸序列系统发育树(NJ法,Bootsrtap1000个循环,n=31)Fig.1 Phylogenetic tree of AEC2 amino acid sequences (NJ method, Bootsrtap1000 cycles, n=31)

图2 AEC2-PD结构域氨基酸序列系统发育树(NJ法,Bootsrtap1000个循环,n=124)Fig.2 Phylogenetic tree of amino acid sequences in ACE2-PD domain (NJ method, Bootsrtap1000 cycles, n=124)
2.3不同物种间ACE2与S蛋白直接相互作用接口氨基酸对比 如表1,相对于人类ACE2的20个残基,猕猴与人类完全一样。家牛和家羊中的这20个残基的变异性仅为3个氨基酸。家猫、双峰骆驼和单峰骆驼中,在该位置有5个氨基酸的差异。蝙蝠的这20个氨基酸中有5个氨基酸与人类不同,而穿山甲的为7个。此外,家犬、野猪和家鼠在该位置的突变分别为6、6、8个氨基酸,而在禽类,如绿头鸭、家鸡以及罗非鱼中的变异性均大于9个氨基酸。
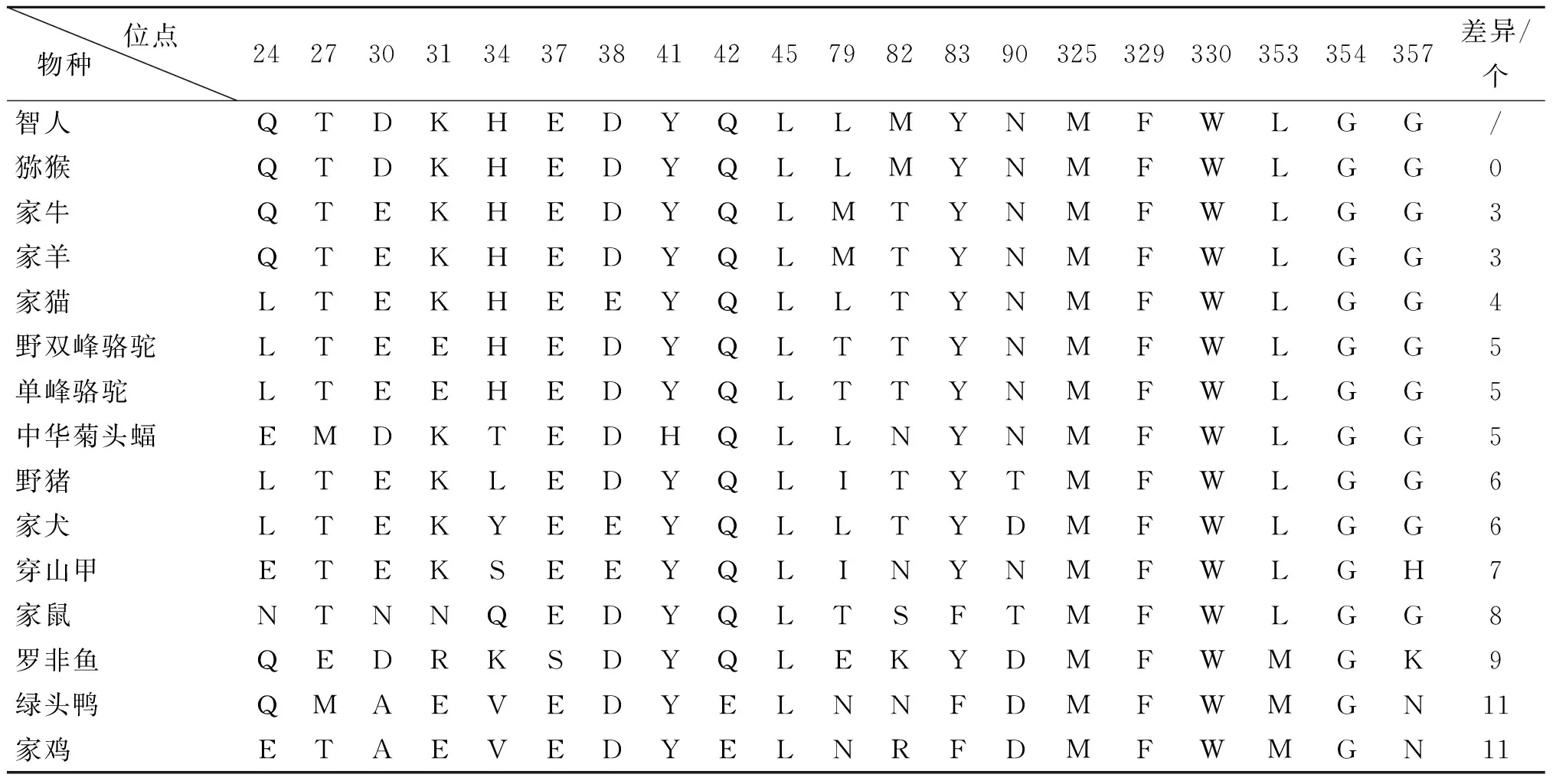
表1 ACE2中与SARS-CoV及2019-nCoV RBD接口的20个氨基酸在不同物种中的保守性比较Tab.1 Comparison of conservation of 20 amino acids in ACE2 interface with SARS-CoV and 2019-nCoV RBD in different species
3 讨 论
ACE2在动物界中广泛表达,在哺乳动物中具有保守的一级结构,ACE2的这种保守性可能反映了其在肾素-血管紧张素系统中的重要作用。ACE2受体与病毒S蛋白RBD直接作用的接口氨基酸中,除部分灵长类动物与人类完全相同外,其他物种均有一定程度的不同(表1)。然而先前的研究表明S蛋白RBD与ACE2相互作用可以“容忍”受体中一定程度的变异。例如,在与SARS-CoV病毒S蛋白RBD相互作用的18个ACE2残基中,猕猴、仓鼠、猫的受体在该区域含有≤4个氨基酸的变异,但仍可被SARS-CoV所识别;而果子狸和浣熊ACE2受体中观察到了大于7个氨基酸的替代,还可被SARS-CoV所识别[11]。
潜在的动物模型:非人灵长类动物与人类ACE2氨基酸全长序列以及ACE2-PD氨基酸序列的同源性最为接近,猕猴与人类ACE2的20个残基完全一样。这提示非人灵长类动物可以作为研究2019-nCoV的良好动物感染模型。事实上,灵长类动物模型如猕猴[13-15]、食蟹猴[16],感染2019-nCoV显示出与人类相似的症状,包括病毒载量的变化、肺部CT影像,研究者们已经使用这些动物模型进行疫苗和药物的筛选实验[17]。氨基酸序列分析表明啮齿类动物与人类同源性高,仅次于非人灵长类动物。小型啮齿类动物具有容易饲养,维护成本低等优点,是医学研域最常用的模型。然而,2019-nCoV在大鼠和小鼠中的复制效率很低,可能是由于小鼠ACE2受体在353位含有赖氨酸到组氨酸的突变,导致缺乏赖氨酸残基所产生的关键亲水相互作用[11];大鼠ACE2也含有这种K353H突变,并且在N82处具有潜在的N-糖基化位点,它消除了受体对SARS-CoV-S的结合能力[11]。我们的研究(表1)也表明家鼠在接口氨基酸突变较多(8个),不太可能通过ACE2受体感染。值得关注的是,金黄色西伯利亚仓鼠模型[18]和人类ACE2转基因小鼠模型[19-20]已显示出了希望的前景。
潜在的中间宿主:有文献报道蝙蝠和穿山甲可能为2019-nCoV的中间宿主[21],因为在这些动物体内发现了与感染人类的2019-nCoV同源性很高(>95%)的冠状病毒[2, 22],但由于不能确定感染的时间顺序,仍存在争议。本文的研究显示穿山甲的20个接口氨基酸中有7个与人类不同,变异性较大。以往的研究表明蝙蝠进化出了一些特殊的免疫机制,机体不会产生强烈的炎症反应以保证免疫系统不对病毒入侵采取过激反应,以达到和多种病毒如埃博拉病毒、狂犬病病毒、MERS-CoV、SARE-CoV等和平相处的状态,是多种病毒的储存库[23]。相比于ACE2氨基酸全长序列分析,蝙蝠与人的识别率在ACE2-PD结构域序列分析中略有下降,20个接口氨基酸中蝙蝠有5个变异。因此,还需要提供更多证据来证明穿山甲和蝙蝠是否为2019-nCoV的中间宿主。
潜在的易感动物:我们的分析表明猫科动物和奇/偶蹄目动物与人类ACE2的一级结构具有较高的同源性。在家猫,双峰骆驼和单峰骆驼(中东呼吸综合征病毒(MERS-CoV)的宿主)中,接口氨基酸只有5个差异,从而提示这些动物有较大感染的可能性。最近,石正丽团队调查了与人类密切接触的动物和雪貂对2019-nCoV的敏感性,发现2019-nCoV在狗、猪、鸡和鸭中的复制能力很差,但在雪貂和猫中却能有效复制,并可以通过呼吸道飞沫在猫之间传播[4]。该团队进一步检测了武汉市COVID-19暴发后的102份猫血清样本,表明在暴发期间已感染武汉的猫群,COVID-19患者拥有的3只猫的中和抗体滴度最高[5]。2020年4月6日美国农业部动植物卫生检验局确认动物园中一只圈养老虎2019-nCoV核酸检测呈阳性,并出现干咳和食欲下降等症状。猫、雪貂、老虎同属于猫科动物,可能是2019-nCoV的易感动物,这与我们的分析结果相吻合(表1)。考虑到猫是与人类接触非常密切的伴侣动物,因此应该引起格外关注。同样值得关注的是,在家牛和家羊中的这20个残基的变异性仅为3个氨基酸,与Joana Damas等[24]的研究相符合,提示反刍动物可能也有利于2019-nCoV的跨物种传播,然而目前的研究却很少关注这类动物。因此,我们建议有关部门应开展易感动物的病原学检测。
其他动物:禽类,如绿头鸭、家鸡以及罗非鱼中的变异性较大,提示与2019-nCoV病毒S蛋白的结合效率可能相对较低,因此这些家禽类动物感染此类病毒的可能性较低。
新冠病毒造成的全球大流行给人类健康和经济发展造成了巨大影响,目前仍无有效的特效药物和预防性疫苗。不同物种间ACE2的同源性分析对建立2019-nCoV动物感染模型、跨物种传播、保护潜在易感动物等提供理论依据。目前,已报了6种有希望的SARS-CoV-2动物感染模型,包括人ACE2转基因小鼠模型[19-20]、金黄色西伯利亚仓鼠模型[18]、雪貂模型[4, 25]、猕猴模型[13-15]和食蟹猴模型[16]等。这些动物模型的开发对于疫苗的评价和药物的筛选至关重要。近年来暴发的一系列新发和再发人兽共患病,严重威胁着人类健康。然而,当前对于动物流行病学的常态化监测和风险评估仍相对滞后。因此,积极推进动物流行病学分析,不仅有助于深刻理解病原体与不同物种间的关系从而为溯源工作奠定基础,也将对建立人类-动物-生态的同一健康理念(One Health)的全方位新型公共卫生防御体系具有重要的指导意义。
利益冲突:无

