优化多核SVM的蛋白质二级结构预测
刘斌 温雪岩
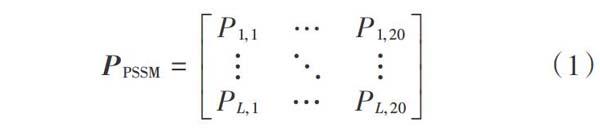
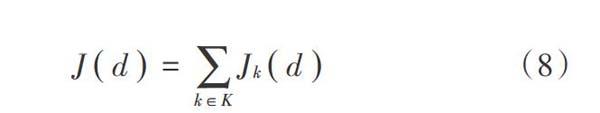

摘 要: 蛋白质序列的不同特征提取方式对蛋白质结构分类有很大的影响。为更好地表达蛋白质结构信息,基于特征融合思想构建特征向量,并使用一种基于多核支持向量机的方法,以多个核函数的线性加权代替传统的单一核函数,在对多类特征进行整合后构造SimpleMKL分类模型;利用梯度下降法迭代求解核函数的权值系数,并校准核函数参数和不同特征表达的融合效果。实验结果表明,该方法提高了蛋白质二级结构分类精度,在分类精度方面有明显优势,有助于准确预测蛋白质的二级结构。
关键词: 蛋白质; 二级结构预测; 多核支持向量机; 特征提取; 特征融合; 線性加权
中图分类号: TN911?34 文献标识码: A 文章编号: 1004?373X(2020)08?0139?04
Protein secondary structure prediction based on optimized multi?kernel SVM
LIU Bin, WEN Xueyan
(College of Electronic Information and Artificial Intelligence, Shaanxi University of Science and Technology, Xian 710021, China)
Abstract: The different feature extraction methods of protein sequence have great influence on protein structure classification. For better expression of protein structure information, the feature vectors are constructed based on feature fusion idea, and the traditional single kernel function is replaced by the linear weighting of multiple kernel functions by means of the method based on multi?kernel SVM (support vector machine); the SimpleMKL classification model is constructed after integrating the multi?class features, the weight coefficient of kernel function is solved iteratively by means of the gradient descent method, and the fusion effects of kernel function parameters and different feature expressions are calibrated. The experimental results show that the proposed method improves the classification accuracy of protein secondary structure, and has obvious advantages in classification accuracy, which is helpful to accurately predict the secondary structure of protein.
Keywords: protein; secondary structure prediction; multi?kernel support vector machine; feature extraction; feature fusion; linear weighting
通过模式识别和机器学习方法来获得蛋白质的结构信息是生物信息学的基本任务。SVM已被广泛用于蛋白质二级结构的预测[1],为扩展SVM分类器性能,有研究利用多层SVM方法增强了单个SVM方案的预测[1]。有研究基于改进模糊支持向量机的预测蛋白质二级结构的新方法[2],通过将位置特定信息和非位置特定信息与更好的核函数相结合,改进了基于SVM的预测[3]。该研究中提出的SVM?PHGS规范SVM核函数的参数,动态校准了不同核函数的融合结果,提高了分类精度,通过多次调整权重来求解最优核参数,其中核权重是线性加权的。但当数据维度过高时,已有文献研究成果的SVM求解速度会受到严重影响。
针对上述问题,本文使用一种自适应的L2范数正则化方法来考虑多核学习问题,即SimpleMKL算法,该算法将核矩阵定义为多个内核的线性组合,可以解决多特征分类问题[4]。和其余多核学习算法比较不同之处在于,SimpleMKL以多个核函数的线性加权代替传统的单一核函数,通过加权L2范数正则化公式解决多核学习问题,并对权重进行额外约束,以鼓励稀疏内核组合,利用梯度下降法不停迭代求解核函数的权值系数,最终达到提高算法的收敛速度与分类精度的目的。有效地探索来自多个非线性特征空间的补充信息对多特征融合的蛋白质序列分类具有较好的适用性。
本文通过对比多类别蛋白质特征向量,尝试不同参数的核函数的线性加权组合,并整合多类特征向量后构造SimpleMKL分类模型。使用SimpleMKL分类模型实现蛋白质结构类型的分类判定,并通过实验证明该模型具有较好的分类精度。
1 蛋白质序列的特征提取
氨基酸是蛋白质的基本组成单元,构成蛋白质的氨基酸主要有20种(A,C,D,E,F,G,H,I,K,L,M,N,P,Q,R,S,T,V,W和Y )。蛋白质二级结构预测是指将一个由 20 种氨基酸组成的序列映射为相应的结构标签序列。氨基酸序列一般是以字符串的形式存储在各大生物數据库中,其表示形式为:[P=R1R2…Ri…RN,Ri∈{A1,A2,…,A20}],[Ri]表示蛋白质的第i个氨基酸残基,蛋白质序列对应的目标二级结构可由[Y=y1y2…yi…yN,yi∈{H,E,C}]表示,其中[yi]是处于第i个位置的氨基酸残基的二级结构类型。
常用的特征表示方法包括位置特异性评分矩阵(Position?Specific Scoring Matrices,PSSM)、氨基酸组成特征(Amino Acid Composition,AAC)和理化性质特征(Physicochemical Property,PP)。位置特异性评分矩阵蕴含进化信息,蛋白质编码特征考虑了蛋白质序列的组成信息,使用氨基酸的理化特性来构建特征表达模型,可以把蛋白质序列中氨基酸的位置信息和不同距离氨基酸间的相互作用包含进去,蛋白质的结构信息能够更好地反映出来。PSSM特征在以往的研究中已用于蛋白质结构和性质预测,并获得了较好的预测效果[5?6]。特征向量构造方法具体如下:
位置特异性评分矩阵(PSSM):运行PSI?BLAST 程序处理序列数据来生成PSSM,参数配置为0.001的E值阈值和3次迭代以搜索UniRef90。该矩阵包含重要的进化信息,具有20×L个元素(20列和L行),其中L是目标序列的长度,序列中每个氨基酸对应一个20维的向量。矩阵的第(i,j)位置上的数值表示蛋白质序列的第i个位置上的氨基酸在进化过程中突变为氨基酸j的可能性得分。
氨基酸理化性质特征常用于蛋白结构和性能预测。氨基酸的8种理化特性包括溶解性、酸碱性质、亲水疏水性、侧链质量等。序列中每个氨基酸对应一个8维的向量。构成蛋白质多肽链的基本氨基酸类型有20种,由于蛋白质序列中可能存在一些未知的氨基酸,在此使用21个元素的独热编码来指示序列中某一位置的氨基酸类型。文中,将来自不同源的特征连接成单个特征向量。训练数据被归一化为具有零均值和单位方差的数据,1个氨基酸残基由49维特征(20维PSSM、8维物理特性和21维蛋白质编码信息)表示,对应的标签是三种结构标签,如表1所示。
为了表示目标氨基酸附近其他氨基酸的特征信息,选择指定单位长度的滑动窗口来提取特征。文献[7]分析了输入特征滑动窗口大小对预测精度的影响,基于蛋白质一级序列,本文选用13个单位的滑动窗口,以每个目标氨基酸为中心,通过在序列上滑动提取窗口范围内的所有氨基酸的以上3种特征。对蛋白质序列两端超出序列范围的窗口位置,其特征向量用零向量代替,即一个氨基酸残基由637维特征(13×(20PSSM+8PP+21AAC))表示。基于此种特征融合方法构建的特征表达模型不仅可以包含蛋白质序列的组成信息,还可以表现出氨基酸的位置信息和相互作用,极大地丰富了特征表达模型中包含的蛋白质序列信息。
2 多核SVM分类模型
支持向量机(SVM)可以通过内核嵌入将输入数据映射到非线性内核空间来建模非线性数据分布,利用非线性映射使数据线性可分,因此核函数是关键。对于蛋白质二级结构预测问题,有研究表明:不同的核函数可以显着改变预测结果,单个内核预测二级结构准确度较低[3]。设计一种融合多个内核空间的方法可以提高PSSP的准确性。然而,用于特定任务的内核的最合适的类型和参数通常是未知的,并且通过穷举搜索来选择最佳内核通常是耗时的,并且有时导致过度拟合。多核学习(MKL)通过加权线性组合学习最优核,引入了预定义的候选内核,并对它们的组合内核进行训练,来学习分类器最佳模型参数。
[xi]表示蛋白质序列第i个位置的特征向量时,用 [(x1,…,xi,…,xL)]表示长度为L的蛋白质序列,将输入数据映射到非线性内核空间后,训练模型以寻找最优核组合。多核学习中的合成核可以表示为多个核函数的加权和,如下:
式中:[dm]≥ 0;[m=1Mdm=1];M为核的个数。决策函数可以表述为:
式中:[α*i]为要学习的系数拉格朗日乘子;[b*]为分离超平面的偏差项;[Kmx,xi] 为第m个核矩阵;[d*m ] 为第m个核矩阵的权重。多核学习的原始目标函数为:
式中:w 是分离超平面的法线;b是偏差项;[ξi]是松弛变量的向量;C是误分类惩罚系数。SimpleMKL算法选择迭代方法确定权系数,求解采用梯度下降法。在式(4)的基础上,SimpleMKL 中的约束优化问题为:
通过简单地推导式(5)中给出的关于[dm]的对偶函数,得:
d可通过梯度下降来更新,更新方案如下:
式中:[Dt]是梯度下降方向的向量;[rt]为步长。可以通过求解该优化问题来得到多核分类器模型参数最优权重d,该方法用核权重的求解与选择来解决核矩阵的表示问题。
SimpleMKL二分类算法可以描述为:
1) 核权重[dlm]初始化:[dlm=1/M],其中M为内核个数。
2) 每一次迭代时利用组合内核来计算目标函数[Jd]。
3) 计算[Jd]对d的偏导、梯度、梯度方向[Dt]和最优步长[rt]。
4) 由式(7)更新d,再用新的d更新核矩阵、梯度、梯度方向[Dt]、最优步长[rt]。
5) 如果不满足迭代终止条件,则返回到步骤2),并重复步骤2)~步骤4);如果满足迭代终止条件,则结束计算。
用SimpleMKL解决多分类问题时,通过组合多个二分类器解决。目标函数可由每个二分类器目标函数求和得到,即:
式中:[Jkd]是第k个二分类器的目标函数值;K表示多个二分类器的集合。考虑到蛋白质序列分类模型的泛化推广能力,本文选用“一对多”方式构造多分类器。
在多核框架下,将样本在特征空间中的表示问题转化为基本核与权系数的选择问题。多核学习的目标是通过最优化方法来求取合成核的参数,为了获得最佳分类精度,多核学习通过求解单个联合优化问题来学习核组合的权重和分类器的参数。
3 实 验
3.1 实验数据
蛋白质二级结构预测为典型的多分类问题,基于其序列特征来判定序列样本的类别。在此使用公开可用的基准数据集RS126和CASP9来训练和测试模型,数据集中每个样本都包含蛋白质序列和结构标签序列。RS126数据集具有126个蛋白质序列,包含26 846个残基,CASP9数据集包含总共24 395个氨基酸残基。模型训练过程中,使用5折交叉验证法,将最好的训练结果进行比较。一轮交叉验证过程完成后,样本被随机分为5个子集,轮流以其中4 个子集的集合作为训练集,最后一个子集作为测试集。
3.2 实验环境
实验所用主机操作系统为WIN7旗舰版64位,CPU型号为Intel? Xeon? Silver 4116 CPU,主频为2.10 GHz,内存为128 GB。
3.3 参数设置
SVM候选内核采用3种常见核函数,包括径向基核、多项式核和Sigmoid核。综合考虑计算机性能和数据规模,使用13个不同类型和不同参数的核函数构成多核。其中包括线性核、2到3维的多项式核和σ属于[0.5,1,2,5,7,10,12,15,17,20]的高斯核。在多核SVM一对多的分类方法中,对于正则化参数C,在区间[0.01,100]上通过普通网格搜索算法来优化参数,得到的最佳参数值为C=0.67。然后使用这13个基核将输入向量映射到一个新的特征空间。本文实验分别对比多类组合内核的分类精度。通过K?means聚类来加速支持向量机训练,SimpleMKL算法的求解由SVM?KM程序来实现。
3.4 评价指标
本文使用蛋白质结构分类预测领域的度量来评估每个蛋白质二级结构分类器的性能,即整体准确率和三态准确率[1]。
3.5 实验结果分析
多次迭代得到一个基于最佳核权重的组合核分类器,并选择每类核函数中最高的核权重作为最终参数值。表2中权系数一栏列出三类核函数的最高核权重参数,参与训练的其他大多是核函数的权重为零,也体现出SimpleMKL算法鼓励稀疏内核组合。模型训练结束后,核矩阵参数显示多个核矩阵加权组合的多核矩阵中只有少量核权重参与学习。为检验提出的多核核函数方法在蛋白质结构分类中的效果,将不同核函数组合的多核分类结果进行比较,结果如表3所示。
所有输入向量进行多核融合训练,不同核函数的组合内核分类性能相差较大,训练不同的候选内核改善了分类结果并补偿误差。由实验结果可看出,通过融合多个不同核矩阵可以明显提升SVM分类器的性能。各类扩展SVM算法分类性能比较如表4所示。
从表4中可以看出,基本核分类器用多核学习方法加以优化可改进其分类效果。针对蛋白质二级结构分类,多个基础核适当线性组合,每个核与特定输入变量相关联,生成的内核是不同输入内核的加权组合,来自不同内核的补充信息被集成以获得更好的准确性。
实验结果证明,相对单核预测,多核方法在与其他扩展SVM算法相比,在分类精度方面有明显优势,多特征组合的分类效果比单一特征的分类效果更好。正确融合互补的特征信息并使用多核学习方法能明显提升SVM的性能。
4 结 语
本文提出了基于多核学习和特征融合的蛋白质二级结构分类方法,将多核学习方法SimpleMKL应用于蛋白质二级结构,并扩展了蛋白质二级结构预测的输入特征,在PSSM特征提取的基础上,通过氨基酸组成和理化特性编码反映出氨基酸之间的长程作用。通过训练多核分类器模型,证明了不同内核的融合使得内核分类效果彼此互补,从而提高结果的准确性。本文的蛋白质二级结构分类方法与其他扩展SVM算法具有相近的分类精度,也可用于分析蛋白质序列特征信息与二级结构类型之间的关联作用。但多核模型训练过程中调整内核参数所涉及的计算成本较大,值得进一步研究蛋白质序列复杂特征的简化。
参考文献
[1] QIAN Jiang, XIN Jin, SHIN?JYE Lee, et al. Protein secondary structure prediction: A survey of the state of the art [J]. Journal of molecular graphics and modeling, 2017(76): 379?402.
[2] XIE S X, LI Z, HU H L. Protein secondary structure prediction based on the fuzzy support vector machine with the hyperplane optimization [J]. Gene, 2018, 642: 74?83.
[3] ZANGOOEI M H, JALILI S. PSSP with dynamic weighted kernel fusion based on SVM?PHGS [J]. Knowledge?based systems, 2012, 27: 424?442.
[4] LI Z F, TONG X. Modeling and parameter optimization for vibrating screens based on AFSA?SimpleMKL [J]. Chinese journal of engineering design, 2016(2): 12.
[5] 郭延哺,李维华,王兵益,等.基于卷积长短时记忆神经网络的蛋白质二级结构预测[J].模式识别与人工智能,2018,31(6):562?568.
[6] 李强,郑宇杰.基于多视角特征融合与随机森林的蛋白质结晶预测[J].现代电子技术,2015,38(8):50?53.
[7] BOUZIANE H, MESSABIH B, CHOUARFIA A. Effect of simple ensemble methods on protein secondary structure prediction [J]. Soft computing, 2015, 19(6): 1663?1678.
[8] KOUNTOURIS P, AGATHOCLEOUS M, PROMPONAS V J, et al. A comparative study on filtering protein secondary structure prediction [J]. IEEE/ACM transactions on computational biology & bioinformatics, 2012, 9(3): 731?739.
[9] YUAN Mingshun, YANG Zijiang, HUANG Guangzao, et al. A novel feature selection method to predict protein structural class, computational [J]. Biology and chemistry, 2018(76): 118?129.
[10] 李红丽,许春香,马耀锋.基于多核学习SVM的图像分类识别算法[J].现代电子技术,2018,41(6):50?52.

