凡纳滨对虾连续3个世代选育群体的遗传多样性分析
彭敏 陈慧芳 李强勇 杨春玲 曾地刚 刘青云 赵永贞 陈晓汉 林勇 陈秀荔

摘要:【目的】明确凡纳滨对虾连续3个世代选育群体的遗传变异情况及近亲繁殖对个体生长速度和抗病性的影响,为凡纳滨对虾种质改良提供理论依据。【方法】从GenBank收录的微卫星(SSR)引物序列中筛选11对能有效扩增的SSR引物,对2016—2018年桂海1号凡纳滨对虾连续3个世代选育群体(合计853个样品)进行遗传多样性分析,监测相邻2个世代选育群体间的遗传变异情况。【结果】11对SSR引物在凡纳滨对虾连续3个世代选育群体中共检测到103个等位基因,各SSR位点的等位基因数(Na)平均为9.3636个,有效等位基因数(Ne)平均为3.8355个,观测杂合度(Ho)平均为0.4794,期望杂合度(He)平均为0.7074,多态信息含量(PIC)平均为0.6708;除TUMXLv10.33位点呈中度多态性(0.25
关键词: 凡纳滨对虾;选育群体;微卫星(SSR)分子标记;遗传多样性;遗传变异
Abstract:【Objective】To clarify the genetic variation of three consecutive generations of Litopenaeus vannamei and the influence of inbreeding on individual growth rate and disease resistance, and provide a theoretical basis for the improvement of L. vannamei germplasm. 【Method】Using 11 pairs of effectively amplified SSR primers selected from the sequence of microsatellite(SSR) primers collected by GenBank, the genetic diversity of L. vannamei Guihai No.1 breeding population for three consecutive generations (2016-2018) (853 samples) was analyzed to monitor the genetic diversity between two adjacent generation breeding populations. 【Result】Using the 11 pairs of SSR primers, 103 alleles were detec-ted in the three consecutive L. vannamei generations. The average number of alleles(Na) at each SSR locus was 9.3636, and the average number of effective alleles(Ne) was 3.8355, the average observed heterozygosity(Ho) was 0.4794, the average expected heterozygosity(He) was 0.7074, and the average polymorphic information content(PIC) was 0.6708; except that TUMXLv10.33 locus was moderately polymorphic(0.25
Key words: Litopenaeus vannamei; breeding population; microsatellite(SSR) molecular marker; genetic diversity; genetic variation
0 引言
【研究意义】凡纳滨对虾(Litopenueus vannamei)又称南美白对虾,原产于秘鲁至墨西哥太平洋沿岸,1988年中国科学院海洋研究所首次引进凡纳滨对虾,并于1992年突破人工繁殖技术难关,之后凡纳滨对虾在全国范围内大面积推广养殖,现已发展成为我国对虾养殖的主导品种(王兴强等,2004;陈锚等,2008;黄薇等,2014)。但目前我国凡纳滨对虾养殖的良种覆盖率较低,大多数对虾养殖场使用的虾苗是从美国进口杂交种的数代甚至十几代孵化苗种,其生长性能和抗逆性严重退化,虾病发生率高,导致对虾养殖经济效益急剧下降。加之世界各国对知识产权保护意识的增强,严格限制原种亲虾出口,致使我国凡纳滨对虾养殖业面临严峻挑战。因此,亟待我国科学家开展凡纳滨对虾品种选育,培育出具有自主知识产权的优良品种。【前人研究进展】微卫星(SSR)分子標记具有简便、快速、稳定性好等优点,在水产生物的亲缘关系鉴定、系统发育进化分析、种群遗传结构、遗传连锁图谱构建及遗传变异等研究领域发挥着重要作用(Wolfus et al.,1997;Valles-Jimenez et al.,2004;Zhang et al.,2007;王霞等,2009;董在杰等,2010)。至今,有关SSR分子标记在凡纳滨对虾上的应用研究已有较多报道。童馨等(2009)采用SSR分子标记对凡纳滨对虾4个世代7个养殖群体进行遗传多样性分析;谢丽等(2009)对凡纳滨对虾4个选育群体进行遗传多样性SSR分析;马春燕等(2011)基于8个高多态性的SSR位点分析凡纳滨对虾引进亲虾群体及引进亲虾子一代、子二代群体的遗传变异情况;包秀凤(2014)采用7对SSR引物对国内3个养殖群体及4个引进亲虾子一代群体的遗传多样性进行分析;Zhang等(2014)利用7个SSR分子标记分析从新加坡和美国引进凡纳滨对虾7个群体的种质特性;吴怡迪等(2016)采用SSR分子标记对凡纳滨对虾4个群体和1个美国引进群体的遗传结构和遗传多样性进行分析,旨在监测各凡纳滨对虾群体的遗传变异情况;杨铭等(2017)基于转录组数据开发出凡纳滨对虾SSR分子标记,进而对凡纳滨对虾进行遗传多样性分析;李东宇等(2017)对凡纳滨对虾选育群体与杂交群体进行遗传多样性分析,同时利用13个SSR分子标记对2个子代群体进行遗传多样性及遗传分化研究,结果表明杂交群体较选育群体具有更丰富的遗传多样性;刘洪涛等(2018)选用15对多态性较高的SSR引物对凡纳滨对虾8个地理群体的遗传多样性和亲缘关系进行分析,以期为海南凡纳滨对虾的育种提供科学依据;陈锦豪等(2019)选用8对SSR引物对厦门市凡纳滨对虾5个亲虾群体进行遗传多样性分析,旨在明确凡纳滨对虾亲虾群体的遗传结构、种质资源状况及近交程度,为该地区凡纳滨对虾种质资源的提纯、保优、复壮及遗传选育提供参考依据;黄小帅等(2019)利用SSR分子标记分析凡纳滨对虾7个引进群体子一代的遗传多样性,为后续挖掘种苗遗传背景与其养殖性能的关联性提供理论参考。【本研究切入点】在人工培育优良品种的过程中,利用分子标记监测相邻2个世代间的遗传变异,或监控近交对个体生长率和抗病力的影响,对保护凡纳滨对虾遗传多态性、防止种质资源退化,以及明确育种亲本间的亲缘关系具有重要意义。其中,利用SSR分子标记对群体间进行遗传多样性和遗传结构差异性分析,不仅有助于对选育群体进行遗传多样性监测,还对亲本群体选择具有指导作用(Cruz et al.,2004)。广西水产科学研究院在引进美国凡纳滨对虾原种的基础上,通过近十年的群体继代和家系选育,选育出桂海1号凡纳滨对虾耐寒家系(赵永贞,2014),但至今鲜见针对其连续世代选育群体进行遗传多样性分析,育种亲本间的亲缘关系尚未明确。【拟解决的关键问题】以广西水产科学研究院凡纳滨对虾育种中心选育建立的凡纳滨对虾耐寒家系为试验材料,利用11对SSR引物对8个耐寒家系2016—2018年连续3年的凡纳滨对虾选育亲本进行群体遗传多样性分析,监测相邻2个世代选育群体间的遗传变异情况及近亲繁殖对个体生长速度和抗病性的影响,为凡纳滨对虾种质改良提供理论依据。
1 材料与方法
1. 1 样品来源及其基因组DNA提取
凡纳滨对虾样本来源于广西水产科学研究院凡纳滨对虾育种中心2016—2018年用于选择育种的亲本群体,分别属于3个世代,合计853个样品(尾)。剪取凡纳滨对虾肌肉组织,经液氮速冻后置于-80 ℃冰箱保存备用。采用醋酸铵法提取凡纳滨对虾肌肉组织DNA:称取肌肉组织100 mg,加入600.0 μL DNA提取缓冲液,剪碎后加入12.0 μL 20 mg/mL蛋白酶K(终浓度400 μg/mL),充分混匀后放入56 ℃水浴锅中温浴5 h或过夜;取出冷却至室温,加入200.0 μL 7.5 mmol/L醋酸铵溶液,颠倒混匀2 min,14000 r/min离心5 min,收集上清液,装入新的离心管中。加入等体积预冷异丙醇,-80 ℃静置15 min后14000 r/min离心5 min,沉淀用70%酒精洗涤2次;自然干燥后,加入100.0 μL灭菌ddH2O,加入1.0 μL 10 mg/mL胰RNA酶,37 ℃水浴30 min。采用1.0%琼脂糖凝胶电泳检测DNA完整性,以蛋白核酸分析仪检测其浓度,并稀释至100 ng/μL,-20 ℃保存备用。
1. 2 SSR引物筛选及合成
根据本课题组的前期研究(彭敏等,2014),从GenBank收录的14对SSR引物中选取11对能有效扩增的SSR引物(表1),用于凡纳滨对虾连续3个世代选育群体的遗传多样性分析。其中,上游引物5'端分别加FAM、HEX和TAMAR等荧光标记。所有SSR引物均委托生工生物工程(上海)股份有限公司合成。
1. 3 PCR扩增及多态性分析
PCR反应体系20.0 μL:2×ES Taq MasterMix 5.0 μL,10 μmol/L上、下游引物各0.4 μL,DNA模板2.0 μL,超纯水补足至20.0 μL。扩增程序:94 ℃预变性3 min;94 ℃ 45 s,48~62 ℃(表1)45 s,72 ℃ 45 s,进行35个循环;72 ℃延伸5 min。PCR扩增产物送至北京基诺博实生物技术有限公司,采用ABI 3130自动测序分析仪GeneScan进行测序分析。在进行大样本测序分析前,每对SSR引物扩增少量样品进行预测序分析。
1. 4 统计分析
采用GeneMarker 3.0.0对测序结果进行分析,记录各SSR位点不同个体的等位基因和基因型。使用PopGene 1.32计算每个SSR位点的等位基因频率(Allele frequency)、等位基因数(Observed number of alleles,Na)、有效等位基因数(Effective allele number,Ne)、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)及Hardy-Weinberg平衡遺传偏离概率(PHWE),根据Nei的方法计算不同群体间的遗传距离、遗传分化系数(Fst)及近交系数(Fis),并以PIC-Calc 0.6对获得的等位基因频率进行多态信息含量(Polymorphism information content,PIC)计算(Nagy et al.,2012)。
2 结果与分析
2. 1 11对SSR引物的特异性扩增结果
采用11对SSR引物对桂海1号凡纳滨对虾DNA混池进行PCR扩增,琼脂糖凝胶电泳结果显示,均能扩增出特异性的目的条带(图1),与预期结果相符。
2. 2 凡纳滨对虾遗传多样性分析结果
11对SSR引物在凡纳滨对虾连续3个世代选育群体中共检测到103个等位基因,各SSR位点的Na为6~14个,平均为9.3636个;Ne为1.9734~4.9414个,平均为3.8355个;Ho为0.1040~0.8070,平均为0.4794;He为0.4935~0.8647,平均为0.7074;PIC为0.4641~0.8509,平均为0.6708(表2)。
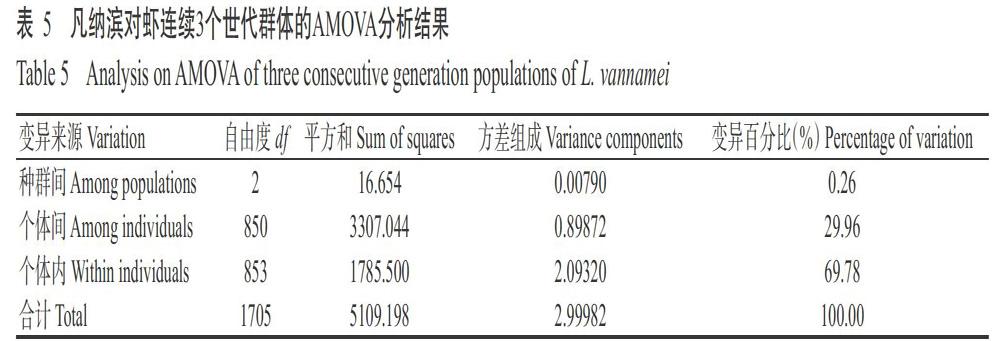
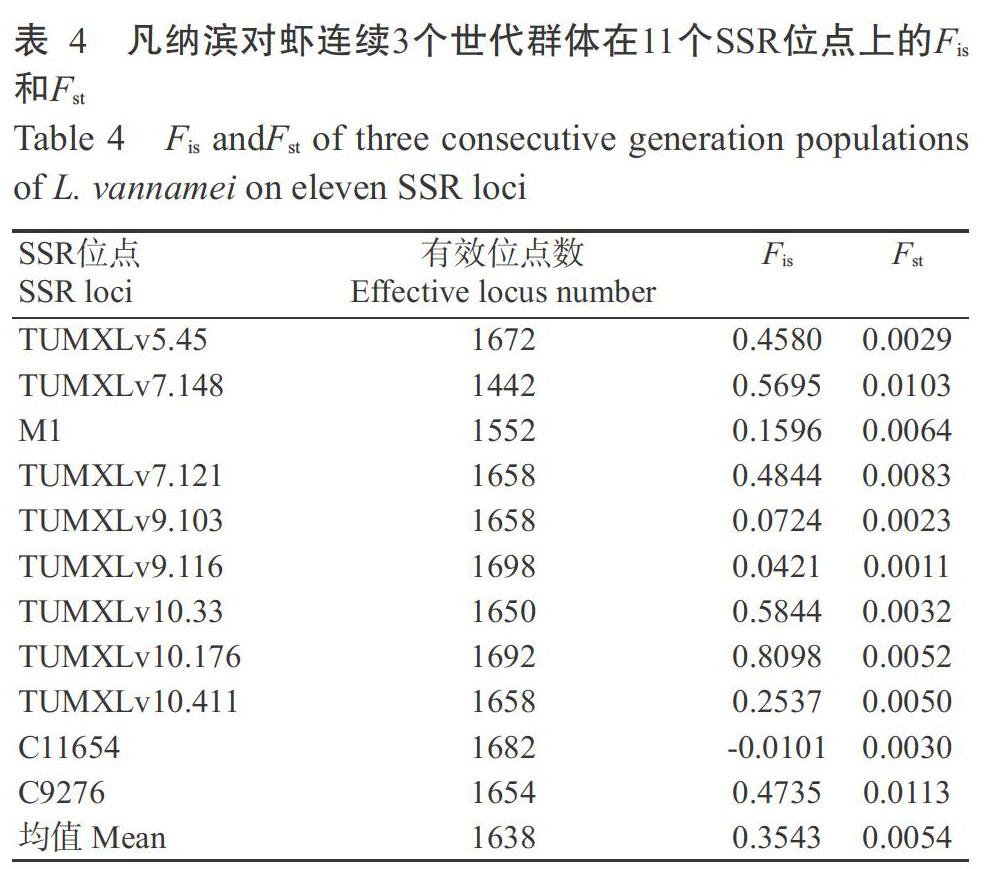
凡纳滨对虾连续3个世代选育群体在11个SSR位点上的遗传多态性分析结果(表3)表明,2016年选育群体的Na为5~13个,平均为8.7272个;Ne介于1.8121~7.1520个,平均为3.8676个。2017年选育群体的Na为5~11个,平均为7.3636个;Ne介于2.1180~7.5242个,平均为3.7654个。2018年选育群体的Na为5~12个,平均为8.0000个;Ne介于1.9335~7.0199个,平均为3.8010个。根据Botstein等(1980)的判断标准,当PIC>0.50时,为高度多态性;当0.25 2. 3 凡纳滨对虾遗传分化分析结果 凡纳滨对虾连续3个世代选育群体在不同SSR位点上的遗传分化参数见表4。其中,凡纳滨对虾连续3个世代选育群体在11个SSR位点上的Fst为0.0011~0.0113,均小于0.0500,表明凡纳滨对虾群体的遗传分化很小;凡纳滨对虾群体内Fis介于-0.0101~0.8098,平均为0.3543,且除C11654位点为负值外,其余SSR位点均为正值,表明凡纳滨对虾连续3个世代选育群体的近交程度较高。 在凡纳滨对虾连续3个世代选育群体中,2016年选育群体与2017年选育群体和2018年选育群体间的Fst分别为0.0056和0.0037,其遗传分化均达极显著水平(P<0.01);2017年选育群体与2018年选育群体间的Fst为0.0007,遗传分化差异不显著(P>0.05)。对凡纳滨对虾连续3个世代选育群体进行分子方差分析(AMOVA),结果发现69.78%的遗传变异来源于凡纳滨对虾个体内,而29.96%的变异来源于凡纳滨对虾个体间(表5),即凡纳滨对虾个体内的遗传变异大于个体间的遗传变异。 2. 4 UPGMA聚类分析结果 基于个体间的遗传距离,通过PHYLIP v3.5对2016年选育群体(234尾凡纳滨对虾)进行UPGMA聚类分析,结果表明234个样品间的最大遗传距离为3.5835,最小遗传距离为0.0000,平均为1.4894。2017年选育群体(341尾凡纳滨对虾)的UPGMA聚类分析结果表明,341个样品间的最大遗传距离为3.6606,最小遗传距离为0.0000,平均为1.0758。2018年选育群体(278尾凡纳滨对虾)的UPGMA聚类分析结果表明,278个样品间的最大遗传距离为3.6632,最小遗传距离为0.0000,平均为1.0099。 3 讨论 遗传多样性是物种适应复杂多变环境、维持生存和进化的基础,遗传变异程度直接影响物种进化潜力及其对环境变化的适应能力(O'Connell and Wright,1997;张文静等,2003;崔蕾等,2012)。Na、PIC及遗传杂合度(H)等遗传参数均是检测种群遗传多样性的主要指标(颉晓勇等,2008;刘海情等,2012;李建林等,2015;叶宁等,2017)。其中,Na是衡量群体遗传变异的重要指标,其数值越接近所检测到的等位基因绝对数,表明等位基因在群体中分布越均匀。PIC是描述SSR位点多态性的重要参数,反映子代所获得的某个等位基因标记来源于其亲代同一个等位标记的可能性(刘丽和刘楚吾,2006),即表征物种的遗传变异程度。本研究结果表明,11个SSR位点在凡纳滨对虾连续3个世代选育群体中的Na为6~14个,平均为9.3636个,达到用于遗传多样性评估的标准,即每个SSR位点至少要有4个等位基因(Wajid et al.,2014)。Botstein等(1980)认为,0.25 H是衡量群体遗传变异的最适参数,是指所检测SSR位点上杂合子基因型占该位点所有基因型的比例,主要反映群体的遗传变异程度(Shikano and Taniguchi,2002;李莉等,2006)。H和PIC均能反映群体内的个体遗传变异程度,且数值高低与遗传变异程度呈正相关。本研究结果表明,2016年选育群体的Ho介于0.1288~0.7632,平均为0.3975,He介于0.4492~0.8621,平均为0.7057;2017年选育群体的Ho介于0.1026~0.8673,平均为0.5224,He介于0.5286~0.8684,平均为0.7057;2018年选育群体的Ho介于0.0846~0.7698,平均为0.4876,He介于0.4837~0.8592,平均为0.7029。说明凡纳滨对虾连续3个世代选育群体具有较高的遗传多样性水平。Ho低于He,则杂合度偏离指数(D)为负值(唐江等,2018),表明凡纳滨对虾连续3个世代选育群体存在杂合子缺失现象,与Valles-Jimenez等(2004)的研究结果一致。杂合子缺失现象可能是研究对象数量有限、近亲杂交或人为干扰等因素引起稀有碱基缺失所致(Antoro et al.,2006)。Hardy-Weinberg平衡检验是评估样本代表性及其基因分型质量的重要工具(Crane et al.,2004;陈静等,2018)。本研究中,凡纳滨对虾连续3个世代选育群体有严重偏离Hardy-Weinberg平衡状态的倾向,表明凡纳滨对虾群体遗传结构处于相对不稳定的状态。由于群体小、近亲交配及等位基因突变等均可导致所检测种群基因型偏离Hardy-Weinberg平衡(宋炜等,2017),因此,凡纳滨对虾连续3个世代选育群体样本小及选育过程中存在近亲繁殖可能是造成偏离Hardy-Weinberg平衡的主要原因。 Fst是反应群体间遗传分化的重要指标(李建林等,2015;于思梦等,2017),而Fis是衡量群体遗传动态的重要指标(刘海情等,2012)。凡纳滨对虾连续3个世代选育群体在11个SSR位点上的Fst为0.0011~0.0113,均小于0.0500,表明凡纳滨对虾群体的遗传分化很小;凡纳滨对虾群体内的Fis介于-0.0101~0.8098,平均为0.3543,且除C11654位点为负值外,其余SSR位点均为正值,表明凡纳滨对虾连续3个世代选育群体的近交程度较高,杂合子缺失可能与近亲交配有关。AMOVA分析结果表明,69.78%的遗传变异来源于凡纳滨对虾个体内,而29.96%的变异来源于凡纳滨对虾个体间,说明凡纳滨对虾个体内的遗传变异大于个体间的遗传变异。综上所述,本研究中的凡纳滨对虾连续3个世代选育群体亲缘关系较近,可能与凡纳滨对虾群体存在近交现象及群体样本小有关,但具体原因有待进一步探究。 4 结论 桂海1号凡纳滨对虾各世代选育群体尚具有较丰富的遗传变异,但群体遗传结构处于不稳定状态。因此,今后应通过人工选育方式保留有利突变、淘汰有害突变,同时避免近亲繁殖,或引进优质品种进行杂交以扩充基因库,保护凡纳滨对虾的遗传多样性,防止种质退化。 参考文献: 包秀凤. 2014. 凡纳滨对虾选育群体遗传多样性分析[D]. 湛江:广东海洋大學. [Bao X F. 2014. Analysis of genetic diversity of selected stocks of Litopenaeus vannamei[D].Zhanjiang:Guangdong Ocean University.] 陈锦豪,郑锦滨,王攀攀,李天骄,毛勇,苏永全,王军. 2019. 凡纳滨对虾养殖亲本群体遗传多样性分析[J]. 渔业研究,41(1):1-10. [Chen J H,Zheng J B,Wang P P,Li T J,Mao Y,Su Y Q,Wang J. 2019. Analysis for genetic diversity of broodstock populations of breeding Litopenaeus vannamei[J]. Journal of Fisheries Research,41(1):1-10.] 陈静,何吉祥,樊佳佳,黄龙,吴本丽,宋光同,汪翔,武松. 2018. 草鱼MyoD基因SNP和Indel标记的筛选及其与生长性状的关联分析[J]. 江苏农业学报,34(3):612-616. [Chen J,He J X,Fan J J,Huang L,Wu B L,Song G T,Wang X,Wu S. 2018. Screening of SNP and Indel mar-ker of MyoD gene and its association with growth traits in Ctenopharyngodon idella[J]. Jiangsu Journal of Agricultural Sciences,34(3):612-616.] 陈锚,吴长功,相建海,黄皓,刘小林,刘翠红,何建国,孙成波. 2008. 凡纳滨对虾的选育与家系的建立[J]. 海洋科学,32(11):5-8. [Chen M,Wu C G,Xiang J H,Huang H,Liu X L,Liu C H,He J G,Sun C B. 2008. Selective breeding and pedigree foundation of Litopenaeus vannamei[J]. Marine Sciences,32(11):5-8.] 崔蕾,谢从新,李艳和,于永耀,刘肖莲,张杰,周小云. 2012. 斑点叉尾鮰4个群体遗传多样性的微卫星分析[J]. 华中农业大学学报,31(6):744-751. [Cui L,Xie C X,Li Y H,Yu Y Y,Liu X L,Zhang J,Zhou X Y. 2012. Analysis of genetic diversity among four different channel catfish populations by using microsatellite markers[J]. Journal of Huazhong Agricultural University,31(6):744-751.] 董在杰,梁政远,刘介奇,苏胜彦,明俊超,曲疆奇,徐跑,谢庄. 2010. 我国新引进吉富品系尼罗罗非鱼群体的遗传多样性分析[J]. 动物学杂志,45(5):129-135. [Dong Z J,Liang Z Y,Liu J Q,Su S Y,Ming J C,Qu J Q,Xu P,Xie Z. 2010. Genetic diversity analysis in a newly introduced population of GIFT strain Nile tilapia(Oreochromis niloticus)[J]. Chinese Journal of Zoology,45(5):129-135.] 黄薇,李剑锋,张根玉,李永权,施永海,张海明,徐嘉波,骆志强. 2014. 池塘温室南美白对虾、罗氏沼虾三茬轮养技术[J]. 中国水产,(6):69-71. [Huang W,Li J F,Zhang G Y,Li Y Q,Shi Y H,Zhang H M,Xu J B,Luo Z Q. 2014. Three-crop rotation culture of Litopenaeus vannamei and Macrobrachium rosenbergii in pond greenhouse[J]. China Fisheries,(6):69-71.] 黄小帅,徐煜,胡晓娟,徐武杰,苏浩昌,文国樑,杨铿,曹煜成. 2019. 利用微卫星标记分析7个凡纳滨对虾引进群体子一代的遗传多样性[J]. 南方水产科学,15(1):54-62.[Huang X S,Xu Y,Hu X J,Xu W J,Su H C,Wen G L,Yang K,Cao Y C. 2019. Genetic diversity analysis of first filial generation of seven introduced Litopenaeus vannamei populations using microsatellite DNA markers[J]. South China Fisheries Science,15(1):54-62.] 颉晓勇,苏天凤,陈文,张志,颜远义,江世贵. 2008. 凡纳滨对虾6个养殖群体遗传多样性的比较分析[J]. 南方水产,4(6):42-49. [Xie X Y,Su T F,Chen W,Zhang Z,Yan Y Y,Jiang S G. 2008. Analysis on genetic diversity of six cultured stocks of Litopenaeus vannamei[J]. South China Fisheries Science,4(6):42-49.] 李东宇,孟宪红,孔杰,栾生,曹寶祥,罗坤. 2017. 凡纳滨对虾(Litopenaeus vannamei)选育群体与杂交群体遗传多样性差异及其在低温条件下生长性能的比较[J]. 渔业科学进展,38(4):69-77. [Li D Y,Meng X H,Kong J,Luan S,Cao B X,Luo K. 2017. The difference of genetic diversity and the comparison of growth performance between selected population and hybridized population of Pacific white shrimp(Litopenaeus vannamei) under low temperature conditions[J]. Progress in Fishery Sciences,38(4):69-77.] 李建林,李红霞,唐永凯,俞菊华,于凡. 2015. 利用微卫星标记分析两个吉富罗非鱼群体的遗传差异[J]. 南方农业学报,46(1):138-143. [Li J L,Li H X,Tang Y K,Yu J H,Yu F. 2015. Genetic difference analysis of two genetically improved farmedtilapia populations by using microsatellite marker[J]. Journal of Southern Agriculture,46(1):138-143.] 李莉,孙振兴,杨树德,常林瑞,杨立红. 2006. 用微卫星标记分析皱纹盘鲍群体的遗传变异[J]. 遗传,28(12):1549-1554. [Li L,Sun Z X,Yang S D,Chang L R,Yang L H. 2006. Analysis of genetic variation of Abalone(Haliotis discus hannai) populations with microsatellite markers[J]. Genetics,28(12):1549-1554.] 刘海情,郭昱嵩,王中铎,刘丽,刘楚吾. 2012. 利用微卫星分析吉富罗非鱼群体的遗传多样性[J]. 南方农业学报,43(1):94-98. [Liu H Q,Guo Y S,Wang Z D,Liu L,Liu C W. 2012. Genetic diversity analysis amongst GIFT strains of Oreochromis niloticus using microsatellites[J]. Journal of Southern Agriculture,43(1):94-98.] 刘洪涛,杨明秋,何玉贵,唐贤明. 2018. 凡纳滨对虾八个地理群体遗传多样性的微卫星分析[J]. 海南大学学报(自然科学版),36(2):146-152. [Liu H T,Yang M Q,He Y G,Tang X M. 2018. Microsatellite analysis of genetic diversity in eight geographical populations of Litopenaeus vannamei[J]. Natural Science Journal of Hainan University,36(2):146-152.] 刘丽,刘楚吾. 2006. 5种笛鲷属鱼类的遗传多样性及分子标记[J]. 农业生物技术学报,14(3):349-355. [Liu L,Liu C W. 2006. Genetic diversity and molecular markers of 5 species of snappers[J]. Journal of Agricultural Biotechnology,14(3):349-355.] 马春艳,马洪雨,马凌波,杨柯. 2011.凡纳滨对虾引进群体和2个养殖群体遗传变异的微卫星分析[J]. 海洋渔业,33(1):1-8. [Ma C Y,Ma H Y,Ma L B,Yang K. 2011. Microsatellite analysis on genetic variation of imported Litopenaeus vannamei population and cultured stocks[J]. Marine Fisheries,33(1):1-8.] 彭敏,陈秀荔,赵永贞,陈晓汉,李咏梅,杨春玲,何苹萍,牛生洋. 2014. 荧光标记微卫星技术用于凡纳滨对虾不同家系亲权关系鉴定[J]. 水生态学杂志,35(4):60-67. [Peng M,Chen X L,Zhao Y Z,Chen X H,Li Y M,Yang C L,He P P,Niu S Y. 2014. Application of fluorescent labeled microsatellite technology in parentage analysis of Litopenaeus vannamei[J].Journal of Hydroecology,35(4):60-67.] 宋炜,孟永永,蒋科技,赵明,任桂静,张凤英,马凌波. 2017. 棘头梅童鱼七个野生群体遗传多样性的微卫星分析[J]. 水产学报,41(1):31-39. [Song W,Meng Y Y,Jiang K J,Zhao M,Ren G J,Zhang F Y,Ma L B. 2017. Analysis of genetic diversity among seven wild Collichthys lucidus populations by using microsatellite marker[J]. Journal of Fisheries of China,41(1):31-39.] 唐江,田永勝,李振通,成美玲,陈张帆,毛东亮,马文辉,王晓梅,李文升,翟介明,李波. 2018. 云纹石斑鱼和鞍带石斑鱼及其杂交后代遗传性状分析[J]. 农业生物技术学报,26(5):819-829. [Tang J,Tian Y S,Li Z T,Cheng M L,Chen Z F,Mao D L,Ma W H,Wang X M,Li W S,Zhai J M,Li B. 2018. Analysis of genetic characters in Epinephelus moara,E. lanceolaus and their hybrids[J]. Journal of Agricultural Biotechnology,26(5):819-829.] 童馨,龚世圆,喻達辉,黄桂菊,杜博,李色东. 2009. 凡纳滨对虾(Litopenaeus vannamei)不同世代养殖群体的遗传多样性分析[J]. 海洋与湖沼,40(2):214-220. [Tong X,Gong S Y,Yu D H,Huang G J,Du B,Li S D. 2009. Genetic diversity of cultured Pacific white shrimp(Litopenaeus vannamei) stocks of different generations in China[J]. Oceanologia et Limnologia Sinica,40(2):214-220.] 王霞,刘小林,张继泉,张成松,黄皓,相建海. 2009. 微卫星用于凡纳滨对虾育种过程中亲权分析及遗传多样性的变化监测[J]. 水产学报,33(5):832-839. [Wang X,Liu X L,Zhang J Q,Zhang C S,Huang H,Xiang J H. 2009. Kinship analysis and genetic variation monitoring in Litopenaeus vanname breeding program using microsatellite DNA markers[J]. Journal of Fisheries of China,33(5):832-839.] 王兴强,马甡,董双林. 2004. 凡纳滨对虾生物学及养殖生态学研究进展[J]. 海洋湖沼通报,(4):94-100. [Wang X Q,Ma S,Dong S L. 2004. Studies on the biology and cultural ecology of Litopenaeus vannamei:A review[J]. Transaction of Oceanology and Limnology,(4):94-100.] 吴怡迪,骆轩,杨章武,黄永春,游伟伟. 2016. 凡纳滨对虾选育系间杂交的生长性状及遗传多样性分析[J]. 厦门大学学报(自然科学版),55(5):646-653. [Wu Y D,Luo X,Yang Z W,Huang Y C,You W W. 2016. Growth performance and genetic diversity analysis of hybrids between selective lines of Litopenaeus vannamei[J]. Journal of Xiamen University (Natural Science),55(5):646-653.] 谢丽,陈国良,叶富良,栗志民. 2009. 凡纳滨对虾4个选育群体遗传多样性的SSR分析[J]. 广东海洋大学学报,29(4):5-9. [Xie L,Chen G L,Ye F L,Li Z M. 2009. Genetic diversity of four selected stocks of Litopenaeus vanna-mei as revealed by SSR marker[J]. Journal of Guangdong Ocean University,29(4):5-9.] 杨铭,于洋,张晓军,王全超,刘敬文,李富花,相建海. 2017. 基于转录组数据的凡纳滨对虾微卫星标记开发[J]. 海洋科学,41(2):96-102. [Yang M,Yu Y,Zhang X J,Wang Q C,Liu J W,Li F H,Xiang J H. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp(Litopenaus vannamei)[J]. Marine Sciences,41(2):96-102.] 叶宁,包秀凤,刘建勇. 2017. 凡纳滨对虾引进群体与养殖群体遗传多样性的AFLP分析[J]. 水产学报,41(3):339-346. [Ye N,Bao X F,Liu J Y. 2017. AFLP analysis of genetic diversity of imported Litopenaeus vannamei po-pulations and cultured stocks[J]. Journal of Fisheries of China,41(3):339-346.] 于思梦,高磊,王伟,赫崇波,鲍相渤,田梅林. 2017. 辽宁黄渤海沿岸长牡蛎遗传多样性分析[J]. 经济动物学报,21(4):215-220. [Yu S M,Gao L,Wang W,He C B,Bao X B,Tian M L. 2017. Genetic diversity of oyster in the Yellow sea and Bohai sea in Liaoning Province[J]. Journal of Economic Animal,21(4):215-220.] 张文静,余育和,沈韫芬. 2003. 微卫星DNA遗传分析在原生动物学中的研究进展[J]. 水生生物学报,27(2):185-190. [Zhang W J,Yu Y H,Shen Y F. 2003. Advances on genetic analysis of microsatellite DNA in protozoology[J]. Acta Hydrobiologica Sinica,27(2):185-190.] 赵永贞. 2014. 凡納滨对虾——桂海1号[J]. 农村百事通,(3):32. [Zhao Y Z. 2014. “Guihai No.1” Litopenaeus vannamei[J]. Rural Affairs,(3):32.] Antoro S,Na-Nakorn U,Koedprang W. 2006. Study of gene-tic diversity of orange-spotted grouper,Epinephelus coioides,from Thailand and Indonesia using microsatellite markers[J]. Marine Biotechnology(New York),8(1):17-26. Botstein D,White R L,Skolnick M H,Davis R. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics,32(3):314-331. Crane P A,Lewis C J,Kretschmer E J,Miller S J,Spearman W J,DeCicco A L,Lisac M J,Wenburg J K. 2004. Cha-racterization and inheritance of seven microsatellite loci from Dolly Varden,Salvelinus malma,and cross-species amplification in Arctic char,S. alpinus[J]. Conservation Genetics,5:737-741. Cruz P,Ibarra A M,Mejia-Ruiz H,Gaffney P M,Pérez-Enríquez R. 2004. Genetic variability assessed by microsatellites in a breeding program of Pacific white shrimp (Litopenaeus vannamei)[J]. Marine Biotechnology(New York),6(2):157-164. Gyapay G,Morissette J,Vignal A,DibC,Fizames C,Millas-seau P,Marc S,Bernardi G,Lathrop M,Weissenbach J. 1994. The 1993-94 Généthon human genetic linkage map[J]. Nature Genetics,7(2):246-339. Nagy S,Poczai P,Cernák I,Gorji A M,Heged?s G,Taller J. 2012. PICcalc:An online program to calculate polymorphic information content for molecular genetic studies[J]. Biochemical Genetics,50(9-10):670-672. O'Connell M , Wright J M. 1997. Microsatellite DNA in fish[J]. Reviews in Fish Biology and Fisheries,7:331-363. Shikano T,Taniguchi N. 2002. Relationships between genetic variation measured by microsatellite DNA markersand a fitness-related trait in the guppy(Poecilia reticulata)[J]. Aquaculture,209(1):77-90. Valles-Jimenez R,Cruz P,Perez-Enriquez R. 2004. Population genetic structure of Pacific white shrimp(Litopenaeus vannamei) from mexico to panama:Microsatellite DNA varia-tion[J]. Marine Biotechnology(New York),6(5):475-484. Wajid A,Wasim M,Yaqub T,Firyal S,Tayyab M,Siddique S,Hussain T. 2014. Assessment of genetic diversity in Balochi and Rakhshani sheep breeds of Balochistan using microsatellite DNA markers[J]. The Journal of Animal & Plant Sciences,24(5):1348-1354. Wolfus G M,Garcia D K,Alcivar-Warren A. 1997. Application of the microsatellite technique for analyzing genetic diversity in shrimp breeding programs[J]. Aquaculture,152(1-4):35-47. Zhang K,Wang W J,Li W Y,Zhang Q Q,Kong J. 2014. Analysis of genetic diversity and differentiation of seven stocks of Litopenaeus vannamei using microsatellite mar-kers[J]. Journal of Ocean University of China,13(4):647-656. Zhang L S,Yang C J,Zhang Y,Li L,Zhang X M,Zhang Q L,Xiang J H. 2007. A genetic linkage map of Pacific white shrimp(Litopenaeus vannamei):Sex-linked microsatellite markers and high recombination rates[J]. Genetica,131(1):37-49. (责任编辑 兰宗宝)

