番茄串联重复SlHSP20基因簇特征及差异表达分析
马 健,李 娇,姜 晶
(沈阳农业大学 园艺学院/设施园艺省部共建教育部重点实验室/辽宁省设施园艺重点实验室,沈阳 110161)
生物在基因组进化和适应环境的过程中,有功能的基因会存在串联重复而形成一类串联重复基因。研究发现,植物基因组中串联重复事件发生频率较高,这带来了植物基因组中基因拷贝数的增加和等位基因的变异[1]。在拟南芥和水稻基因组中,串联重复机制对胁迫耐受性和膜功能基因的扩张有很大的影响[2-3]。NBS-LRR抗病基因家族在茄科和十字花科植物中都存在串联重复,这对家族进化起着非常重要的作用[4]。对于复杂的功能串联重复序列-串联重复基因的研究,可以揭示植物的重要性状及表型相关的基因/基因家族的形成机制或演化规律。
小热激蛋白(sHSPs)最为人所知的是对细胞应激的早期反应,作为不依赖ATP 的分子伴侣,可延迟有害蛋白质聚集体的形成,以响应应激源[5]。sHSPs 的分子量大小均在12~42kDa 之间,因此也将其称为HSP20。该家族成员包含ACD 结构域,这个结构域是以具有典型的β-sandwich 结构的低聚物组成的一个空心球,作为分子伴侣的功能,有助于蛋白质折叠和防止其靶蛋白的聚集[6]。此外,sHSPs 本身非常敏感,它们的活性以及它们的蛋白质水平会被细胞所处环境激活[7]。最初sHSPs 是在热应激过程中上调的蛋白质中发现并且组成型表达。sHSPs 在热激蛋白中含量最为丰富,与植物耐热性关系密切,正常生长条件下,大多数sHSPs 在植物组织中不积累,而在热激或其他胁迫下,sHSPs 含量迅速增加。研究表明,高温胁迫下sHSPs 能自发形成大小为200~300kDa 的同源寡聚体,并与其他分子伴侣相互作用,帮助受损蛋白重新折叠[8]。在分子水平上,sHSPs 基因可对细胞活性进行瞬时的重新编程,其特征是伴随热激蛋白(HSP)的合成进而停止合成正常蛋白质。HSP 的一个共同特征是它们以剂量依赖性方式进行积累,足以达到保护细胞的水平。HSP 基因的表达主要受转录水平控制,并且需要转录水平机制与特定转录因子相互作用,热应激因子(HSF)在控制HS 反应中起关键作用[9]。位于5′末端的转录调节序列包括HS 响应元件(HSEs)和AT-rich 区域分别负责基因的热诱导性和感应热诱导性。已有关于拟南芥和水稻的sHSPs 基因家族基因表达数据表明,双子叶植物和单子叶植物之间的I 类细胞质sHSPs 基因成员具有明显的时空分布特异性[10-11]。
番茄sHSPs 基因家族42 个成员中存在有两组串联重复基因,其中分别是SlHSP49.3/Sl-HSP39.4 和SlHSP17.7A/SlHSP17.6A/SlHSP17.6B/SlHSP17.6C[5,13]。其中 SlHSP17.7A/SlHSP17.6A/SlHSP17.6B/SlHSP17.6C 的基序排列高度一致,而这组氨基酸序列具有高度保守性的串联基因簇的功能尚不清楚。本研究系统分析番茄中四个串联重复基因Sl-HSP20 基因的表征和特性,根据基因的5′末端富含对激素响应的顺式作用元件,比较该基因簇在激素处理、糖分响应的差异以及该基因簇在番茄果实发育和成熟阶段的共表达基因,以期为进一步分析sHSPs 的进化起源及其功能奠定基础。
1 材料与方法
1.1 材料
供试Micro-Tom 番茄,取自沈阳农业大学蔬菜分子生物学番茄课题组。种子催芽时分别在水浴锅55℃热激20 min,待水温降为室温后于28℃恒温箱培养,早晚控水洗种。培养48h 后,待70%以上的种子露白后,播于50 孔穴盘。培养于沈阳农业大学园艺学院蔬菜分子生物学组培室,温度25℃,光周期为16h/8 h(昼/夜),光照强度 120 μmol·m-2·s-1。
1.2 方法
1.2.1 番茄SlHSP20 基因簇的鉴定及理化性质 利用tBLASTn 程序在番茄基因组中鉴定SlHSP20 基因簇成员,Solyc06g076520,Solyc06g076540,Solyc06g076560 和 Solyc06g076570(分别命名为 SlHSP17.7A,SlHSP17.6A,SlHSP17.6B 和SlHSP17.6C)。运用ExPASy(http://expasy.org/)在线工具预测鉴定番茄SlHSP20 基因簇氨基酸序列蛋白质长度、分子质量等理化性质。根据SGN(http://solgenomics.net/)鉴定染色体定位、染色体分布、分子量、等电点和β 折叠数量等。
1.2.2 番茄SlHSP20 基因簇系统系统发育进化树的构建及基序分析 将获得的番茄SlHSP20 基因簇的氨基酸序列保存为FASTA 格式文件,通过MEGA6.0 软件的邻接法(Neighbor-Joining)对基因簇进行系统进化树分析。模式采用“Poisson correction”,缺口设置为“Pairwise Deletion”,校验参数为 Bootstrap=1000,去除 bootstrap 支持率低于60%的节点。利用ClustalX 软件对番茄SlHSP20 成员进行多重序列比对,并人工校正。利用MEME(http://meme-suite.org/)软件分析番茄SlHSP20 基因家族成员基序类型和排列顺序,获得番茄SlHSP20 基因家族基序特点。
1.2.3 番茄SlHSP20 基因簇染色体定位分析 根据获得番茄SlHSP20 基因组信息,利用番茄基因组数据(SGN,http://solgenomics.net,release v2.50)和软件MapChart 2.3 定位分析番茄SlHSP20 基因簇染色体。
1.2.4 番茄SlHSP20 基因簇顺势作用元件分析 分析启动子区域顺式作用元件,从NCBI 数据库(http://www.ncbi.nlm.nih.gov/) 中下载番茄SlHSP20 基因簇ATG 上游2.0kb 序列作为启动子序列,利用在线工具PLANT CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行顺式作用元件分析。
1.2.5 SlHSP20 基因簇在番茄中表达分析 根据番茄表达数据库(http://ted.bti.cornell.edu/)分析SlHSP20 基因簇在不同组织及果实生长发育不同时期的表达量。在番茄转录组综合分析在线网站(http://tomexpress.toulouse.inra.fr/query) 中以SlHSP20 基因簇为诱饵使用对数标度的转录计数值和r 绝对值≥0.98 的阈值的皮尔逊相关系数进行基因的共表达分析[12]。
1.2.6 激素和外源糖诱导SlHSP20 基因簇表达分析 取果实绿熟期的番茄果实,用不锈钢打孔器取果肉(直径4 mm,厚度2 mm),进行果肉孵育试验,果肉圆片(包含果实维管束)放入20 mL 基础缓冲液(MES 50 mmol·L-1pH5.5),含 CaC125 mmol·L-1,MgC125 mmol·L-1,EDTA 5 mmol·L-1,抗坏血酸 5 mmol·L-1和甘露醇 200 mmol·L-1中平衡 30 min 后,在缓冲液中做以下处理:分别加入 IAA 50 mg·L-1、GA 50 mg·L-1、ABA 50 μmol·L-1、MeJA 50 μmol·L-1、ETH 50 μmol·L-1、SA 50 μmol·L-1、果糖(F) 50mg·L-1、葡萄糖(G) 50mg·L-1、蔗糖(S) 50mg·L-1,在室温下孵育8h 和24h。取果肉圆片,用滤纸吸干表面水分,液氮速冻保存(-80℃冰箱),用于后续基因表达分析。
总RNAs 的提取使用天根公司的植物总RNA 提取试剂盒(Tiangen,中国),具体步骤参照制造商的说明。总cDNAs 的获得使用天根公司的Fast King RT Kit 反转录试剂盒,具体步骤参照制造商的说明。实时荧光定量PCR 使用SYBR Green PCR Master Mix 试剂盒 (Tiangen,中国),sHSPs 基因的实时荧光定量PCR 引物使用Primer Express 软件设计,并委托鸿讯生物公司合成,纯合方式为OPC 级,质量为2 OD。使用仪器为Bio-Rad CFX ManagertTM Software 实时定量PCR 仪,具体步骤参照制造商的说明。选择内参基因为Actin,所有反应进行3 次生物学重复,做阴性对照并且添加溶解曲线来验证PCR 产物的特异性。
SlHSP17.6A 正向引物 5′-GAAGGAGGAAGTGAAAGT-3′和反向引物 5′-ATCCATCTTTGCGTTCTC-3′,SlHSP17.6B 正向引物 5′-TTTGCCAACACACGAATA-3′和反向引物 5′-GTAACAGTAAGCACTCCG-3′,SlHSP17.6C 正 向引物 5′-ATTAGGCTTCACAGTTTC-3′和 反向引物 5′-AGTAACAGTAAGCACTCC-3′,SlHSP17.7A 正向引物 5′-TTTGCCAACACACGAATA-3′和反向引物 5′-AGTAACAGTAAGCACTCC-3′。
2 结果与分析
2.1 番茄串联重复SlHSP20基因簇鉴定
利用tBLASTn 在番茄基因组中鉴定4 个SlHSP20 基因,分析番茄基因簇中4 个串联基因的氨基酸长度、分子质量、不稳定指数及等电点等,根据氨基酸的分子量大小分别对基因簇成员命名为:SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A。基因簇成员均具有典型的β 折叠结构的保守ACD 结构域。根据基因组序列分析显示番茄SlHSP20 蛋白大小一致,番茄 SlHSP20 基因簇(SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A) 蛋白编码154 个氨基酸,分子质量变化范围为17.6~17.7kDa,其中SlHSP17.7A 分子量最大,为17.7kDa,番茄sHSPs 基因簇成员等电点变化为5.57~5.84 之间,等电点最低的是SlHSP17.6C,等电点为5.57,等电点最高的是SlHSP17.6B、SlHSP17.7A,等电点为5.84,说明番茄基因簇SlHSP20 蛋白为酸性(表1)。
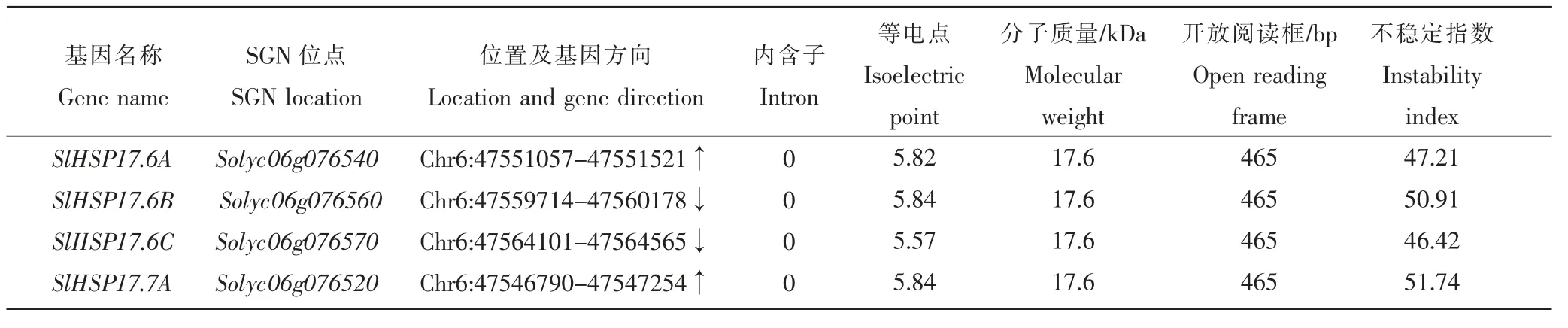
表1 番茄SlHSP20 基因簇成员的鉴定Table 1 The identification of SlHSP20 gene members in tomato
根据同源性序列分析发现,基因簇成员基因序列具有高度的相似性,同源性达到95.74%。碱基序列比对发现4 个串联重复的基因具有相同的基因结构,均不含内含子,且CDS 序列与基因序列基本一致(图1)。根据番茄SlHSP20 基因簇成员染色体的定位情况可知,在番茄第6 条染色体上发现4 个串联重复的基因(SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和 SlHSP17.7A)形成了 1 个基因簇。

图1 番茄SlHSP20 基因簇成员序列比对Figure 1 Sequence alignment of tomato SlHSP20 gene cluster members
2.2 番茄SlHSP20的4 个串联重复基因系统进化及基序分析
对番茄SlHSP20 基因簇成员的进化树分析显示,SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A 定位于CI 类亚族(细胞质或细胞核),说明这4 个串联重复基因编码的蛋白分布在细胞质或细胞核中。根据SlHSP20基因簇进行系统进化分析发现SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A 为4 个平行同源基因对(图2)。运用MEME 软件在番茄sHSPs 蛋白中检测到SlHSP20 保守基序个数为5 个,其基因簇成员的基序类型及排列顺序高度一致,并且SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A 基序类型相同和排列顺序一致(图2),由图3 可知在其基序中包含 8 个保守的 β 折叠结构(β2~β9)。
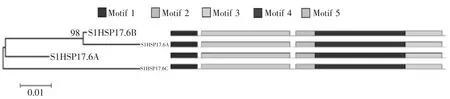
图2 番茄SlHSP20 基因簇进化树分析及蛋白基序类型Figure 2 Charging module circuit diagram

图3 番茄SlHSP20 基因簇基序及保守结构域β 链分析Figure 3 Analysis of motif and β-chain region of conserved domain in tomato SlHSP20 gene cluster
根据氨基酸同源性分析,基因簇成员具有高度的同源性(图4 和表2)。所有的SlHSP20 在C 端均具有共同保守的热激序列,热激序列又分为保守序列I 和保守序列II,中间有不同长度的亲水结合域。保守序列I 都含有共同的M 残基:Pro-X(14)-Gly-Val-Leu;保守序列II 的相似氨基酸残基为:Pro-X(14)-X-Val/Leu/Ile-Val/Leu/Ile;保守序列 I 和 II 是植物 sHSPs 所共有的[7]。

图4 番茄SlHSP20 基因簇氨基酸序列的多序列比对及保守结构域分析Figure 4 Multiple sequence alignment and conserved domain analysis of tomato SlHSP20 gene cluster
2.3 番茄SlHSP20基因簇顺式作用元件分析
对番茄SlHSP20 的4 个串联重复片段基因的启动子序列进行分析,截取番茄 SlHSP20 基因簇(SlHSP17.6A、SlHSP17.6B、SlHSP17.6C、SlHSP17.7A)ATG 上游2000bp 序列,运用PLANTCARE 网站作顺式作用原件功能预测,ABRE,ARE,CGTCA-motif,TGACG-motif,CAT-box,LTR,MBS,TGA-element,site,GARE-motif与生物胁迫相关,4 个串联基因均含有脱落酸应答元件(ABRE),茉莉酸应答元件(CGTCA-motif),抗氧化应答元件(ARE),其中 SlHSP17.7A、SlHSP17.6B 和 SlHSP17.6C含有低温胁迫应答元件(LTR),SlHSP17.7A 和SlHSP17.6A 含有干旱胁迫应答元件(MBS),而SlHSP17.7A、SlHSP17.6A 和SlHSP17.6C 含有生长素应答元件 (TGA-element),SlHSP17.6A 含有防御和压力应答元件 (TC-rich repeats),SlHSP17.6B 和SlHSP17.6C 含有赤霉素应答元件(GARE-motif)。SlHSP20 基因簇成员在响应非生物胁迫和抗性胁迫过程中起到了明显的作用,说明该基因簇在响应生物进程中起着关键作用[7]。
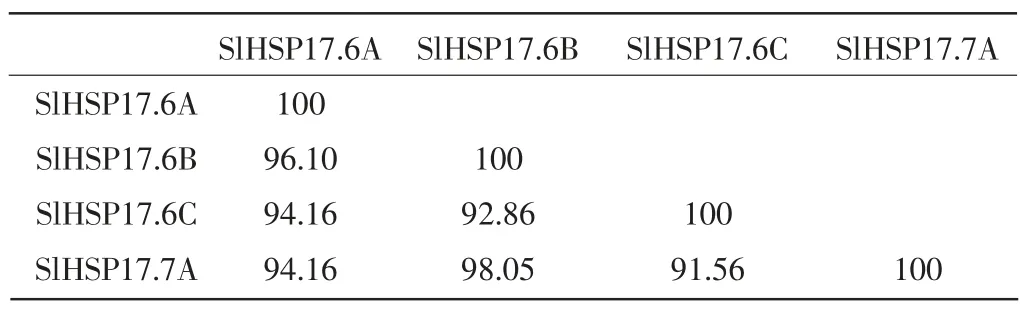
表2 番茄SlHSP20 基因簇氨基酸序列两两比对Table 2 Comparison of amino acid sequence of SlHSP20 gene cluster in tomato %
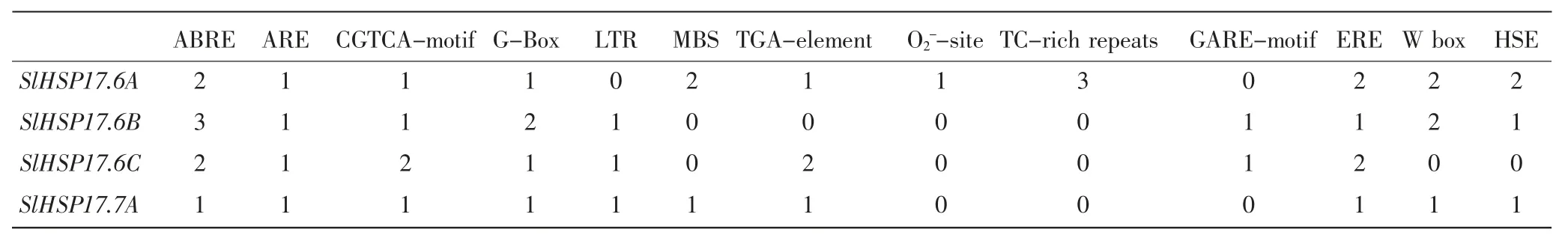
表3 番茄SlHSP20 基因簇启动子顺式响应元件种类Table 3 Type of cis-response element in the promoter of SlHSP20 gene cluster
2.4 串联重复SlHSP20基因簇在番茄中的表达分析
通过番茄基因数据库(http://ted.bti.cornell.edu/)分析SlHSP20 基因簇在不同组织及果实生长发育不同时期的表达量(图5)。SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和SlHSP17.7A 在叶根和花器官中表达量均很低,但在果实发育与成熟时期表达量呈现明显的差异。在果肉部位SlHSP17.6A 表达较低,而SlHSP17.6B 和SlHSP17.6C在绿熟期(MG_35DPA)到红熟期(Red_44DPA)表达量逐渐增强,而SlHSP17.7A 在果实绿熟期后一直处于较高的表达水平。在果实的外果皮和种子部位SlHSP17.7A、SlHSP17.6A、SlHSP17.6B 和SlHSP17.6C 随着果实的发育和成熟表达量也随之增强,其中SlHSP17.6A 较其他3 个基因的表达要弱。相比较而言,SlHSP17.7A 在果实中的表达量相对于SlHSP17.6A,SlHSP17.6B,SlHSP17.6C 明显提高,其中在绿果期中SlHSP17.7A 的表达量是SlHSP17.6A 的表达量的20 倍。

图5 SlHSP20 基因簇在番茄生长发育中的表达分析Figure 5 Expression analysis of SlHSP20 gene cluster in tomato
根据基因表达的相似性分析基因表达产物可能的相互作用关系,了解基因间相互作用网络,利用转录组综合分析在线网站(http://tomexpress.toulouse.inra.fr/query)信息,分析SlHSP20 基因簇成员在番茄果实发育过程中的共表达差异 (图6)。SlHSP20 基因簇4 个成员在果实发育的共表达网络中处于不同的作用区间。其中Solyc06g076570 (SlHSP17.7A) 可能的正相关互作基因有11 个,负相关互作基因2 个;Solyc06g076560(SlHSP17.6C)可能的正相关互作基因有4 个,负相关互作基因1 个;Solyc06g076540(SlHSP17.6A)可能的正相关互作基因有1 个,Solyc06g076520(SlHSP17.6B)可能的负相关互作基因有1 个。研究结果表明SlHSP20 基因簇成员氨基酸序列虽高度同源,但其结合的互作基因的蛋白产物却不同,说明氨基酸的不同可能导致蛋白的空间构型改变进而影响了其功能的差异。
2.5 激素对番茄果实SlHSP20基因簇的表达分析
通过PLANTCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/)在线分析软件中分析其顺式作用元件。结果表明,SlHSP17.6A、SlHSP17.6B、SlHSP17.6C 和 SlHSP17.7A,这 4 个 SlHSP20 基因的启动子含有 12 种已知响应逆境胁迫的若干相关作用元件,其中一半以上为响应激素相关的顺式作用元件,尤其是参与响应脱落酸(ABA)、生长素(IAA)、赤霉素(GA3)、乙烯(ETH)和茉莉酸甲酯(MeJA)。这说明 SlHSP20 在番茄对逆境胁迫的应激反应中起着重要作用。果实发育成熟过程中,生长素、脱落酸等发挥重要作用。为了探究激素对番茄果实特异SlHSP20 基因的调控作用,本研究利用番茄果实圆片进行了激素处理。通过外源施加脱落酸(ABA)、生长素(IAA)、赤霉素(GA3)、乙烯(ETH)、水杨酸(SA)和茉莉酸甲酯(MeJA)等激素处理,对 4 个串联 SlHSP20 基因的表达变化进行分析(图7)。
结果表明,在激素处理的绿熟期果实圆片中,ETH 能够显著诱导SlHSP17.6A 的表达,其表达量与对照相比增加了近60%;而ABA 可显著抑制SlHSP17.6A 的表达,其表达量下降约6 倍;其他激素处理对SlHSP17.6A的表达影响不大。SlHSP17.6B 在IAA 和MEJA 处理后表达受到明显诱导,其中IAA 处理后SlHSP17.6B 相对表达量增加约6 倍,MEJA 处理后表达量增加约3 倍;在ABA 处理后,SlHSP17.6B 表达量降低63%;而其他激素处理下SlHSP17.6A 的表达没有显著变化。SlHSP17.6C 在ABA、ETH 和SA 处理后表达量受到明显抑制,它们分别下降约为96%、81%和91%。SlHSP17.7A 在GA 和IAA 处理后,表达量显著升高,分别增加约2.5 倍和6倍;而ABA、ETH 和SA 可显著抑制SlHSP17.7A 的表达,受抑制程度分别下降约98%、51%和83%。以上结果表明,虽然SlHSP20 基因簇成员在序列上高度同源,但在番茄果实成熟期受激素调控的表达形式却明显不同。SlHSP20 基因簇能够响应脱落酸、生长素、赤霉素、茉莉酸甲酯、乙烯和水杨酸等激素的调控,说明其在相关激素调控通路上发挥着重要作用,并且在不同的外源激素处理下基因簇成员展现出明显的差异性。
2.6 糖对番茄果实SlHSP20基因簇成员的表达调控
课题组前期对番茄的HSP20 基因家族成员的启动子序列分析发现,SlHSP20 基因簇的4 个成员均含有一定数量的糖响应元件有SURE(糖响应元件)和W-box(蔗糖诱导蛋白元件)[5]。因此本研究进一步分析外源葡萄糖、果糖和蔗糖处理对番茄绿熟期果实圆片SlHSP20 基因簇成员的表达影响。由图8 可知,绿熟期果糖处理8h后,SlHSP17.6A 表达量明显增加,其相对表达量增加约 2.5 倍,而 SlHSP17.6B,SlHSP17.6C,SlHSP17.7A 均受明显抑制。在葡萄糖处理后,SlHSP20 基因簇成员的表达均受到明显抑制。在蔗糖处理后,SlHSP17.6A 表达量没有显著变化,而SlHSP17.6B,SlHSP17.6C 和SlHSP17.7A 的表达量也显著降低。研究结果表明,虽然基因簇成员具有着高度的同源性,但在对糖的响应下基因簇成员表现出明显不同的基因表达水平变化。
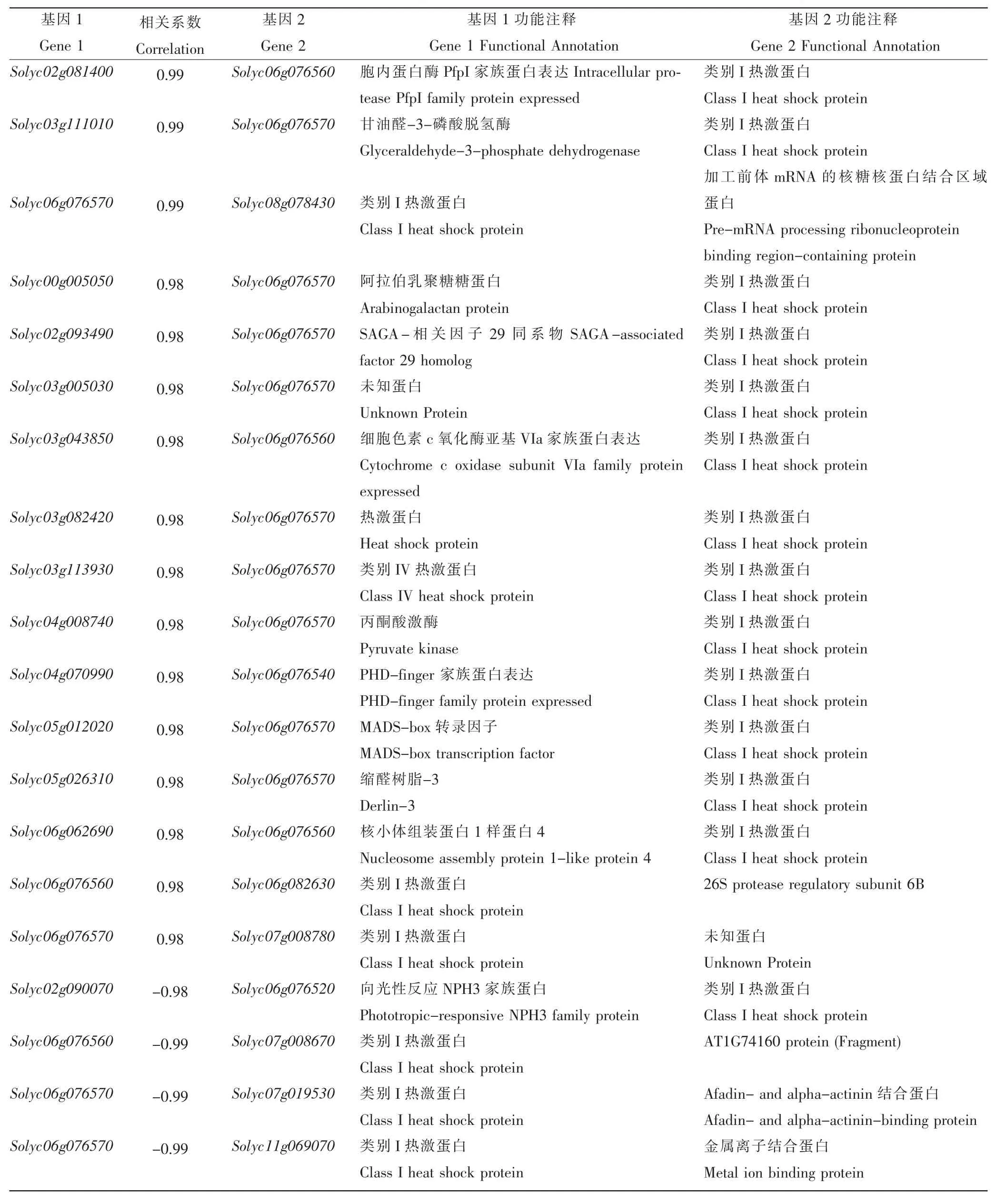
表4 SlHSP20 基因簇在番茄果实发育过程中共表达基因的相关性分析Table 4 Correlation analysis of gene cluster co-expressed SlHSP20 gene cluster during the development of tomato fruit
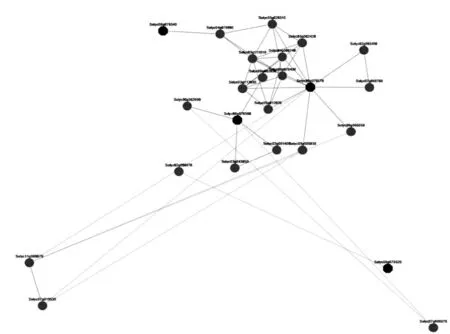
图6 SlHSP20 基因簇在番茄果实发育过程中的共表达网络分析Figure 6 Co-expression network analysis and expression of SlHSP20 gene cluster in the development of tomato fruit
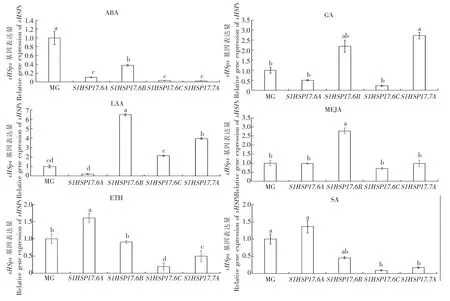
图7 激素对番茄果实SlHSP20 基因簇的表达影响Figure 7 Effects of hormones on the expression of SlHSP20 gene cluster in the fruits of tomato
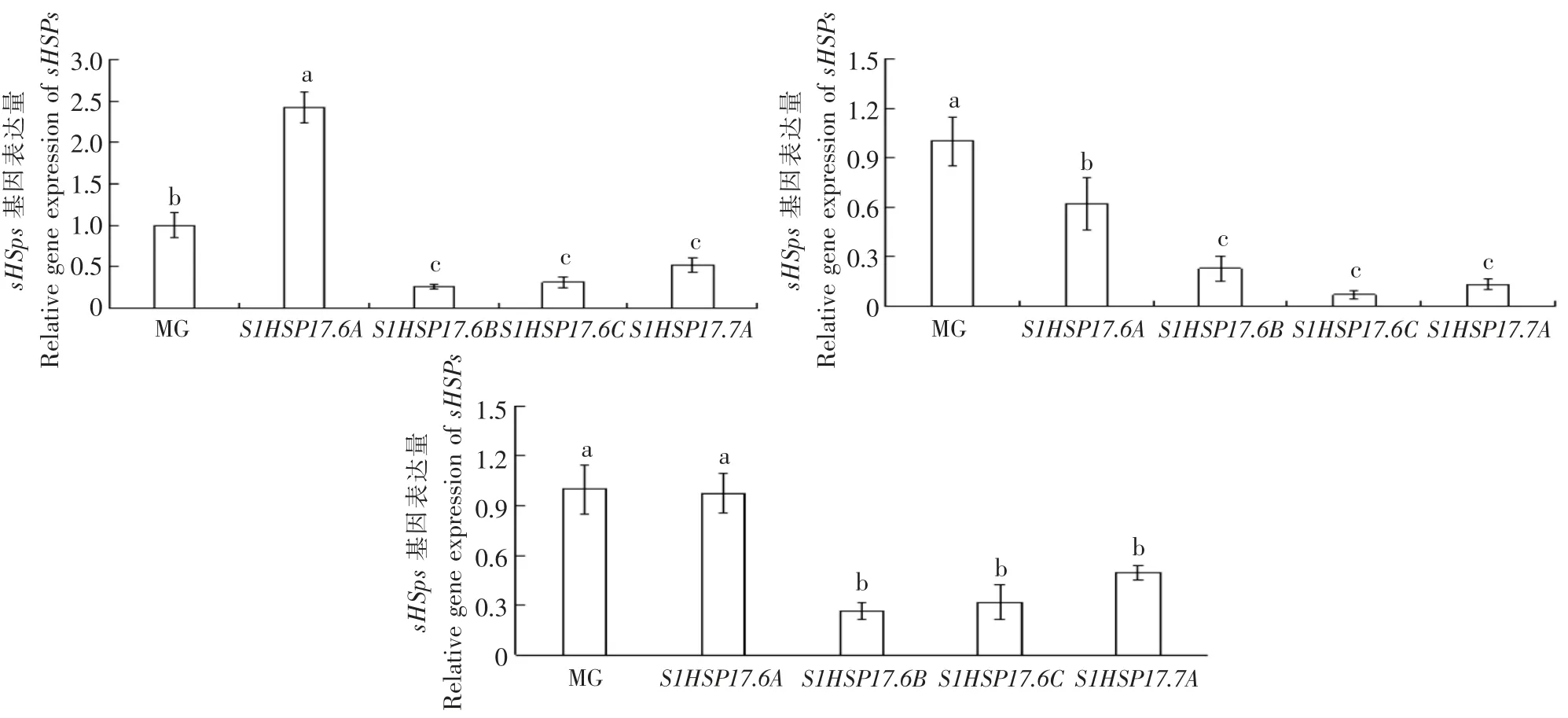
图8 糖对番茄果实SlHSP20 基因簇成员的表达影响(A.果糖;B.葡萄糖;C.蔗糖)Figure 8 Effect of sugar on expression of SlHSP20 gene cluster in tomato fruit(A.Fructose; B.Glucose; C.Sucrose)
3 讨论与结论
本研究CI 类小热激蛋白(sHSP)定位于番茄第6 条染色体上的无内含子的基因簇SlHSP20,属于低分子质量的HSP20 家族。其数量和细胞定位的复杂性是高等植物热激响应的独特特征[7,9]。其中基因的复制转化和倒置被认为是不同sHSP 基因起源的主要机制[7]。到目前为止,所有测序的基因组中都有基因重复的痕迹,并且基因重复被看做是导致生物进化进程复杂化的依据[14-16]。根据番茄数据库Sl4.0 版本对SlHSP20 基因簇4 个串联重复基因的结构和表达进行了分析,结果表明该基因簇在番茄基因组中以两两相反的方向编码CI 类家族sHSP,其ORF 的长度完全相同,基因串联排列。该基因簇具有序列高度相似性的事实表明他们是通过基因重复或者倒置而产生的。
对SlHSP20 基因簇中串联的4 个基因的结构分析发现,该基因簇成员均具有保守的ACD 结构,且结构中带有典型的β-sandwich 结构,这与前人在模式作物拟南芥、水稻的sHSPs 家族成员分布特点相同[10-11]。基因簇成员具有高度同源的cDNA 序列和氨基酸序列,通过系统进化分析发现SlHSP20 基因簇成员定位于细胞质/细胞核的CI 类亚族。分析番茄SlHSP20 基因簇的进化关系发现基因簇成员间存在直系同源关系。这些结果说明SlHSP20 基因簇成员之间存在着广泛的同源性和相似性,为深入研究SlHSP20 基因的功能提供了理论基础。
GOYAL 等[2]曾对该段基因簇中的3 个基因的结构进行分析,但其结果中3 个基因转录方向相同,这与最新注释的番茄数据库的数据不符。本研究结果表明,该基因簇是在番茄基因组第六染色体中以两两相反的方向编码CI 类家族sHSP。因此本研究根据新的基因注释转录方向分析了5’-UTR 侧翼区域顺式作用元件,并利用绿熟期的果实圆片进行了激素处理。结果表明,SlHSP20 基因簇成员的表达对激素的响应都不同。事实上,真核生物中基因调控的主要机制是转录的激活,这主要是由于顺式调控元件与转录因子相互作用的结果。因此,在对基因簇成员进行启动子序列的顺式作用元件分析发现,在4 个基因的启动子区域中发现了几种(脱落酸,乙烯,茉莉酸,水杨酸,赤霉素和各种非生物和生物胁迫以及糖相关)的调控元件,这些元件与响应不同的胁迫诱导和影响基因表达有关[5,17]。基因的共表达分析网络分析结果也表明,SlHSP20 基因簇成员的可能互作蛋白亦不同。SlHSP17.6A 互作的Solyc04g070990 编码PHD-finger 蛋白,是一类在基因转录和染色质状态调控方面有重要作用的锌指蛋白[17]。与SlHSP17.6B 互作的Solyc02g081400、Solyc03g043850、Solyc06g062690 的基因分别编码胞内蛋白酶PfpI 家族蛋白、细胞色素c 氧化酶亚基VIa 家族蛋白、核小体组装蛋白等,主要作为伴侣蛋白和抗氧化因子等发挥作用。而与SlHSP17.6C 互作的可能基因产物类别更为广泛,包括Solyc03g111010(编码甘油醛-3-磷酸脱氢酶)、Solyc00g005050(编码阿拉伯乳聚糖糖蛋白)、Solyc02g093490(编码SAGA-相关因子)等多条途径发挥作用。这说明高度同源的SlHSP20 基因簇成员,其在植物体中的功能或参与的信号转导途径可能也存在不同。但对于SlHSP20 基因簇成员的功能尚需通过基因编辑等手段进行深入的研究。

