剑麻酵母双杂交cDNA表达文库的构建及与AhKNOX2相互作用蛋白的筛选
杨子平 孙艺桓 杨倩 鹿志伟 张燕梅 李俊峰 周文钊

摘 要:植物KNOX(Knotted1-like homeobox)可通过蛋白质相互作用参与调控落果、纤维细胞发育、次生细胞壁发育、开花等事件。为筛选剑麻叶中与AhKNOX2相互作用的蛋白,采用SMART同源重组技术构建剑麻叶片酵母双杂交表达文库,利用AhKNOX2为诱饵筛选与之相互作用的蛋白。结果表明:文库总容量为3.9106 CFU,转化效率为9.15105 CFU/μg pGADT7-Rec,插入片段大小主要分布于0.5~3.0 kb之间,重组率为92%,保存的文库细胞密度为1.35108/mL,文库滴度为2.22107 CFU/mL。构建的pGBKT7-AhKNOX2诱饵载体存在自激活性,5 mmol/L的3-氨基- 1,2,4-三唑(3-Amino-1,2,4-triazole,3-AT)能抑制诱饵载体的自激活,通过筛选获得了16个与AhKNOX2相互作用的蛋白。本研究成功构建剑麻叶片酵母表达文库,并筛选获得与AhKNOX2相互作用的蛋白,该结果为进一步研究AhKNOX2在剑麻中的功能奠定前期基础,为培育高纤维产量的剑麻新品种提供基因资源和理论基础。
关键词:剑麻;AhKNOX2;酵母双杂交;cDNA文库;蛋白质相互作用
中图分类号:S563.8;Q78 文献标识码:A
Abstract: To obtain the proteins interacting with AhKNOX2 in Agave hybrid No.11648, a full length-expression cDNA library was constructed in yeast strain Y187 using homologous recombination-mediated SMART technology. The capacity of the cDNA library was 3.9×106 CFU and the transformation efficiency was about 9.15×105 CFU/μg pGADT7-Rec. The PCR amplification of 24 clones randomly selected from the cDNA library indicated that the length of most inserts was ranged from 0.5 kb to 3.0 kb, with a recombination rate of 92%. The cell density was 1.35×108/mL and the titer was up to 2.22×107 CFU/mL. The bait vector (pGBKT7-AhKNOX2) was transformed into yeast strain Y2HGold, colonies appeared on SD/-Trp/-His media and was inhibited by 5 mmol/L 3-AT. After screening the library using bait plasmids,a total of 16 proteins interacting with AhKNOX2 were obtained by sequencing and homology analysis. In conclusion, a high-quality cDNA library was constructed and 16 proteins interacting with AhKNOX2 were screened from cDNA library.
Keywords: Agave hybrid; AhKNOX2; yeast two-hybrid; cDNA library; protein-protein interaction
DOI: 10.3969/j.issn.1000-2561.2020.09.004
同源异型盒基因(homeobox genes)广泛存在于真核生物中,编码一类含有保守同源异型盒结构域(homeodomain,HD)的转录因子,在生物个体形态建成、生殖发育、细胞命运、非生物胁迫等生命活动中发挥着重要的调控作用[1-6]。KNOX亞家族几乎存在于所有植物中,其编码蛋白N端至C端依次存在MEINOX、GSE、ELK、HD 4个保守结构域,MEINOX(包含KNOX1和KNOX2结构域)和ELK结构域与蛋白质间的相互作用有关,核定位信号位于ELK结构域,GSE结构域涉及泛素降解途径调节蛋白的稳定性,HD位于蛋白的C端[7]。由于在植物中的表达模式和基因中内含子位置存在差异,KNOX亚家族被进一步分为ClassⅠ和ClassⅡ 2大亚家族[8]。ClassⅠ类亚家族主要在分生组织中表达,是分生组织发生与维持所必需的关键转录基因,参与侧生器官的形态建成[9-10]、调控激素[11-12]和木质素合成[13-14];ClassⅡ类亚家族在所有植物组织均有表达,目前发现调控植物次生细胞壁形成和纤维的发育[15-17]。
现有研究发现KNOX成员可通过蛋白质相互作用,形成复合物调控植物细胞壁的形成、木质素的合成、细胞壁的扩张和加固[18-21]。Abraham- Juárez等[22]首次报道了剑麻中2个Ⅰ类KNOX基因。接着Zhou等[23]研究发现一个Ⅰ类KNOX基因在茎尖分生组织和幼龄的叶片中高表达。Gross等[24]的研究结果则表明剑麻Ⅰ类KNOX基因在叶片基部表达,在叶片中上部不表达。剑麻KNOX2调控珠芽的发育,在珠芽发育的起始阶段,剑麻KNOX2随着珠芽的增大,表达量逐渐增加;在拟南芥中过表达剑麻KNOX2,导致叶出现裂片的表型;进一步发现剑麻KNOX2能够回补拟南芥knat1突变表型[22]。植物KNOX基因参与叶片的形态建成和纤维的发育,初步推测剑麻KNOX2基因参与调控剑麻叶片和纤维的发育。因此,本课题组克隆了国内主栽品种H.11648(Agave hybrid No.11648)的AhKNOX2基因,构建剑麻叶片酵母双杂交cDNA表达文库,筛选与AhKNOX2相互作用的蛋白,从蛋白质相互作用的角度研究AhKNOX2基因在剑麻叶片中的功能和调控网络,结果为培育高纤维产量的剑麻新品种提供基因资源和理论基础。
1 材料与方法
1.1 材料
剑麻H.11648采自中国热带农业科学院南亚热带作物研究所剑麻种质保存圃;RNA提取试剂盒购自北京华越洋生物科技有限公司;反转录试剂盒、Hi-Fusion试剂盒、Nde I和BamH I购自江苏愚公生命科技有限公司;双链cDNA纯化、酵母转化和文库构建试剂盒购自Clontech公司;用于配制酵母培养基的试剂和其他试剂购自Sigma- Aldrich公司。
1.2 方法
1.2.1 总RNA的提取 采集H.11648品种5龄植株叶样品,液氮速冻。将样品研磨成粉,按照华越洋多糖多酚RNA提取试剂盒操作,取50~100 mg样品提取其总RNA。
1.2.2 RNA的反转录 反转录按照愚公生物M- MLV GIII First-Strand Synthesis Kit操作。反应体系中加入1.0 μL CDS Ⅲ和1.0 μL SMART Ⅲ引物,以提取的总RNA为模板,反转录获得带同源重组接头的单链cDNA。反转录程序为:55 ℃30 min;85 ℃ 1 min。CDS Ⅲ和1.0 μL SMART Ⅲ引物序列见表1。
1.2.3 双链cDNA的合成 以反转录的单链cDNA为模板,加入5 PCR引物和3 PCR引物(表1),利用Advantage 2 Polymerase Mix合成双链cDNA。PCR扩增条件为:95 ℃ 30 s;95 ℃ 10 s,68 ℃ 6 min,68 ℃ 5 min,26个循环。
1.2.4 双链cDNA的纯化 预先将CHROMA SPINTM TE-400纯化柱中胶体和缓冲液混合均匀,瞬时离心弃液体。将93 μL双链cDNA加入纯化柱胶体,700?g离心5 min。向收集液中加入2.5倍體积无水乙醇和1/10体积的3 mol/L乙酸钠,置于20 ℃沉淀DNA。用20 μL无菌水溶解沉淀物,并测定双链cDNA浓度。
1.2.5 文库构建 按照YeastmakerTM Yeast Trans-formation System 2操作制备酵母感受态细胞。取纯化的双链cDNA(2~5 μg)与3 μg线性化pGADT7-Rec质粒共转化Y187酵母菌,转化菌体用6 mL生理盐水悬浮,每个平板100 μL,将悬浮液涂布于不含亮氨酸的综合培养基(SD/-Leu)平板上,于30 ℃培养5 d。将培养平板于4 ℃放置 4 h,向平板加入5 mL液体酵母膏胨葡萄糖琼脂培养基(YPDA)(含25%的甘油),用涂布棒刮离酵母菌,收集菌体悬浮液。浓缩文库体积,血球板计数法计算细胞密度,使文库细胞密度大于2×107/mL。按每管1 mL,将浓缩液分装于1.5 mL无菌管中,保存于80 ℃。
1.2.6 文库质量检测 取100 μL悬浮液用生理盐水逐步稀释10倍和100倍,取100 μL稀释液涂布SD/-Leu平板,30 ℃培养5 d。采用ImageJ软件统计生长菌落数。按照如下公式分别计算文库容量和转化效率:
挑取SD/-Leu平板上的菌落,利用pGADT7- Rec的载体引物PCR鉴定插入片段。PCR扩增条件为:95 ℃ 10 min;95 ℃ 45 s,58 ℃ 45 s,72 ℃ 3 min,55 个循环;72 ℃ 5 min。用1%琼脂糖凝胶电泳检测。按照如下公式计算重组率:
取10 μL文库保存液悬浮于1 mL YPDA液体培养基中,混合均匀得稀释液A(稀释度102)。从稀释液A中吸取10 μL悬浮液加入1 mL YPDA液体培养基中,混合均匀得稀释液B(稀释度104)。分别从稀释液A和稀释液B中取10 μL和50 μL悬浮液涂布于SD/-Leu平板,30 ℃培养5 d。采用ImageJ软件统计生长菌落数。按照如下公式计算文库滴度:
1.2.7 诱饵载体构建 设计携带Nde I和BamH I酶切位点的特异引物(表1),扩增AhKNOX2编码框。将AhKNOX2编码框插入pGBKT7载体,构建诱饵载体。诱饵载体双酶切检测并送测序。
1.2.8 诱饵载体自激活检测及3-氨基-1,2,4-三唑(3-AT)抑制浓度筛选 将pGBKT7-AhKNOX2载体转化Y2HGold酵母菌株,转化液分别涂布于不含色氨酸的综合培养基(SD/-Trp)、SD/-Leu、不含色氨酸和腺嘌呤的综合培养基(SD/-Trp/-Ade)和不含色氨酸和组氨酸的综合培养基(SD/-Trp/-His)平板,30 ℃培养5 d,以检测诱饵载体是否具有自激活性。将携带有诱饵载体的Y2HGold酵母菌株涂布于含不同浓度的3-AT的SD/-Trp/-His平板,30 ℃培养5 d,以确定3-AT的抑制浓度。
1.2.9 互作蛋白筛选 使用50 mL SD/-Trp液体培养基扩大含诱饵载体的Y2HGold酵母菌株,收集菌体与1 mL文库菌体混合于50 mL 2×YPDA液体培养基中,30 ℃培养20~24 h。收集菌体,使用10 mL 0.5×YPDA液体培养基悬浮共培养的菌体,并涂布于含3-AT的SD/-Trp/-Leu/-His平板,30 ℃培养5 d。筛选平板长出的菌落进行挑取,转接3次到SD/-Trp/-Leu/-His平板上。挑取剩下活的菌落,接种于SD/-Trp/-Leu液体培养基中,30 ℃、250 r/min培养20~24 h,收集菌体提取酵母质粒。质粒转化DH5α大肠杆菌并提取质粒送测序。
1.2.10 阳性克隆cDNA的注释 采用本地Blast的方式,将测序获得的阳性克隆cDNA序列与剑麻H.11648的转录组序列进行比对,根据转录组的注释信息,获得候选互作蛋白的功能等信息。
2 结果与分析
2.1 总RNA的提取
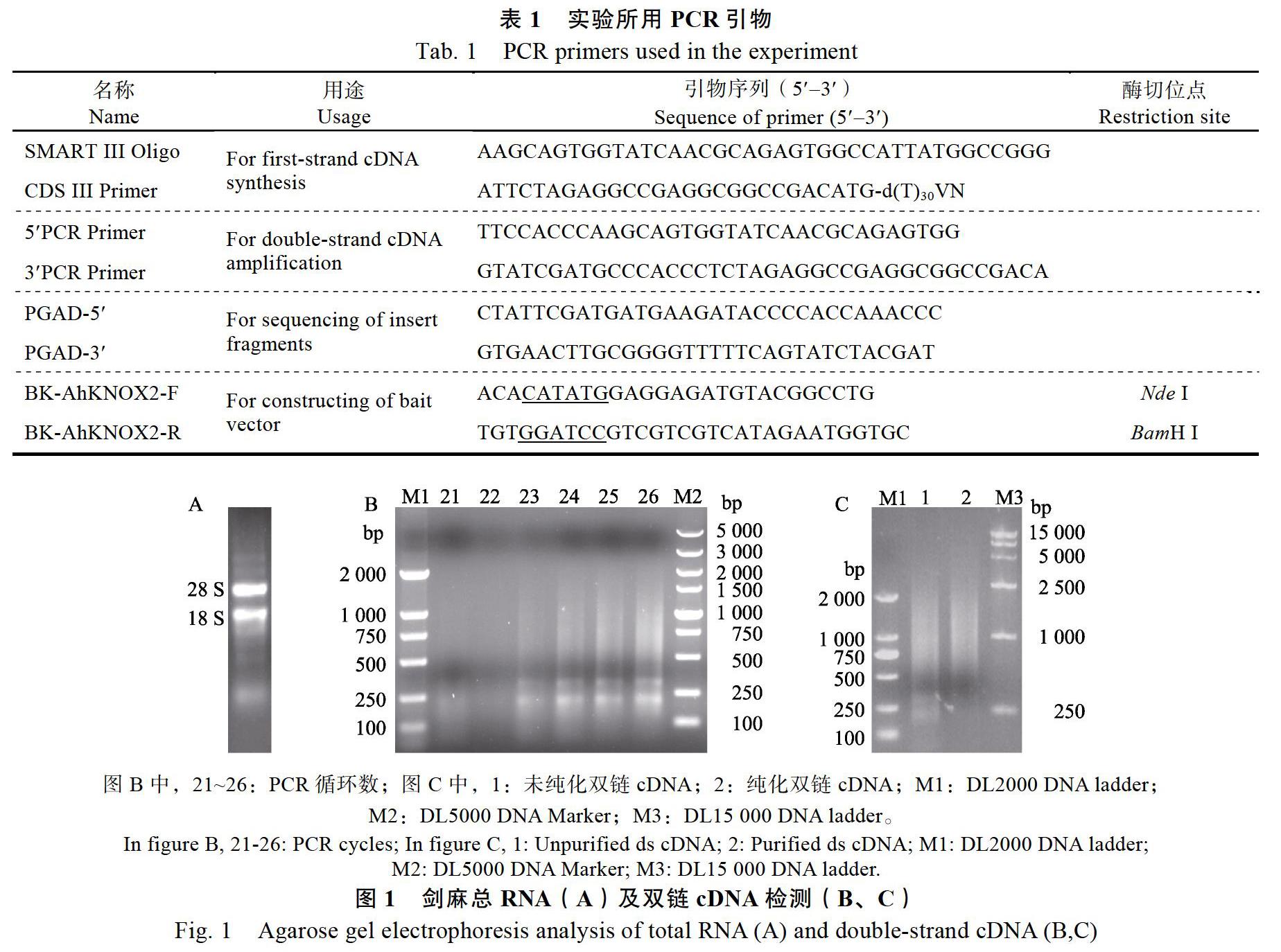
提取的剑麻总RNA经1%琼脂糖凝胶检测,结果显示28S和18S条带清晰,RNA未降解(图1A);OD260/OD280比值为1.95,表明提取的总RNA纯度和质量较高,可用于后续实验。
2.2 cDNA的合成及纯化
分别在合成双链cDNA的第21~26个循环取5 μL PCR产物,经1%琼脂糖凝胶检测,结果显示,第21和第22个循环的PCR产物不明显,从第23个循环开始,PCR产物明显且浓度逐渐增加(图1B)。合成的cDNA片段分布于0.2~5.0 kb之间,且主要集中在1.0 kb左右。对第26个循环合成的双链cDNA进行纯化,纯化的双链cDNA中0.5 kb以下小片段含量明显降低(图1C),纯化的双链cDNA总量为4.26 μg(213 ng/μL)。
2.3 文库构建及质量检测
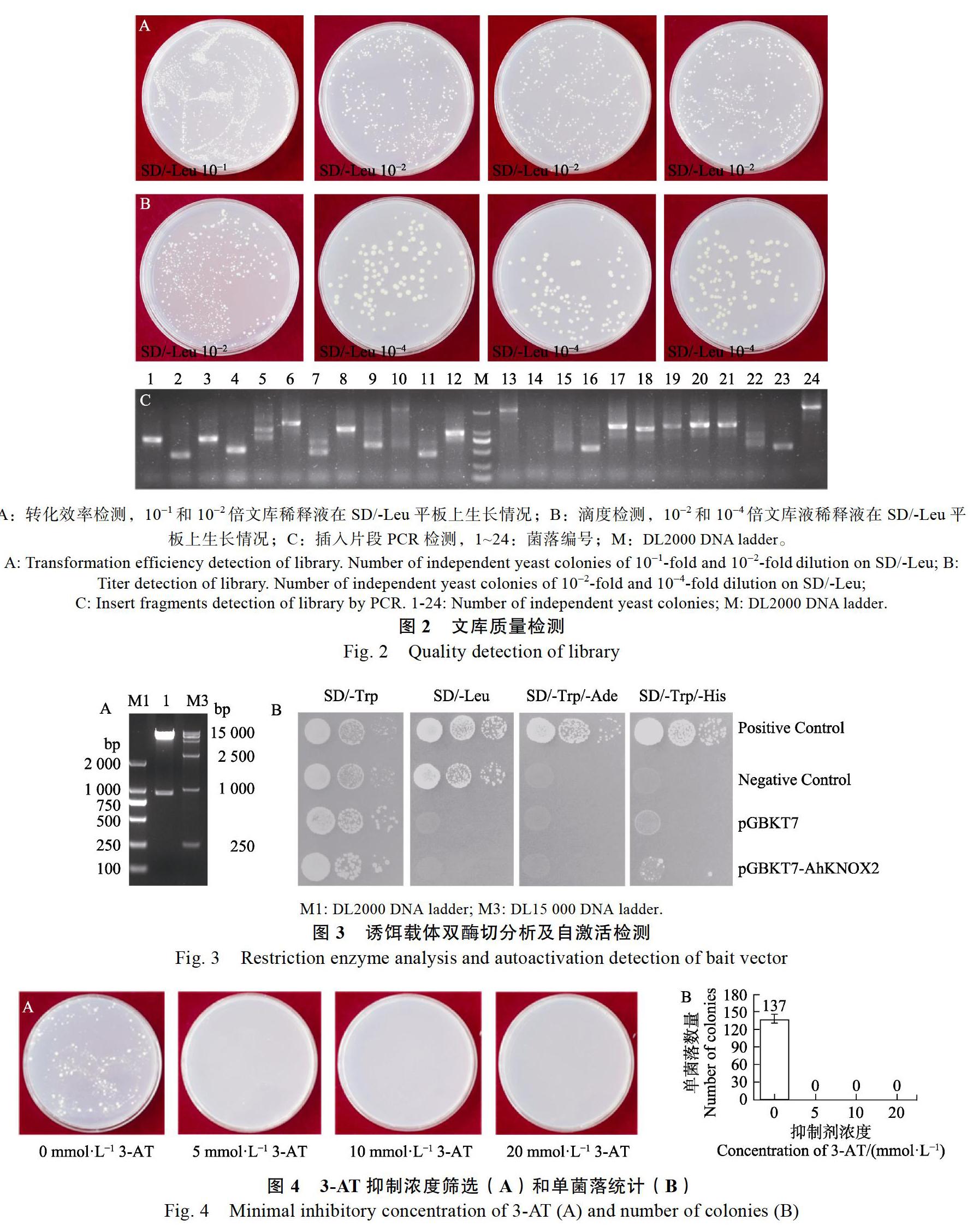
Image J软件统计100 μL 102倍的文库稀释液在SD/-Leu平板上平均长出了650个单菌落(图2A),文库总容量为3.9106 CFU,转化效率为9.15105 CFU/μg pGADT7-Rec。100倍稀释液经镜检,80个小格共计27个酵母细胞,文库细胞密度为1.35×108/mL。Image J软件统计50 μL 104倍的文库稀释液在SD/-Leu平板上平均长出了111个单菌落(图2B),文库滴度为2.22107 CFU/ mL。菌落PCR产物经1%琼脂糖电泳检测,结果显示插入片段在0.5~3.0 kb之间,少量酵母菌含有多个重组质粒(图2C),重组率为92%。
2.4 诱饵载体构建
pGBKT7-AhKNOX2载体经Nde I和BamH I内切酶双酶切出现900 bp左右条带(图3A),测序结果表明,插入片段即为AhKNOX2的編码框,双酶切和测序结果说明诱饵载体构建成功。
2.5 自激活检测
阳性对照可在4个培养基上生长(p53蛋白和T-antigen相互作用)。阴性对照仅在SD/-Trp和SD/-Leu培养基上生长(Lam和T-antigen不相互作用)。空载体对照(pGBKT7)仅能在SD/-Trp培养基上生长。含有pGBKT7-AhKNOX2载体的Y2HGold酵母可在SD/-Trp培养基生长,在SD/-Leu和SD/-Trp/-Ade培养基上不生长,但在SD/-Trp/- His培养基上生长了少量的菌落(图3B),结果表明,诱饵载体没有激活报告基因ADE2的表达,但激活了报告基因HIS3的表达,存在自激活性。
2.6 3-AT抑制浓度筛选
由于KNOX2存在自激活性,能激活His的表达,使Y2HGold酵母能在缺His(组氨基酸)的培养基生长,因此,采用在培养基中加入3-AT来抑制自激活。含有pGBKT7-AhKNOX2载体的Y2HGold酵母能在不含3-AT的SD/-Trp/-His培养基上生长,而在加了5 mmol/L及更高浓度3-AT的SD/-Trp/-His培养基上不能生长(图4)。由此可知,5 mmol/L的3-AT就能抑制诱饵载体的自激活性。
2.7 互作蛋白筛选
经SD/–Trp/–Leu/–His/+5 mmol/L 3-AT培养基的筛选,获得16个与AhKNOX2相互作用的阳性克隆,将阳性克隆cDNA测序序列与剑麻转录组数据比对,获得与AhKNOX2相互作用蛋白的cDNA注释信息(表2)。根据候选蛋白的功能,可分为蛋白/DNA结合蛋白、蛋白/RNA结合蛋白、核苷酸结合蛋白、蛋白质代谢、蛋白质加工、碳水化合物代谢、光形态建成和未知蛋白8大类。
3 讨论
酵母双杂交是目前能够高通量筛选相互作用蛋白的技术之一。但大多数转录因子具备转录激活功能,这就导致构建的诱饵载体往往存在自激活,因此抑制诱饵载体的自激活是降低假阳性的关键之一。本研究采用的是Clontech公司的Matchmaker? Gold酵母双杂交系统,该系统采用3个异源的Gal4响应启动子元件(G1、G2和M1)控制HIS3、ADE2、MEL1/AUR1-C 4个报告基因的表达,目的是通过增加报告基因来降低筛选结果的假阳性。3-AT是HIS3蛋白的竞争性抑制剂,能够抑制组氨酸的合成[25-26]。如果在实验过程中发现诱饵载体存在自激活,可通过在筛选培养基中增加3-AT来抑制由诱饵蛋白激活表达的HIS3蛋白的活性,减少自激活背景[27]。本实验发现构建的pGBKT7-AhKNOX2存在自激活性,启动了HIS3报告基因的表达,向SD/–Trp/–His培养基中添加5 mmol/L的3-AT能抑制其自激活能力,因此将SD/–Trp/–Leu/–His/+5 mmol/L 3-AT培养基作为文库筛选培养基。
KNOX蛋白能形成同源或者异源二聚体,通过改变亚细胞定位实现调控作用。从KNOX蛋白的结构域功能看,MEINOX结构域对形成二聚体起重要作用,ELK结构域参与蛋白质之间的相互作用,而HD结构域参与DNA的结合和同源二聚体的形成[7],表明KNOX蛋白能形成同源或者异源二聚体,而蛋白质相互作用往往会改变复合体的亚细胞定位。研究发现,KNOX与BELL相互作用形成异源二聚体,进入细胞核,从细胞质移动到细胞核依赖于二者形成复合物[28]。拟南芥OFPs(Arabidopsis thaliana ovate family proteins)与KNAT1蛋白相结合,将KNAT1蛋白从细胞核中移动至细胞质中的微管上[29]。KNATM与KNOX的结合,阻止了KNOX进入细胞核[30]。本研究通过酵母双杂交筛选获得16个与AhKNOX2相互作用的候选蛋白,16个候选蛋白中没有同源异型盒基因家族蛋白,表明AhKNOX2与候选蛋白形成异源二聚体发挥作用。
KNOX可通過蛋白质相互作用参与调控植物发育的多个事件,目前发现与KNOX相互作用的蛋白主要是转录因子,如与SH5相互作用抑制木质素的合成,调控落果[20];与OFP和MYB转录因子相互作用,调控纤维细胞和次生细胞壁发育[16-17, 19];与NAC直接相互作用,抑制其下游基因CESAs等的表达,调控细胞壁加厚[21];与水稻生长调控因子GRF4互作,抑制其下游基因Expansin等的表达,调控细胞扩展[21];与KNOX同源蛋白间相互作用,调控开花[31-32]。本研究通过筛选,一共获得16个阳性克隆,其中3个为转录因子(MADS、VOZ、bHLH),AhKNOX2与这些蛋白的相互作用在剑麻中的具体功能还有待深入研究。
参考文献
[1] Li P H, Ponnala L, Gandotra N, et al. The developmental dynamics of the maize leaf transcriptome[J]. Nature Genetics, 2010, 42(12): 1060-1067.
[2] 孙海龙, 侍 婷, 章 镇, 等. KNOXⅠ类基因在雌蕊发育中的作用[J]. 江苏林业科技, 2012, 39(5): 39-44.
[3] Zhao Y, Ma Q, Jin X L, et al. A novel maize homeodomain-leucine zipper (HD-Zip) I gene, Zmhdz10, positively regulates drought and salt tolerance in both rice and Arabidopsis[J]. Plant Cell Physiology, 2014, 55(6): 1142-1156.
[4] Shafiullah M D, Lacroix C R. Extended expression of MaKN1 contributes to the leaf morphology in aquatic forms of Myriophyllum aquaticum[J]. Botany, 2015, 93(9): 611- 621.
[5] 朱钦士. 植物和动物的殊途同归(2)[J]. 生物学通报, 2017, 52(11): 12-15.
[6] Du F, Guan C F, Jiao Y L. Molecular mechanisms of leaf morphogenesis[J]. Molecular Plant, 2018, 11(9): 1117-1134.
[7] Sano R, Juárez C M, Hass B, et al. KNOX homeobox genes potentially have similar function in both diploid unicellular and multicellular meristems, but not in haploid meristems[J]. Evolution & Development, 2005, 7(1): 69-78.
[8] Kerstetter R, Vollbrecht E, Lowe B, et al. Sequence analysis and expression patterns divide the maize knotted1-like homeobox genes into two classes[J]. The Plant Cell, 1994, 6(12): 1877-1887.
[9] Ramirez J, Bolduc N, Lisch D, et al. Distal expression of knotted1 in maize leaves leads to reestablishment of proximal/distal patterning and leaf dissection[J]. Plant Physiology, 2009, 151(4): 1878-1888.
[10] Kalve S, De V D, Beemster G T S. Leaf development: a cellular perspective[J]. Frontiers in Plant Science, 2014, 5: 362.
[11] Bolduc N, Hake S. The maize transcription factor KNOTTED1 directly regulates the gibberellin catabolism gene ga2ox1[J]. The Plant Cell, 2009, 21(6): 1647-1658.
[12] Ma C, Meir S, Xiao L T, et al. A KNOTTED1-LIKE HOMEOBOX protein, KD1, regulates abscission in tomato by modulating the auxin pathway[J]. Plant Physiololgy, 2015, 167(3): 844-853.
[13] Townsley B T, Sinha N R, Kang J. KNOX1 genes regulate lignin deposition and composition in monocots and dicots[J]. Frontiers in Plant Science, 2013, 4: 121.
[14] Woerlen N, Allam G, Popescu A, et al. Repression of BLADE-ON-PETIOLE genes by KNOX homeodomain protein BREVIPEDICELLUS is essential for differentiation of secondary xylem in Arabidopsis root[J]. Planta, 2017, 245(6): 1079-1090.
[15] Li E Y, Bhargava A, Qiang W Y, et al. The Class II KNOX gene KNAT7 negatively regulates secondary wall formation in Arabidopsis and is functionally conserved in Populus[J]. New Phytologist, 2012, 194(1): 102-115.
[16] Bhargava A, Ahad A, Wang S, et al. The interacting MYB75 and KNAT7 transcription factors modulate secondary cell wall deposition both in stems and seed coat in Arabidopsis[J]. Planta, 2013, 237(5): 1199-1211.
[17] Gong S Y, Huang G Q, Sun X, et al. Cotton KNL1, encoding a class II KNOX transcription factor, is involved in regulation of fibre development[J]. Journal of Experimental Botany, 2014, 65(15): 4133-4147.
[18] Kuijt S J, Greco R, Agalou A, et al. Interaction between the GROWTH-REGULATING FACTOR and KNOTTED1- LIKE HOMEOBOX families of transcription factors[J]. Plant Physiology, 2014, 164(4): 1952-1966.
[19] Liu Y, Douglas C J. A role for OVATE FAMILY PROTEIN1 (OFP1) and OFP4 in a BLH6-KNAT7 multi- pro?tein complex regulating secondary cell wall formation in Arabidopsis thaliana[J]. Plant Signaling Behavior, 2015, 10(7): e1033126.
[20] Yoon J, Cho L H, Antt H W, et al. KNOX protein OSH15 induces grain shattering by repressing lignin biosynthesis genes[J]. Plant Physiology, 2017, 174(1): 312-325.
[21] Wang S H, Yang H L, Mei J S, et al. A rice homeobox protein KNAT7 integrates the pathways regulating cell expansion and wall stiffness[J]. Plant Physiology, 2019, 181(2): 669-682.
[22] Abraham-Juárez M J, Martínez-Hernández A, Leyva-Gon-zález M A, et al. Class I KNOX genes are associated with organogenesis during bulbil formation in Agave tequilana[J]. Journal of Experimental Botany, 2010, 61(14): 4055-4067.
[23] Zhou W Z, Zhang Y M, Lu J Y, et al. Construction and evaluation of normalized cDNA libraries enriched with full-length sequences for rapid discovery of new genes from sisal (Agave sisalana Perr.) different developmental stages[J]. International Journal of Molecular Sciences, 2012, 13(10): 13150-13168.
[24] Gross S M, Martin J A, Simpson J, et al. De novo transcriptome assembly of drought tolerant CAM plants, Agave deserti and Agave tequilana[J]. BMC Genomics, 2013, 14: 563.
[25] Fields S. The two-hybrid system to detect protein-protein interactions[J]. Methods, 1993, 5(2): 116-124.
[26] Durfee T, Becherer K, Chen P L, et al. The retinoblastoma protein associates with the protein phosphatase type 1 catalytic subunit[J]. Genes Development, 1993, 7(4): 555-569.
[27] Shan C M, Shangguan X X, Zhao B, et al. Control of cotton fibre elongation by a homeodomain transcription factor GhHOX3[J]. Nature Communications, 2014, 5: 5519.
[28] Kumar R, Kushalappa K, Godt D, et al. The Arabidopsis BEL1-LIKE HOMEODOMAIN proteins SAW1 and SAW2 act redundantly to regulate KNOX expression spatially in leaf margins[J]. The Plant Cell, 2007, 19(9): 2719-2735.
[29] Hackbusch J, Richter K, Müller J, et al. A central role of Arabidopsis thaliana ovate family proteins in networking and subcellular localization of 3-aa loop extension homeodomain proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 29, 102(13): 4908-4912.
[30] Magnani E, Hake S. KNOX lost the OX: the Arabidopsis KNATM gene defines a novel class of KNOX transcriptional regulators missing the homeodomain[J]. The Plant Cell, 2008, 20(4): 875-887.
[31] Smith H M, Hake S. The interaction of two homeobox genes, BREVIPEDICELLUS and PENNYWISE, regulates internode patterning in the Arabidopsis inflorescence[J]. The Plant Cell, 2003, 15(8): 1717-1727.
[32] Ragni L, Belles-Boix E, Günl M, et al. Interaction of KNAT6 and KNAT2 with BREVIPEDICELLUS and PENNYWISE in Arabidopsis in?orescences[J]. The Plant Cell, 2008, 20(4): 888-900.

