凝结芽孢杆菌次级代谢挖掘与泛基因组分析
钱文江 汪步青 李葳茜 杨雪苗 刘洪伟 张丽萍
(1. 河北工业大学化工学院,天津 300130;2. 河北省科学院生物研究所,石家庄 050081)
凝结芽孢杆菌(Bacillus coagulans)是一类革兰氏阳性菌,在发酵培养过程中能分解糖类生成乳酸,也被称作乳酸发酵菌[1]。凝结芽孢杆菌在自然界中广泛分布,其在生长繁殖过程中产生一种抗菌肽-凝结素,对多种致病菌都具有杀菌活性[2]。凝结芽孢杆菌不仅具有乳酸菌和双歧杆菌相同的营养特征,还具有很强的耐酸、耐热、耐盐等性状[3]。1998年,Hyronimus等[4]研究发现,凝结芽孢杆菌I4能产生凝结素,它是一类抗菌肽类物质,能够抑制沙门菌、小球菌等诸多致病菌的生长。2006年,Huszcza等[5]发现凝结芽孢杆菌生长过程中会有多种表面活性素产生,其中surfactin是一种强大的脂肽类表面活性剂。同时,2009年,Kodali等[6]在凝结芽孢杆菌RK-02中分离出一种胞外多糖(EPS)具有乳化活性。2015年,赵钰等[7]发现在凝结芽胞杆菌LL1103发酵液中存在可以抑制革兰氏阳性菌生长的细菌素。刘全永等[8]发现凝结芽胞杆菌LU-B02发酵液对白色念珠菌生长有抑制作用。此外,也有研究表明凝结芽孢杆菌在生长过程中也会产生了乳糖酶、β-半乳糖苷酶、木聚糖酶、纤溶活性酶等物质。
到目前为止,在NCBI上可以查询到的凝结芽孢杆菌共有33株并且都完成了基因组测序。属于同一菌种的细菌菌株的基因含量之间差异很大,而泛基因组的遗传信息要比单个菌株的遗传信息含量大得多[9]。泛基因组的概念是由Tettelin等[10]在2005年提出,泛基因组是指某一个物种所有存在的基因,它包括核心基因组(在全部菌株中都包含的基因),非必须基因组(只有部分菌株都含有的基因)和菌株具有的特有基因[11]。最近几年,泛基因组分析在细菌和真菌功能基因挖掘中应用广泛[12]。根据泛基因总数与基因组个数的关系,细菌的泛基因组可以分为两种类型,分别是开放型和闭合型[13]。开放型泛基因组是指随着预测的基因组个数的增多,泛基因总数也相应增加,而闭合型泛基因组是指在预测的基因组个数增加到某一数值时泛基因总数趋于不变[14]。对细菌的次级代谢产物进行分析时,传统的分析方法可能会有局限性,但是分析细菌中已知的基因组数据,可能会挖掘出崭新的次级代谢基因簇,并会有潜在的活性物质出现[15]。
本研究从NCBI上找到了33株凝结芽孢杆菌的基因组,首先对其中11株有完整基因组水平的凝结芽孢杆菌进行了泛基因组分析,找出了其泛基因组的大小;随后利用antiSMASH软件对33株凝结芽孢杆菌的次级代谢基因簇进行挖掘,发现了其最可能产生的活性物质[16]。本研究旨在对凝结芽孢杆菌的基因组信息进行探索,为以后研究凝结芽孢杆菌的进化,适应和种群结构的方式奠定一定的基础。
1 材料与方法
1.1 材料
从NCBI基因组数据库(https://www.ncbi.nlm.nih.gov/ genome)中查找到33株凝结芽胞杆菌的NCBI登录号,在以GenBank格式下载获得基因组信息。本研究中使用的33株凝结芽孢杆菌的基因组数据全部来自2020年1月5日之前在GenBank数据库中提交的基因组信息,详细信息如表1所示。
1.2 方法
1.2.1 ANI值和DDH值分析 平均核苷酸一致性(ANI)可以用来判断菌株是否为同一个种或亚种,而ANI值达到95%以上的菌株认为同一个种。本研究中使用在线软件(http://enve-omics.ce.gatech.edu/g-matrix/)中的ANI/AAI matrix对33株凝结芽孢杆菌的基因组ANI值进行计算[17]。
DDH值是指基因组与基因组之间的距离,通常将DDH值大于70%的菌株认为是同种菌株使用。使用在线软件(http://ggdc.dsmz.de/)中的GGDC计算33株凝结芽孢杆菌基因组的DDH值[18]。
1.2.2 基因组系统发育分析 对表1中33株凝结芽孢杆菌以genbank格式在NCBI数据库上下载基因组数据,利用REALPHY(基于参考序列比对的系统发生构建器)在线软件进行全基因组数据比对,登录http://realphy.unibas.ch网 址,以GenBank格式进行上传基因组数据,使用默认参数运行[19]。获得的数据结果使用FigTree软件构建进化树[20]。
1.2.3 核心基因组和泛基因组分析 从NCBI数据库中下载如表1中11株组装到完整基因组水平的凝结芽孢杆菌的基因组序列文件(.fna)和基因组注释文件(.ppt)作为上传数据,PGAweb软件的PGAP-X模块选择GeneFamily Method(GF)算法对11个基因组进行分析,使用默认值运行[21]。下载结果文件中,选择Orthologs_Cluster.txt文件,使用PanGP软件进行泛基因组数据拟合[22]。
1.2.4 次级代谢产物合成基因簇分析 利用antiSMASH5.0(https://antismash.secondarymetabolites.org)在线软件的细菌分析模块中[22],输入菌株NCBI登录号,选择relaxed预测模式对33株凝结芽孢杆菌次级代谢产物生物合成基因簇进行预测注释,参数选用默认值[23]。
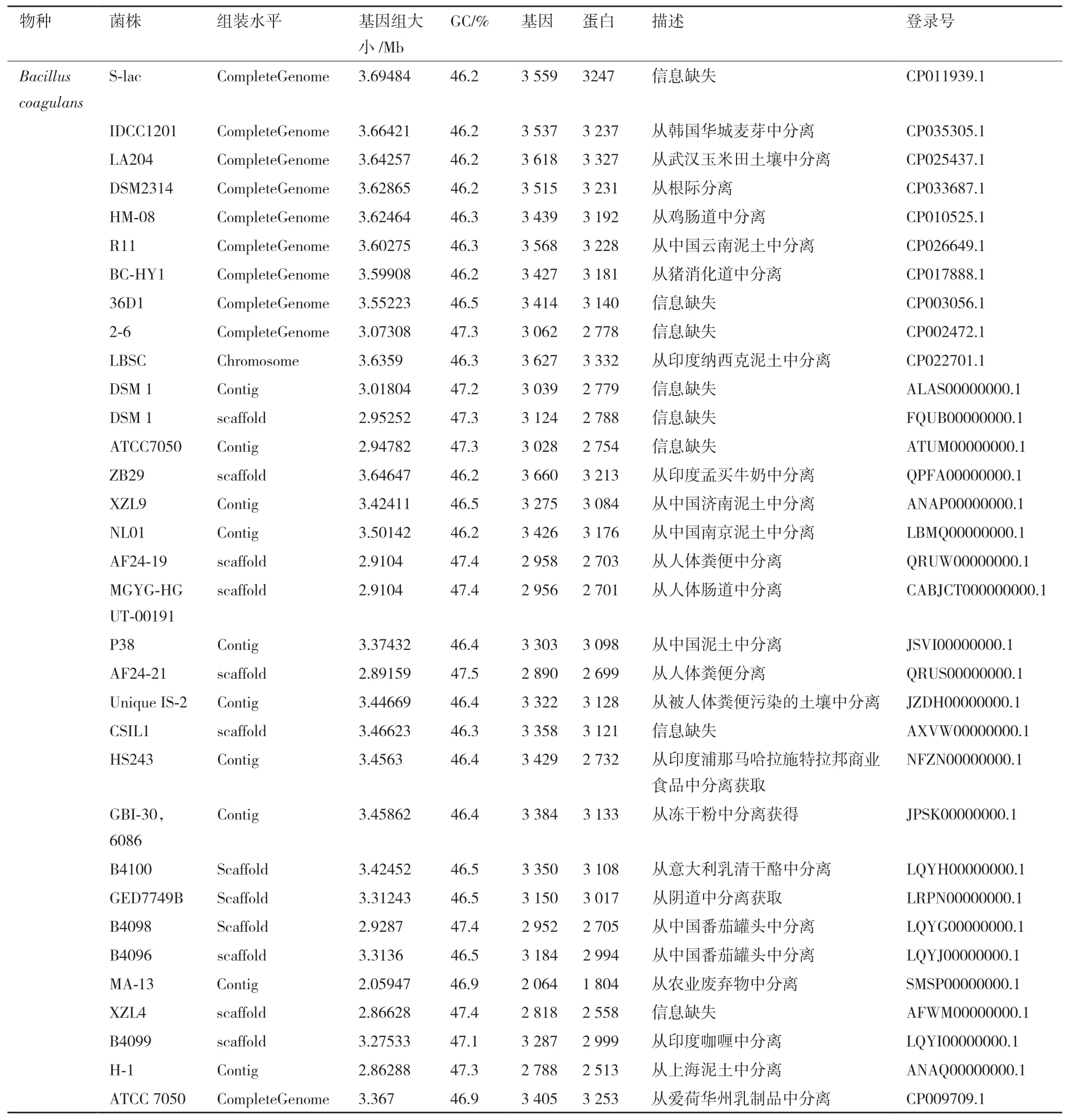
表1 研究分析中所使用的菌株
2 结果
2.1 凝结芽孢杆菌基因组基本数据分析
如 表1所 示,到2020年1月12日 为 止,在NCBI数据库中查找到共有33株凝结芽孢杆菌的基因组,其中共有11株凝结芽孢杆菌基因组装到完整基因组水平。由基因组数据统计发现,33株环状芽孢杆菌的基因组大小范围是2.059 47-3.694 84 Mb,而相应的GC含量范围为46.2%-47.5%,预测到的基因数范围是2 064-3 660个。
2.2 ANI值和DDH值分析和基因组系统发育分析
通过使用ANI值和DDH值对33株凝结芽孢杆菌的序列同源性进行了评估。本研究以Bacillus coagulan P38的全基因组序列为参考,计算了菌株两两间DDH值(图1-A)和ANI值(图1-B)。结果显示,其中20株凝结芽孢杆菌两两间ANI 值均>95%,DDH值均>70%是同一种凝结芽孢杆菌,而剩余的13株菌两两间的DDH值≤70%和ANI值≤95%,这13株菌不是同一种凝结芽孢杆菌[24]。通过使用REALPHY在线软件将33株凝结芽孢杆菌基因组进行了比对,然后使FigTree软件构建进化树。全基因组系统发育显示(图1-C),33株凝结芽孢杆菌在基因组系统进化树上被归为两个分支,一个支包 括 有2-6、B4098、H-1、MA-13、XZL4、B4099、ATCC7050、DSM_1、DSM_1_1、ATCC7050_1、AF24-21、MGYG-HGUT-00191、AF24-19,其余菌株是另一支。最后,全基因组系统发育树结果与ANI值和DDH值的观察结果一致。
2.3 核心基因组和泛基因组分析�
对11株组装完整基因组水平的凝结芽孢杆菌进行了泛基因组分析。由图2可知,共有34 647个蛋白质编码的基因用于聚类分析,被分为5 899个基因家族中,每个基因家族可能代表一个的同源基因。核心基因组是指在全部基因组中均包含的基因,如图3所示,在11株凝结芽孢杆菌的基因组中,核心基因组是由2 152个基因家族组成,共有的核心基因组约占凝结芽孢杆菌泛基因组的36.48%。发现特有基因2 255个,占凝结芽孢杆菌泛基因组的38.22%。
基于全基因组基因聚类结果,我们使用PanGP软件计算了泛基因组、核心基因组和基因组数目之间的关系。如图3所示,泛基因组大小(T)与基因组数(X)关系的拟合方程为T=1 801.66X0.38+1 417.71(R2=0.999 98),拟合方程表明凝结芽孢杆菌的泛基因组中的基因数是随着基因组个数的增加而增多。核心基因的个数(D)与基因组数(N)关系的拟合方程为D=1 751.08e-0.53N+2 151.5(R2=0.980 9)。根据公式推测出凝结芽孢杆菌的核心基因组有2 152个基因组成。不同的基因组中有相同的基因家族,当每个基因家族覆盖的基因组个数增多时,这个基因家族中的基因就相对开放[25]。在曲线中显示了泛基因数和核心基因数随基因组增加而发生的变化。在该曲线中可以看出凝结芽孢杆菌的泛基因组是开放性的。
推测出每增加一个新的凝结芽孢杆菌基因组,大约有150个新基因被发现。如图4所示,PanGP软件计算了新基因家族数量(M)与基因组数(F)的关系,并计算出拟合方程为M=876.572F-0.71(R2=0.996 822)。
2.4 次级代谢产物合成基因簇
AntiSMASH是一种强大而全面的生物信息学工具,可用于识别和注释编码次生代谢产物的生物合成基因簇,并已在该领域中广泛使用[26]。利用antiSMASH软件对33株凝结芽孢杆菌基因组中的次级代谢基因簇进行在线预测,预测结果总共注释到8类、79个与次级代谢相关的基因或基因簇。与基因组大小无关,所测试的33株凝结芽孢杆菌中存在24株凝结芽孢杆菌都携带大量基因簇,并且这些基因簇编码不同的潜在生物活性物质。鉴定出有细菌素的基因簇平均在每个菌株中介于一到两个之间。如图5和表2所示,可以注释到的次级代谢基因簇中有11个糖类(Saccharide)、30个细菌素(Bacteriocin)、2个脂肪酸(Fatty acid)、10个Ⅲ型聚酮化合物合酶(T3PKS)、18个内酯(Betalactone)、3个LAP、2个萜烯(terpene)和3个硫肽(Thiopeptide)基因簇。Riazi等[27]在2009年研究发现了凝结芽孢杆菌ATCC 7050可以产生一种抗菌蛋白-乳酸菌素(Lactosporin),乳酸菌素和表2中对凝结芽孢杆菌ATCC 7050预测的次级代谢产物T3PKS都属于核糖体途径生成的抗菌蛋白类物质。在线预测结果表明,目前预测凝结芽孢杆菌的主要次级代谢产物可能是细菌素、T3PKS、硫肽、内酯和糖类等化合物。
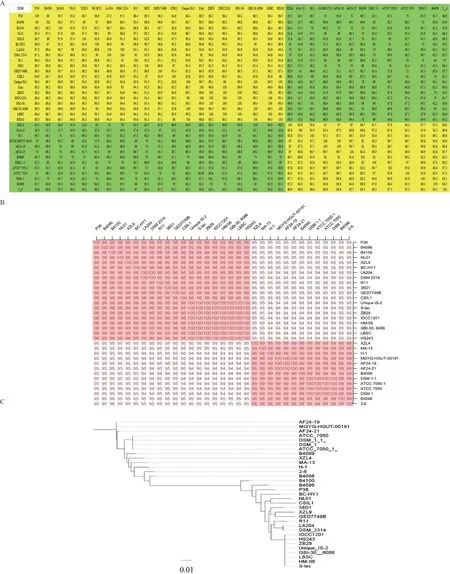
图1 33株凝结芽孢杆菌ANI值和DDH值分析及全基因组系统发育树分析
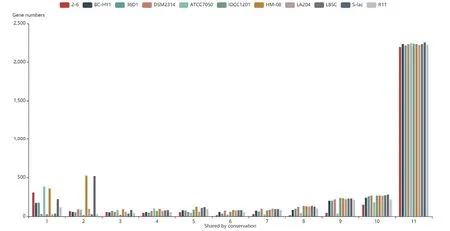
图2 凝结芽孢杆菌基因组中核心基因和非必须基因
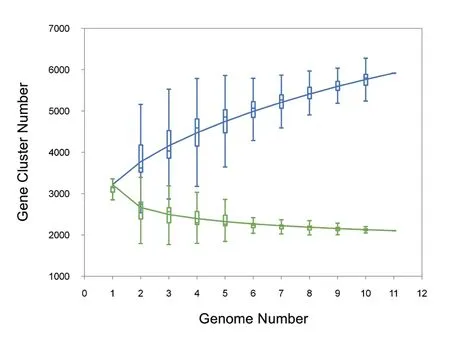
图3 凝结芽孢杆菌的泛基因组分析
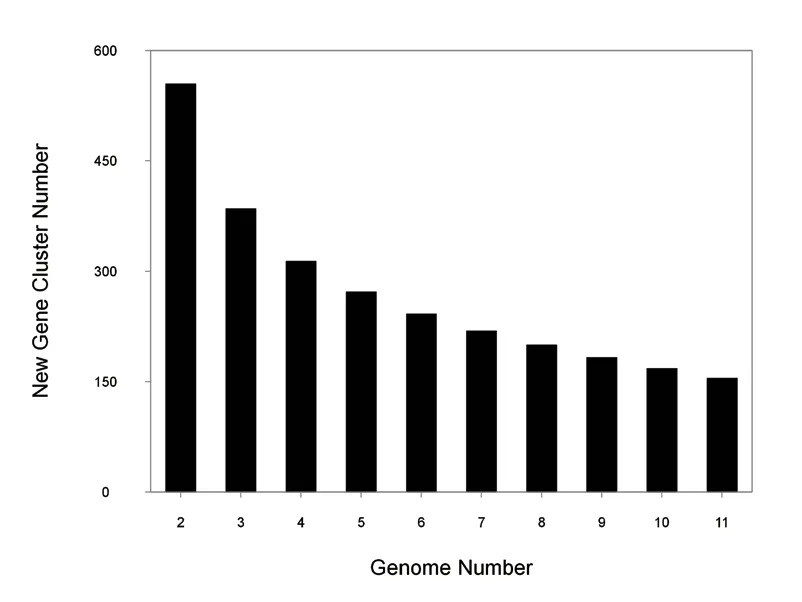
图4 凝结芽孢杆菌新基因数量与基因组的关系
对于上述预测的基因簇中,如图6和表3所示,共有43个基因簇和与已知基因簇具有一定的同源性。在预测到的43个基因簇中,有11个预测基因簇与Amylocyclicin基因簇同源相似度达到66%,另外还有1个预测基因簇与Amylocyclicin基因簇的同源相似度为50%,此外,还有18个预测基因簇与Fengycin基因簇的同源相似度为40%,有3个预测基因簇与Listeriolysin S 基因簇的同源相似度为37%,剩余10个预测基因簇与已知基因簇的同源相似度均低于30%,其中基因Cluster26和Cluster76与Kanamycin基因簇的同源相似度最低为1%。预测结果表明,凝结芽孢杆菌中预测基因簇与已知基因簇可能会有不同的产物。
3 讨论
在NCBI数据库中,有33株凝结芽孢杆菌菌株具有基因组相关数据,其中有11株组装到完整基因组水平。对这其中的11株凝结芽孢杆菌基因组进行了的泛基因组分析,泛基因组中包含5 899个基因,具体是包括2 152个核心基因、2 255个特有基因和1 492个非必须基因,并且核心基因的个数占凝结芽孢杆菌泛基因总数的36.48%。通过计算泛基因组、核心基因组和基因组个数之间的公式,发现随着基因组个数的增加,凝结芽孢杆菌的泛基因总数为上升的趋势,说明凝结芽孢杆菌的遗传物质具有开放性,同时也说明凝结芽孢杆菌具有相对较高的遗传多样性。
通过对33株凝结芽孢杆菌的次级代谢产物合成基因簇分析,共注释到8类、79个次级代谢基因簇,平均每株凝结芽孢杆菌有2-3个次级代谢基因簇,其中重复出现最多的代谢通路是细菌素、T3PKS、硫肽、糖类和内酯类化合物合成。此外,共有43个基因簇与已知基因簇具有一定同源性,其中有11个预测基因簇与Amylocyclicin基因簇同源相似度最高。这些结果表明,凝结芽孢杆菌可能具有相似的代谢产物合成途径,最有可能的活性物质包 括 有Fengycin、Amylocyclicin、Rhizocticin A和exopolysaccharide。孙天拥[28]对635株细菌的基因组进行了挖掘,共注释出有40种、6 174个次级代谢基因簇,平均每株细菌有9-10个次级代谢基因簇。Jeske等[29]对13株浮霉状菌的基因组进行了挖掘,共挖掘到102个次级代谢基因簇,平均每个基因组有7-8个次级代谢基因簇。凝结芽孢杆菌与这些细菌相比,发现的次级代谢基因簇数量较少,有新型物质合成的可能性较低。
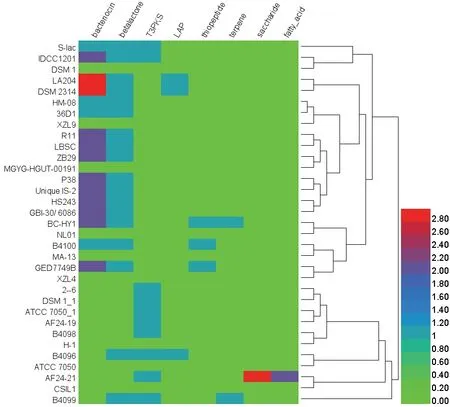
图5 33株凝结芽孢杆菌中预测的次级代谢产物类型热图
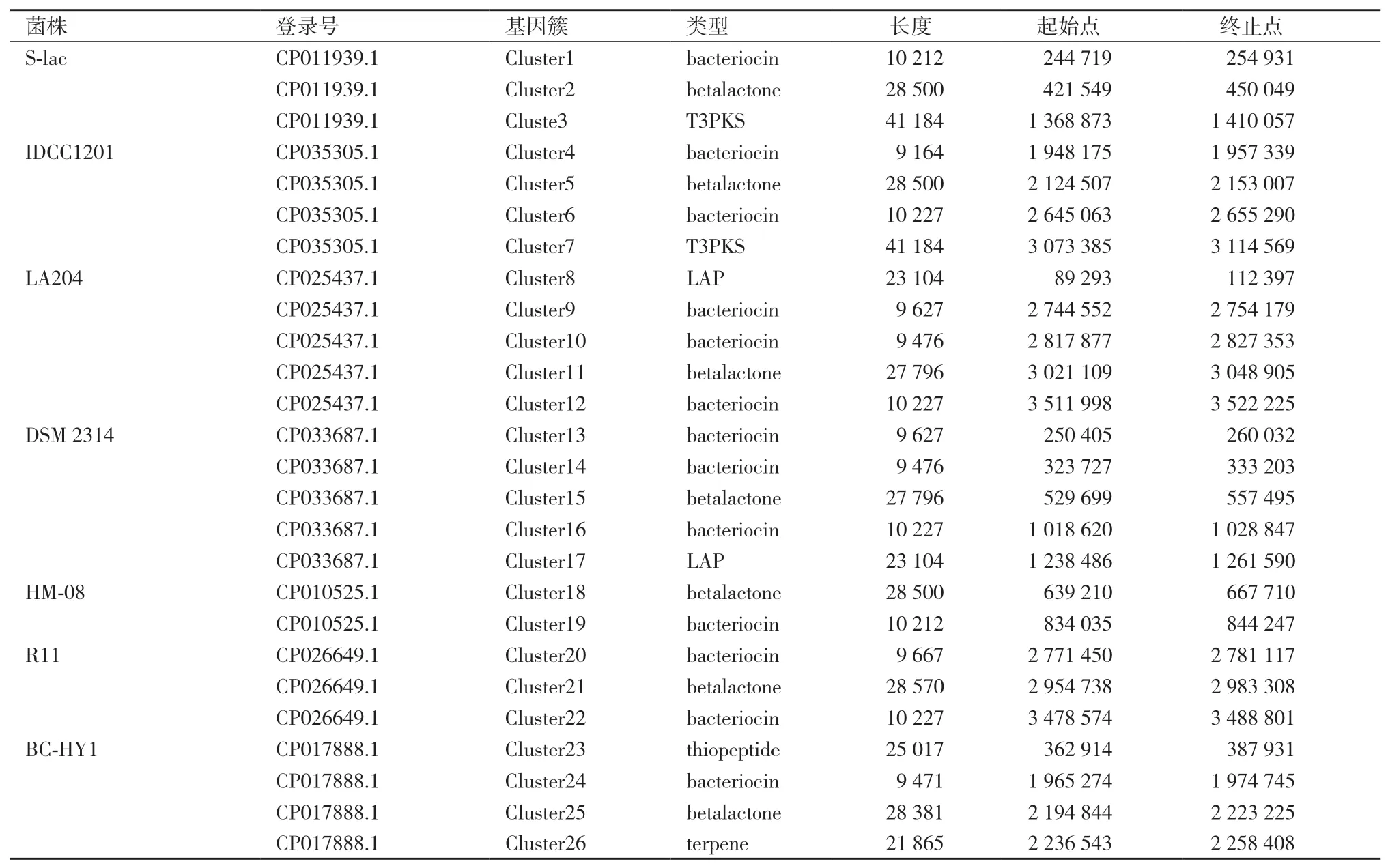
表2 凝结芽孢杆菌中预测存在的次级代谢基因簇
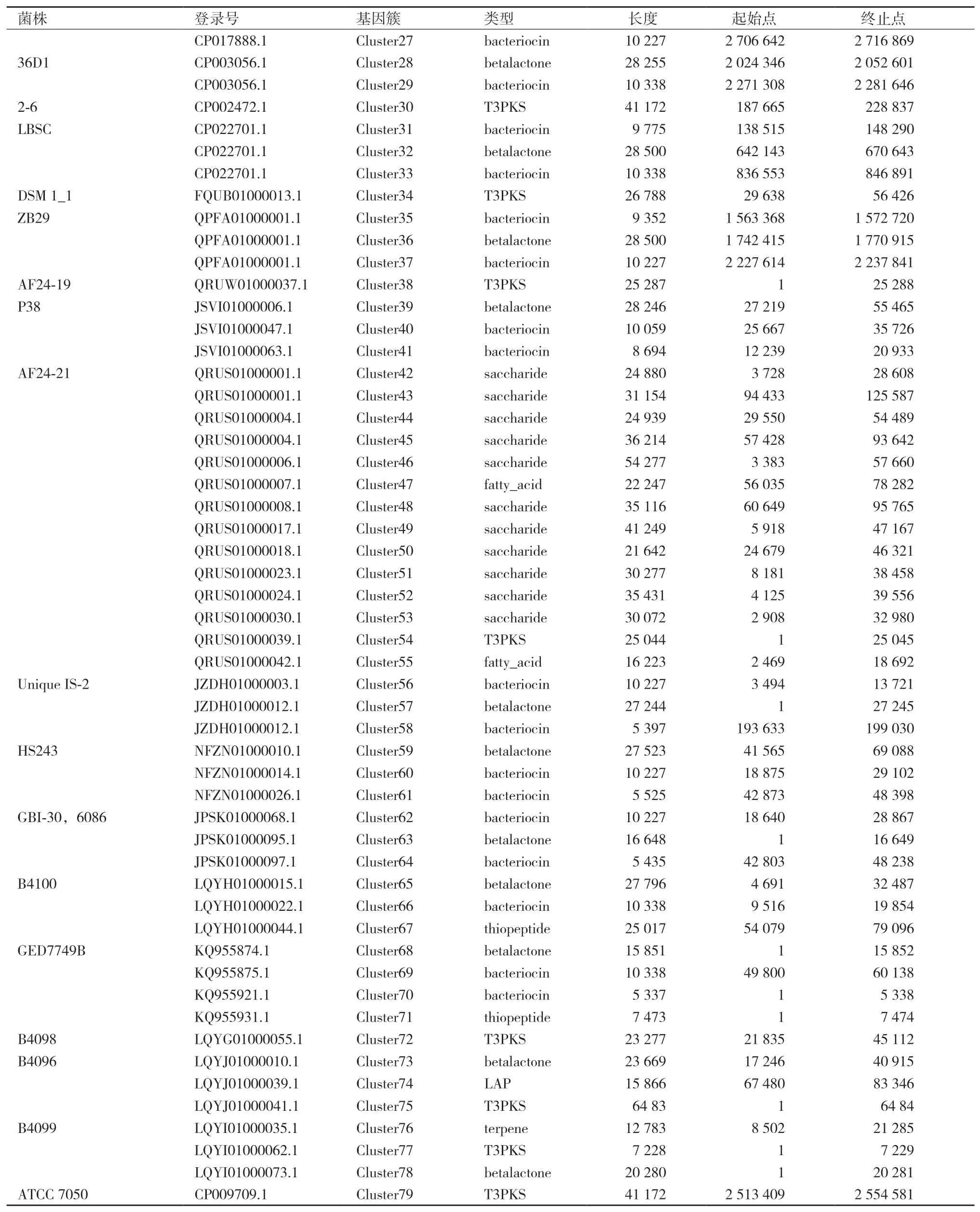
表2 续表
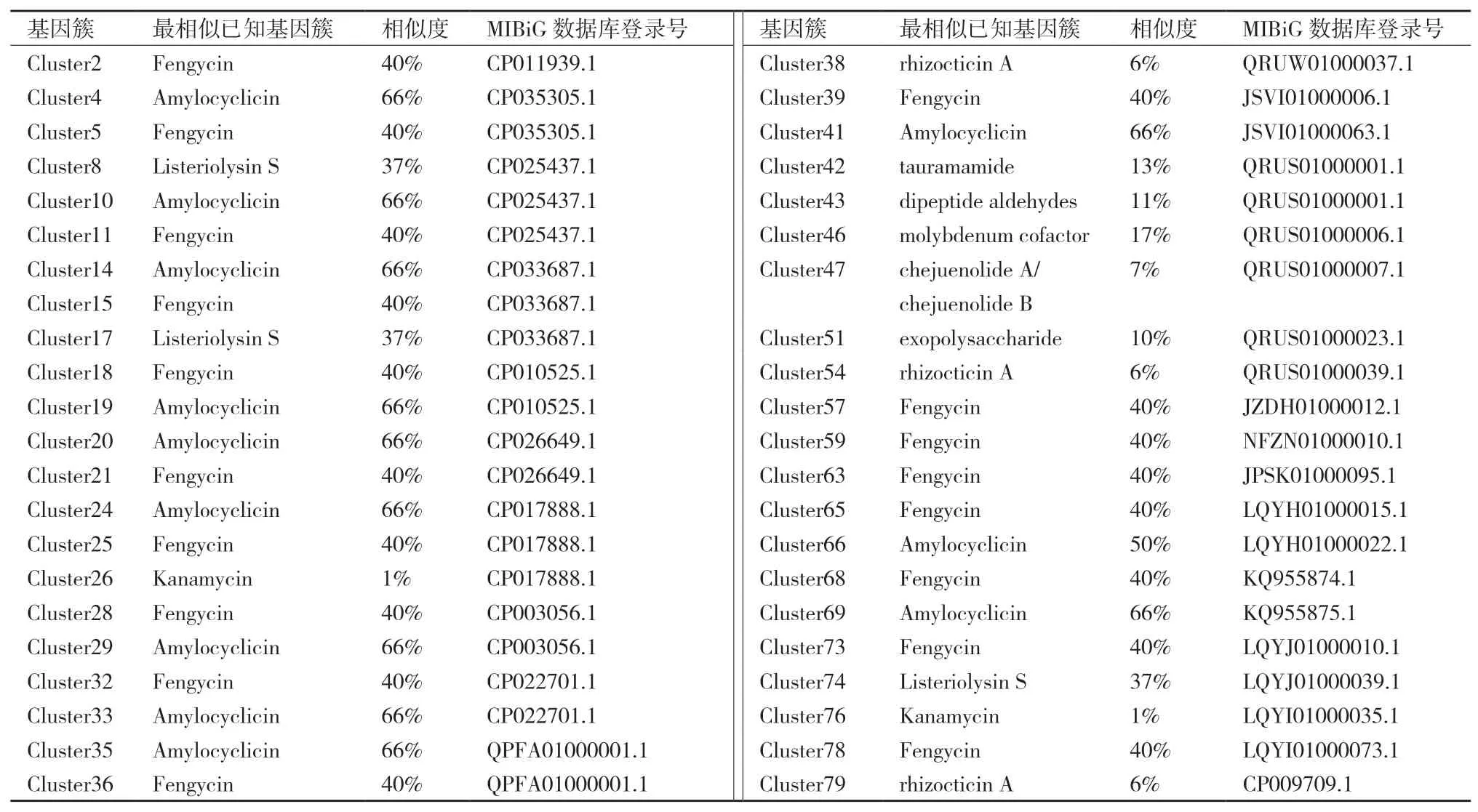
表3 凝结芽孢杆菌中预测基因簇与已知基因簇的相似度
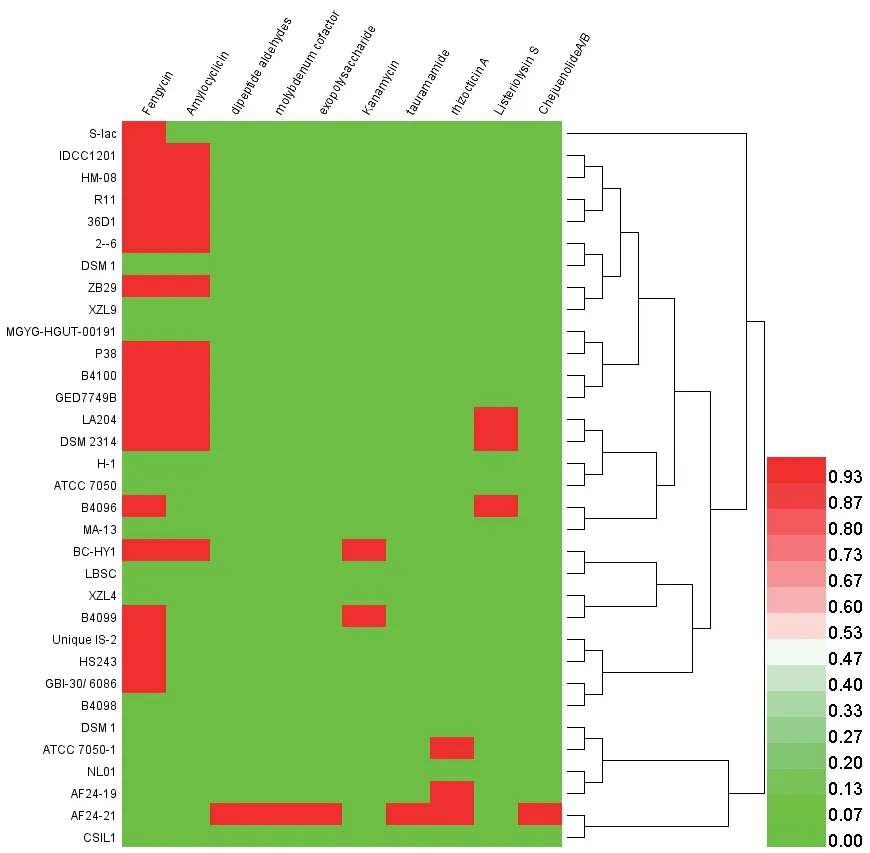
图6 33株凝结芽孢杆菌中已知基因簇类型热图
4 结论
本研究对33株凝结芽孢杆菌中的11株具有完整基因组的凝结芽孢杆菌进行泛基因组分析,检索到其泛基因组含有5 899个基因,其中特有基因有2 255个,核心基因组有2 152个基因;此外,通过对33株凝结芽孢杆菌使用antiSMASH软件进行了次级代谢基因簇挖掘,共注释到8类、79个次级代谢基因簇,其中主要的次级代谢产物合成基因簇是关于内酯、细菌素和糖类化合物。

