基于GEO数据库分析类风湿关节炎及骨关节炎的关键基因
王辰飞 姚雪婷 邵同聚 刘波
【摘 要】目的:探讨类风湿关节炎及骨关节炎中的共同关键基因及其功能意义。方法:通过GEO数据库分析类风湿关节炎及骨关节炎的差异基因,并使用DAVID数据库对差异基因进行通路富集分析和功能注释,通过STRING数据库构建差异基因的蛋白-蛋白相互作用(PPI)网络,并下载结果进一步分析。结果:GEO数据库分析表达谱数据集GSE55235和GSE55457共得到112个差异基因,DAVID数据库富集分析表明,这些差异基因主要富集在免疫应答、整合在质膜、DNA序列特异性结合以及IgA生成的肠道免疫网络。STRING数据库分析显示,共有72个基因存在于PPI网络中,而JUN、MYC、VEGFA、ATF3、CD19、EGR1、CXCL10、TLR7、TLR8和NR4A1为网络中的关键基因。结论:JUN、MYC、VEGFA、ATF3、CD19、EGR1、CXCL10、TLR7、TLR8和NR4A1在类风湿关节炎及骨关节炎中表达失调,有望成为类风湿关节炎及骨关节炎的生物标志物及治疗靶点。
【关键词】 关节炎,类风湿;骨关节炎;GEO数据库;DAVID数据库;STRING数据库
【ABSTRACT】Objective:To investigate the common key genes in rheumatoid arthritis and osteoarthritis and their functional significance.Methods:The GEO database was used to analyze the differential genes of rheumatoid arthritis and osteoarthritis,and DAVID database was used to make pathway enrichment analysis and function annotation of the differential genes.The protein-protein interaction network of the differential genes was constructed by the STRING database,and the results were downloaded for further analysis.Results:A total of 112 deferentially expressed genes were obtained from the expression profile data sets GSE55235 and GSE55457 from GEO database analysis.The enrichment analysis of DAVID database showed that these differential genes were mainly concentrated in immune response,integrated in plasma membrane,and were in the DNA sequence specific binding and intestinal immune network generated by IgA.The STRING database analysis showed that there were 72 genes in the protein-protein interaction network,and JUN,MYC,VEGFA,ATF3,CD19,EGR1,CXCL10,TLR7,TLR8 and NR4A1 were the key genes in the network.Conclusion:The expressions of JUN,MYC,VEGFA,ATF3,CD19,EGR1,CXCL10,TLR7,TLR8 and NR4A1 were dysregulated in rheumatoid arthritis and osteoarthritis,which may become their biomarkers and therapeutic targets.
【Keywords】 arthritis,rheumatoid;osteoarthritis;GEO database;DAVID database;STRING database
類风湿关节炎(rheumatoid arthritis,RA)是一种以关节病变为主的慢性自身免疫性疾病,主要特征为持续性滑膜炎、全身性炎症和自身抗体(特别是类风湿因子和瓜氨酸化肽)的产生。RA的病因约50%与遗传因素有关,病情控制不理想会导致关节损伤、残疾、生活质量下降、心血管疾病和其他并发症[1-2]。目前RA仍缺乏高效的治疗方法,因此探索RA的生物标志物及治疗靶点将更为迫切。
骨关节炎(osteoarthritis,OA)是目前最常见的慢性疾病之一,患病率和发病率逐年上升。OA发病原因可能与遗传、生物学和生物力学等因素有关,也是影响人类生活质量的一大困扰[3-4]。OA的治疗方法主要包括药物、物理和手术治疗等,但效果仍较差。因此,研究与骨关节发病及进展相关的生物标志物,将为其治疗方向提供新思路。
先前研究显示,RA和OA可能存在着共同的发病机制且受共同的因子调节[5-6],另外,某些药物(如他汀类)对它们的治疗也存在共同益处[7]。因此,本研究拟通过基因表达数据库(GEO数据库)提取RA及OA基因芯片数据进行差异基因表达谱分析,找出与这两种疾病相关的共同差异基因,并对筛选得到的差异基因进行功能注释,为研究RA及OA的共同发病机制及治疗靶点提供一定的参考依据。
1 材料与方法
1.1 GEO数据库 GEO数据库由美国国立生物技术信息中心(NCBI)创建并维护,该数据库收录了多种疾病的多组学原始数据可供用户下载及研究。本研究通过GEO数据库搜索获取RA及OA患者基因表达谱数据集GSE55235和GSE55457。GSE55235和GSE55457均包含了30例患者的基因表达谱数据集,其中10例为正常滑膜对照组织,10例为RA滑膜组织,10例为OA滑膜组织。采用R语言软件(https://www.r-project.org/)进行基因芯片质量分析和筛选差异基因。差异基因筛选采用R语言中的limma包对得到的基因表达谱矩阵文件进行差异基因筛选,以满足log2FC > 1或log2FC < -1,P < 0.05为候选差异基因。
1.2 DAVID数据库 DAVID数据库(https://david.ncifcrf.gov/)是一个生物信息数据库,其整合了多种生物学数据和分析工具,为大量的基因或蛋白列表提供系统的生物功能注释信息,帮助用户从中提取生物学信息。本研究通过DAVID数据库对筛选得到的差异基因进行基因本体(GO)、京都基因与基因组百科全书(KEGG)富集分析和功能注释,下载结果数据进行进一步整理分析。
1.3 STRING数据库 STRING数据库(https://string-db.org/)是一个在线搜索已知的蛋白-蛋白相互作用(PPI)关系的数据库,提供了超过2000个物种的9万多种蛋白的互作信息。本研究通过该数据库对筛选得到的差异基因进行构建PPI网络[8]。
1.4 Cytoscape软件 使用Cytoscape软件对差异基因的PPI网络进行模块分析和筛选关键基因。
2 结 果
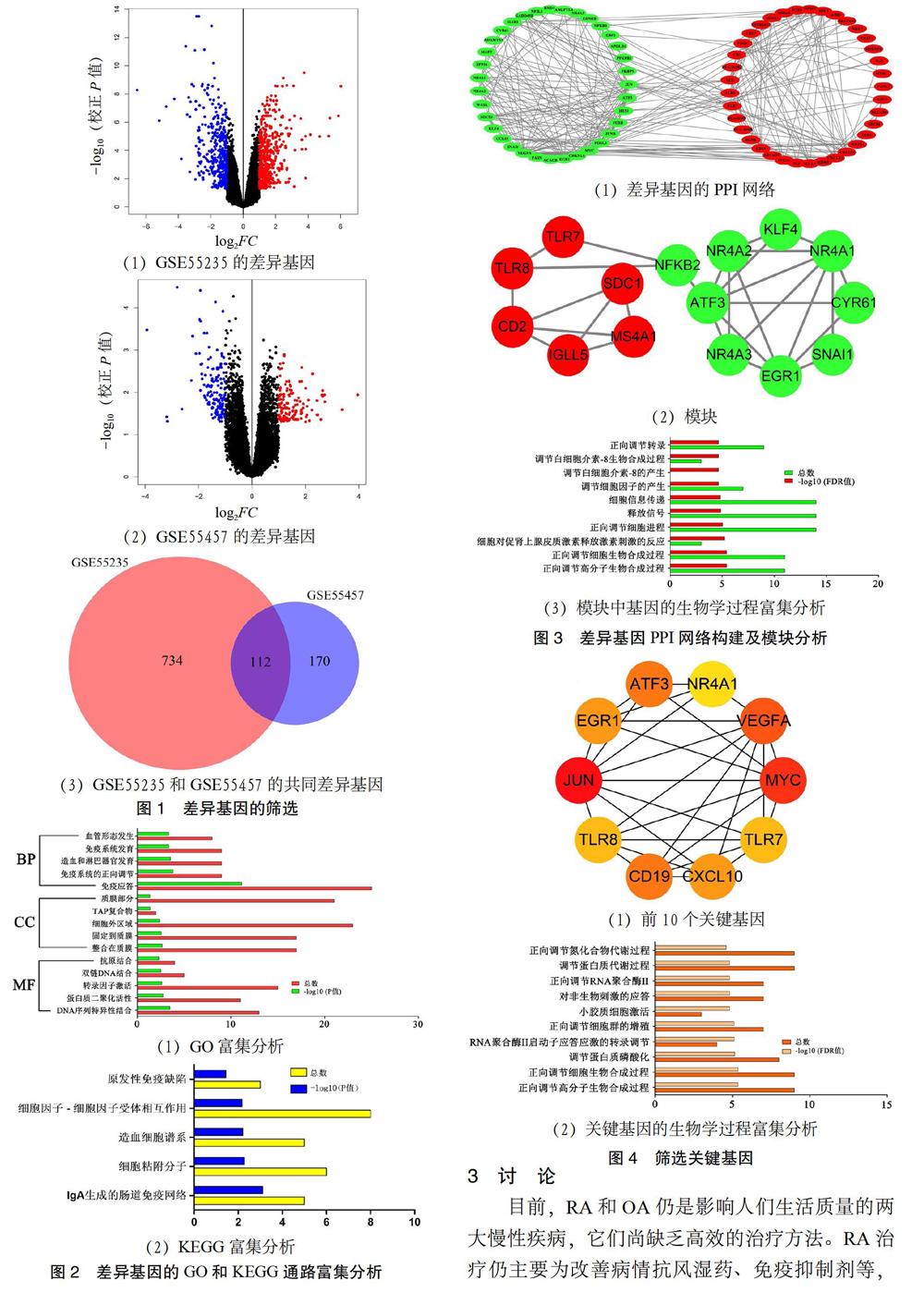
2.1 RA与OA差异基因的筛选 使用R语言对基因表达谱数据集GSE55235[9]和GSE55457[9]进行标准化处理后再进一步分析差异基因。其中GSE55235和GSE55457数据集的平台文件均为GPL96([HG-U133A]Affymetrix Human Genome U133A Array)。分析GSE55235数据集共筛选出846个差异基因,其中上调基因462个,下调基因384个,见图1(1)。分析GSE55457数据集共筛选出282个差异基因,其中上调基因133个,下调基因149个,见图1(2)。最后对这两个数据集筛选出的差异基因取交集,共得到112个差异基因,见图1(3)。
2.2 差异基因的富集分析及功能注释 使用DAVID数据库对筛选出来的112个差异基因进行GO和KEGG富集分析,结果显示,这些差异基因在生物学过程(BP)中主要集中在免疫应答,细胞组分(CC)中主要集中在整合在质膜,分子功能(MF)中主要集中在DNA序列特异性结合,KEGG通路分析主要集中在IgA生成的肠道免疫网络,见图2。
2.3 差异基因PPI网络构建及模块分析 为了进一步明确这些差异基因之间的相互作用关系,使用STRING数据库在线工具对这些差异基因进行PPI网络构建及模块分析。Cytoscape软件分析结果显示,PPI网络共有72个节点和266条边,见图3(1)。PPI网络可以分为4个模块,笔者选择最重要的模块进行生物学过程分析,见图3(2)。结果表明,模块中的基因主要富集在正向调节高分子生物合成过程,见图3(3)。
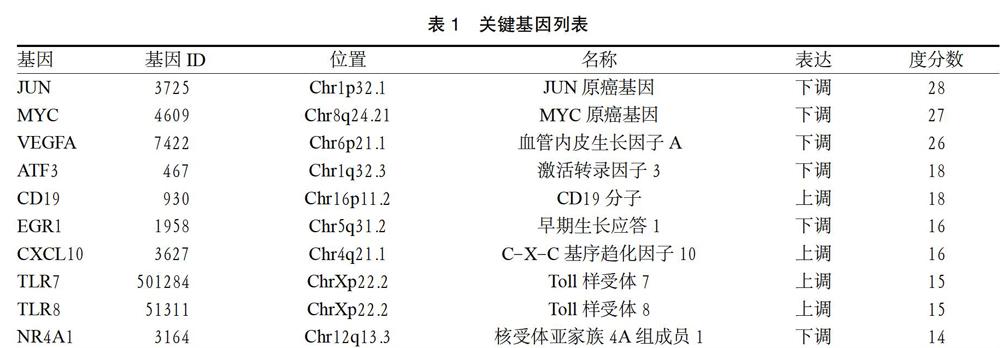
2.4 筛选关键基因 使用Cytoscape软件分析差异基因的PPI网络中的关键基因,根据度数算法选择前10个关键基因(JUN、MYC、VEGFA、ATF3、CD19、EGR1、CXCL10、TLR7、TLR8和NR4A1),見图4、表1,对这10个关键基因进行生物学过程功能注释,结果表明,这些基因主要集中在正向调节高分子生物合成过程。
3 讨 论
目前,RA和OA仍是影响人们生活质量的两大慢性疾病,它们尚缺乏高效的治疗方法。RA治疗仍主要为改善病情抗风湿药、免疫抑制剂等,而OA也主要以药物治疗及手术治疗为主。本研究通过利用生物信息学技术分析GSE55235和GSE55457数据集辨别RA和OA中的共同差异表达基因,结果发现,112个基因在这两种疾病中存在共同差异表达(log2FC > 1,P < 0.05);进一步对这些差异基因进行功能注释和KEGG通路富集分析,结果表明,这些差异基因主要集中在免疫应答(生物学过程)、整合在质膜(细胞组分)、DNA序列特异性结合(分子功能)和IgA生成的肠道免疫网络(KEGG通路分析)。这些结果表明,差异基因可能通过这些功能及通路影响RA及OA的进展。研究表明,免疫应答在RA中具有重要作用,间充质干细胞可以通过调节免疫应答调控RA的进展[10],另外,多巴胺也可以通过调控免疫应答抑制RA的进展[11]。免疫应答也在OA的发生进展中具有重要作用[12],HARRELL等[13]报道间充质干细胞可以通过抑制一些免疫细胞的免疫应答来延缓和控制OA病情的进展。IgA生成的肠道免疫网络信号通路也是一条重要的通路,它在多种疾病中发挥着重要的作用。在肝癌中,IgA生成的肠道免疫网络信号通路的激活可以促进肝癌细胞的增殖和迁移[14]。在RA及OA中,IgA生成的肠道免疫网络信号通路可能是RA及OA病情进展的共同信号通路[15-17]。
PPI网络分析结果表明,在112个差异基因中,有72个差异基因的蛋白质具有相互作用关系,进一步对PPI网络进行分析显示,该网络可分为4个功能模块,笔者对最重要的功能模块进行了生物学过程功能分析,结果显示,模块中的差异基因主要富集在正向调节高分子生物合成过程。分析网络中的关键基因结果显示,JUN、MYC、VEGFA、ATF3、CD19、EGR1、CXCL10、TLR7、TLR8和NR4A1为PPI网络中前10个关键基因。对这10个关键基因进行生物学过程功能注释显示,它们主要集中在正向调节高分子生物合成过程。先前的研究亦表明这10个关键基因均为RA与OA的共同差异基因,并参与RA与OA的病程进展[18-19]。如VEGFA,研究显示其核苷酸的多态性是人患RA的一个危险因素[20-21],并且受一些miRNA调节来促进RA的进展[22]。同样,在OA病程进展中VEGFA也发挥着重要作用,其受一些miRNA(miR-15a-5p)调节,miR-15a-5p的高表达可抑制VEGFA的表达并延缓OA的进展,而其抑制表达可导致VEGFA的表达升高并促进OA的进展[23]。
CXCL10也是調节RA及OA病程进展的关键基因。在OA中C5aR1和TLR2可以通过激活p38-MAPK信号通路并增加CXCL10的表达来促进OA的进展[24]。而在RA中,TXNDC5的高表达可抑制CXCL10的表达并抑制RA进展,沉默TXNDC5可导致CXCL10表达显著上调并促进RA进展[25]。
这些结果进一步揭示了RA和OA疾病发展的共同分子机制,将为研究治疗RA和OA的靶点提供一个初步理论依据。然而,本研究仍有一些不足之处,仍需要结合大样本并进行相关的体内外及分子机制实验进一步验证,才能完全明确这些差异基因在RA及OA中的具体作用机制。
参考文献
[1] SCOTT DL,WOLFE F,HUIZINGA TWJ.Rheumatoid arthritis[J].Lancet,2010,376(9746):1094-1108.
[2] KATZ P.Causes and consequences of fatigue in rheumatoid arthritis[J].Curr Opin Rheumatol,2017,29(3):269-276.
[3] GLYN-JONES S,PALMER AJR,AGRICOLA R,et al.Osteoarthritis[J].Lancet,2015,386(9991):376-387.
[4] PEREIRA D,RAMOS E,BRANCO J.Osteoarthritis[J].Acta Med Port,2015,28(1):99-106.
[5] POPP J,SCHICHT M,GARREIS F,et al.Human Synovia Contains Trefoil Factor Family(TFF)Peptides 1-3 Although Synovial Membrane Only Produces TFF3:Implications in Osteoarthritis and Rheumatoid Arthritis[J].Int J Mol Sci,2019,20(23):6105.
[6] HUANG W,ZHANG LL,CHENG C,et al.Parallel comparison of fibroblast-like synoviocytes from the surgically removed hyperplastic synovial tissues of rheumatoid arthritis and osteoarthritis patients[J].BMC Musculoskelet Disord,2019,20(1):591.
[7] SARMANOVA A,DOHERTY M,KUO C,et al.Statin use and risk of joint replacement due to osteoarthritis and rheumatoid arthritis:a propensity-score matched longitudinal cohort study[J].Rheumatology,2020,59(10):2898-2907.
[8] VON MERING C,HUYNEN M,JAEGGI D,et al.STRING:a database of predicted functional associations between proteins[J].Nucleic Acids Res,2003,31(1):258-261.
[9] WOETZEL D,HUBER R,KUPFER P,et al.Identification of rheumatoid arthritis and osteoarthritis patients by transcriptome-based rule set generation[J].Arthritis Res Ther,2014,16(2):R84.
[10] LUQUE-CAMPOS N,CONTRERAS-L?PEZ RA,JOSE PAREDES-MART?NEZ M,et al.Mesenchymal Stem Cells Improve Rheumatoid Arthritis Progression by Controlling Memory T Cell Response[J].Front Immunol,2019,10(1):798.
[11] CAPELLINO S.Dopaminergic Agents in Rheumatoid Arthritis[J].J Neuroimmune Pharmacol,2019,15(1):48-56.
[12] KALAITZOGLOU E,GRIFFIN TM,HUMPHREY MB.Innate Immune Responses and Osteoarthritis[J].Curr Rheumatol Rep,2017,19(8):45.
[13] HARRELL CR,MARKOVIC BS,FELLABAUM C,et al.Mesenchymal stem cell-based therapy of osteoarthritis:Current knowledge and future perspectives[J].Biomed Pharmacother,2019,109(1):2318-2326.
[14] YANG Z,TAO Y,XU X,et al.Bufalin inhibits cell proliferation and migration of hepatocellular carcinoma cells via APOBEC3F induced intestinal immune network for IgA production signaling pathway[J].Biochem Biophys Res Commun,2018,503(3):2124-2131.
[15] XUE F,ZHANG C,HE Z,et al.Analysis of critical molecules and signaling pathways in osteoarthritis and rheumatoid arthritis[J].Mol Med Rep,2013,7(2):603-607.
[16] LIU GY,JIANG YS,CHEN XG,et al.Measles contributes to rheumatoid arthritis:evidence from pathway and network analyses of genome-wide association studies[J].PLoS One,2013,8(10):e75951.
[17] LIN J,WU GW,ZHAO ZS,et al.Bioinformatics analysis to identify key genes and pathways influencing synovial inflammation in osteoarthritis[J].Mol Med Rep,2018,18(6):5594-5602.
[18] LI WC,BAI DL,XU Y,et al.Identification of differentially expressed genes in synovial tissue of rheumatoid arthritis and osteoarthritis in patients[J].J Cell Biochem,2019,120(3):4533-4544.
[19] CAI PA,JIANG TM,LI B,et al.Comparison of rheumatoid arthritis(RA)and osteoarthritis(OA)based on microarray profiles of human joint fibroblast-like synoviocytes[J].Cell Biochem Funct,2019,37(1):31-41.
[20] RAM?REZ-BELLO J,CADENA-SANDOVAL D,FRAGOSO JM,et al.The VEGFA-1154G/A polymorphism is associated with reduced risk of rheumatoid arthritis but not with systemic lupus erythematosus in Mexican women[J].J Gene Med,2018,20(6):e3024.
[21] PARADOWSKA-GORYCKA A,PAWLIK A,ROMANOWSKA-PROCHNICKA K,et al.Relationship between VEGF Gene Polymorphisms and Serum VEGF Protein Levels in Patients with Rheumatoid Arthritis[J].PLoS One,2016,11(8):e0160769.
[22] YANG SZ,YANG YG.Downregulation of microRNA-221 decreases migration and invasion in fibroblast?like synoviocytes in rheumatoid arthritis[J].Mol Med Rep,2015,12(2):2395-2401.
[23] CHEN HW,TIAN Y.MiR-15a-5p regulates viability and matrix degradation of human osteoarthritis chondrocytes via targeting VEGFA[J].Biosci Trends,2017,10(6):482-488.
[24] M?DINGER Y,RAPP A,PAZMANDI J,et al.C5aR1 interacts with TLR2 in osteoblasts and stimulates the osteoclast-inducing chemokine CXCL10[J].J Cell Mol Med,2018,22(12):6002-6014.
[25] XU B,LI J,WU CS,et al.CXCL10 and TRAIL Are Upregulated by TXNDC5 in Rheumatoid Arthritis Fibroblast-like Synoviocytes[J].J Rheumatol,2018,45(3):335-340.
收稿日期:2019-12-19;修回日期 :2020-05-04