基于TCGA 数据库确定宫颈癌预后免疫相关性长链非编码RNA 并构建预后模型
底斐瑶,王一鹤,底泽亚,袁瑞(通讯作者)
(1.重庆医科大学附属第一医院妇产科,重庆;2.郑州大学第一附属医院泌尿外科,河南 郑州;3.北京大学信息科学技术学院,北京)
0 引言
宫颈癌是女性最常见的恶性肿瘤之一,发病率和死亡率均居女性常见恶性肿瘤的第四位;在世界范围内,据统计,每年大约有53 万新发病例,27 万死亡病例[1-3]。其病理类型最常见的是鳞状细胞癌(squamous cell carcinoma,SCC),约占所有宫颈癌的70%[3,4],其他类型的宫颈癌相对少见。早期宫颈癌的标准治疗方法为手术治疗,晚期宫颈癌主要是放射治疗和化学治疗,由于耐药性和复发性,治疗效果有限[5,6],有文献报道晚期宫颈癌五年生存率仅为16.8%[7],严重危害妇女身心健康。
近几年,免疫治疗逐渐兴起。癌症免疫治疗是通过增强或触发患者的免疫系统诱发机体抗肿瘤的治疗总称[8]。越来越多的研究表明免疫疗法在癌症治疗中的可行性[9-11],这种治疗方法为癌症患者带来了希望。特别是程序性死亡-1(PD-1)/(PD-L1)抑制剂在包括宫颈癌在内的多种实体肿瘤中取得了疗效,已获得美国食品和药物管理局(FDA)的批准[8]。
长链非编码RNA(long non-conding RNA,LncRNA)是转录长度大于200 个核苷酸的非编码RNA,不具备编码蛋白的能力[6],但是LncRNA 的过表达、缺失或者突变等对肿瘤的恶性生物学行为有驱动作用[12,13]。例如,LncRNA-H19、LncRNAXIST、LncRNA-HOTAIR、LncRNA-NCK1-AS1、LncRNA-IGF2等的异常表达已被证明与宫颈癌的发生、发展及预后相关[14-21]。癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库是美国国家癌症研究所和美国国家人类基因组研究组于2006共同发起建立的癌症数据库,该数据库收集了常见肿瘤的临床数据、长链非编码RNA、差异表达基因、拷贝数变异、甲基化等,是重要的权威癌症数据库之一[22]。TCGA 数据病理诊断明确、来源可靠、免费、公开,目前已被癌症研究者广泛使用。本研究基于TCGA 数据库寻找与宫颈癌预后相关的免疫相关性LncRNA,并将确定的免疫相关性LncRNA 构建宫颈癌预后模型,为宫颈癌的预后提供新的研究思路。
1 资料与方法
1.1 研究方法
从TCGA 数据库中下载宫颈癌转录数据及临床数据,从Molecular Signatures Database 下载免疫相关基因,利用Strawberry Perl 软件(版本 5.30.1,64-bit)对数据进行分析与合并,通过R 软件对数据进行处理和分析。首先通过构建免疫基因-LncRNA 共表达网络,筛选出免疫相关性LncRNA。通过单因素COX 回归筛选具有预后价值的LncRNA。通过多因素COX 回归分析构建预后模型,根据赤池信息量准则(Akaike information criterion,AIC)值确定最佳模型。根据风险评分的中位数将患者分为高风险组和低风险组,运用R 软件对两组患者进行生存分析。我们使用受试者接受特征(receiver operating characteristic,ROC)曲线对模型进行评价,并与T 等级、N 等级、Grade 等级、年龄等临床特征进行比较。最后将得到的关键LncRNA 与宫颈癌临床特征进行相关性分析。
1.2 数据收集及处理
2020年5 月,从癌症基因组图谱(TCGA)数据库(https://tcga-data.nci.nih.gov/tcga/)CSCC 项目下载宫颈癌患者的转录表达数据及临床资料。利用strawberry perl(版本 5.30.1,64-bit)脚本语言软件用于获取整合转录组LncRNA 表达数据和相应的临床资料;其中基因表达数据提取包括基因名称、样本编号、和表达值。
患者纳入标准:1)肿瘤原发于宫颈;2)病理证实为鳞状细胞癌;3)数据类型为转录组;4)数据类型:基因表达量;5)可获得患者的总体生存时间;
由于本研究制定的模型与预后相关,因此排除标准如下:
1)排除生存时间≤30 天的患者;2)总体生存时间未知或者生存状态未知的患者。
从Molecular Signatures Database 获取免疫相关基因即Immune System process 基 因 集,编 号:M13664, Immune Response 基因集,编号:M19817。
1.3 确定显著差异的免疫相关性LncRNA
获得表达数据后,在R 软件中(版本 3.6.2),使用“limma package” 和 “cor.test” 函数进行共表达分析,其中“cor.test”函数可得到LncRNA 与免疫基因的相关系数,从而确定有显著差异的免疫相关性LncRNA(设置标准:P<0.001,相关系数COR 绝对值大于0.4)。
1.4 确定与预后相关的LncRNA 并建立风险评估模型
为筛选与宫颈癌预后相关的免疫相关性LncRNA,我们在R 软件中使用“Survival package”进行单因素Cox 回归分析(P<0.01 认为有统计学差异)筛选出影响宫颈癌预后的免疫相关性LncRNA。我们根据上一步筛选出的LncRNA 在每位患者中的表达量,通过Cox 回归分析获得每个LncRNA 的风险系数,计算模型AIC 值并对其进行调整,AIC 值最高者即为最优模型,计算得到患者的风险评分Risk,计算公式如下;其中exp 代表LncRNA 的表达量。
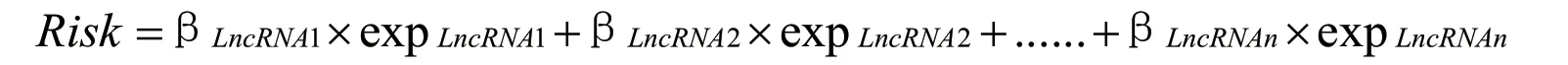
根据风险评分的中位值,Risk ≥中位值的患者为高风险组,Risk<中位值低风险组,利用R 包“Survival package”进行生存分析(P<0.05 认为有统计学差异)。根据每位患者的风险值利用R 软件绘制风险状态图,风险热图。风险热图使用的R 包“Pheatmap”。为了验证模型,我们在R 软件中使用ROC 工作曲线评估每个危险因素的价值。
1.5 LncRNA 与临床的关系
为了确定LncRNA 与临床之间的关系,我们将从TCGA中下载的临床数据进行筛选,剔除临床情况未知的患者。在R 软件中,使用“ggpubr package”评估LncRNA 表达量与T分期,M 分期,N 分期和Grade 分级的相关性,并将其可视化(P<0.05 认为有统计学差异)。
2 结果
2.1 创建免疫基因-LncRNA 共表达网络,筛选免疫相关性LncRNA
首先我们从TCGA 数据库中获取255 例宫颈癌样本,通过构建免疫-LncRNA 共表达网络来筛选有显著差异的免疫相关性LncRNA,最终筛选出637 个LncRNA。
2.2 确定与宫颈癌预后相关的免疫相关性LncRNA
我们利用R 包 “Survival Package”将上一步鉴定的LncRNA 与TCGA 数据库中下载的255 例宫颈癌患者的生存时间及生存状态进行单因素COX 回归分析(P<0.01 认为有统计学差异)筛选出34 个LncRNA(图1)。
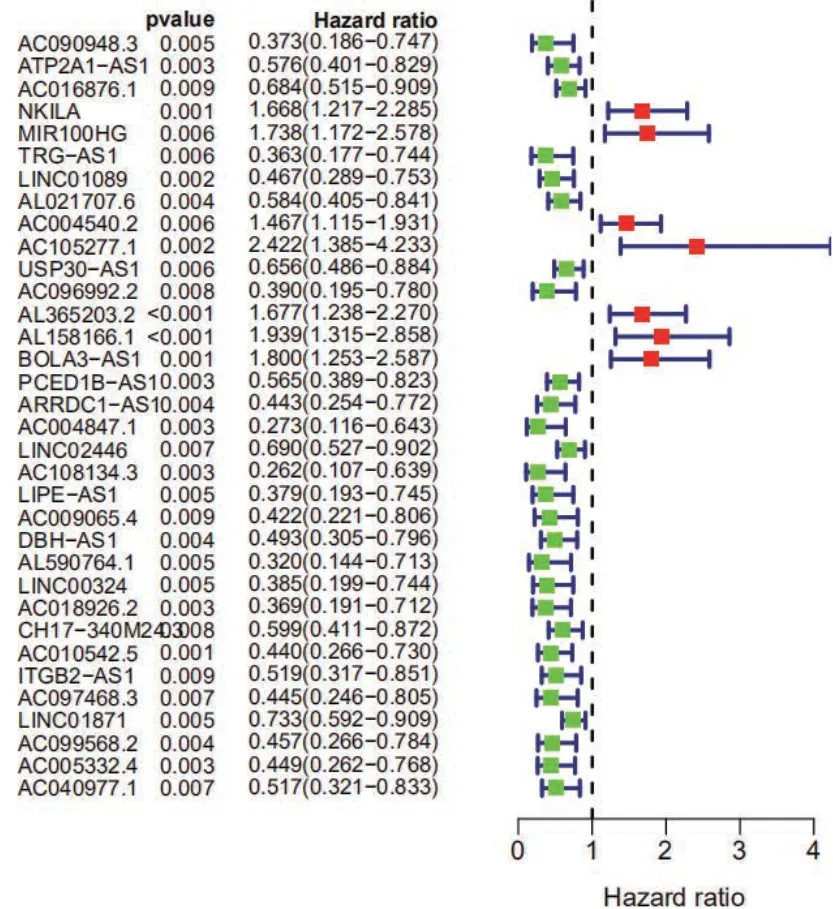
图1 单因素COX 回归筛选出和宫颈癌预后相关的LncRNA
2.3 构建宫颈癌预后有关的模型并进行生存分析和绘制风险曲线图
基于多因素COX 回归和AIC 值筛选出构成最优模型的8 个关键的免疫相关性LncRNA(表1),根据风险评分模型,计算每个患者的预后风险评分Risk,并根据中位数将患者分为高风险组和低风险组以构建预后模型。为了进一步验证模型的预测能力,将高风险组和低风险组进行生存分析显示,高风险组较低风险组生存率有差异(P<0.05 认为有统计学意义)(图2)。根据每位患者的风险值利用R 软件绘制风险状态图显示:随着风险值增加,死亡人数逐渐增加(图3A);风险热图显示:AC004540.2、AL365203.2 表达量随着风险值增加逐渐增加;相反ATP2A1-AS1、AC096992.2、AC004847.1、AC097468.3、AC099568.2 表达量随着风险值增加逐渐降低。LncRNA;AC105277.1 表达量随着风险值增高变化不明显(图3B)。

表1 多因素COX 回归筛选出用于构建模型的免疫相关性lncRNA

图2 将高风险组和低风险组进行K-M 生存分析

图3 A:风险状态图
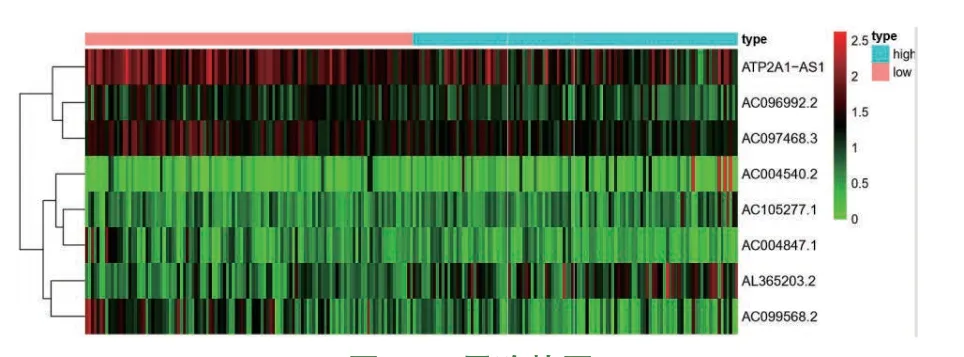
图3 B:风险热图
2.4 利用ROC 曲线评价模型
为了进一步评估模型的准确性,将风险值及临床性状共同绘制ROC 曲线(图4)显示:风险评分模型AUC 值(ROC曲线下面积)为0.758,较其他临床特征作为预后因素准确性高。

图4 ROC 曲线评价宫颈癌预后模型
2.5 确定LncRNA 与临床的关系
我们将从TCGA 中下载的临床数据进行筛选,剔除临床情况未知的患者。在R 软件中,评估LncRNA 表达量与T 分期的相关性,并将其可视化。结果显示AC099568.2、AC105277.1 与T 分期具有相关性(P<0.05 认为有统计学差异),其表达量随着T 分期增加逐渐降低(图5)。
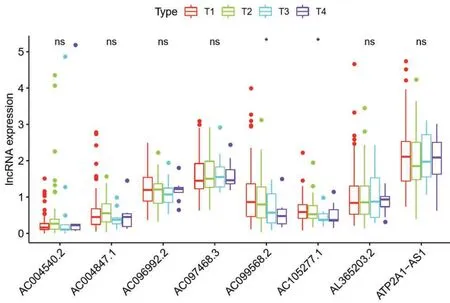
图5 将8 个LncRNA 与宫颈癌临床T 分期进行临床相关性分析,*代表P<0.05,ns 代表P>0.05
3 讨论
晚期宫颈癌患者一般采取传统的放射治疗和化学治疗,由于耐药性和易复发性,治疗效果欠佳[23,24],文献报道宫颈癌患者的五年生存率仍低[7,23],这个问题亟需解决。随着免疫治疗在多种实体肿瘤的兴起,宫颈癌的免疫治疗也成为当前研究热点。越来越多的研究显示LncRNA 的异常如突变、缺失或过表达等对肿瘤的发生、发展、转移、复发等有驱动作用[12,25-28],这为宫颈癌的研究提供了新的思路。夏艳[29]等研究显示LncRNA TUG1 高表达与宫颈癌预后不良密切相关。Huang[30]等发现LncRNA PANDAR 在宫颈癌组织和细胞中的表达水平上调,能促进肿瘤的生长,与宫颈癌的不良预后相关,提出PANDAR 可能是宫颈癌早期诊断的生物标志物,并且是逆转肿瘤恶性表型的潜在治疗靶标。HaoBo[31]等发现,AFAP1-AS1 在宫颈癌中表达升高且甲基化不足,并且与宫颈癌患者预后不良相关。因此,本文利用生物信息学方法从TCGA 数据库中提取宫颈癌相关信息,寻找与宫颈癌预后相关的免疫相关性LncRNA,构建预后模型,为宫颈癌治疗提供新的靶点。
我们从TCGA 数据库下载宫颈癌转录组及临床数据,从Molecular Signatures Database 下载免疫相关基因,通过免疫基因-LncRNA 共表达筛选出637 个显著差异的免疫相关性LncRNA。我们将这些LncRNA 与TCGA 数据库中下载的宫颈癌患者的生存时间及生存状态行单因素COX 回归分析初步筛选出34 个与宫颈癌预后相关的LncRNA,基于多因素COX 回归和AIC 值筛选出构成最优模型的8 个关键的LncRNA,分别为AC004540.2、AL365203.2、ATP2A1-AS1、AC096992.2、AC004847.1、AC097468.3、AC099568.2、AC105277.1。我们根据每个病人的LncRNA 表达量构建预后风险模型,根据风险中位值将病人分为高风险组及低风险组。通过绘制风险热图可见AC004540.2、AL365203.2 表达量随着风险值增加逐渐增加,考虑为危险性LncRNA;相反ATP2A1-AS1、AC096992.2、AC004847.1、AC097468.3、AC099568.2 表达量随着风险值增加逐渐降低,考虑为保护性LncRNA;AC105277.1 表达量随着风险值增高变化不明显,待临床相关性实验进一步验证。将高、低风险组进行K-M 生存分析、绘制ROC 曲线评估模型的准确性及预测能力。在K-M生存分析中,高风险组患者生存率显著低于低风险组;ROC 曲线下面积为0.758,两者结果一致,验证了预后模型的准确性,联合检测这八个LncRNA 所创建的模型能较准确地预测宫颈癌患者的三年和五年生存率,而且其有可能成为宫颈癌治疗的潜在靶点。将确定的LncRNA 与患者临床特征(T、N、M)进行相关性分析,结果显示AC099568.2、AC105277.1 与T 分期具有相关性(P<0.05 认为有统计学差异),且其表达量随着T 分期增加逐渐降低,提示其可能为宫颈癌预后的保护因素。
ZHOU[32]等人建立了舌鳞状细胞癌(SCCT)患者的生存风险评估模型,通过生存分析显示,AC105277.1 与SCCT患者整体生存率显著相关,认为该LncRNA 有可能是预测SCCT 预后的生物标志物之一。同样地,在我们的研究中也发现AC105277.1 与宫颈癌的预后显著相关,因此,我们推测其可能成为宫颈癌的潜在治疗靶点。我们的研究结果表明,AC004540.2、AL365203.2、ATP2A1-AS1、AC096992.2、AC004847.1、AC097468.3、AC099568.2 均与宫颈癌的预后密切相关,但目前尚未发现关于它们的研究。在未来的研究中,这些LncRNA 在临床上的价值仍然需要探索,我们认为它们可能作为宫颈癌预后标志物。我们计划收集自己的临床数据对本研究结果进一步验证,并从分子生物学角度对其发生机制进行研究探讨。
综上所述,我们通过TCGA 数据库挖掘相关宫颈癌相关的生物学信息,创建免疫基因-LncRNA 共表达网络,确定了8 个与宫颈癌预后相关的免疫相关性LncRNA,为宫颈癌患者提供可能的免疫治疗靶点。并基于此8 个LncRNA 构建了宫颈癌预后模型,预测准确性中等。将8 个LncRNA 与宫颈癌患者临床特征进行相关性分析,其中AC099568.2、AC105277.1 表达量随着T 分期增加逐渐降低,提示其可能为宫颈癌预后的保护因素。但它们在宫颈癌中的作用机制有待进一步研究和验证。

