辽宁省野生羊肚菌资源调查与遗传多样性分析*
王 红 ,曹 君 ,张 敏 ,张士义 ,张季军 ,刘俊杰 ,杨 镇 ,吕立涛 **
(1.辽宁省农业科学院食用菌研究所,辽宁 沈阳 110161;2.辽宁省食用菌优质栽培重点实验室,辽宁 沈阳 110161)
羊肚菌属(Morchella Dill.ex Pers.:Fr.),隶属于子囊菌门(Ascomycata) 盘菌亚门(Pezizomycotina)盘菌纲(Pezizomycetes) 盘菌目(Pezizales) 羊肚菌科(Morchellaceae)[1],因其菌盖有不规则的凹陷褶皱,状如羊肚而得名,是一种珍贵的食(药)用真菌。自羊肚菌属建立以来,科研人员针对其生态分布、分类鉴定、菌种保育及栽培技术等开展了大量的研究。羊肚菌在亚洲、欧洲、北美洲等均有分布[2-5]。目前,已记录羊肚菌种和变种的数据多达344个[6],其中中国分布有羊肚菌30种[7],以温带落叶阔叶林、高海拔暗针叶林及针阔混合林为羊肚菌的主要植被生境[8-9]。羊肚菌主要在3月~5月出菇[2],仅有少数物种在7月及9月~10月出菇[8,10]。
根据传统的形态学分类方法,羊肚菌分为黄色羊肚菌(yellow morels)、黑色羊肚菌(black morels)和半开羊肚菌(semifree capped moerls),而发育过程中,其形态特征会受到环境的影响,且物种间宏观显微特征有限,会引起同物异名和同名异物的现象[11-14]。2000年,Taylor等[15]提出了多基因系谱一致性系统发育种识别法(genealogical concordance phylogenetic species recognition,GCPSR),为羊肚菌精准分类提供了方向。2011年,O’Donnell等[16]首次利用GCPSR的方法,基于ef1-α、LSU、rpb1和rpb2的核苷酸序列,将羊肚菌分为Elata Clade(黑色羊肚菌支系)、Esculenta Clade(黄色羊肚菌支系) 和Rufobrunnea Clade(变红羊肚菌支系)。目前,国内外已有许多地区采用GCPSR对羊肚菌资源进行系统分类的报道[17-22],以从分子系统分类学角度,纠正或弥补羊肚菌分类上的错误和不足。
1987年开始,郭浩等[23]对辽宁省陆续进行实地考察,对野生羊肚菌的生态分布、分类鉴定、驯化栽培等进行了研究。而野生资源分类鉴定方面,仅根据形态特征分类[23-25],除本课题组对辽宁省康平地区羊肚菌资源[26-27]及羊肚菌多基因序列模标数据库(Morchella MLST database)[28]中涉及的辽宁省沈阳市和丹东市两个采样点采用分子生物学的方法进行分类鉴定外,未见依据分子生物学标记专门针对辽宁省野生羊肚菌进行分类鉴定的报道。
通过调查辽宁省的羊肚菌资源及其发生环境,确定其种属关系,探索其发生的适宜条件,旨在确定辽宁省拥有的羊肚菌种质资源,为辽宁省野生羊肚菌的应用提供科学依据。
1 材料与方法
1.1 材料
根据文献记载[23-25]及实地调研,辽宁省野生羊肚菌主要分布在辽宁省东部山区和辽西的大凌河流域,出菇时间在每年的4月初~5月末,通常分布在杨树林或杨树林缘草地、河滩草等地。
根据出菇特征,2016年~2018年,在辽宁省的沈阳市、抚顺市、鞍山市、朝阳市、丹东市、本溪市、阜新市7个市布点采样。3年间,共采集羊肚菌子实体53份,分布于16个羊肚菌发生地。样品采集地点以杨树林为主,分布在东部山区的羊肚菌多发生在杨树林中,树林中腐叶较多,土壤多为褐色粘质土壤;分布在大凌河流域的羊肚菌多发生在树林缘草地、河滩草中,杂草丛生,土壤为黑褐色沙质土,在树林附近有小溪或水沟。试验采集样品保存于辽宁省农业科学院食用菌研究所食用菌所,采集样本信息见表1。

表1 羊肚菌子实体样本采集信息及居群Tab.1 Collection information and populations of Morchella spp.samples collected
1.2 形态鉴定
将采集的子实体,参照《羊肚菌生物学与栽培技术》[29]按照子实体颜色、形状和脊背等特征进行形态学分类,然后采用组织分离法获得菌丝体进行显微形态鉴定。
1.3 分子鉴定
1.3.1 总DNA提取
采用E.Z.N.A.TM Fungal DNA Kit试剂盒提取菌丝体的总DNA,将提取的总DNA用1%的琼脂糖凝胶电泳检测,-20℃保存备用。
1.3.2 ITS序列扩增
建立20 μL的PCR反应体系,其中2×Easy Taq Mix 10 μL,引物(10 μmol·L-1)各1 μL,模板DNA 0.8 μL,ddH2O 补至 20 μL。
ITS-PCR扩增反应程序为95℃预变性4 min,95℃变性 30 s,57℃退火 30 s,72℃延伸 60 s,30个循环,72℃延伸7 min。引物序列[29]见表2。
PCR产物用1.0%琼脂糖凝胶电泳检测,目的条带测序。
1.3.3 多基因序列扩增
建立20 μL的PCR反应体系,其中2×Easy Taq Mix 10 μL,引物(10 μmol·L-1)各0.8 μL,模板DNA 0.8 μL,ddH2O 补至 20 μL。
多基因扩增反应程序为94℃预变性3 min,94℃变性60 s,50℃退火30 s,72℃延伸 60 s,35个循环,72℃延伸10 min。引物序列见表2。PCR产物用1.0%琼脂糖凝胶电泳检测,目的条带测序。

表2 PCR引物序列Tab.2 PCR Primer sequences
1.4 ISSR序列分析
利用1.3.1提取的总DNA,建立15 μL的ISSRPCR反应体系,其中2×Easy Taq Mix 7.5 μL,引物(10 μmol·L-1)各0.6 μL,模板DNA 0.6 μL,ddH2O补至15 μL。引物序列见表3。

表3 ISSR-PCR引物序列Tab.3 ISSR-PCR Primer
ISSR-PCR扩增反应程序为94℃预变性5 min,94℃变性 45 s,53℃退火 45 s,72℃延伸 60 s,38个循环,72℃延伸10 min。
PCR产物用2.0%琼脂糖凝胶,80 V电压下电泳90 min,电泳检测,用凝胶成像仪记录条带数量及位置。
2 结果与分析
2.1 形态鉴定
不同地区羊肚菌子实体形态特征见图1、图2、表4。
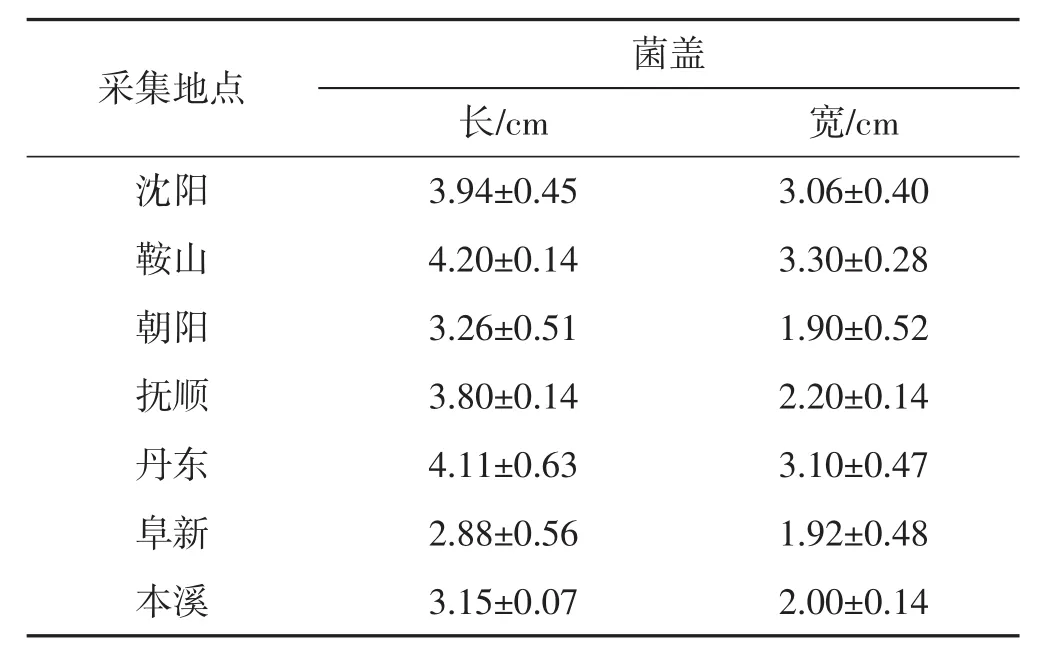
表4 不同地区羊肚菌子实体的大小Tab.4 The size of Morchella spp.in different areas

图1 不同发生地采集的羊肚菌Fig.1 Morchella spp.collected from different habitats

图2 羊肚菌菌丝显微结构Fig.2 Micromorphology of Morchella spp.
由图1,图2,表4可知,辽宁野生羊肚菌子实体偏小。子囊果4.9 cm~6.0 cm,菌盖高2.88 cm~4.20 cm,最宽部位宽1.90 cm~3.30 cm,通常为圆锥形;主脊和横脊交错组成不规则的凹坑;子囊果为棕黄色至深褐色;菌盖与菌柄合生;菌柄长0.8 cm~1.7 cm,宽0.4 cm~1.1 cm,近乎圆柱形;菌柄和菌肉同色,白色,中空。孢子椭圆型,表面光滑,孢子印深灰色。
2.2 分子鉴定
2.2.1 ITS分子鉴定
提取子实体总DNA,ITS序列扩增测序,通过羊肚菌多基因序列模标数据库Morchella MLST[27]比对,53份样本为黄色系羊肚菌。采用MEGE 7.0进化树分析软件,以黑色系羊肚菌Mel-10作为外群,采用Maximum Likelihood Tree法,构建系统发育树见图3。
由图3可知,53份羊肚菌样本,通过NCBI网站查询比较,均归类为Esculenta Clade。其中8X01~8X06、 8C04 ~8C19、 8C91、 7S02 ~7S06、 7C01 ~7C02、7C05~7C06、6F01~6F02、6S02,共 35 份样本与Mes-6同为一种;8D01~8D04,共4份样本与Mes-21为同一种;8B01~8B02,共2份样本与 Mes-8 为同一种;8C01~8C03、8D05~8D08、6A01~6A02、6S01、 6S04~6S05,共 12 份样本与Mes-20和Mes-9聚在一起,共分为4个区域。基于ITS序列构建的辽宁省野生羊肚菌系统发育树无法将第4区域中的12份样本完全区分开来。因此,还需要采用GCPSR法构建系统发育树,明确其种间关系。

图3 基于ITS序列构建的羊肚菌系统发育树Fig.3 Phylogentic tree based on rDNA ITS sequence
2.2.2 系统发育树构建
利用GCPSR的方法,基于ef1-α、LSU、rpb1和ITS这4个基因的核苷酸序列,对53份羊肚菌样品进行了系统发育分析。多基因序列经逐一多重比对后,利用Sequencematrix进行多源数据合并,数据文件进行同质性检验(partition homogeneity test,PHT),采用ILD Test检验方法,P值为0.06,大于数据集可联合的阈值0.05,表明4种基因序列可以联合分析。采用最大简约法(most parsimony tree,MP)构建系统发育树,见图4。

图4 辽宁省黄色羊肚菌系统发育树Fig.4 Phylogenetic tree of yellow morels in Liaoning province
由图4可知,与ITS系统发育树一致,但略有差异。GCPSR法,将与Mes-9和Mes-20聚在一起的 12 份样本区分开来,8C01~8C03、8D05~8D08、6A01~6A02,9份样本与Mes-20聚成一类,6S01、6S04~6S05,3份样本与Mes-9聚成一类。而8D01~8D04被分成两部分,8D01~8D02与Mes-21聚成一类,8D03~8D04与Mes-7聚成一类,因此8D03~8D04的种属关系还需进一步验证。
2.3 ISSR的统计分析
2.3.1 ISSR-PCR扩增
以辽宁省分布最多的Mes-6为研究对象,32份样本进行扩增,11条引物共获得56个位点,片段大小分布在300 bp~2 000 bp,其中具多态性的位点51个,占总位点数的91.07%。
2.3.2 遗传多样性分析
利用遗传多样性分析软件POPGEN version,辽宁省野生羊肚菌遗传多样性分析见表5。
由表5可知,居群水平上,平均每个位点的有效等位基因数 (Ne) 为 1.211 6±0.082 2,Nei’s基因多样性(H) 为0.120 1±0.046 6,各居群多样性信息指数为0.106 6~0.287 0,平均指数为0.177 4±0.069 5。居群之间,遗传多样性有显著性差异,其中P8遗传多样性水平最高,采样点位于朝阳北票市速生杨人工林中。P10遗传多样性水平最低,采样点位于朝阳喀左老爷庙地区。

表5 野生羊肚菌遗传多样性水平Tab.5 Genetic diversity of Morchella
2.3.3 遗传多结构分析
利用Arlequin软件分析辽宁野生羊肚菌居群的基因多样性Nei’s结果表明,辽宁省Mes-6野生羊肚菌种居群遗传有一定分化。6个居群总基因多样度(Ht)为0.306 2±0.028 5,其中居群内多样度(Hs) 为0.120 1±0.009 8,居群间基因多样度为0.186 1,居群间遗传分化系数(Gst)为0.607 9,表明在物种水平上,居群间存在60.79%的遗传变异,而居群内存在39.21%的遗传变异,居群间的遗传分化大于居群内。基因流(Nm)为0.322 5,表明辽宁省野生羊肚菌在各地区存在非常有限的交流或短距离移动。
2.3.4 居群聚类分析
Nei’s遗传一致度与遗传距离见表6。
由表6可知,运用POPGEN version软件分析获得6个居群间的遗传距离(D)和遗传一致度(I),遗传距离为0.084 6~0.492 1,遗传一致度为0.611 4~0.918 9。

表6 Nei’s遗传一致度与遗传距离Tab.6 Nei’s unbiased measures of genetic identity and genetic distance
3 讨论
羊肚菌资源种类丰富,营养和药用建价值高,对羊肚菌种属关系的鉴定是羊肚菌资源保育及其开发利用的基础。传统羊肚菌研究是基于羊肚菌形态学和生态学,包括子实体形态观察和显微结构观察等。收集的53份辽宁省野生羊肚菌子实体,表面多凹坑,凹坑形状不规则,棕黄色至深褐色;菌柄短且较细,乳白色,圆柱状,表面光滑,符合羊肚菌的形态特征,可确定为羊肚菌,但很难对其进行准确的分类鉴定。野生羊肚菌在生长发育过程中,形态特征受环境条件影响较大,由于连续3年春季降雨比较少,全年干旱,采摘时地面上菌帽清晰可见,菌柄高度小于1 cm,菌帽采摘时已失水严重,子实体大小也比往年小[24]。
羊肚菌种属丰富、物种多样,采用单一的分子标记技术(如PCR-ITS,PCR-RAPD) 进行鉴定,会出现同物异名和同名异物现象[14]。对收集的53份样品ITS rDNA进行测序分析,通过Morchella MLST数据库比对,均属于黄色羊肚菌支群,以Mes-10为外群,构建羊肚菌系统发育树[28,30];Mes-20、Mes-9和 12 份样本 (8C01~8C03、8D05~8D08、6A01~6A02、6S01、 6S04~6S05,) 聚在一类,但无法精确鉴定这12份样本种属关系,可能导致同物异名现象发生。
多基因系谱一致性系统发育种识别法(GCPSR法),是通过多基因构建系统发育树进行物质界定,因此也被称为多基因联合分析法。通过对ef1-α、LSU、rpb1核酸序列测序[7,20],结合ITS序列,采用MP法,构建羊肚菌进化树,获得辽宁省羊肚菌遗传多样性图谱。通过GCPSR法,将ITS法精确鉴定的12份样本与Mes-20和Mes-9清晰分开。
在基于ITS序列的物种多样性的基础上,结合ef1-α、LSU、rpb1三个片段,联合分析,更准确、全面地对辽宁省羊肚菌属进行了分析,并界定到具体的物种并正确命名。53份羊肚菌样本被鉴定为5种,即 Morchella sp.(Mes-6)、Morchella sp.(Mes-8)、Morchella sp.(Mes-9)、Morchella sp.(Mes-20)、Morchella sp.(Mes-21),其中, Mes-20 和Mes-21为辽宁省新记录种。8X01~8X06、8C04~8C19、8C91、7S02~7S06、7C01~7C02、7C05~7C06、6F01~6F02、6S02 鉴定为 Mes-6,8B01~8B02鉴定为Mes-8,6S01、 6S04~6S05 鉴定为 Mes-9,8C01~8C03、 8D05~8D08、 6A01~6A02 鉴 定 为 Mes-20,8D01~8D04鉴定为Mes-21。在羊肚菌多基因序列模标数据库Morchella MLST[28]中,有辽宁省2个采样点的数据,丹东采样点收集的羊肚菌鉴定为Mes-8,康平采样点收集的羊肚菌鉴定为Mes-6。对辽宁省康平地区羊肚菌资源[26]收集鉴定后,确定康平存在2个羊肚菌种,即Mes-6和Mes-9。收集的53份样本,鉴定为5个种,经比对,确定Mes-20和Mes-21为辽宁省新记录种。该研究成果不仅丰富了辽宁省野生羊肚菌多样性的菌种资源库信息,还为野生羊肚菌的人工栽培及进一步开发利用提供科学依据。辽宁省羊肚菌物种多样性丰富,还需加强羊肚菌资源分类研究。
4 结论
利用11条ISSR引物对Mes-6的6个居群进行分析,结果显示,辽宁省羊肚菌在居群水平上的遗传多样性较低。虽然辽宁省东部山区、大凌河流域特有的地理特征、生境特征,为丰富遗传资源提供了孕育场所,但近些年人们对羊肚菌认识越来越广泛,人为滥采滥伐,造成了生态环境破坏和居群片段化,对辽宁省羊肚菌资源调查的准确性造成了一定的影响。因此,需加强野生羊肚菌资源的保护,进一步研究辽宁省羊肚菌种群进化活跃区域,探索辽宁省羊肚菌种群、居群间的演化过程。

