Luminal型乳腺癌胞膜蛋白相关预后模型的构建与验证
朱钊雯,贾卫娟,刘洁琼
胞膜蛋白(plasma membrane proteins,PMPs)癌细胞增殖、转移和免疫逃逸等过程有关,随着这些基因表达水平的改变,细胞无限增殖、粘附细胞外基质、侵入血管或淋巴管、于靶器官定植的能力也发生了相应的变化[1-3]。乳腺癌现已成为危害女性健康最常见的癌症[4],而luminal型乳腺癌占所有乳腺癌的70%以上[5]。
由于PMPs在肿瘤发生发展中经过受体配体结合发挥相应的生物效应,成为许多抗癌药物研发的目标靶点[6],如多种细胞表面受体和离子通道[7-9]。然而,近来除了CDK4/6抑制剂外,应用于Luminal型乳腺癌的新型PMPs相关治疗靶向有限[10]。同时,鉴于PMPs表达于细胞膜上,便于临床上的定性定量检测,更能快速有效地获取每一位患者的相应肿瘤表达特点,进而运用于临床工作。本研究旨在获得一种有效的Luminal型乳腺癌PMPs相关预后预测模型,同时尽可能为Luminal型乳腺癌患者筛选出潜在的PMPs相关治疗靶点。
1 材料和方法
1.1 临床样本及数据采集
从TCGA数据库中下载Luminal型乳腺癌转录组RNA测序数据和这些患者的临床信息作为训练集,包括769例Luminal型乳腺癌和111例正常组织样本。排除标准包括:患者①无乳腺癌分子分型信息;②不具备完整的临床数据:包括总生存期(overall survival,OS)、生存状态、诊断年龄、肿瘤大小(T)和淋巴结转移情况(N)。另外,我们从GEO数据库下载了两个微阵列数据集GSE20685和GSE37751中的RNA测序数据及临床信息作为外部验证集。最终,246例Luminal型乳腺癌患者被纳入验证集。同时我们从人类蛋白质图谱数据库中获得了相应的PMPs基因列表。
1.2 训练集差异表达基因分析
利用R软件中Limma包将肿瘤组织与正常组织进行比较,得到所有差异表达基因(deferentially expressed genes,DEGs)和差异表达PMPs(DEPMPs)。筛选条件为logFC>0.5,P<0.05。
1.3 PMPs预后模型
首先,将DEPMPs的基因表达量与相应患者的OS和生存状态相结合,筛选出影响预后的DEPMPs,进一步运用多因素Cox回归分析建立PMPs相关预后预测模型。将构建模型中相关基因表达水平与各自的Cox回归系数相乘,得到每个患者的风险评分(RiskScore)根据RiskScore中位数将训练集中的患者划分为高危组和低危组。两组生存差异采用Kaplan-Meier生存分析及log-rank检验,并计算ROC曲线下面积,验证RiskScore的有效性和预测模型的准确性。
1.4 多基因预后模型验证
利用验证集中246例Luminal型乳腺癌患者的数据验证上述模型的临床有效性。计算风险评分,根据中位风险评分将患者分为高、低风险组,构建K-M生存分析和ROC曲线验证预后预测模型的有效性。
1.5 统计分析
使用R软件通过Survival ROC包计算ROC曲线的AUC以检验预后模型的有效性。对于K-M曲线,通过log-rank和单因素Cox风险回归分析得到95%置信区间的P值和风险比(HR)。所有的统计检验都是双向的。P<0.05被认为是具有统计学差异的。
2 结果
2.1 筛选差异表达基因DEGs
首先,从训练集中得到所有的DEGs(图1A),进一步发现Luminal型乳腺癌的DEPMPs基因中有59个上调基因和85个下调基因(图1B)。
2.2 预后模型的建立
通过单因素Cox回归分析,从DEPMPs中筛选出30个预后相关基因(图2),进一步通过多变量Cox回归分析,构建出一个包含11个基因的预后预测模型,模型计算公式如下:[ADRA1B的基因表达量*(0.164496)]+[CD99L的基因表达量*(0.35221)]+[EZR的基因表达量*(0.33013)]+[IYD的基因表达量*(0.119785)]+[RGS9BP的基因表达量*(0.125277)]+[SLC16A2的基因表达量*(0.165859)]+[DUS1L的基因表达量*(-0.57798)]+[KIT的基因表达量*(-0.07974)]+[MS4A1的基因表达量*(-0.06685)]+[PI3的基因表达量*(-0.0783)]+[SUSD2的基因表达量*(-0.07928)]。由图2可知,ADRA1B、CD99L2、EZR、IYD、RGS9BP及SLC16A2高表达和DUS1L、KIT、MS4A1、PI3及SUSD2的低表达均与患者预后不良有关。据此计算所有患者的风险评分,根据风险评分中位数将患者分为高危组和低危组(图3A)。同时我们绘制得到了患者的生存状态图(图3B)和11个预后基因的生存热图(图3C)。单因素Cox回归分析显示,包括诊断年龄、分期、T、N在内的临床特征和风险评分都不同程度地影响患者的预后(图4A)。多因素Cox风险回归分析表明诊断年龄、分期和风险评分的P值具有统计学意义(图4B)。为了预测患者的OS,我们得到了高危组和低危组之间的K-M生存曲线,发现高危组的总生存期明显低于低危组(图4C)。此外,风险评分的AUC为0.825,表明该模型可能具有较好的预后预测潜力(图4D)。

图1 DEGs基因(图1A)与DEPMPs基因(图1B)
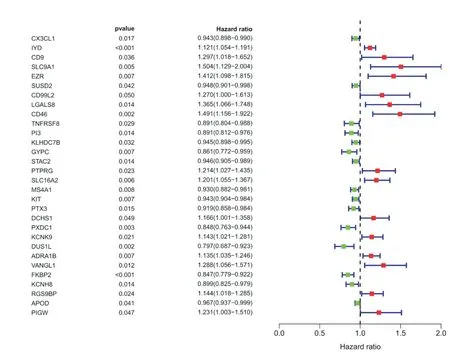
图2 预后相关DEPMPs基因红色为HR>1,绿色为HR<1
2.3 多基因预后模型的验证
利用验证集的RNA测序数据对预后预测模型进行验证,计算验证集中所有患者的风险评分并将患者分为高危组和低危组。结果提示高危组患者预后较低危组差(图5A),且风险评分ROC曲线下面积为0.861(图5B),进一步证明该模型具有一定的临床应用价值。
3 讨论
本研究中,我们建立并验证了可行的Luminal型乳腺癌PMPs预后预测模型。通过分析Luminal型乳腺癌PMPs预后相关基因,我们构建了一个包含高风险相关基因ADRA1B、CD99L2、EZR、IYD、RGS9BP和SLC16A2,以及低风险相关基因DUS1L、KIT、MS4A1、PI3和SUSD2的预后预测模型。
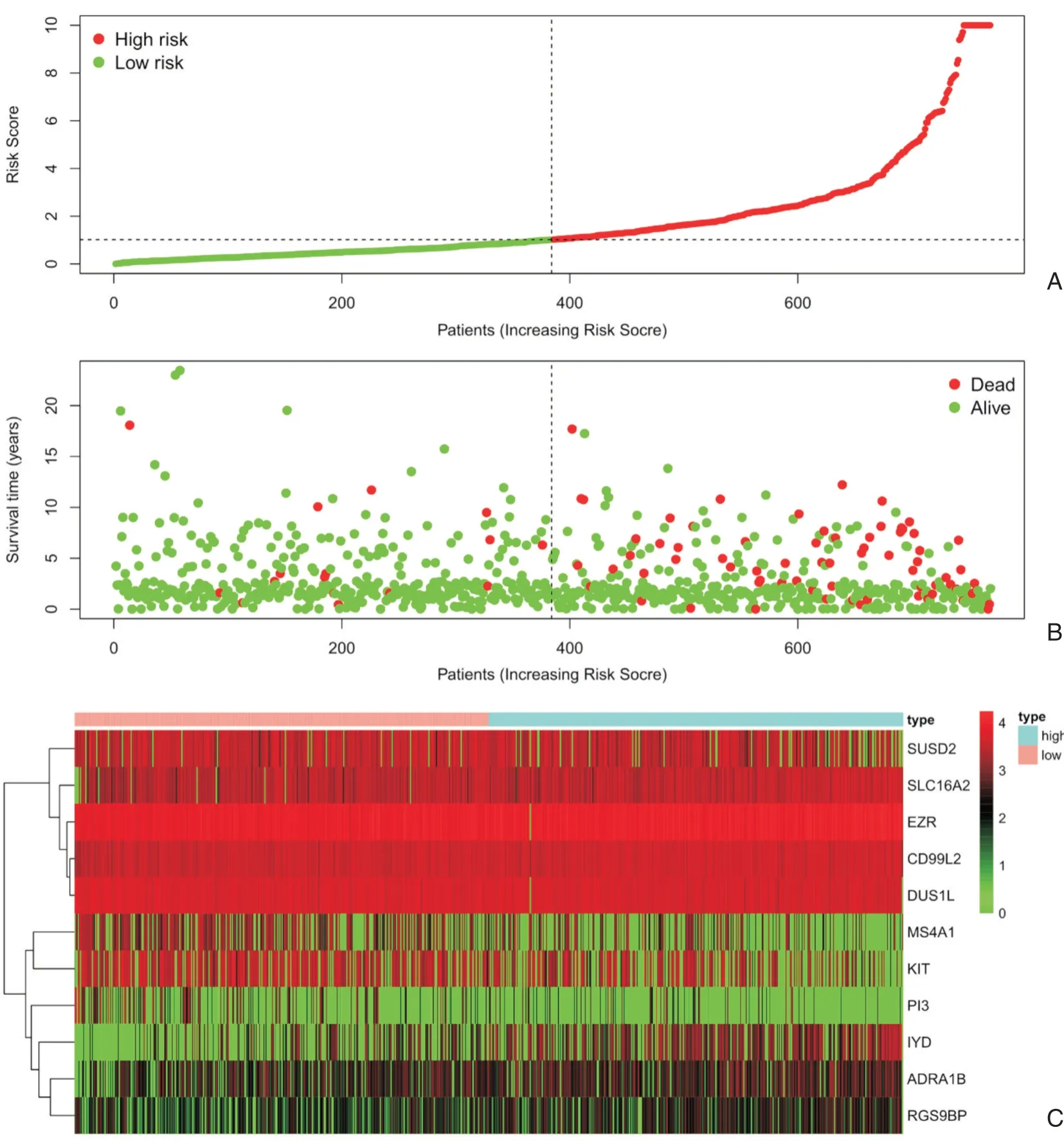
图3 预后模型的特征患者风险评分(图3A)、生存状态图(图3B)和模型中预后基因的生存热图(图3C)
据报道,ADRAIB(肾上腺素受体α1B)增加细胞增殖的同时减少细胞的凋亡,导致较长的肿瘤生存时间和肿瘤复发[11]。CD99L2可以帮助中性粒细胞突破内皮基膜并迁移到炎症组织中[12]。EZR已被证实在多种癌症的增殖、迁移和侵袭中发挥重要作用[13,14]。Ezrin由EZR编码,交联胞膜和构建细胞骨架,Ezrin的磷酸化依赖于TGF-β和MAPK通路的激活,参与癌症的生长和转移,导致疾病进展和低生存率[15,16]。有研究报道IYD能够抑制肝癌细胞糖酵解,进而通过增加有氧糖酵解支持癌细胞的存活和增殖,并导致癌症患者预后较差[17]。RGS9BP在趋化因子诱导的淋巴细胞迁移中发挥作用,负调控naive和调节T细胞迁移的能力,从而影响T淋巴细胞的免疫应答[18]。作为SLC16基因家族中与生存相关的代谢基因,编码MCT8的SLC16A2的表达减少与部分头颈部癌症患者生存的改善相关[19]。
在乳腺癌中,KIT编码的CD117+造血祖细胞,可在肿瘤发生远处转移之前,便被富集至肺部诱导细胞外基质重构,进而为肿瘤的肺部转移与定植提供条件[20]。而在血液系统疾病中,抗人CD117嵌合抗原受体T细胞可有效治疗骨髓增生异常综合征与急性髓系白血病,这也为乳腺癌肺转移患者的靶向治疗提供了启示[21]。SUSD2编码含822个氨基酸的蛋白,与半凝集素-1相互作用,促进乳腺癌细胞的免疫逃逸与转移、肿瘤血管新生,并显著减少CD4肿瘤浸润淋巴细胞,这表明SUSD2可能是乳腺癌甚至其他癌症的新靶点[22,23]。
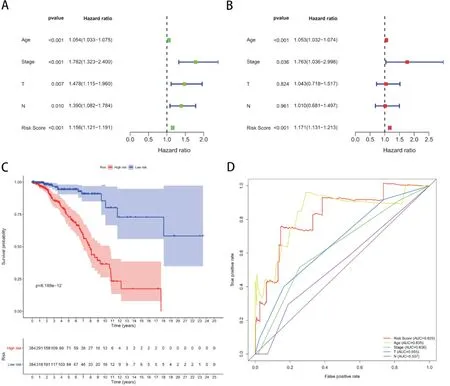
图4 模型的预后价值单因素Cox回归分析(图4A);多因素Cox回归分析(图4B);高低风险组患者生存曲线(图4C)和ROC曲线(图4D)
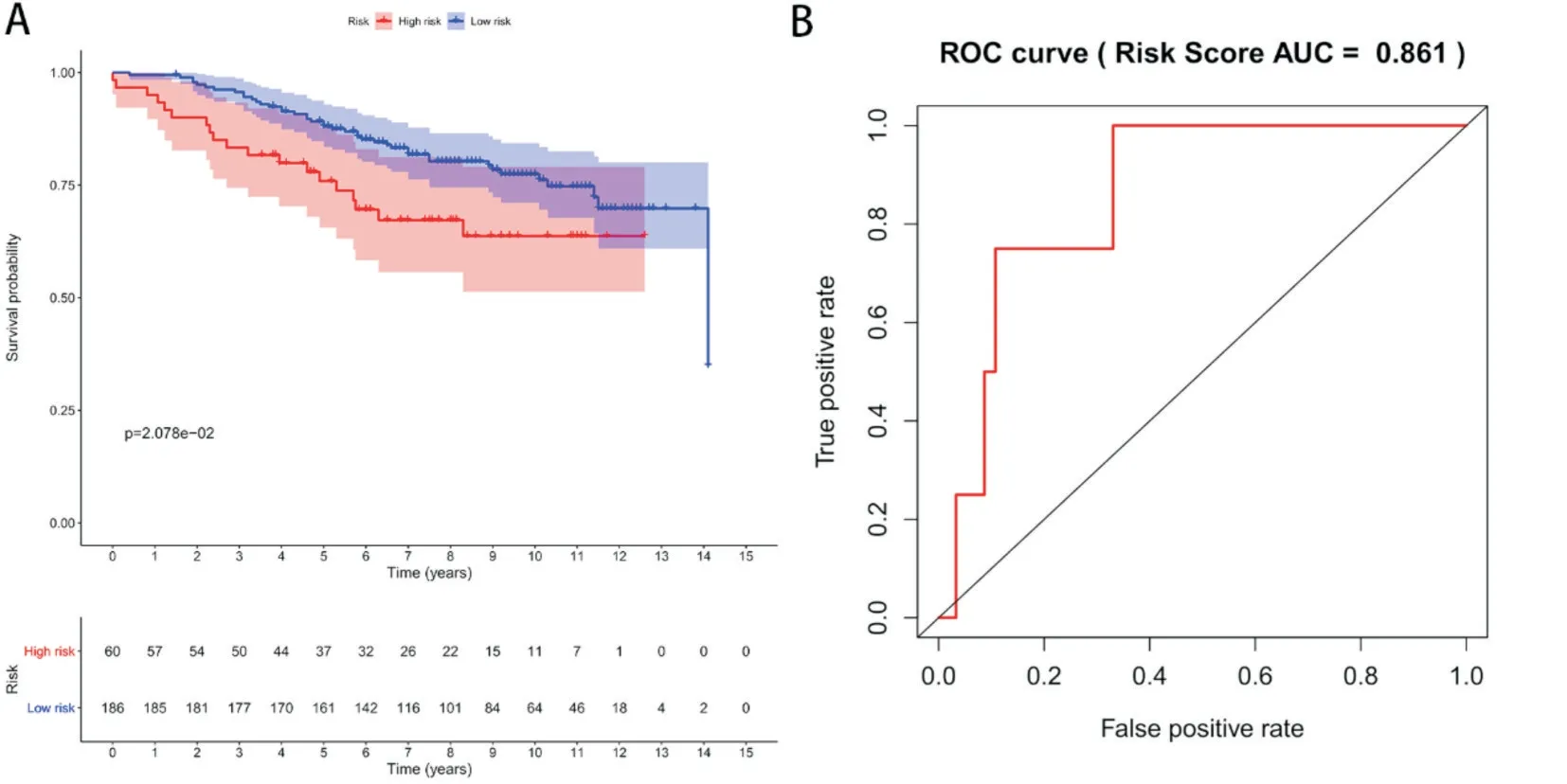
图5 预后模型效能的验证:高低风险组患者生存曲线(图5A)和ROC曲线(图5B)
无论在训练集还是验证集中,高危患者的预后都明显较差。进一步采用多因素Cox回归分析,风险评分的AUC均大于0.8。在类似的研究中,肿瘤突变负荷、自噬、不同表达的mRNA或免疫相关预后信号的AUC分别为0705、0.742、0.785、0.83[24,25]。因此,我们的基于11个基因的预后预测模型可能有更好的临床应用潜力,而其中一些基因甚至可能成为有效的治疗靶点。基于11个预后模型相关基因,我们认为可以为深入的实验研究提供一些新的研究思路。
同时,本研究也存在一些局限性。首先,我们还需要更多的功能性实验来探索和验证我们的PMPs预后预测模型的有效性,以及它们是否有可能成为精准治疗的治疗靶点。其次,基于研究现状,我们暂且仅能关注于已被发现及验证的在PM上表达的蛋白质,而不是所有的PMPs。

