一个鉴别低温刺激型秀珍菇的ISSR-SCAR标记建立与应用
陈雪凤 吴圣进 王灿琴 韦仕岩 王晓国 郞宁 张雯龙

摘 要:为了快速、准确地鉴别低温刺激型秀珍菇菌株,在对18个供试菌株进行拮抗试验、现蕾出菇试验、ISSR图谱分析的基础上获得了秀珍菇菌株的ISSR特异性标记,将其克隆、测序。根据测序结果,应用Primer 6.0软件设计SCAR引物进行引物筛选,获得1对引物并成功转化为低温刺激型秀珍菇的稳定SCAR标记。采用该引物对供试秀珍菇菌株的基因组DNA 进行PCR扩增,低温刺激型秀珍菇菌株均能扩增出大小为205 bp的特异性DNA片段。该ISSR-SCAR标记能快速、有效地鉴别出低温刺激型秀珍菇菌株,准确率达100%,为秀珍菇种质资源的鉴别和分类提供了技术支撑。
关键词:秀珍菇;低温刺激;ISSR特异性标记;SCAR分子标记
Abstract: A specific ISSR marker was obtained based on the antagonism test, fruiting test and ISSR spectrum analysis of 18 strains to identify the low-temperature stimulating type of Pleurotus geesteranus rapidly and accurately. The specific ISSR marker was cloned and sequenced. SCAR primers (designed by Primer 6.0) for the specific ISSR marker were screened, than a pair specific primers were obtained, which could convert the specific ISSR marker to stable SCAR mark. Verification was conducted by PCR using the screened specific primers to amplify the genomic DNA. Results showed that 205 bp specific fragment was amplified only in the low-temperature stimulating type of P. geesteranus strains. This ISSR-SCAR marker could distinguish the low-temperature stimulating type of P. geesteranus strains rapidly with accurasy of 100%. This study would provide a technological support for the identification and classification of P. geesteranus germplasm resources.
Keywords: Pleurotus geesteranus; low-temperature stimulating; specific ISSR marker; SCAR marker
秀珍菇(Pleurotus geesteranus)隸属于真菌门、担子菌纲、伞菌目、侧耳科、侧耳属,商品名或俗称凤尾菇,其味道鲜美、营养丰富,是一种深受广大消费者喜爱的食用菌。近年来随市场的需求,秀珍菇的栽培规模日益增大,且国内栽培的秀菇菌株品种繁多。秀珍菇根据出菇温度可分为高温型品种和低温型品种,根据出菇是否需要低温刺激可分为自然出菇型(俗称假秀珍)和低温刺激型(也称反季节秀珍菇或真秀珍)。自然出菇型秀珍菇特性是菌丝长满袋且生理成熟后不需要低温刺激即可出菇。低温刺激型秀珍菇特性是菌丝长满袋且生理成熟后,需要低温刺激方能整齐出菇。真秀珍菇和假秀珍由于出菇时对低温温差要求程度不一样,因而栽培管理方法差异较大。生产中如不了解秀珍菇出菇时所需要的温度特性,采取不恰当的栽培管理方法,会造成出菇差或不出菇、产量低等问题。鉴别秀珍菇出菇是否需要低温刺激,在菌丝生长阶段无法判断,只有在栽培出菇后才能判别。但栽培出菇周期长,耗时耗力,出菇时还会受到各种气候因素的影响,判定结果会存在较大误差。因此,迫切需要一种简便快速的方法来鉴别不同温型的秀珍菇品种。
DNA分子标记由于检测手段简单、高效,被应用到许多领域。近年来,利用分子标记进行食用菌品种种间的遗传多样性分析、遗传育种、检测和基因克隆等方面研究在很多品种上都有广泛应用。分子标记种类很多,如RAPD、SSR、SRAP、ISSR等,其中SCAR标记(Sequence characterized amplified region)重复性好、可靠性高、对反应条件不敏感、不同材料的DNA差异可通过单一条带的出现与否加以判断,是一种非常方便、快捷的可用于快速检测大量个体的方法。目前,在香菇、黑木耳、蘑菇、草菇、侧耳、羊肚菌等种质资源上已建立了稳定的SCAR标记[1-8]。在秀珍菇研究上,已有ISSR[9-12]、SRAP[13]、RAPD[14-15]、EST-SSR[14]等分子标记应用于种质资源遗传多样性和亲缘关系分析方面的报道,但能成功转化成SCAR稳定标记的研究较少。有学者发现秀珍菇出菇温型与ISSR分子标记分类相关性较高[11],但没有与温度相关的SCAR分子标记的报道。
目前,我国秀珍菇品种繁多且各地相互引种,异种同名、同物异名现象较多,菌种混乱,为了更便捷、准确、科学地进行低温刺激型与非低温刺激型秀珍菇菌株的鉴定分类,本研究采用拮抗、出菇试验、ISSR分子标记技术对18个秀珍菇菌株进行了测定,旨在构建可以鉴别低温刺激型秀珍菇菌株的SCAR分子标记,为加快秀珍菇菌株的鉴别和定向选育提供分子技术支撑。
1 材料与方法
1.1 材料
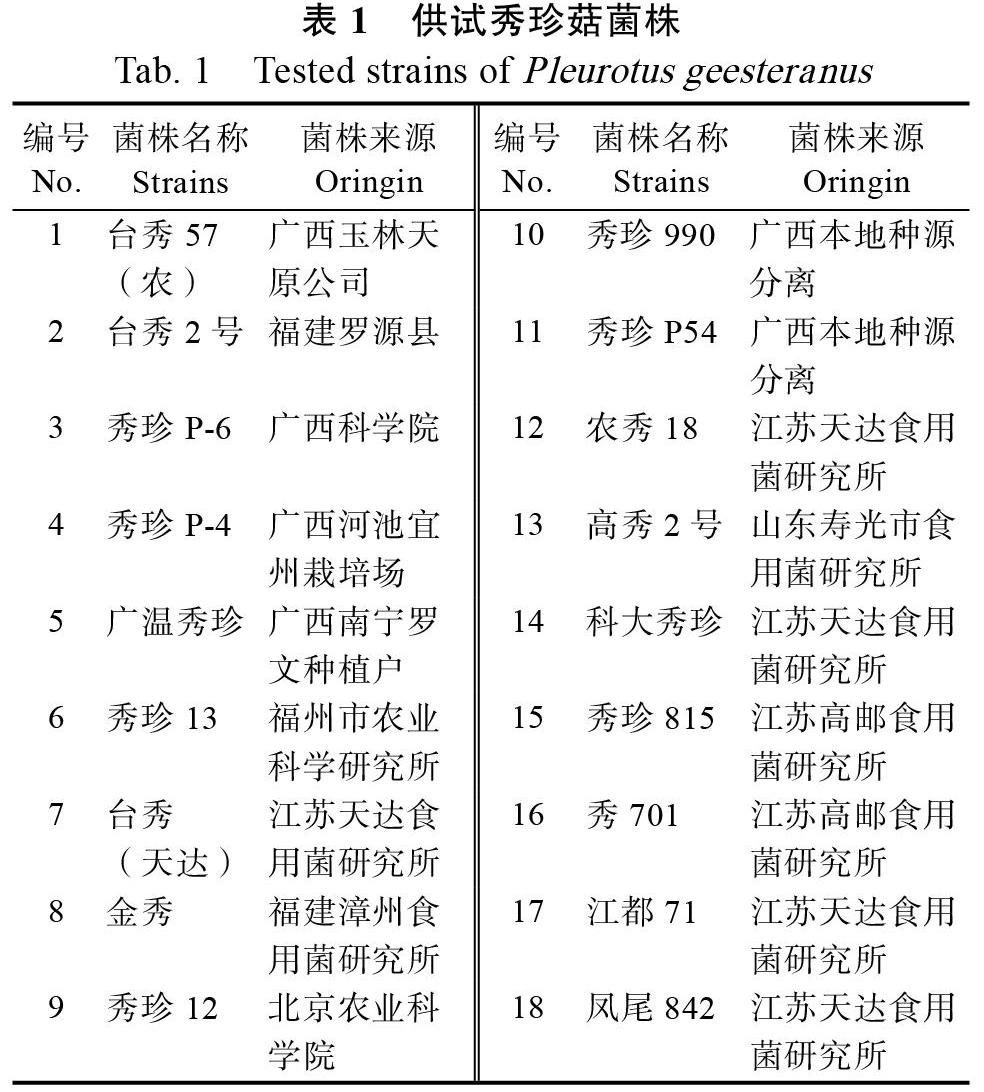
1.1.1 供试菌株 供试菌株为18个秀珍菇菌株,具体名称和来源见表1。
1.1.2 培养基 出菇试验培养料配方:棉籽壳20%、桑枝30%、木屑30%、麦麸18%、轻钙1%、石灰1%。PDA综合培养基:马铃薯200 g、葡萄糖20 g、硫酸镁2 g、磷酸二钾2 g、琼脂20 g、水1000 mL。
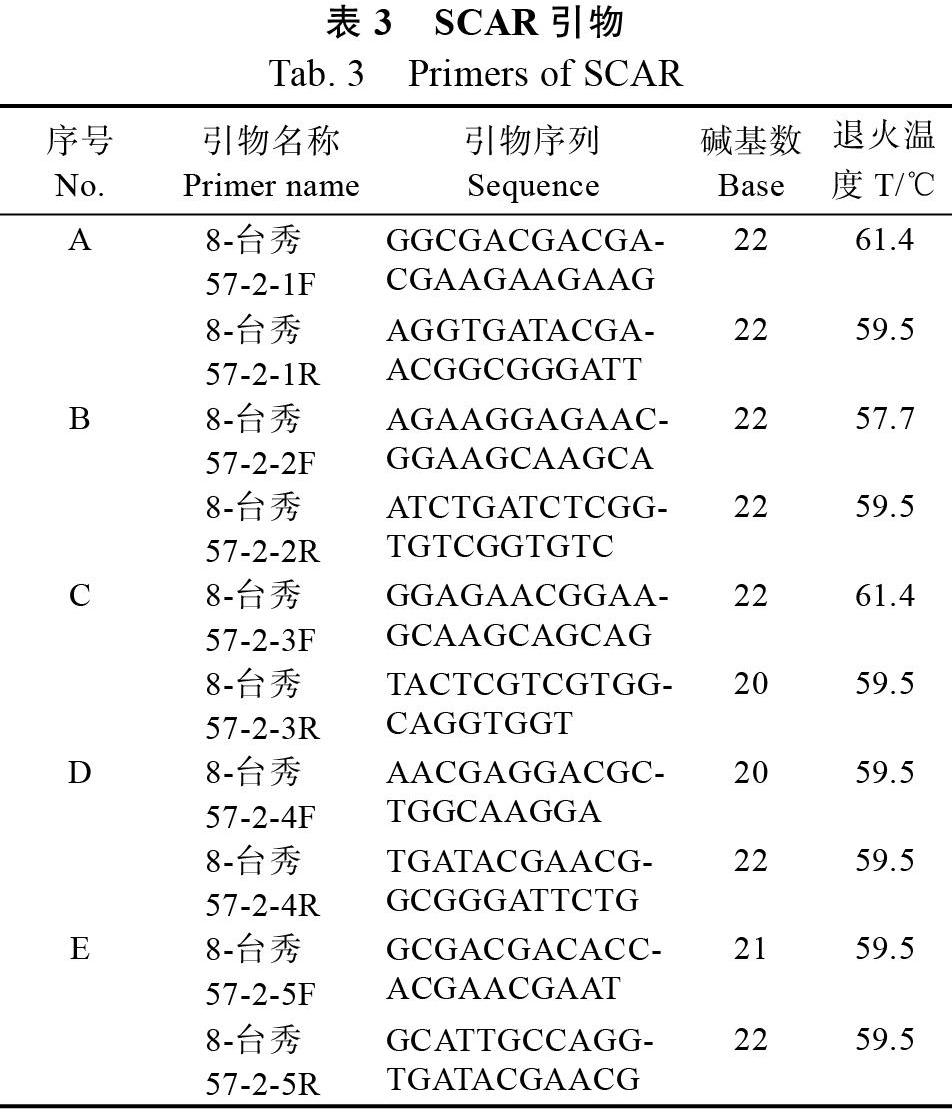
1.1.3 试剂 基因组DNA提取试剂盒、2×Es TaqMasterMix (Dye)、Marker DL2000购自北京康为世纪生物有限公司;通用型DNA纯化回收试剂盒购自北京天根生化科技公司;GeLRed核酸凝胶染料购自上海玉博生物科技有限公司;DNA连接试剂盒购自TaKaRa公司;ISSR、SCAR引物由北京擎科生物技术有限公司合成,引物的名称及核苷酸序列见表2~表3。
1.2 方法
1.2.1 供试菌株出菇温差类型的初步判定 18个秀珍菇菌棒菌丝长满后,进行出菇试验。出菇分2种方式进行:①菌棒菌丝生理成熟后在25~28 ℃下自然放置进行出菇,4~6 d观察现蕾出菇情况;②菌棒菌丝生理成熟后放入8~10 ℃的冷库中放置12 h后,拿出放置室温下2~3 d,观察菌棒现蕾出菇情况。
1.2.2 18个菌株的拮抗试验 利用PDA综合培养基对18个菌株进行拮抗试验。接种后的培养皿放置于28 ℃的培养箱中培养,培养10 d左右观察菌株之间的拮抗反应[16]。
1.2.3 基因组DNA的提取与检测 将菌丝块接种到PDA综合培养基的培养皿上,于27 ℃培养6~8 d,用解剖刀刮取菌丝。使用DNA提取试剂盒提取菌丝DNA,用1%琼脂糖凝胶电泳检测质量,质量合格的DNA储藏于?20 ℃冰箱备用。
1.2.4 具有多态性、特异性ISSR引物的筛选 利用前期研究[9]筛选获得18个对秀珍菇具有多态性的ISSR引物(引物序列见表2)对供试的18个秀珍菇菌株基因組DNA进行扩增。PCR产物用1%琼脂糖凝胶检测,观察ISSR图谱,选出具有特异性片段的ISSR引物。ISSR-PCR反应体系及条件:2×Es TaqMasterMix(Dye) 12 μL,ddH2O 11 μL,引物(10 μmol/L)1 μL,DNA模板1 μL,总体积25 μL;94 ℃预变性5 min;94 ℃变性30 s,45~55 ℃退火30 s,72 ℃延伸1 min,共35个循环;最后72 ℃终延伸10 min,16 ℃保存30 min。
1.2.5 秀珍菇特异性DNA片段的获取及序列测定 从18个菌株中选出4个代表菌株(低温刺激型菌株台秀57(农)、金秀和非低温刺激型菌株秀珍P54、农秀18),利用1.2.4筛选出的特异性引物分别对4个代表菌株的基因组DNA进行PCR扩增,并进行1.2%琼脂糖凝胶电泳检测。紫外光下查看在琼脂糖凝胶的特异条带,利用通用型DNA纯化回收试剂盒对特异片段进行DNA纯化回收。将回收纯化的DNA片段连接到载体pMD18-T Vector上,进行转化培养,培养后对克隆菌落进行PCR扩增。然后用1.2%的琼脂糖凝胶对ISSR-PCR产物、回收产物、克隆-PCR产物进行电泳检测,对正确的克隆子进行测序。克隆子PCR扩增反应体系及条件:2×Es Taq Master Mix(Dye) 10 μL,M13(47)0.5 μL,M13(48)0.5 μL,克隆子稀释液模板0.5 ?L,ddH2O 8.5 μL,总体积20 μL;94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸90 s,25个循环;最后72 ℃延伸5 min。
1.2.6 SCAR引物合成及SCAR标记的验证 根据克隆子测序结果,用Primers 6.0设计SCAR引物,由北京擎科生物技术有限公司进行引物合成。利用合成引物对4个代表菌株基因组DNA扩增,进行引物筛选。利用筛选出来的合成引物对18个秀珍菇菌株基因组DNA扩增验证,并对特异片段进行测序。SCAR扩增反应体系:DNA模板l μL,2×Es Taq MasterMix(Dye)12 μL,前引物(10 μmol/L)1 μL,后引物(10 μmol/L)1 μL,ddH2O 10 μL,总体积为25 μL;94 ℃预变性5 min;94 ℃变性30 s,55~65 ℃退火30 s,72 ℃延伸l min,35个循环;72 ℃终延伸10 min。
2 结果与分析
2.1 菌株出菇类型的初步判定
通过18个菌株出菇试验,台秀57(农)、台秀2号等8个菌株的菌丝长满生理成熟后,在25~28 ℃条件下,菌棒不会直接出菇,而在较大的低温刺激(8~10 ℃)条件下能很快现蕾出菇,且出菇整齐;秀珍12、秀珍990等10个菌株菌丝满袋后在25~28 ℃温度条件下直接现蕾出菇,同时在低温条件下刺激也能现蕾出菇,但出菇不整齐,具体情况见表4。
2.2 供试菌株的拮抗情况
拮抗试验显示,低温刺激型秀珍菇菌株与非低温刺激型菌株拮抗程度较大,同类菌株之间无拮抗或拮抗程度较低(表5、图1~图2)。
2.3 具有多态性、特異性ISSR引物的筛选
通过18个ISSR通用引物对18个供试菌株基因组DNA的PCR扩增产物图谱发现(图3),ISSR通用引物8对供试菌株具有多态性和特异性,即不同类型的菌株能扩增出较一致且有别于其他菌株的特异性谱带。
2.4 秀珍菇特异性DNA片段的获取及序列测定
利用ISSR通用引物8分别对4个菌株的基因组DNA进行PCR扩增,获得特异性谱带(图4)。回收利用台秀57的特异谱带,通过ISSR-PCR产物、胶回收产物、克隆子-PCR产物电泳检测,观察检测图谱,3个产物扩增条带位点一致(图5),说明阳性克隆子克隆正确。选出正确的克隆子进行测序,所得台秀57(农)胶回收DNA片段序列长1312 bp(图6A)。
2.5 SCAR引物的筛选及验证
根据台秀57获得的1312bp DNA序列共设计合成5对SCAR引物(表3)。利用这5对引物分别对4个代表菌株的基因组DNA进行PCR扩增。由图7可见,低温刺激型菌株台秀57、金秀利用引物A能扩增出特异性片段,而利用其他SCAR引物扩增,没有获得特异性片段。利用引物A对18个供试菌株的基因组DNA进行扩增,由图8可见,8个低温刺激型菌株均能扩增出特异性片段,而10个非低温刺激型菌株未能扩增出特异片段。通过验证,可以说明引物A是低温刺激型秀珍菇的特异性标记,鉴定准确率达100%。特异性片段经胶回收测序,片段大小为205 bp(图6B)。
3 讨论
用于真菌菌种鉴定的拮抗反应是指同种真菌不同个体之间的一种相互识别、相互排斥的现象,准确的应该叫做体细胞不亲和性或营养不亲和性。长期以来,拮抗反应被广泛用于真菌菌株鉴别、群体遗传学研究中,但是,由于非拮抗基因
的差异也造成了亲和菌株间的遗传差异,拮抗反应程度如弱,拮抗与无拮抗之间的界定受实验者主观影响比较大,培养基成分也影响拮抗的有无和程度强弱。拮抗反应鉴别亲和菌株的遗传关系存在着不确定性,对于遗传关系比较近的菌株来说,拮抗反应只能作为菌种鉴定的一个辅助方法[17-18]。出菇试验是种质资源鉴定评价所必须的,但在区别温差型品种时,受外界自然条件影响较大,如在秋冬或冬春栽培时,有时外界环境早晚温差大,全部菌株均会正常出菇,导致错误判断某些菌株是低温刺激型或是非低温型菌株。DNA是物种中较稳定的遗传物质,其反映遗传关系较形态学和生理生化指标更客观准确,SCAR分子标记技术操作简单、快速、准确可靠,不受外界环境条件的影响,是较为可靠的秀珍菇种源鉴定分析的方法,但有时因DNA酶降解等一些因素,偶尔会产生一些不稳定性。因此,在种质资源鉴定中,三种方法结合使用则更为科学、准确。
ISSR检测手段简单、高效,常用于揭示食用菌遗传多态性和区分种内菌株间亲缘关系,是一种快速、可靠的工具。冯伟林等[11]、陆娜等[10]、林原等[12]、熊芳[7]利用ISSR技术对秀珍菇菌株进行了种群分类,即种内菌株间的亲缘性鉴定。在几位学者的研究中,大部分只是将他们所研究的菌株分为几个类群,只有冯伟林等[11]发现农艺性状中出菇温度与ISSR分子标记有相关性,但没有找到与温度相关的准确分子标记报道。熊芳[7]利用RAPD、SRAP、ISSR分子技术进行了秀珍菇种质资源种群分析,并建立6个SCAR标记,但这几个分子标记未说明与温型相关。笔者在前期研究中,利用ISSR技术对8个秀珍菇菌株多样性研究分析[9],发现在遗传相似系数0.59时8个菌株明显分为2个群,即不需要大温差刺激出菇的菌株类群与需要大温差刺激出菇的菌株类群,2个类群亲缘关系较远,这也进一步说明了出菇温型与ISSR分子标记分类相关性较高。但ISSR扩增易受反应条件影响,结果存在重复性差的问题,而且由于图谱条带数多、背景模糊,给判断会带来一定的误差。与ISSR标记相比,SCAR分子标记对反应条件不敏感,容易获得稳定的、可靠的、可重复的条带,因此,笔者在ISSR分析的基础上,将ISSR标记成功转化成SCAR标记,提高了鉴定的稳定性、准确性。
本次研究的供试菌株选择了近几年在广西范围内推广应用的菌株,验证效果明显,但国内当前栽培的秀珍菇菌株众多,且各地交叉引种频繁,异种同名、同物异名现象较多,本研究构建的SCAR标记是否能广泛应用于所有的秀珍菇菌株鉴定,尚有待于在更多样品中进行检验。
参考文献
张美彦, 尚晓冬, 谭 琦, 等. 香菇135菌株特异SCAR标记的分布和遗传特性[J]. 食用菌学报, 2008, 15(4): 1-5.
马庆芳, 张丕奇, 戴肖东, 等. 黑木耳Au185菌株一个SCAR标记的建立[J]. 菌物研究, 2009, 7(2): 104-108, 115.
李媛媛, 隋玉龙, 牛淑力, 等. SCAR标记在黑木耳栽培菌株分类鉴定中的应用[J]. 菌物研究, 2013, 11(3): 182-185, 189.
史灵燕, 张云龙, 刘保卫, 等. 基于SRAP分子标记的24个黑木耳栽培菌株遗传分析[J]. 食用菌, 2019, 41(6): 20-23, 33.
张 静, 陈文炳, 邵碧英, 等. 双孢蘑菇SCAR标记的建立及在菌株群鉴定中的应用[J]. 中国食品学报, 2011, 11(4): 194-202.
傅俊生, 刘新锐, 谢宝贵, 等. 草菇SCAR遗传标记建立及其杂种鉴定应用[J]. 中国农学通报, 2010, 26(17): 41- 46.
熊 芳. 分子标记鉴别侧耳属10种食用菌种植资源的研究[D]. 福州: 福建农林大学, 2008.
刘 伟, 兰阿峰, 张倩倩, 等. 羊肚菌栽培菌株遗传多样性分析及种特异性RAPD-SCAR标记开发[J]. 菌物学报, 2018, 37(12): 1650-1660.
陈雪凤, 郎 宁, 韦仕岩, 等. 8个反季节秀珍菇菌株栽培性状评价与遗传差异分析[J]. 西南农业学报, 2019, 32(8): 1898-1903.
陆 娜, 王伟科, 宋吉玲, 等. 秀珍菇种质遗传多样性分析与优质菌株筛选[J]. 中国食用菌, 2018, 37(6): 20-23.
冯伟林, 蔡为明, 金群力, 等. 秀珍菇菌株主要农艺性状比较及ISSR分子标记鉴定[J]. 食用菌学报, 2014, 21(2): 14-18.
林 原, 陈 剑, 赵光辉, 等. 7个秀珍菇菌株栽培特性及ISS R遗传分析[J]. 福建农业学报, 2015, 30(4): 339- 343.
卢政辉. 秀珍菇及其近缘22株菌株的SRAP分析[J]. 江西农业学报, 2008, 20(11): 8-10, 13.
忻 雅, 阮松林, 王世恒, 等. 基于RAPD和EST-SSR标记的秀珍菇菌株聚类分析[J]. 食用菌学报, 2008, 15(4): 20-25.
朱 坚, 卢启泉, 谢宝贵. 秀珍菇种质资源分子多态性研究[J]. 菌物学报, 2007, 26(增刊): 226-231.
陈雪凤, 韦仕岩, 吴圣进, 等. 广西野生灵芝菌株的遗传多样性分析[J]. 北方园艺, 2016(7): 136-139.
张金霞. 中国食用菌菌种学[M]. 北京: 中国农业出版社, 2011: 76-81.
张美彦. 香菇135菌株特异SCAR标记的遗传规律[D]. 南京: 南京农业大学, 2007.
责任编辑:黄东杰

