基于SRAP分子标记的51份西瓜抗、感病毒病种质资源遗传多样性分析
高宁宁 李晓慧 康利允 常高正 梁慎 徐小利 李海伦 王慧颖 赵卫星


摘要:【目的】利用SRAP分子标记对51份西瓜抗、感病毒病种质资源遗传多样性分析,为加速西瓜抗病毒病新品种的选育进程提供理论参考。【方法】利用筛选出的多态性引物对51份抗、感病毒病西瓜种质进行多态性扩增,利用NTsys-pc 2.1e中的非加权组平均法(UPGMA)计算获得遗传相似系数,利用PopGene Version 3.2计算不同类型西瓜的遗传多样性参数。【结果】从500对SRAP引物组合中筛选出18对扩增条带清晰稳定且多态性较高的引物,共扩增出402条清晰可辨的条带,其中多态性条带271条;平均每对引物可扩增出稳定清晰的条带22.33条,其中多态性条带15.06条,平均多态性比率達67.41%。观测等位基因数(Na)为1.6766、有效等位基因数(Ne)为1.3033、Neis基因多样性指数(H)为0.1878、Shannons信息指数(I)为0.2931,除感病毒型西瓜种质的Na高于抗病毒型西瓜种质外,感病毒型西瓜种质的其他遗传多样性指数均低于抗病毒型西瓜种质,表明抗病毒型西瓜种质的遗传多样性较感病毒型西瓜种质丰富。抗病毒型与感病毒型西瓜种质的遗传一致度为0.8924,遗传距离为0.1138。供试西瓜种质的遗传相似系数为0.62~0.95。在遗传相似系数0.78处,可将51份供试西瓜种质划分为四大类群,抗病毒型西瓜种质分布在Ⅰ和Ⅱ类群中,感病型种质均集中在第Ⅲ、Ⅳ类群中,野生西瓜种质与其他西瓜种质的亲缘关系最远。遗传相似系数矩阵和UPGMA聚类矩阵之间的相关性明显(r=0.914),可准确地体现供试西瓜种质之间的遗传关系。【结论】筛选出的SRAP引物组合具有较好的品种鉴别能力,可用于西瓜种质的遗传多样性分析。抗病毒型与感病毒型西瓜种质的亲缘关系较近,遗传多样性丰富度不高,应加大发掘优良抗病种质资源力度,以拓宽西瓜的遗传基础。
关键词: 西瓜;SRAP;遗传多样性;抗病毒;分子标记
中图分类号: S651.034 文献标志码: A 文章编号:2095-1191(2021)05-1174-09
Abstract:【Objective】To provide theoretical referencefor acceleratingthe breeding process of antivirus varieties of watermelon,the genetic diversity of 51 germplasm resources of antivirus watermelon were analyzed by SRAP molecular markers. 【Method】The polymorphic primers were selected to amplify 51 antivirus and susceptible germplasm resources of watermelon.The genetic similarity coefficients were calculated by unweighted pair group method with arithmetic avera-ge(UPGMA) by NTsys-pc 2.1e. Genetic diversity parameters of different types of watermelon were calculated by PopGene Version 3.2. 【Result】Eighteenpairs of primers with clear and stable bands and high amplified polymorphism were selected from 500 pairs of SRAP primers,and a total of 402 clearly identifiable bands were amplified,among which 271 were polymorphic bands. And 22.33 stable and clear bands were amplified on average with each pair of primers,including 15.06 polymorphic bands,with an average polymorphism rate of 67.41%. The observed number of alleles(Na) was 1.6766,the effective number of alleles(Ne) was 1.3033,the diversity index of Neis gene(H) was 0.1878,and the Shannons information index(I) was 0.2931. Except the Na of susceptible watermelon germplasmswas higher than that of antivirus watermelon germplasms,the other genetic diversity indexes of antivirus watermelon germplasms was higher than that of susceptible watermelon germplasms. The results showed that the genetic diversity of antivirus watermelon germplasms was higher than that of virus susceptible watermelon germplasms. The genetic consistency and genetic distance of the virus resistant and susceptible watermelon germplasms were 0.8924 and 0.1138 respectively. The genetic similarity coefficients of the experimental watermelon germplasms were between 0.62-0.95. With the genetic similarity coefficient of 0.78 as the boundary,51 watermelon germplasms were divided into four groups. Antiviral type of watermelon germplasms were in groupsⅠ and Ⅱ,and virus susceptible type of watermelon germplasms were concentrated in groups Ⅲ and Ⅳ,and wild watermelon materials and other watermelon varieties showed the most distantly related.The correlation between the genetic similarity coefficient matrix and the UPGMA clustering matrix was obvious(r=0.914),which accurately reflected the genetic relationship among the watermelon germplasms. 【Conclusion】The selected SRAP primer combinations have good ability of variety identification and can be used for the analysis of watermelon germplasm genetic diversity. The germplasm of antivirus and virus susceptible type are closely related, so the genetic diversity is not high. Excellent di-sease-resistant germplasms should be explored to broaden the genetic base of watermelon.
Key words: watermelon; SRAP; genetic diversity; antivirus; molecular markers
Foundation item: National Melon Industrial Technology System Construction Project(CARS-25); Henan Key Research and Promotion Special Project(192102110036,202102110041); Innovation Project of Henan Academy of Agricultural Sciences(2020ZC21)
0 引言
【研究意义】西瓜是我国重要的经济作物,病毒病是其重要病害(吴洋等,2017)。随着我国西瓜种植面积增大,病毒种类也逐渐增加,病毒病发生可造成30%~50%的经济损失,甚至绝收,其果实品质也大幅降低(Kang et al.,2005;王凯娜,2018)。常规的化学防治无法有效地控制西瓜病毒病的发生,选育抗病品种是最经济有效的手段之一。但目前制约西瓜抗病毒病育种研究的最重要因素是其抗病毒种质资源的遗传基础较狭窄。因此,收集、保存、筛选抗病毒西瓜种质资源,并进行其遗传多样性分析对抗病毒西瓜种质资源的利用及抗病毒育种具有重要意义。【前人研究进展】目前已利用分子生物学手段对收集保存的抗病毒西瓜种质资源进行基因挖掘,从而找出未被发现的抗病毒基因的潜在种质资源成了我国西瓜育种工作的重要部分(Xu et al.,2004;张永兵等,2013)。SRAP(Sequence-related amplified polymorphism)分子标记是一种基于PCR的标记系统,具有简便快捷、多态性高、稳定等特点(Li and Quiros,2001),已广泛应用在番茄(王燕等,2007)、甘蓝(张羽等,2017)、甜瓜(陈芸等,2010;高宁宁等,2019b)等作物相关研究上。据报道,已利用SRAP分子标记进行西瓜品种指纹图谱构建(赵胜杰等,2009)、基因关联分析(羊杏平等,2013)、种子纯度鉴定(韩宏伟等,2017)等。利用SRAP分子标记分析西瓜遗传多样性的研究主要集中在对杂交种遗传多样性分析(李严和张春庆,2005)、西瓜不同地域种质资源遗传多态性分析(赵胜杰等,2010)、地方品种遗传差异分析(Solmaz et al.,2016)及其在品种DUS测试中的应用(刘丽娟等,2011)等方面。【本研究切入点】目前西瓜抗病毒病研究多集中在抗性基因定位(刘洁,2019)及其转基因研究(吴会杰等,2009)等方面。鲜见利用SRAP分子标记对西瓜抗感病毒病种质资源進行分类、鉴定及遗传多样性分析等方面的研究报道。【拟解决的关键问题】利用SRAP分子标记对51份抗、感病毒病西瓜种质资源进行遗传多样性分析,为抗病毒型西瓜种质资源的高效利用提供有力保障,加速西瓜抗病毒型新品种的选育进程。
1 材料与方法
1. 1 试验材料
供试的51份西瓜种质为河南省农业科学院园艺研究所西甜瓜研究室从全国各地收集并经过多年种植观察的品种(系),其中,1~9号为抗病毒型品种,10~51号为感病毒型品种(表1)。2×Taq MasterMix和DL2000 DNA Marker购自BioTeke公司。主要仪器设备:Thermal Cycler2720 PCR扩增仪、台式高速离心机和多功能紫外分光光度计。
1. 2 试验方法
1. 2. 1 DNA提取 将51份西瓜种质于温室穴盘中育苗,待西瓜幼苗长出2~3片真叶时,取少量鲜嫩的叶片,用破碎仪进行破碎。采用改良的CTAB方法提取西瓜基因组DNA(李金路等,2013),用紫外分光光度计测其浓度,将提取的DNA样品稀释成30 ng/μL工作液后放置于-20 ℃保存备用。
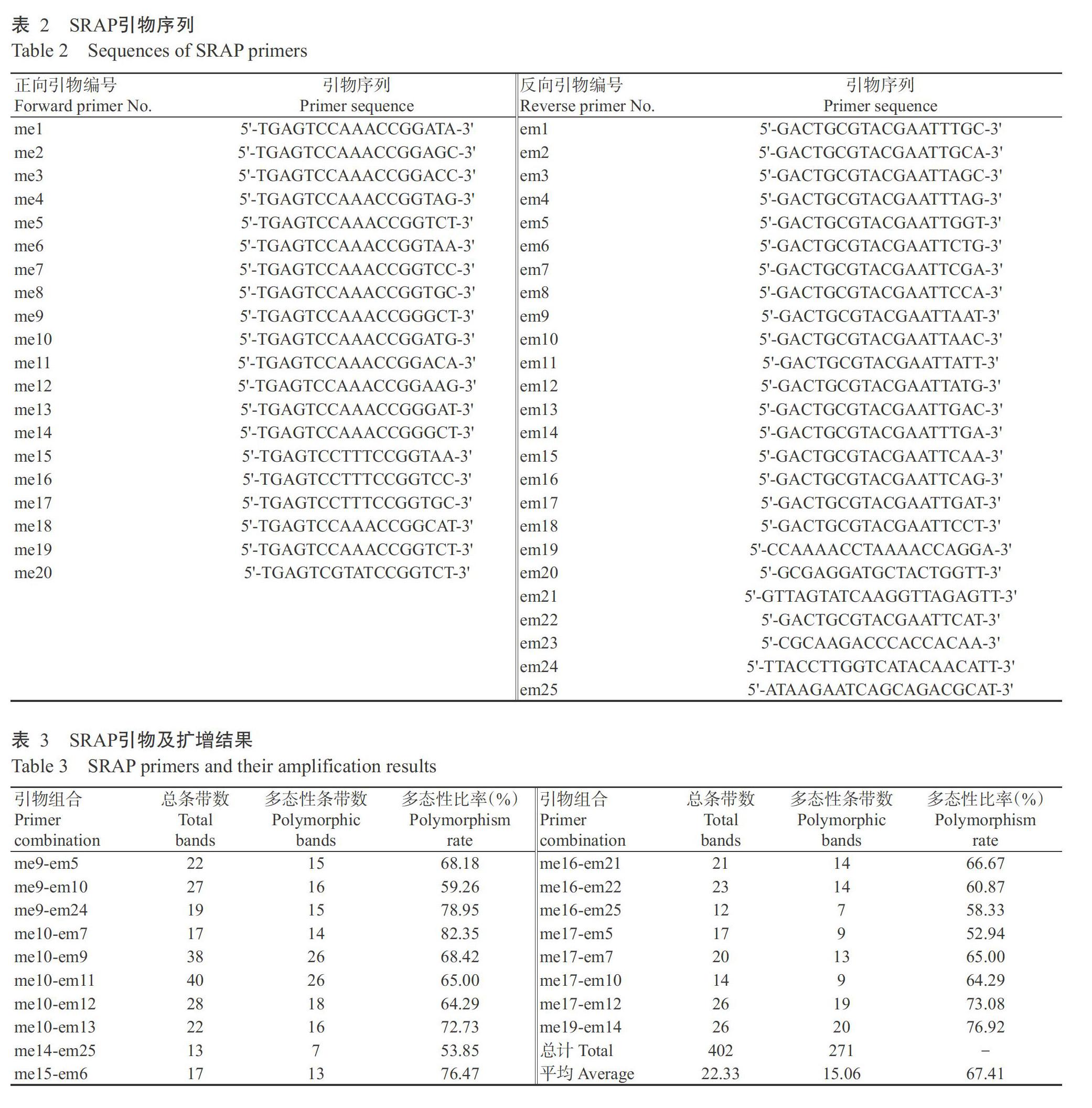
1. 2. 2 SRAP引物筛选 SRAP反应体系15.0 μL:2×Taq MasterMix 7.5 μL,30 ng/μL DNA模板1.0 μL,0.4 μmol/L正、反向引物各0.4 μL,ddH2O补足至15.0 μL,扩增程序参考黄春琼等(2015)的方法。利用抗病毒型西瓜品种野生-2和感病毒型西瓜品种18-268对500对SRAP引物进行筛选(表2),利用8%非变性聚丙烯酰胺凝胶进行电泳检测(梁宝萍等,2012),筛选出扩增条带清晰稳定且多态性丰富的引物对。
1. 2. 3 统计分析 将电泳图谱上出现条带的记为“1”,相同位置没有条带的记为“0”,形成原始数字矩阵。利用NTsys-pc 2.1e中的非加权组平均法(UPGMA)计算获得遗传相似系数,使用Treeplot模块进行聚类分析;用PopGene Version 3.2计算不同类型西瓜的遗传多样性参数,包括遗传一致度、遗传距离、观测等位基因数(Na)、有效等位基因数(Ne)、Neis基因多样性指数(H)、Shannons信息指数(I),并用Cophenetic相关性方法对聚类分析结果的准确性进行检验。
2 结果与分析
2. 1 SRAP引物筛选及多态性分析结果
选用抗病毒型西瓜品种野生-2和感病毒型西瓜品种18-268,从500对引物组合中筛选出18对扩增条带清晰稳定且扩增多态性较高的引物组合(表3)。利用筛选出的多态性引物组合对51份西瓜种质进行SRAP分子标记扩增,结果共扩增出402条清晰可辨的条带,其中多态性条带271条;平均每对引物组合可扩增出22.33条条带,其中多态性条带15.06条,平均多态性比率达67.41%。每对引物组合扩增出的条带数为13~40条;me10-em9和me10-em11引物组合扩增出的多态性条带数最多,为26条;me14-em25和me16-em25引物组合扩增出的多态性条带数最少,仅有7条。18对引物组合多态性比率为52.94%~82.35%,其中,引物组合me10-em7扩增多态性比率最高,me17-em5扩增多态性比率最低。表明筛选出的SRAP引物组合具有较好的品种鉴别能力,SRAP分子标记方法可用于西瓜种质材料的遗传多样性分析。图1为引物组合me10-em11对51份西瓜种质的扩增结果。
2. 2 西瓜抗病毒病种质资源的遗传多样性分析结果
51份西瓜种质资源按照抗病情况分为抗病毒型和感病毒型,通过软件PopGene Version 32计算不同类型西瓜的遗传多样性参数(表4),结果发现抗病毒型和感病毒型西瓜种质的Na分别为1.5224和1.5796,总体均值为1.6766;Ne分别为1.2685和1.2579,总体均值为1.3033;H分别为0.1637和0.1593,总体均值为0.1878;I分别为0.2513和0.2484,总体均值为0.2931。由此可知,供试51份西瓜种质具有较丰富的遗传多样性,除感病毒型西瓜种质的Na高于抗病毒型西瓜种质外,感病毒型西瓜种质的其他遗传多样性指数均低于抗病毒型西瓜种质,表明抗病毒型西瓜种质的遗传多样性较感病毒型西瓜种质丰富。抗病毒型与感病毒型西瓜种质的遗传一致度为0.8924,遗传距离为0.1138(表5),表明2种类型西瓜种质亲缘关系较近。
2. 3 西瓜抗病毒病种质资源的聚类分析
根据SRAP扩增图谱的矩阵,利用NTsys-pc 2.1e计算遗传相似系数,结果发现,51份供试西瓜种质的遗传相似系数为0.62~0.95(表6)。抗病毒型西瓜品种野生-2与感病毒型西瓜品种17Y6、17Y15、18-121、18-247、18-268、18-277、18-424、18-512、18-516和18-642间,以及抗病毒型野生-1与感病毒型18-512间的遗传相似系数最小,为0.62,表明这11对种质间的亲缘关系最远,同为感病毒型西瓜品种的18-516与18-602间的遗传相似系数最大,为0.95,表明二者的亲缘关系最近。
聚类分析结果(图2)显示,在遗传相似系数0.78处可将51份供试西瓜种质划分为四大类群。其中,Ⅰ类群包含2份种质,均为抗病毒型野生西瓜,Ⅱ类群包含7份种质,均为抗病毒型品种(系);Ⅲ和Ⅳ类群分别包含3和39份种质,均为感病毒型西瓜品种(系),结果表明抗病毒型与感病毒型西瓜种质的亲缘关系较远,抗病毒型野生西瓜种质与其他抗病毒型种质亲缘关系较远,感病毒型西瓜种质间的亲缘关系较近,遗传多样性较低。
利用Cophenetic相关性方法检验和评价遗传相似性系数矩阵和聚类结果转化矩阵的相关性及吻合度,结果发现2个矩阵相关系数r为0.914,说明遗传相似性数据矩阵与聚类结果转化的矩阵间呈明显相关性,聚类结果可较好地体现供试西瓜种质的遗传关系。
3 讨论
SRAP分子标记引物设计快捷简单,且稳定性好、多态性高,已广泛应用于多种作物的遗传多样性研究。本研究从500对引物组合中筛选出18对扩增条带清晰稳定且多态性较高的引物,对51份西瓜种质进行SRAP分子标记扩增,结果扩增出402条清晰可辨的条带,其中多态性条带271条,平均每对引物组合可扩增出条带22.33条,其中多态性条带15.06条,引物组合多态比率为52.94%~82.35%,平均多态比率达67.41%。Wang等(2015)利用25对SRAP引物对61份不同地域西瓜种质的遗传多样性进行研究,平均每对引物扩增条带12.5条,其中多态性条带6.5条,多态性比率52.24%,低于本研究平均每对引物扩增出的条带数、多态性条带数及多态性比率,说明本研究供试的西瓜种质多态性较丰富。但本研究测得的多态性比率低于前人利用SRAP分子标记对甘薯(张凯等,2013)(78.4%)、西瓜(Solmaz et al.,2016;Pandey et al.,2019)(97.3%、95.4%)、甜瓜(杨永等,2017)(96%)等测得的多态性比率,可能与供试材料的数量和来源地的丰富度有关。
目前Na、Ne、H和I是衡量物种群居变异程度和多态性的重要遗传多样性参数(Nei,1978)。供试的51份西瓜种质的Na、Ne、H和I分别为1.6766、1.3033、0.1878、0.2931,均高于高宁宁等(2019a)研究获得的26份抗早衰甜瓜种质基于SRAP分子标记的遗传多样性参数(1.5418、1.3007、0.1795、0.2714),表明供试西瓜种质的遗传多样性较丰富。但本研究遗传多样性参数均低于陈芸等(2016)研究获得的42份新疆甜瓜种质基于SRAP分子标记的遗传多样性参数(2.0000、1.4750、0.2876、0.4435)、郭禄芹等(2018)对167份西瓜种质基于SSR分子标记的的遗传多样性参数(4.86、2.20、0.27、0.94)。与陈芸等(2016)的结果相比,本研究供试材料多态性比率较高,但遗传多样性参数较低,可能与选择样本种类和样本来源地域丰富度有关。感病毒型西瓜种质的Na(1.5796)高于抗病毒型西瓜种质的Na(1.5224),但对于其他遗传多样性参数Ne、H和I,抗病毒型西瓜种质(1.2685、0.1637和0.2513)均高于感病毒型西瓜种质(1.2579、0.1593和0.2484),表明抗病毒型西瓜种质的遗传多样性较感病毒型西瓜种质丰富。抗病毒型和感病毒型西瓜种质的遗传一致度为0.8924,遗传距离为0.1138,说明2种类型西瓜種质亲缘关系较近,同时也表明SRAP标记能区别亲缘关系较近的种质。综上所述,供试西瓜种质的变异类型和遗传多样性丰富度不高,表明供试西瓜种质的遗传背景较狭窄单一。
明确西瓜种质资源的亲缘关系和遗传变异信息,从而有目的地选配亲本,才能加快优良品种的培育进程(罗世杏等,2018)。本研究供试西瓜种质的遗传相似系数为0.62~0.95,表明种质间遗传差异度小,其中,抗病毒型西瓜品种野生-2和感病毒型的10个西瓜种质及野生-1和抗病毒型西瓜品种18-512间的遗传相似系数最小,表明野生-1和野生-2与其他供试西瓜种质的亲缘关系较远。本研究聚类分析结果显示,可将51份供试西瓜种质资源划分为四大类群,其中Ⅰ类群包含2份种质,均为抗病毒型野生西瓜;Ⅱ类群包含7份种质,均为抗病毒型西瓜品种;Ⅲ和Ⅳ类群类群分别包含3和39份种质,均为感病型西瓜品种(系),表明抗病毒型野生西瓜与其他供试西瓜种质亲缘关系较远,与遗传相似系数分析结果一致;且抗病毒型和感病毒型西瓜种质的亲缘关系较远,遗传差异较大,感病毒型西瓜种质间的亲缘关系较近,遗传多样性较低。遗传相似系数矩阵和UPGMA聚类结果转化矩阵之间的相关系数(r=0.914)呈明显相关,说明18对SRAP引物能较好地体现供试西瓜的遗传多样性信息。
利用SRAP分子标记和关联性分析法可准确地反映供试材料间的亲缘关系和遗传差异(庞新华等,2019),从而得到科学可靠的结果,便于育种工作者科学合理地选配亲本。我国西瓜种质资源遗传背景较狭窄,应加大发掘优良抗病毒型种质资源的力度,选择遗传差异度大的杂交亲本,拓宽西瓜的遗传基础,提高抗病种质创新效率。同时,今后还应注重对野生资源和地方品种的利用,提高西瓜抗病种质资源的遗传多样性。
4 结论
筛选出的SRAP引物组合具有较好的品种鉴别能力,可用于西瓜种质的遗传多样性分析。抗病毒型西瓜种质与感病毒型西瓜种质的亲缘关系较近,遗传多样性丰富度不高,应加大发掘优良抗病毒型种质资源力度,以拓宽西瓜的遗传基础。
参考文献:
陈芸,李冠,王贤磊. 2010. 甜瓜种质资源遗传多样性的SRAP分析[J]. 遗传,32(7):744-751. doi:10.3724/SP.J.1005. 2010.00744. [Chen Y,Li G,Wang X L. 2010. Genetic diversity of a germplasm collection of Cucumis melo L. using SRAP markers[J]. Hereditas,32(7):744-751.]
陈芸,王继莲,丁晓丽,马刘峰. 2016. 新疆石榴种质资源遗传多样性的SRAP分析[J]. 西北植物学报,36(5):916-922. doi:10.7606/j.issn.1000-4025.2016.05.0916. [Chen Y,Wang J L,Ding X L,Ma L F. 2016. Genetic diversity of germplasm collection of pomegranate in Xinjiang using SRAP makers[J]. Acta Botanica Boreali-Occidentalia Sinica,36(5):916-922.]
高宁宁,常高正,康利允,李晓慧,梁慎,李海伦,王琰,徐小利,赵卫星. 2019a. 甜瓜抗早衰种质资源SRAP分子标记分析[J]. 分子植物育种,17(19):6390-6397. doi:10.13271/ j.mpb.017.006390. [Gao N N,Chang G Z,Kang L Y,Li X H,Liang S,Li H L,Wang Y,Xu X L,Zhao W X. 2019a. Use of SRAP markers to analyze anti-premature germplasm resources of melon[J]. Molecular Plant Bree-ding,17(19):6390-6397.]
高宁宁,常高正,康利允,李晓慧,梁慎,李海伦,赵卫星. 2019b. 基于SRAP标记的甜瓜耐盐种质资源遗传多样性分析[J]. 西北植物学报,39(1):68-75. doi:10.7606/j.issn. 1000-4025.2019.01.0068. [Gao N N,Chang G Z,Kang L Y,Li X H,Liang S,Li H L,Zhao W X. 2019b. Genetic diversity analysis of salt tolerance germplasm resources of melon based on SRAP[J]. Acta Botanica Boreali-Occidentalia Sinica,39(1):68-75.]
郭禄芹,赵世豪,朱华玉,胡建斌,孙守如,马长生,杨路明. 2018. 167份西瓜种质材料的遗传多样性分析[J]. 中国瓜菜,31(1):5-11. doi:10.3969/j.issn.1673-2871.2018. 01.002. [Guo L Q,Zhao S H,Zhu H Y,Hu J B,Sun S R,Ma C S,Yang L M. 2018. The genetic diversity analysis of 167 watermelon germplasms[J]. Chinese Cucurbits and Vegetables,31(1):5-11.]
韓宏伟,王浩,佘建华,王强,庄红梅,刘会芳,李宁. 2017. 利用SRAP标记快速检测西瓜杂交种子纯度[J]. 新疆农业科学,54(9):1621-1626. doi:10.6048/j.issn.1001-4330. 2017.09.007. [Han H W,Wang H,She J H,Wang Q,Zhuang H M,Liu H F,Li N. 2017. Using SRAP markers for rapid detection of Zaojia(84-24) watermelon hybrid seed purity[J]. Xinjiang Agricultural Sciences,54(9):1621-1626.]
黄春琼,刘国道,白昌军. 2015. SRAP标记在落花生属种质资源遗传多样性上的利用[J]. 基因组学与应用生物学,34(3):622-627. doi:10.13417/j.gab.034.000622. [Huang C Q,Liu G D,Bai C J. 2015. Application of SRAP markers in the genetic diversity of Arachis Linn. germplasm resour-ces[J]. Genomics and Applied Biology,34(3):622-627.]
李金璐,王硕,于婧,王玲,周世良. 2013. 一种改良的植物DNA提取方法[J]. 植物学报,48(1):72-78. doi:10.3724/SP.J.1259.2013.00072. [Li J L,Wang S,Yu J,Wang L,Zhou S L. 2013. A modified CTAB protocol for plant DNA extraction[J]. Chinese Bulletin of Botany,48(1):72-78.]
李严,张春庆. 2005. 西瓜杂交种遗传多态性的SRAP标记分析[J]. 园艺学报,32(4):643-647. doi:10.3321/j.issn:0513-353X.2005.04.015. [Li Y,Zhang C Q. 2005. Stu-dies on genetic diversity with a molecular marker SRAP of watermelon hybrids[J]. Acta Horticulturae Sinica,32(4):643-647.]
梁宝萍,原玉香,朴凤植,张晓伟,蒋武生,梁爽,姚秋菊,张强,赵艳艳. 2012. 大白菜非变性聚丙烯酰胺凝胶电泳技术的优化[J]. 河南农业科学,41(5):129-132. doi:10.3969/ j.issn.1004-3268.2012.05.033. [Liang B P,Yuan Y X,Piao F Z,Zhao X W,Jiang W S,Liang S,Yao Q J,Zhang Q,Zhao Y Y. 2012. The optimization of technique of native polyacrylamide gel electrophoresis(Native-PAGE)[J]. Journal of Henan Agricultural Sciences,41(5):129-132.]
刘洁. 2019. 西瓜抗黄瓜绿斑驳花叶病毒病遗传分析与抗性基因定位研究[D]. 杭州:浙江大学. [Liu J. 2019. Genetic analysis and gene mapping of watermelon resistance to cucumber green mottle mosaic virus[D]. Hangzhou:Zhe-jiang University.]
刘丽娟,舒烈波,罗利军,陈海荣. 2011. SRAP标记与形态学标记在西瓜DUS测试中的比较[J]. 植物遗传资源学报,12(5):790-795. doi:10.13430/j.cnki.jpgr.2011.05.027. [Liu L J,Shu L B,Luo L J,Chen H R. 2011. Comparison of morphological and SRAP markers inwatermelon(Citrullus lanatus) DUS test[J]. Journal of Plant Genetic Resources,12(5):790-795.]
罗世杏,唐玉娟,黄国弟,宋恩亮,何江,宁琳,赵英. 2018. 利用SRAP分子标记分析杧果种质遗传多样性[J]. 热带作物学报,39(12):2369-2376. doi:10.3969/j.issn.1000-2561.2018.12.007. [Luo S X,Tang Y J,Huang G D,Song E L,He J,Ning L,Zhao Y. 2018. Genetic diversity analysis of mango germplasm using SRAP markers[J]. Chinese Journal of Tropical Crops,39(12):2369-2376.]
庞新华,檀小辉,韦丽君,梁芳,张继,吕平,程琴,黄秋伟,周全光. 2019. 35份甘蔗种质的遗传多样性分析及DNA指纹图谱构建[J]. 南方农业学报,50(6):1157-1164. doi:10. 3969/j.issn.2095-1191.2019.06.0 1. [Pang X H,Tan X H,Wei L J,Liang F,Zhang J,Lü P,Cheng Q,Huang Q W,Zhou Q G. 2019. Genetic diversity analysis for 35 sugarcane germplasm resources and construction of DNA fingerprint[J]. Journal of Southern Agriculture,50(6):1157-1164.]
王凱娜. 2018. 番茄、马铃薯及西瓜病毒病的病原鉴定[D]. 北京:中国农业科学院. [Wang K N. 2018. Identification of viral pathogens from tomato,potato and watermelon[D]. Beijing:Chinese Academy of Agricultural Sciences.]
王燕,龚义勤,赵统敏,刘广,郁樊敏,叶海龙,柳李旺. 2007. 番茄SRAP-PCR体系优化与品种分子鉴定[J]. 南京农业大学学报,30(1):23-29. [Wang Y,Gong Y Q,Zhao T M,Liu G,Yu F M,Ye H L,Liu L W. 2007. Optimization of SRAP-PCR system and cultivar molecular identification in tomato(Lycopersicon esculentum L.)[J]. Journal of Nanjing Agricultural University,30(1):23-29.]
吴会杰,刘丽锋,彭斌,田莉莉,田延平,古勤生. 2009. 西瓜抗病毒RNAi植物表达载体的构建[J]. 果树学报,26(4):525-531. [Wu H J,Liu L F,Peng B,Tian L L,Tian Y P,Gu Q S. 2009. Construction of RNAi plant expression vectors in transgenic watermelon resistant to viruses[J]. Journal of Fruit Science,26(4):525-531.]
吳洋,李俊香,彭斌,古勤生. 2017. 河南、甘肃和新疆西瓜甜瓜病毒检测[J]. 中国蔬菜,(8):31-35. doi:10.ssss/j.issn. 1000-6346.2017.8.006. [Wu Y,Li J X,Peng B,Gu Q S. 2017. Detection of viruses infecting watermelon and me-lon in Henan,Gansu Provinces and Xinjiang autonomous region[J]. China Vegetables,(8):31-35.]
羊杏平,刘广,侯喜林,徐锦华,张曼. 2013. 西瓜核心种质枯萎病抗性与SRAP分子标记的关联分析[J]. 园艺学报,40(7):1298-1308. doi:10.16420/j.issn.0513-353x.2013.07. 011. [Yang X P,Liu G,Hou X L,Xu J H,Zhang M. 2013. Association analysis of fusarium wilt resistance of core collection of watermelon germplasms based on SRAP markers[J]. Acta Horticulturae Sinica,40(7):1298-1308.]
杨永,王豪杰,张学军,李寐华,伊鸿平,张永兵. 2017. 新疆甜瓜地方种质资源遗传多样性的SRAP分析[J]. 植物遗传资源学报,18(3):436-448. doi:10.13430/j.cnki.jpgr.2017. 03.008. [Yang Y,Wang H J,Zhang X J,Li M H,Yi H P,Zhang Y B. 2017. Genetic diversity analysis of melon landraces(Cucumis melo) in Xinjiang based on SRAP markers[J]. Journal of Plant Genetic Resources,18(3):436-448.]
张凯,罗小敏,蒋玉春,吴正丹,王季春,唐道彬. 2013. 甘薯种质资源的SRAP鉴定及遗传多样性分析[J]. 核农学报,27(5):568-575. doi:10.11869/hnxb.2013.05.0568. [Zhang K,Luo X M,Jiang Y C,Wu Z D,Wang J C,Tang D B. 2013. Genetic diversity among main sweetpotato germplasm resources using SRAP markers[J]. Journal of Nuclear Agricultural Sciences,27(5):568-575.]
张永兵,伊鸿平,马新力,李寐华,吴明珠. 2013. 新疆甜瓜地方品种资源核心种质构建[J]. 植物遗传资源学报,14(1):52-57. doi:10.3969/j.issn.1672-1810.2013.01.008. [Zhang Y B,Yi H P,Ma X L,Li M H,Wu M Z. 2013. Construction of core collection of melon landraces in Xinjiang[J]. Journal of Plant Genetic Resources,14(1):52-57.]
张羽,张晓娟,陈进,孙晓敏,张成兵. 2017. 43份油菜菌核病抗性资源的SCoT,SSR与SRAP标记分析[J]. 西北农林科技大学学报,45(1):45-52. doi: 10.13207/j.cnki.jnwafu.2017.01.007. [Zhang Y,Zhang X J,Chen J,Sun X M,Zhang C B. 2017. Germplasm analysis of 43 sclerotinia sclerotiorum resistant Brassica napus by SCoT,SSR and SRAP markers[J]. Journal of Northwest Scientific and Technology University of A & F,45(1):45-52.]
赵胜杰,刘文革,阎志红,何楠,包文风. 2010. 西瓜种质资源遗传差异的SRAP和EST-SSR分析[J]. 华北农学报,25(3):76-79. doi:10.7668/hbnxb.2010.03.018. [Zhao S J,Liu W G,Yan Z H,He N,Bao W F. 2010. Studies of watermelon genetic diversity using SRAP and EST-SSR[J]. Acta Agricultrue Boreali-Sinica,25(3):76-79.]
赵胜杰,刘文革,阎志红,何楠. 2009. 基于SRAP的三倍体无子西瓜品种指纹分析和杂种纯度鉴定[J]. 果树学报,26(5):687-691. doi:10.13925/j.cnki.gsxb.2009.05.018. [Zhao S J,Liu W G,Yan Z H,He N. 2009. Studies on fingerprinting of seedless triploid watermelon cultivars and hybrid purity identification by SRAP markers[J]. Journal of Fruit Science,26(5):687-691.]
Kang B C,Yeam I,Jahn M M. 2005. Genetics of plant virus resistance[J]. Annual Review of Phytopathology,43(1):581-621. doi:10.1146/annurev.phyto.43.011205.141140.
Li G,Quiros C F. 2001. Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics,103(2):455-461.
Nei M. 1978. Estimation of average heterozygosis and gene-tic distance from a small number of individuals[J]. Gene-tics,89(3):583-590.
Pandey A,Khan M K,Isik R,Turkmen O,Acar R,Seymen M,Hakki E E. 2019. Genetic diversity and population structure of watermelon(Citrullus sp.) genotypes[J]. 3 Biotech,9(6):1-14. doi:10.1007/s13205-019-1736-2.
Solmaz ?,Kacar A Y,Sari N,Imsek O. 2016. Genetic diversity within Turkish watermelon(Citrullus lanatus(Thunb.) Matsumura & Nakai) accessions revealed by SSR and SRAP markers[J]. Turkish Journal of Agriculture and Forestry,40(3):407-419. doi:10.3906/tar-1511-26.
Wang P Q,Li Q,Hu J B,Su Y. 2015. Comparative analysis of genetic diversity among Chinese watermelon germplasms using SSR and SRAP markers,and implications for future genetic improvement[J]. Turkish Journal of Agriculture and Forestry,39(2):322-331. doi:10.3906/tar-1407-29.
Xu Y,Kang D,Shi Z,Shen H,Wehner T. 2004. Inheritance of resistance to zucchini yellow mosaic virus and watermelon mosaic virus in watermelon[J]. The Journal of Heredity,95(6):498-502. doi: 10.1093/jhered/esh076.
(責任编辑 陈 燕)

