QTL-Seq定位粳稻整精米率QTL
陈喜娜 袁泽科 胡珍珍 赵全志 孙红正
QTL-Seq定位粳稻整精米率QTL
陈喜娜#袁泽科#胡珍珍 赵全志 孙红正*
(河南农业大学 农学院,郑州 450046;#共同第一作者;*通信联系人,E-mail:sunhongzheng@foxmail.com)
【目的】稻米整精米率是加工品质的重要评价指标之一,其QTL定位将为提升稻米品质提供理论依据。【方法】通过两个粒型相近、整精米率差异极大的粳稻材料构建的F2分离群体为材料,利用QTL-Seq方法对分离群体中的高整精米率单株和低整精米率单株进行混池重测序以定位粳稻整精米率QTL位点。【结果】经过QTL-Seq分析发现在第8和第12染色体区间存在控制粳稻整精米率的QTL位点。进一步通过200个F2分离群体单株对第8和12染色体进行InDel分子标记QTL作图,发现粳稻中控制整精米率的QTL位点:、和。其中,位于第8染色体21.8−23.2 Mb的表型贡献率达到10.80%,其他两个QTL的贡献率较小。位于第8染色体24.2−25.2 Mb,表型贡献率为3.26%。位于第12染色体的2.9−4.5 Mb的表型贡献率为4.06%。【结论】本研究定位了1个控制粳稻整精米率的主效QTL位点,对克隆粳稻整精米率控制基因以及在品质育种中提高粳稻整精米率有一定的参考价值。
水稻; 稻米加工品质; 整精米率; QTL定位
整精米率是稻米加工品质评价指标之一,不同品种间的整精米率有很大差异,直接影响稻米的商品价值和生产效益,因而提高整精米率对于稻米品质育种有重要意义[1]。遗传研究表明,整精米率是受多基因控制的数量性状,且易受环境影响[2]。分子标记技术的发展为复杂性状的分子改良提供了可能,迄今已利用遗传群体对稻米整精米率进行了QTL定位分析,检测到多个QTL位点[3-10]。2002年梅捍卫等[3]利用籼粳杂交重组自交系群体定位4个主效QTL,其中位于第6和第7染色体的和具有较大遗传效应。2007年刘家富等[4]利用籼稻与野生稻回交渗入系在第1染色体发现QTL位点,该QTL可解释13%的整精米率表型变异。2007年翁建峰等[2]利用具有籼稻染色体片段的粳稻染色体片段置换系(CSSL)群体在第3和第6染色体定位到主效QTL位点和,平均表型贡献率分别为34.6%和22.3%,来自籼稻亲本IR24的减效等位基因分别降低整精米率9.1%和9.0%。胡霞等[5]利用回交导入系发现6个整精米率相关的QTL,分别位于第1、2、5、10、12染色体。梅德勇等[6]利用重组自交系在第3和第5染色体定位到控制籼稻整精米率的QTL和。张上都等[7]利用籼稻和爪哇稻的重组自交系在第1染色体发现QTL,对整精米率表型贡献率为8.7%。李承欣等[8]利用旱稻和水稻的重组自交系在第6、7、8染色体上定位了5个整精米率QTL。Qiu等[9]利用272份籼稻材料进行DArTseqTM测序获取基因型信息,然后进行全基因组关联分析,定位了位于第3、4、5、6、8、12染色体的6个QTL。方雅洁等[10]利用400份籼稻种质资源材料的芯片检测高密度SNP基因型进行的全基因组关联分析发现4个整精米率QTL,分别位于第2、6、7、11染色体,其表型贡献率为6.5%~7.9%。
近年来随着测序技术的发展,重要农艺性状QTL因测序技术的应用而得以快速定位,仅在水稻中就有大量QTL及基因使用QTL-Seq方法进行了定位和克隆[11-13]。前人的研究中,整精米率QTL位点大多是籼粳杂交或籼稻全基因组关联分析定位的QTL,粳稻基因组背景下整精米率的QTL位点少有研究。在我们前期的资源筛查中发现,有两个粳稻种质资源拉木加和水晶3号在花期、籽粒大小、长宽比等性状上均差异不大,而两者的整精米率差异较大。因此,我们利用这两个材料构建了F2分离群体并采用QTL-seq方法对分离群体的极端个体进行分池测序,对分析得到的基因组区域利用传统QTL定位方法进行验证,最终发现了位于第8染色体与整精米率相关的主效QTL。本研究对克隆粳稻整精米率控制基因以及提高粳稻整精米率品质育种有一定的参考意义。
1 材料与方法
1.1 材料
以粳稻材料水晶3号为父本,拉木加为母本进行杂交得到F1代,F1代自交得到的F2群体单株以及两亲本材料于2019年种植于河南农业大学原阳试验基地(N35°11′, E113°94′),常规大田管理,并于拔节期取亲本和F2单株叶片用于提取DNA。
1.2 单株整精米率表型分析及QTL-Seq测序
收获的F2群体459个单株籽粒晾晒后于室温条件下放置3个月后脱粒脱壳,采用小型碾米机使用相同碾磨时间碾成精米,根据大于完整精米平均长度3/4的整精米占稻谷比例计算整精米率。根据单株整精米率挑选整精米率小于30%和大于64%的两端极端个体单株各25株,CTAB法提取极端个体单株DNA后等量混合成高整精米率池和低整精米率池,混池及两亲本DNA分别建库采用Illumina NovaSeq6000测序仪2×150 bp双端重测序。测序数据去除低质量序列数据以后,使用BWA和GATK软件对序列进行比对和SNP读取[14-15]。QTL位点搜索采用QTL-seqr R软件包中的ΔSNP指数和ED (Euclidean distance)方法进行计算[16]。
1.3 局部的遗传图谱构建及QTL分析验证
根据重测序结果选取水晶3号与拉木加之间有多态性的InDel标记,选用200个F2群体单株进行作图验证。将硅胶干燥保存的亲本及F2群体单株叶片采用CTAB法提取DNA,PCR扩增后以6%聚丙烯酰胺凝胶电泳分离片段读取基因型。多态InDel标记与整精米率的连锁分析使用QTL IciMapping软件进行[17]。
2 结果与分析
2.1 水晶3号与拉木加及F2群体的整精米率表型
粳稻材料亲本水晶3号连续两年的整精米率都在75%左右,而另一粳稻材料亲本拉木加的整精米率连续两年都在50%左右,整精米率年际间表现较为稳定(图1-A)。两者均为粳稻,在粒形上也差异不大。水晶3号与拉木加精米长度分别为4.82、4.88 mm,宽度分别为2.72、2.80 mm,长宽比分别为1.8、1.7。F2群体的整精米率分布于11.2%~71.7%,中值偏向于高整精米率(图1-B)。
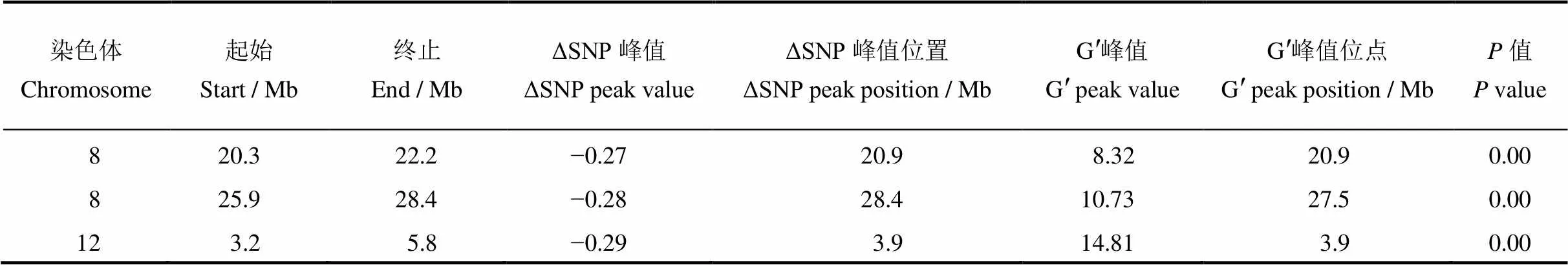
表1 QTL-Seq检测到的控制整精米率的QTL区间
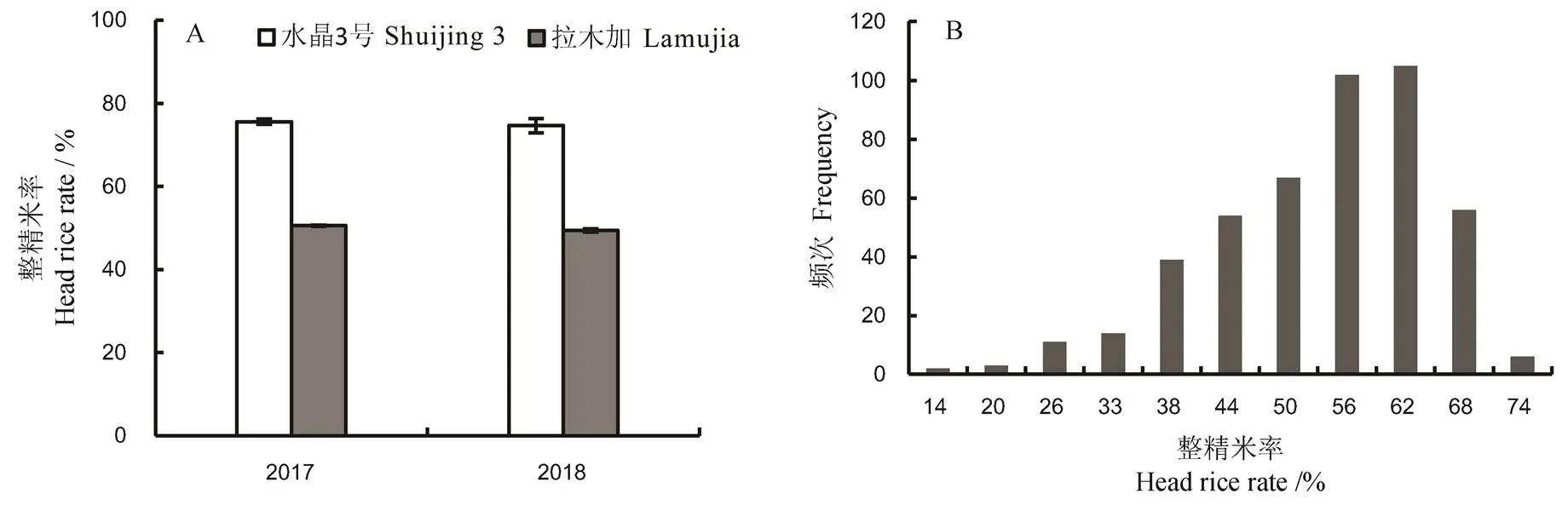
图1 亲本整精米率表型(A)及F2群体(B)整精米率频次分布
Fig. 1. Phenotype of head rice rates of parents(A) and histogram distribution of the F2population(B).
2.2 QTL-seq分池重测序定位整精米率QTL位点
对F2群体中整精米率分布在两端的极端个体各25个单株进行混池重测序,高整精米率混池25个单株的整精米率均在64%以上,低整精米率混池的25个单株整精米率均在30%以下。全基因组重测序中两亲本测得的数据量分别为24.1 Gb和23.8 Gb,测序深度分别为65倍、64倍。高低混池分别得到19.46 Gb和19.47 Gb高质量碱基序列,基因组覆盖深度约为51倍。经过序列比对和SNP位点提取,将两个极端混池共得到的SNP和InDel位点与两亲本进行比较,过滤掉不一致的位点后,共有1 178 288个SNP和InDel位点用于QTL-Seq分析。经过R软件包QTLseqr分析,采用ΔSNP指数法没有得到统计上显著的QTL,而使用ED方法分析后,在第8、12染色体上共得到3个QTL,第8染色体检测到2个QTL,第12染色体检测到1个QTL(表1),其中第12染色体上检测到的QTL位点ED峰值较高(图2-B)。进一步对全基因组12条染色体上测序深度超过50次的SNP和InDel位点在高低混池的SNP指数进行作图,发现第8和第12染色体上均有较长的染色体区间呈现出高整精米率混池的SNP指数高于低整精米率混池的,与QTL-seq分析结果较为一致(图3)。因此,进一步针对第8和第12染色体设计多态性引物,采用传统QTL定位方法进行验证。
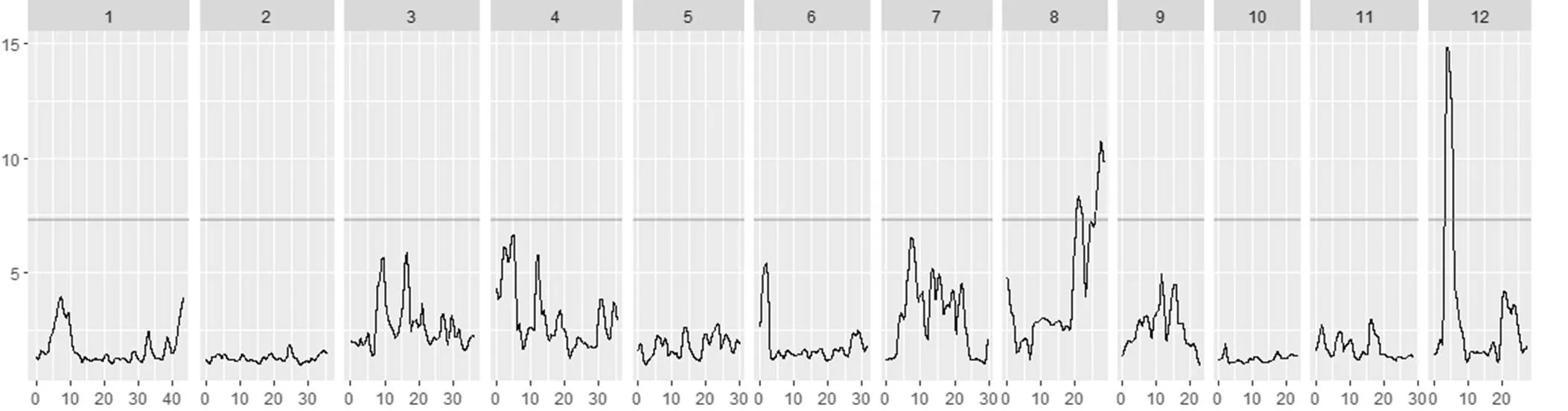
灰色横线代表95%置信区间,横轴为染色体位置(Mb),纵轴为ED值。
Fig. 2. Results of the QTL-seq analysis.
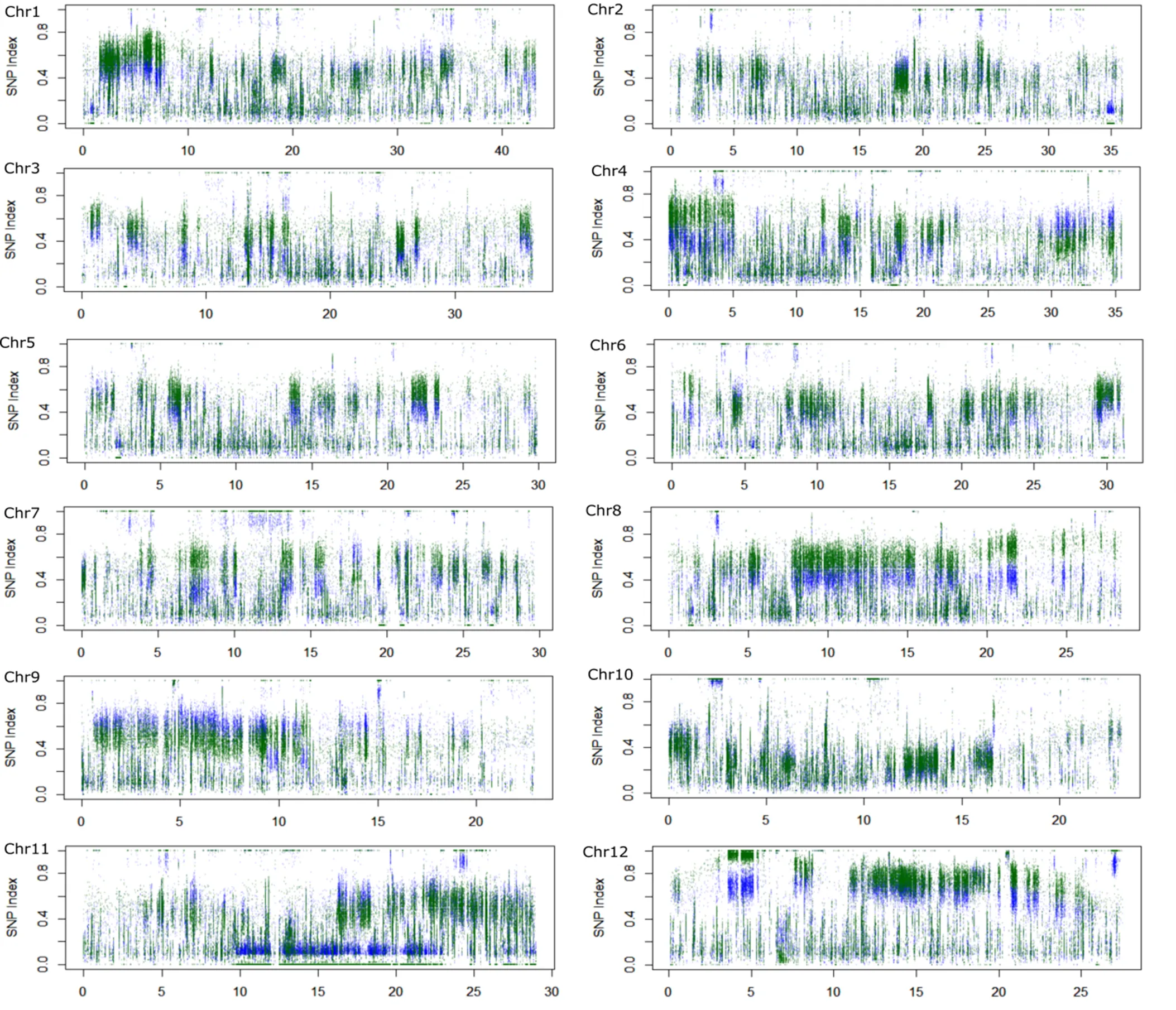
蓝色点代表高整精米率混池SNP指数,绿色点代表低整精米率混池SNP指数。纵轴为SNP指数,横轴为染色体位置(Mb)。
Fig. 3. SNP-index of all the polymorphic sites on 12 chromosomes with sequencing depth greater than 50.
2.3 第8和第12染色体整精米率QTL位点分子标记定位验证
根据全基因组重测序结果得到的InDel位点在第8和第12染色体设计引物,共获得有多态性的引物26对(表S1),其中第8染色体14对,第12染色体12对,引物覆盖整条染色体,引物间物理距离为1~4 Mb。利用这些多态性InDel标记对200个F2群体单株进行基因型鉴定,基因型与表型的连锁作图发现在第8染色体和第12染色体检测到3个QTL,其位置与QTL-seq分析得到的QTL位点一致。其中第8染色体检测到的位于21.8−23.2 Mb区间,LOD值5.11,对整精米率表型贡献率为10.80%。位于24.2−25.2 Mb区间,LOD值2.15,对整精米率表型贡献率为3.26%。第12染色体检测到的QTL位于2.9−4.5 Mb区间,LOD值2.61,表型贡献率为4.45%(表2,图4)。
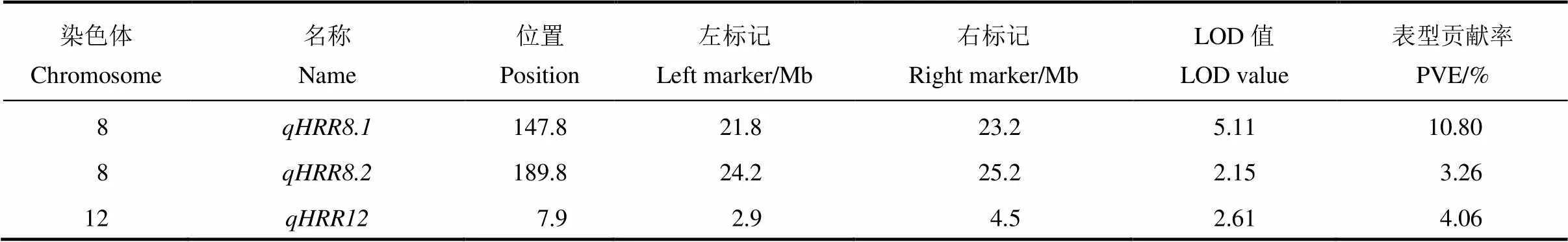
表2 第8和第12染色体定位到的整精米率QTL
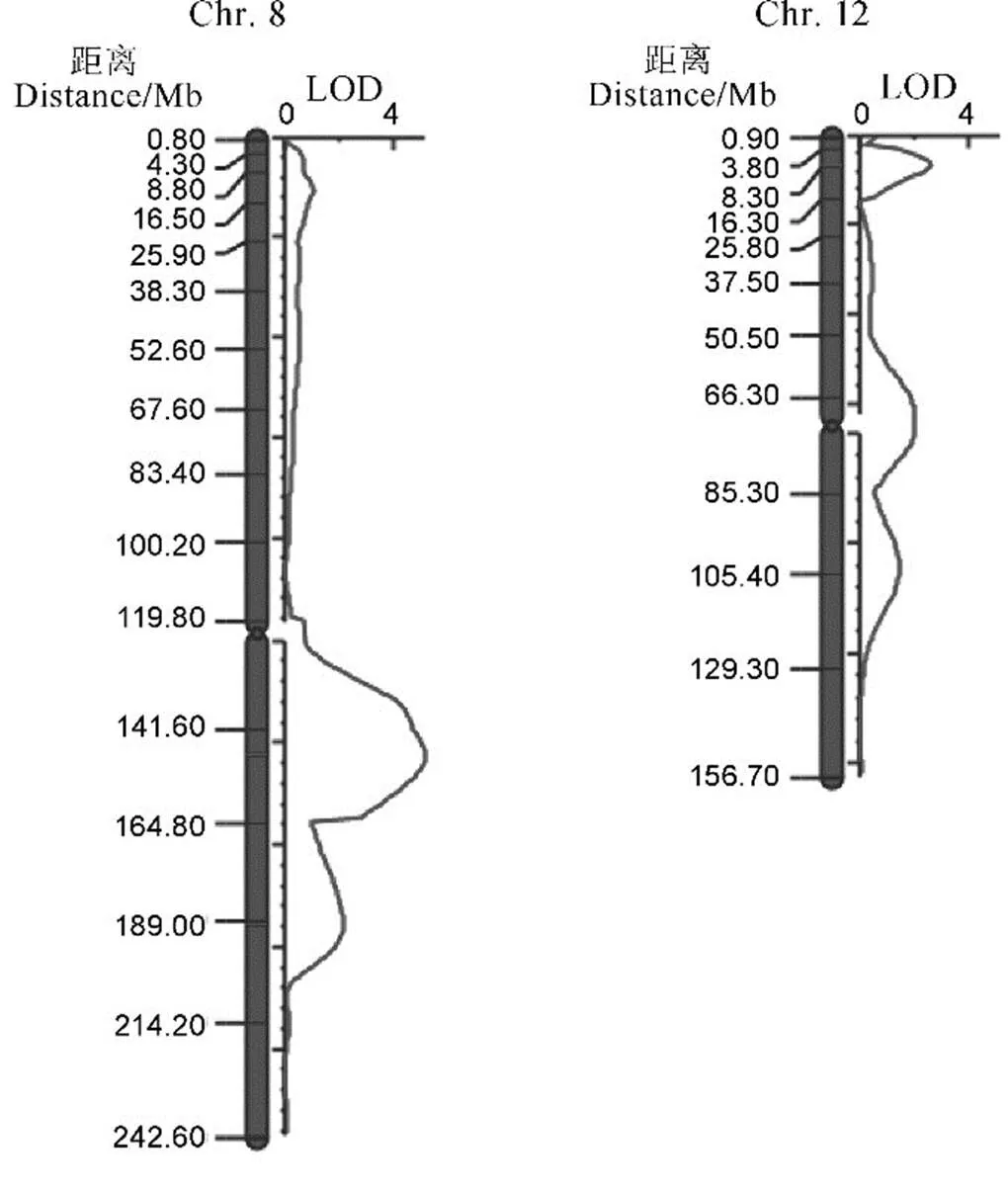
图4 整精米率QTL位点第8和第12染色体定位结果
Fig. 4. Results of head rice rate QTL mapping on chromosomes 8 and 12.
3 讨论
稻米整精米率是重要的加工品质性状,除了环境、水分含量、加工方式等影响因素外,遗传因素是最重要的影响因素,不同粳稻种质资源材料之间的整精米率差异极大,整精米率可低至18%,高整精米率材料可达到73%[18]。稻米粒形与整精米高度相关,长宽比较大的稻米在碾磨过程中较易产生碎米,而粒形短圆的稻米则不易产生碎米,因此在对全国五千余份稻谷样品进行品质分析时发现,粳稻的平均整精米率比籼稻高19%左右[19]。而前人在对整精米率QTL进行定位多是采用籼粳杂交构建分离群体,而籼稻的粒形细长,粒长及长宽比一般比粳稻大,因此不能排除粒形对整精米率的影响,而本研究中所用的两个亲本材料均为粳稻,籽粒大小及长宽比接近,因此,相对于籼粳杂交,粳粳杂交构建的分离群体更具优势。
随着测序成本的降低,QTL-Seq方法定位数量性状QTL在作物研究中被广泛应用,在本研究中,对整精米率的QTL-Seq分析中采用了两种方法进行分析,即ΔSNP指数和ED方法[12, 20]。在使用ΔSNP指数方法进行分析时,虽然也有染色体区段的ΔSNP指数偏离0假设,但是均未达到统计上的显著水平,而在使用ED方法进行分析时发现有3个QTL的ED值达到显著水平。后经实验验证发现表型贡献率最大的所处位置的ED值峰值却不是最高的,而第8染色体另外一个贡献率较小的定位的位置与ED方法得到的结果也差别较大。虽然QTL-Seq定位的QTL位置存在偏差,但是由于高通量测序的便利性,该方法可快速定位目标QTL的区间,为进一步的精细定位提供良好的基础,因此该方法在数量性状QTL位点的定位和克隆中得到了广泛应用。
本研究中发现的整精米率主效QTL位点、位于第8染色体21.3−25.2 Mb区间,之前报道的整精米率QTL位点中,Qiu等[8]通过GWAS发现的QTL位点位于第8染色体2.6 Mb附近,与、相距较远。李承欣等[7]利用陆稻与粳稻的RIL群体定位的整精米率QTL位点位于第8染色体RM1345−RM1384区间,相当于第8染色体12−26 Mb区间,但由于区间过大,无法确定是否与本研究中的QTL位点为同一位点。位于第12染色体2.9−4.5 Mb区间,之前的报道仅Qiu等[9]发现的位点位于第12染色体,但该QTL位点位于26 Mb附近,与本研究中发现的位点明显不同。本研究中发现的控制粳稻整精米率的QTL位点对于整精米率控制基因的克隆及粳稻品质育种有一定的参考价值。
在线辅助性信息:有1个辅助性表S1放在《中国水稻科学》网上,网址为http://www.ricesci.cn。
[1] Xu Q, Chen W F, Xu Z J. Relationship between grain yield and quality in rice germplasms grown across different growing areas[J]., 2015, 65(3): 226-232.
[2] 翁建峰, 万向元, 郭涛, 江玲, 翟虎渠, 万建民. 利用CSSL群体研究稻米加工品质相关QTL表达的稳定性[J]. 中国农业科学, 2007, 40(10): 2128-2135.
Weng J F, Wan X Y, Guo T, Jiang L, Zhai H Q, Wan J M. Stability analysis of QTL for milling quality of rice (L) using CSSL population[J]., 2007, 40(10): 2128-2135. (in Chinese with English abstract)
[3] 梅捍卫, 罗利军, 郭龙彪, 王一平, 余新桥, 应存山, 黎志康. 水稻加工品质数量性状基因座(QTLs)分子定位研究[J]. 遗传学报, 2002, 29(9): 791-797.
Mei H W, Luo L J, Guo L B, Wang Y P, Yu X Q, Ying C S, Li Z K. Molecular mapping of QTLs for rice milling yield traits[J]., 2002, 29(9): 791-797. (in Chinese with English abstract)
[4] 刘家富, 奎丽梅, 朱作峰, 谭禄宾, 王桂娟, 黎其万, 束继红, 孙传清. 普通野生稻稻米加工品质和外观品质性状QTL定位[J]. 农业生物技术学报, 2007, 15(1): 90-96.
Liu G F, Kui L M, Zhu Z F, Tan L B, Wang G J, Li J M, Shu J H, Sun C Q. Identification of QTLs associated with processing quality and appearance quality of common wild rice (Griff.)[J], 2007, 15(1): 90-96. (in Chinese with English abstract)
[5] 胡霞, 石瑜敏, 贾倩, 徐琴, 王韵, 陈凯, 孙勇, 朱苓华, 徐建龙, 黎志康. 影响水稻穗部性状及籽粒碾磨品质的QTL及其环境互作分析[J]. 作物学报, 2011, 37(7): 1175-1185.
Hu X, Dan Y M, Jia Q, Xu Q, Wang Y, Chen K, Sun Y, Zhu L H, Xu J L, Li Z K. Analyses of QTLs for rice panicle and milling quality traits and their interaction with environment[J]., 2011, 37(7): 1175-1185. (in Chinese with English abstract)
[6] 梅德勇, 朱玉君, 樊叶杨. 籼稻稻米碾磨与外观品质性状的QTL定位[J]. 遗传, 2012, 34(12): 1591-1598.
Mei D Y, Zhu Y J, Fan Y Y. Mapping QTL for rice milling and appearance quality traits in indica rice[J]., 2012, 34(12): 1591-1598. (in Chinese with English abstract)
[7] 张上都, 彭强, 张大双, 吴健强, 杨林, 朱速松. 利用RIL群体进行稻米加工品质QTL定位分析[J]. 贵州农业科学, 2016, 44(6): 8-10.
Zhang S D, Peng J, Zhang D S, Wu J J, Yang L, Zhu S S. QTL analysis for milling quality of rice by using RIL population[J]., 2016, 44(6): 8-10. (in Chinese)
[8] 李承欣, 王敬国, 刘化龙, 孙健, 王江旭, 赵宏伟, 邹德堂. 水、旱条件下稻米品质相关性状的QTL定位及其与环境互作分析[J]. 农业生物技术学报, 2016, 24(10): 1491-1499.
Li C X, Wang J G, Liu H L, Sun J, Wang J X, Zhao H W, Zou D T. QTL mapping and QTL×environment interaction analysis of grain quality-related traits in rice() under water-and dry-cultivation conditions[J]., 2016, 24(10): 1491-1499. (in Chinese with English abstract)
[9] Qiu X, Pang Y, Yuan Z, Xing D, Xu J, Dingkuhn M, Li Z, Ye G. Genome-wide association study of grain appearance and milling quality in a worldwide collection of indica rice germplasm[J]., 2015, 10(12): e0145577.
[10] 方雅洁, 朱亚军, 吴志超, 陈凯, 申聪聪, 石英尧, 徐建龙. 全基因组关联定位籼稻种质资源外观和加工品质QTL[J]. 作物学报, 2018, 44(2): 32-42.
Fang Y J, Zhu Y J, Wu Z C, Chen K, Shen C C, Dan Y Y, Xu J L. Genome-wide association study of grain appearance and milling quality in a worldwide collection of indica rice germplasm[J]., 2018, 44(2): 32-42. (in Chinese with English abstract)
[11] Nguyen K L, Grondin A, Courtois B, Gantet P. Next-generation sequencing accelerates crop gene discovery[J]., 2019, 24(3): 263-274.
[12] Takagi H, Abe A, Yoshida K, Kosugi S, Natsume S, Mitsuoka C, Uemura A, Utsushi H, Tamiru M, Takuno S. QTL-seq: Rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations[J], 2013, 74(1): 174-183.
[13] Zegeye W A, Zhang Y, Cao L, Cheng S. Whole genome resequencing from bulked populations as a rapid QTL and gene identification method in rice[J], 2018, 19(1): 4000.
[14] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J], 2009, 14(1): 1754-1760.
[15] Mckenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M. The genome analysis toolkit: A mapreduce framework for analyzing next-generation DNA sequencing data[J], 2010, 20(9): 1297-1303.
[16] Mansfeld B N, Grumet R. QTLseqr: An R package for bulk segregant analysis with next-generation sequencing[J], 2018, 11(2): 1-5.
[17] Lei M, Li H, Zhang L, Wang J. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations[J]., 2015, 3(3): 269-283.
[18] 胡珍珍, 陈喜娜, 袁泽科, 杜彦修, 张静, 李俊周, 彭廷, 孙红正, 赵全志. 135份粳稻种质资源在河南省沿黄稻区的品质性状表现及相关分析[J]. 河南农业科学, 2020, 49(8): 6-15.
Hu Z Z, Chen X N, Yuan Z K, Du Y X, Zhang J, Li J Z, Peng T, Sun H Z, Zhao Q Z. Grain quality evaluation and correlation analysis 135 ricegermplasm in Henan Huanghe River rice planting area[J], 2020, 49(8): 6-15. (in Chinese with English abstract)
[19] 罗玉坤, 朱智伟, 陈能, 段彬伍, 章林平. 中国主要稻米的粒型及其品质特性[J]. 中国水稻科学, 2004, 18(2): 135-139.
Luo Y K, Zhu Z W, Chen N, Duan B W, Zhang L P. Grain types and related quality characteristics of rice in China[J], 2004, 18(2): 135-139. (in Chinese with English abstract)
[20] Magwene P M, Willis J H, Kelly J K, Siepel A. The statistics of bulk segregant analysis using next generation sequencing[J], 2011, 7(11): e1002255.
QTL-Seq Mapping of Head Rice Rate QTLs inRice
CHEN Xina, YUAN Zeke, HU Zhenzhen, ZHAO Quanzhi, SUN Hongzheng*
(College of Agronomy, Henan Agricultural University, Zhengzhou 450046, China;#These authors contributed equally to this work;*Corresponding author, E-mail: sunhongzheng@foxmail.com)
【Objective】Rice milling quality is largely measured by head rice rate. So QTL mapping for head rice rate will lay a theoretical basis forrice quality improvement.【Method】Using an F2segregated population from tworice cultivars with similar grain shape but different head rice rates, QTL-Seq bulked segregated sequencing method was used to locate the QTLs controlling head rice rate inrice. 【Result】 Three QTLs were found on chromosomes 8 and 12 by QTL-Seq analysis, and the target regions were further verified by traditional QTL mapping method using 200 F2individuals. Of the three QTLs,was located at 21.8−23.2Mb with 10.80% phenotype variation contribution. The QTLlocated on chromosome 8 at 24.2−25.2 Mb andlocated on chromosome 12 at 2.9−4.5 Mb had smaller contributionof 3.26% and 4.06%, respectively. 【Conclusion】The head rice rate controlling QTLwas mappedon chromosome 8, which lay a theoretical foundation for further clone of head rice rategenes, and grain quality improvement inrice.
rice; grain milling quality; head rice rate; QTL mapping
10.16819/j.1001-7216.2021.201105
2020-11-16;
2021-02-05。
国家重点研发计划资助项目(2017YFD0300100);河南省现代农业产业技术体系项目(S2012-04-G02)。

