AFEchidna—动植物育种数据遗传评估分析的R程序包*
肖丽娜 魏瑞研 谭芷茵 张卫华 林元震
(1.广东省森林培育与保护利用重点实验室/广东省林业科学研究院,广东 广州 510520;2.华南农业大学/广东省森林植物种质创新与利用重点实验室,广东 广州 510642)
混合线性模型(mixed linear model)被广泛应用于动植物子代测试数据的分析,包括树木在内[1-2]。混合线性模型是指含有固定效应和随机效应的线性模型,来解释目标性状的变异程度,例如奶牛产奶量或林分材积增长量。育种者的主要兴趣是预测某种动物或作物特定基因型在环境中的未来表现,他们往往将影响目标性状的潜在遗传因素视为待预测的随机效应。相比之下,试验地点或试验地点内的重复一般视为固定效应[3]。因此,混合线性模型非常适合于遗传数据分析,在预测随机遗传效应时通过固定效应来调整表型数据。现在多数遗传分析软件采用限制性最大似然(Restricted Maximum Likelihood, REML)法来估算随机效应的方差分量,进而估计固定效应和预测随机效应。
几乎所有的动植物遗传改良项目都广泛使用子代测定试验。在子代试验数据分析中,育种者主要关注加性效应。在一个给定的群体中,加性效应是亲本可以稳定遗传给子代的效应,因此,将加性方差与表型方差的比例定义为性状的狭义遗传力(h2)。加性方差和遗传力的数值大小对遗传增益有着较大影响[4-5]。现在的子代测定数据,往往具有谱系数据、表型数据以及标记数据,专业遗传分析软件可基于谱系数据形成A 矩阵或基于标记数据形成G 矩阵,用于方差分量和随机效应BLUP 的估计[6-9]。这类软件包括ASReml[10]、Echidna[11]、BLUPf90[12]和R 程 序 包sommer[13]、breedR[14]等。上述软件中的免费软件,存在功能有限或者用法复杂。因此,本文的目的是提供一个免费的R 程序包AFEchidna,并以半同胞家系测定数据为例,演示如何使用该包基于混合线性模型产生方差分量、遗传参数的估计,BLUP 值与准确性的估算,随机效应显著性的检验,以及单性状的批量分析。
1 材料与方法
1.1 开发背景
对于植物试验来说,尤其是林木试验,由于试验环境因子比较复杂,因此需要专业遗传分析软件可以拟合复杂的方差结构[15-18]。目前,商业软件ASReml 被公认为植物遗传评估的先锋软件,但其费用昂贵。breedR 包虽然是针对林木试验开发的,但其版本偏旧且方差结构类型偏少,难以满足林木试验的分析需求。Echidna 是ASReml 主要研发者Gilmour 教授于2018 年开发的免费软件,其也是使用REML 法估算参数值,功能非常接近ASReml,是目前动植物遗传评估免费软件中功能最强大的软件,但其用法稍复杂,一般用户难以适应。因此,我们使用R 语言基于Echidna 软件开发AFEchidna 包,降低用户进行育种数据遗传分析的使用难度,另外,该包不仅保留了Echidna软件的专业遗传评估功能,还可扩展使用R 语言强大的数据管理、统计分析与图形绘制功能。
1.2 AFEchidna 包开发策略
Echidna 单机版通过.es 程序进行混合线性模型分析,并生成一系列结果文件。但其语法稍复杂,结果文件数较多,且数据处理和可视化功能较弱,一般用户使用不太方便。因此,我们使用R 语言开发一个程序包,通过该包来辅助Echidna软件,让代码运行和结果提取更直观和更便利。
开发思路分3 个层次:第一层次是数据和es0模版程序的处理,主要是各类数据读取和es0 模版程序生成,其通过get.es0.file()函数来完成;第二层次是混合线性模型的指定与运行,主要是将指定的混合线性模型通过R 语言传递给Echidna 运行,然后将生成的结果传回到R 语言中,其通过主函数echidna()来实施,传回的结果存为R 对象object;第三层次是模型结果的提取与运用,通过各种函数从R 对象object 中提取相应结果,例如方差分量、模型解等,或者基于R 对象object 做进一步的遗传参数计算或模型比较等。
图1 展示了AFEchidna 包的运行流程。AFEchidna 包通过主函数echidna()结合模板程序.es0、表型数据、谱系甚至标记关系矩阵等文件进行混合线性模型分析,将运行结果存为R 对象object,然后通过函数Var()提取方差分量、coef()获取模型解、predict()获取模型预测值、pin()计算遗传参数值、update()运行新模型和model.comp()比较不同模型。表1 列出了AFEchidna 包隶属函数所需的R 依赖包、功能和简易用法。

图1 AFEchidna 包的运行流程Figure 1 The running process of AFEchidna package

表1 AFEchidna 包隶属函数的汇总Table 1 The summary of listed functions in AFEchidna package
1.3 AFEchidna 的在线安装
我们已将AFEchidna 包上传至github 官网,并免费对外开放。感兴趣的读者可通过在R 语言 中 运 行remotes::install_github(‘yzhlinscau/AFEchidna’)来安装程序包AFEchidna,然后运行AFEchidna::checkPack()检测R 依赖包是否安装,如R 依赖包未安装,将自动进行安装。首次使用Echidna 或AFEchidna,需要先进行软件注册,具体请见AFEchidna 包github 官网主页(https://github.com/yzhlinscau/AFEchidna)。
1.4 示例数据
数据来源于文献[2]中的例4.5,该数据集为来自4 个种源的某松树36 个半同胞家系测定数据,试验设计为完全随机区组设计,5 个区组,每个小区中测量2~6 棵树,测量性状为树高height、胸径diameter 和材积volume。
1.5 统计方法
以树高为示例性状,假定混合线性模型如下:

狭义单株遗传力公式如下:

其中,h2是狭义单株遗传力,Va是加性方差,Vp是表型方差,Vf是母本方差,Vbf是区组与母本互作方差,Ve是误差方差。
母本育种值及其准确性公式如下:

其中,bv是母本育种值,gca 是母本一般配合力,μ是总体均值;r是母本育种值准确性,SE 是母本一般配合力的标准误差,Vf是母本方差。
上述统计分析将采用我们开发的AFEchidna,并与ASReml 软件结果[2]进行比较。
2 结果与分析
2.1 方差分量与遗传力的估计
AFEchidna 包和ASReml 软件估计的方差分量如表2 所示,包括母本方差Vf等四个方差分量及其标准误差的估计值均一致,具体地,区组方差Vb为0.107,母本方差Vf为0.190,区组与母本互作方差Vbf为0.198,误差方差Ve为2.527,且各项方差值(除了区组外)均大于其标准误差值的1.5倍,说明这两个软件估计的方差分量准确性高。基于上述方差分量进一步估算的单株遗传力等遗传参数结果(表3)显示AFEchidna 包和ASReml软件的结果也完全一致,且估算的加性方差、表型方差与遗传力值均大于其标准误差值的1.5 倍。这表明AFEchidna 包估计混合线性模型的方差分量值等效于ASReml 软件。
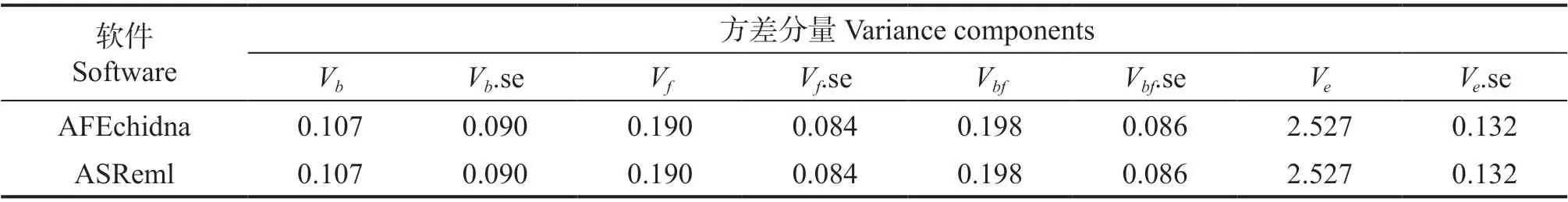
表2 AFEchidna 和ASReml 估计的方差分量Table 2 The estimation of variance components from AFEchidna and ASReml
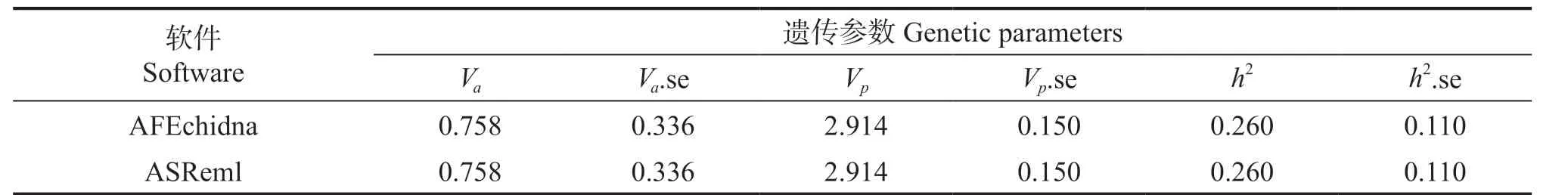
表3 AFEchidna 和ASReml 估算的遗传参数Table 3 The estimation of genetic parameters from AFEchidna and ASReml
2.2 固定效应估计
本研究中,固定效应包含总体均值和种源Prov,它们的无偏估计值如表4 所示,在AFEchidna 中,将总体均值固定为0,直接给出种源各个水平的效应值;在ASReml 中,将种源首个水平的效应值固定为0,然后求解出总体均值和种源其它水平值。由此可见,AFEchidna 和ASReml 对于固定效应的求解方法稍有不同,结果也稍有差异。不过,ASReml 得到的种源效应值是相对值,当它们加上总体均值后,与AFEchidna 的结果基本一致。

表4 固定效应的估计Table 4 The estimate of fixed effects
2.3 母本育种值及其准确性
如表5 所示,罗列了前6 个母本的一般配合力、育种值及其准确性,可以看出AFEchidna 和ASReml 得到的母本一般配合力及其标准误差结果是一致的,总体均值稍有差异,但其标准误差也是相同的。由于总体均值的轻微不同,导致估计的母本育种值在两软件间也稍有不同,但它们准确性结果是一致的,不过两软件估计育种值之间的相关达到了0.999(P<.001, 图2),因此,可以认为AFEchidna 和ASReml 估计的母本育种值也是等效的。
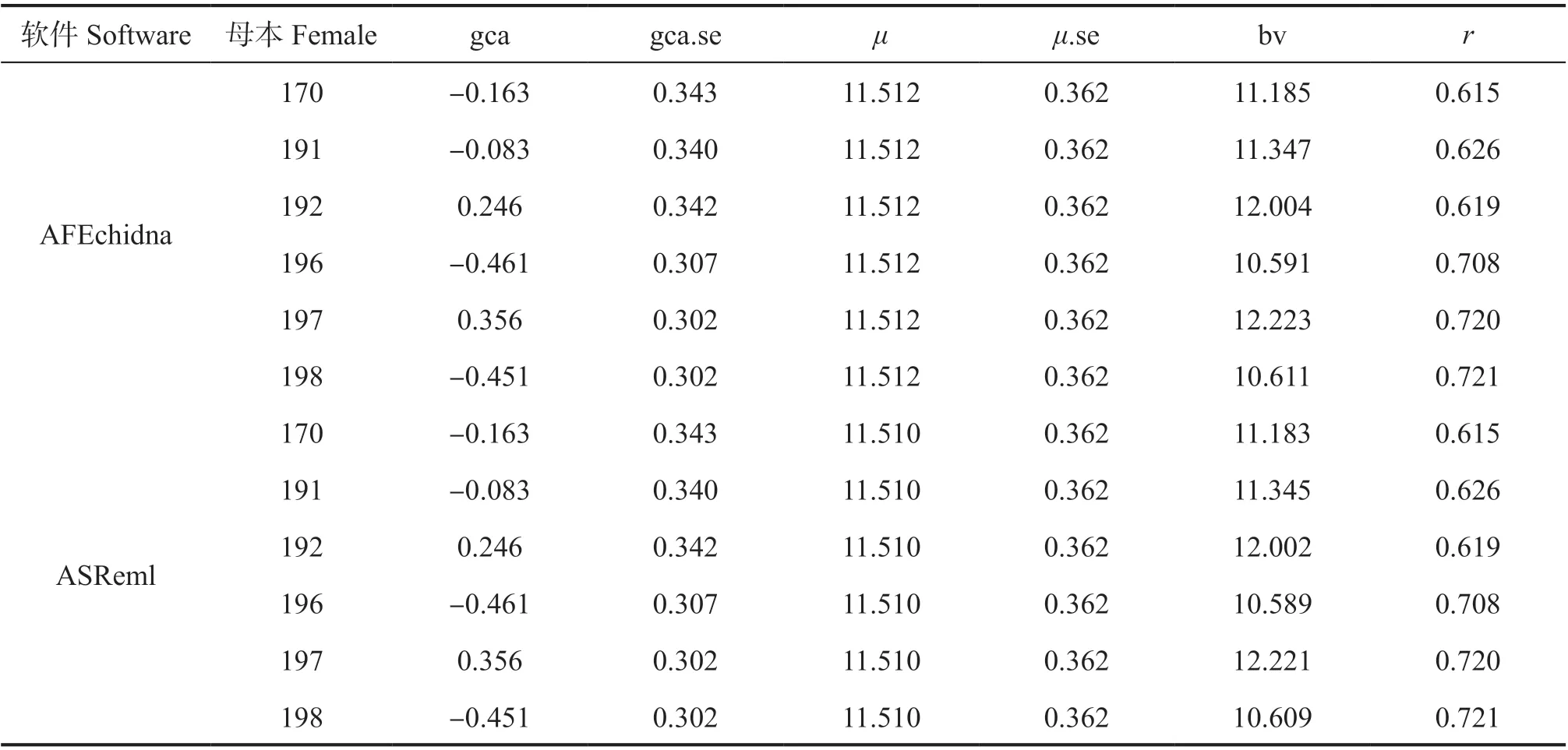
表5 估计的前6 个母本育种值及其准确性Table 5 The first six female breeding values and their accuracy

图2 来自ASReml 和AFEchidna 估计的母本育种值的散点图Figure 2 Scatter plot of female breeding values from ASReml and AFEchidna
2.4 随机效应的显著性检验
将本研究的混合线性模型视为全阶模型,其随机效应含有区组效应Block、母本效应Female以及区组与母本互作效应BlockFemale,依次删除它们中的一项作为降阶模型,然后采用LRT 法来检验相应随机效应的显著性[2]。AFEchidna 包中的model.comp()函数可以进行LRT 检验。LRT 检验结果(表6)显示,区组效应Block、母本效应Female 以及区组与母本互作效应BlockFemale 对树高的影响均为极显著(P<0.01)。
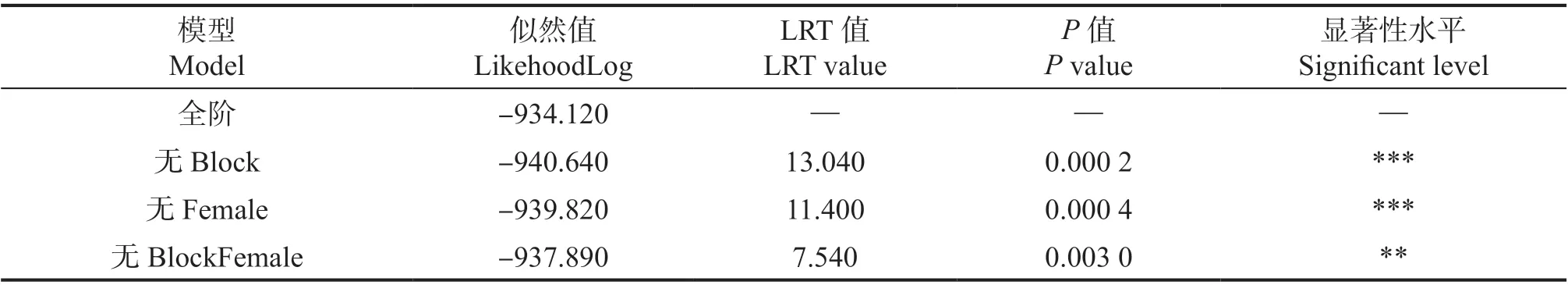
表6 随机效应的显著性检验Table 6 Significant test for random effects
2.5 性状批量分析
所谓批量分析即混合线性模型中,固定效应、随机效应和残差效应均相同条件下,将待分析的多个性状依次传入程序中进行分析并输出结果,其可是单性状的批量运行,也可是多性状的批量运行。尤其是待分析的性状数较多时,通过批量分析可大大减少分析工作量。AFEchidna 包中的主函数echidna()可通过参数batch=TRUE 来实现性状批量分析。本研究中,采用同样的混合线性模型,对树高height、胸径diameter 和材积volume做了单性状的批量分析,得到的方差分量结果如表7 所示。
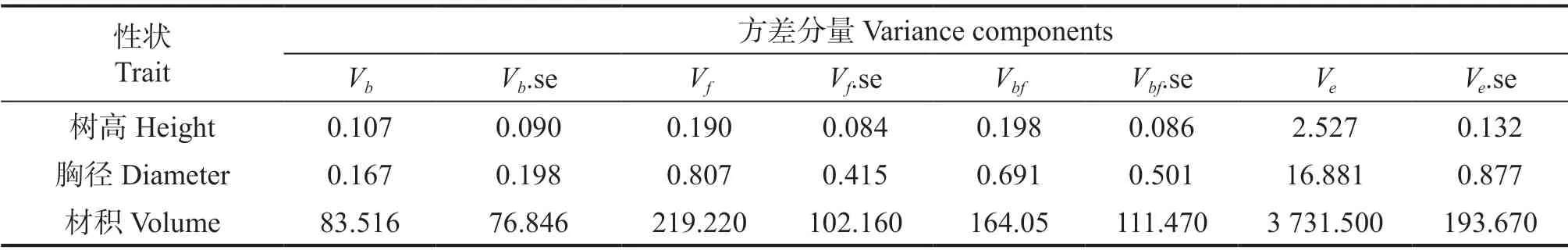
表7 基于单性状批量分析的方差分量估计值Table 7 The estimation of variance components from single trait batch analysis
3 讨论与结论
本研究利用R 语言4.0.2 基于Echidna 单机版(V1.54)开发了R 程序包AFEchidna,以半同胞数据为例,使用混合线性模型进行了随机效应方差分量与遗传力的估计、固定效应和育种值及其准确性的估算,这些结果与商业软件ASReml 的结果高度一致。此外,还演示了随机效应显著性的检验和单性状的批量分析。
王德源和童春发[19]开发了R 程序包HalfsibMS,用于分析林木多地点半同胞子代测定数据,采用他们指定的数据变量格式直接录入数据进行遗传分析。这种做法虽然看似方便,但实则应用范围很窄。由于植物(包含林木)的遗传测定类型广,不仅田间试验设计类型繁多,而且遗传材料来源复杂,简单的一个固定混合模型,根本不足以囊括,更别说涉及不同的统计方法,比如空间分析模型[15]、因子分析模型[16,18]和基因组选择模型[20]。因此,主流的遗传评估软件(例如ASReml、Echidna、SAS、breedR 等)均未采用固定的混合模型。基于这种理念,我们开发AFEchidna包时,在主函数echidna()中通过参数fixed、random 和residual 保留了混合线性模型可以指定任意试验因子及其方差结构的灵活性,延续了Echidna分析混合模型的强大功能。
Isik 等[2]指出商业软件ASReml 采用平均信息(average information, AI)和稀疏矩阵(sparse matrix)算法求解大量的混合模型方程组,其比SAS Proc Mixed 模版(采用牛顿-拉夫逊代数法,Newton-Raphson algorithm)速度更快、效率更高。ASReml 由于可拟合多样化的方差结构,其可轻松处理不同交配设计、不同田间设计、多变量模型等分析。ASReml 的不足之处是它缺乏数据管理、综合性统计和图形可视化功能。Echidna 作为ASReml的免费姊妹版软件,同样继承了ASReml 的优缺点,因此,我们借助R 语言开发AFEchidna 包,加入R 语言在数据管理、综合性统计和图形可视化方面的优势。例如,LRT 检验法,用于检验模型随机项的显著性,或者比较不同模型,我们根据LRT 检验原理编写了函数model.comp()。再比如,随着育种进程的推进与目标性状数量的累积,对性状的批量分析需求是必然的,Echidna 软件虽可以处理单性状的批量分析(麻烦是结果文件偏多,分析结果批量提取难度大),但不能处理多性状模型的批量分析。于是,我们在主函数echidna()中通过参数batch 来运行单性状或多性状的批量分析,并可直接输出结果。此外,主函数echidna()中还有参数batch.G 和batch.R,可用于多种G 结构和多种R 结构的批量分析,这是我们首次提出的方法,在其它遗传评估软件中均未涉及。
简而言之,AFEchidna 包不仅继承了Echidna软件的优点,可拟合不同交配设计(测交、巢式交配、双列杂交等)模型、空间分析模型、多变量模型、多地点模型和基因组BLUP 模型,还延续了R 语言的优势,可自编函数批量分析性状、检验随机项的显著性等,并可利用R 语言管理数据、综合统计和图形可视化的功能。未来我们会陆续介绍AFEchidna 包如何拟合空间分析模型、多变量模型、多地点模型和基因组BLUP 模型。

