转录组揭示自养和混合培养栅藻油脂代谢途径差异
位文倩,孙昕,黄峰,李鹏飞,李盟
(西安建筑科技大学环境与市政工程学院,西北 水资源与环境生态教育部重点实验室,陕西西安,710055)
目前,能源危机是国内外面临的主要危机之一[1]。生物燃料作为一种可再生能源,可以有效削减化石燃料的使用量,减少碳排放,生物燃料(特别是生物柴油)的产量增加能极大地缓解能源危机[2]。微藻具有生物质产率和油脂产率高、环境友好、成熟期短等优点,被认为是一种最具潜力的生物柴油原料[3~6]。但微藻较高的生产成本限制了其规模化应用。
在能源和水资源危机加重的大背景下,利用城市污水培养微藻,有效利用污水中的氮磷养分,既能实现污水资源化,也能降低微藻生物质能源的成本[7~9]。Shen 等[10]在二沉池出水中培养Scenedesmus obliquus,其油脂产率达到了9 mg·L-1·d-1。Han 等[11]在初沉池出水中培养Scenedesmus obliquus,其生物质产率为123 mg·L-1·d-1,油脂产率达到了32.78%。Manzoor 等[12]在甘蔗废水中培养Scenedesmus dimorphusNT8c,生物量为119.5 mg·L-1·d-1,蛋白质含量和脂肪酸含量分别达到了34.82%、15.41%。
转录组学作为一种重要的分子生物学方法[13],有助于研究生物基因结构及基因表达,以便更好地进行生物基因组水平的调控。目前转录组学已应用于分析废水中微藻、蓝细菌等对不同环境条件的基因应答[14]。由于贮藏脂质的生物合成受到多种途径的调控,因此转录组学分析有助于揭示微藻高产脂质的调控机制[15]。本研究将栅藻Desmodesmussp.分别在培养基和生活污水中进行自养和混合培养,使用转录组分析Desmodesmussp. 在不同培养条件下的代谢途径差异和基因的差异表达。
1 材料与方法
1.1 栅藻的培养
栅藻Desmodesmussp.(FACHB-2042)购买于中国科学院水生生物研究所淡水藻种库。用蒸馏水洗涤以消除介质影响,进一步在含有琼脂粉的BG11培养基进行分离纯化。
自养采用含有BG11 培养基的锥形瓶,在恒温光照培养箱(MGC-P,上海一恒)中进行,光照强度80 μmol 光子·m-2·s-1,光暗比为12h∶12h,生长温度在25℃下进行培养,每天手动摇藻3~4次,随机更换位置以保证光照均匀。混合培养采用的生活污水来源于西安建筑科技大学雁塔校区逸夫楼污水,对污水静沉12 h后取上清液、再用0.45 μm滤膜过滤,然后取滤液高压灭菌。对灭菌后污水进行接种,初始吸光度OD680为0.1,培养时间为20 d左右,其他培养条件同自养。自养和混合培养,均取了两组平行样,分别记为P_0、P_00(自养)和YG_1、YG_11(混合培养)。
1.2 RNA提取与检测
使用无RNase 的枪头吸取对数期(OD680=0.5~0.8)的微藻样品于10mL 无酶管中。在8000 r·min-1、4℃下离心5 min充分去除上清,重复操作取藻泥约0.3~0.6 g 迅速放入液氮中冷冻15 min,放入-80℃冰箱保存。使用RNAprep Pure Plant kit 多糖多酚植物试剂盒(DP441)对RNA 进行提取。对RNA 提取后使用RNA 专用琼脂糖凝胶电泳对浓度及纯度进行检测。通过mRNA 特有的poly A 结构纯化总RNA 中的mRNA,通过离子打断的方式将mRNA打断到200~300 bp片段。
1.3 转录组测序及分析
由上海派森诺生物科技有限公司利用Illumina HiSeq 测序平台完成,使用Trinity 软件对高质量序列(Clean Reads)进行拼接得到转录本后再进行后续分析。Trinity 为针对转录组拼接的De Novo 组装软件,基于DBG(De Bruijn Graph)拼接原理对高质量序列进行拼接。拼接完成后,可获得FASTA 格式的Transcript 序列文件。提取每个基因下最长的转录本作为该基因的代表序列,称为Unigene。N50 是将所有序列从长到短排列,将序列长度按照该顺序依次相加,当相加的长度达到序列总长度的50%时,最后一条序列的长度。N90 为将所有序列从长到短排列,将序列长度按照该顺序依次相加,当相加的长度达到序列总长度的90%时,最后一条序列的长度。一般认为N50 大于1000 bp 则拼接结果合格。
1.4 Unigene功能注释
对Unigene 进行基因功能注释。基因功能注释所用到的数据库主要有NR (NCBI non-redundant protein sequences)、GO (Gene Ontology)、KEGG (Kyoto Encyclopedia of Genes and Genome)、eggNOG (evolutionary genealogy of genes: Non-supervised Orthologous Groups)等。
1.5 表达量分析
使用转录组表达定量软件RSEM,以转录本序列为参考,分别将每个样品的Clean Reads 比对到参考序列上。然后统计每个样品比对到每一个基因上的Reads 数,并使用如下公式计算每个基因的FPKM值:
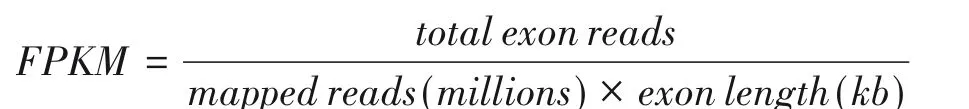
1.6 差异表达及差异基因富集分析
采用DESeq 对基因表达进行差异表达分析,筛选差异表达基因条件为表达差异倍数|log2Fold-Change|>1,显著性P-value<0.05。差异基因富集分析主要是通过GO 富集分析和KEGG 富集分析。使用topGO 进行GO 富集分析,利用GO term 注释的差异基因对每个term 的基因列表和基因数目进行计算,然后通过超几何分布方法计算P-value(显著富集的标准为P-value<0.05),找出与整个基因组背景相比,差异基因显著富集的GO term,从而确定差异基因行使的主要生物学功能。选择每个GO 分类中P-value 最小即富集最显著的前10 个GO term 条目。KEGG 富集分析是通过该pathway 中富集到的差异基因个数与注释到的差异基因个数的比值(Rich factor)、FDR 值和富集到此通路上的基因个数来衡量富集的程度。选择FDR 值最小的前20 条,通过KEGG 注释进一步了解基因在代谢途径中的功能。对样本分别进行KO 注释,即将分子网络的相关信息进行跨物种注释,和KEGG Pathway 注释,即代谢通路注释,获得物种内分子间相互作用和反应的网络。
2 结果与分析
2.1 数据质量评估及转录本拼接结果评估
样品经过上机测序,得到图像文件,由测序平台自带软件进行转化,生成FASTQ 的原始数据(Raw Data),即下机数据。对每个样品的下机数据(Raw Data)分别进行统计,包括Q30、模糊碱基所占百分比、以及Q20(%)和Q30(%)。统计结果见表1。Reads 总数都在4 千万条,碱基总数都超过了60 亿bp,碱基识别准确率在99.9%以上的碱基总数所占百分比(Q30)在90%以上,测序错误率为0.1%;碱基识别率在99%以上的碱基总数所占百分比(Q20)均超过了95%,测序错误率为1%。一般来说,Q20不低于90%,Q30不低于80%被认为数据质量较好,数据可用。
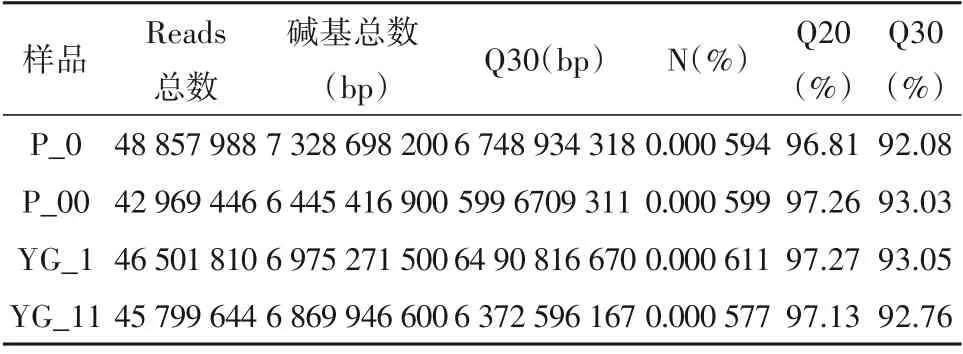
表1 原始数据统计Table 1 Raw data statistics
对Transcript 和Unigene 序列进行统计,结果如表2 所示。拼接完成后的Transcript 序列总数(Sequence Number)在5 万以上,Unigene 的序列总数在2 万以上,最长Transcript 和Unigene 的N50 分别为2069 bp和1979 bp。
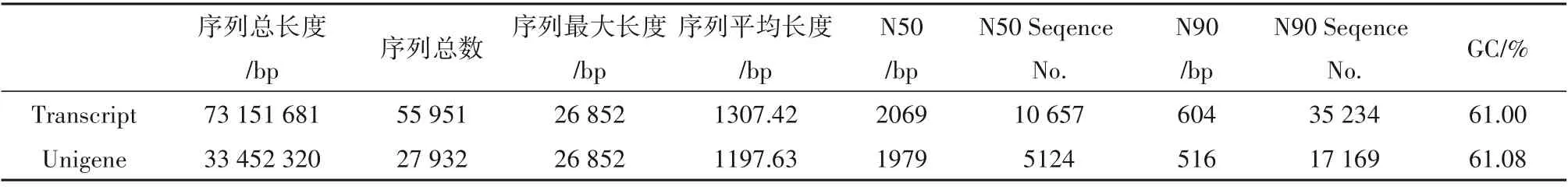
表2 序列整体统计表Table 2 Sequence overall statistics table
2.2 注释结果统计
对原始数据进行无参拼接后的Unigene 进行基因功能注释,无参拼接后共获得了27 932 个Unigene,在GO, KEGG, Pfam, Swiss-prot, eggNOG, NR 数据库注释的结果如图1 所示。其中9.54%的Unigene 在上述6 个数据库中都有注释,在NR、eggNOG、KEGG 数据库中分别注释到11 886、9690、6382 个Unigene。
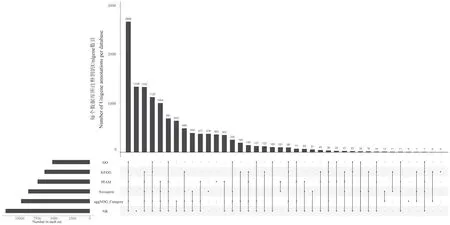
图1 Unigene注释结果统计信息Fig.1 Unigene annotation result statistics upset chart
2.3 表达量分析
FPKM 密度分布能整体考察样品所有基因的表达量模式,一般来说,中等表达的基因占绝大多数,低表达和高表达的基因占一小部分。所有基因在各个样品中的表达量特征统计结果见图2。由图可知,自养组和混合培养中等表达的基因都是占绝大多数,低表达和高表达的基因占比较小。
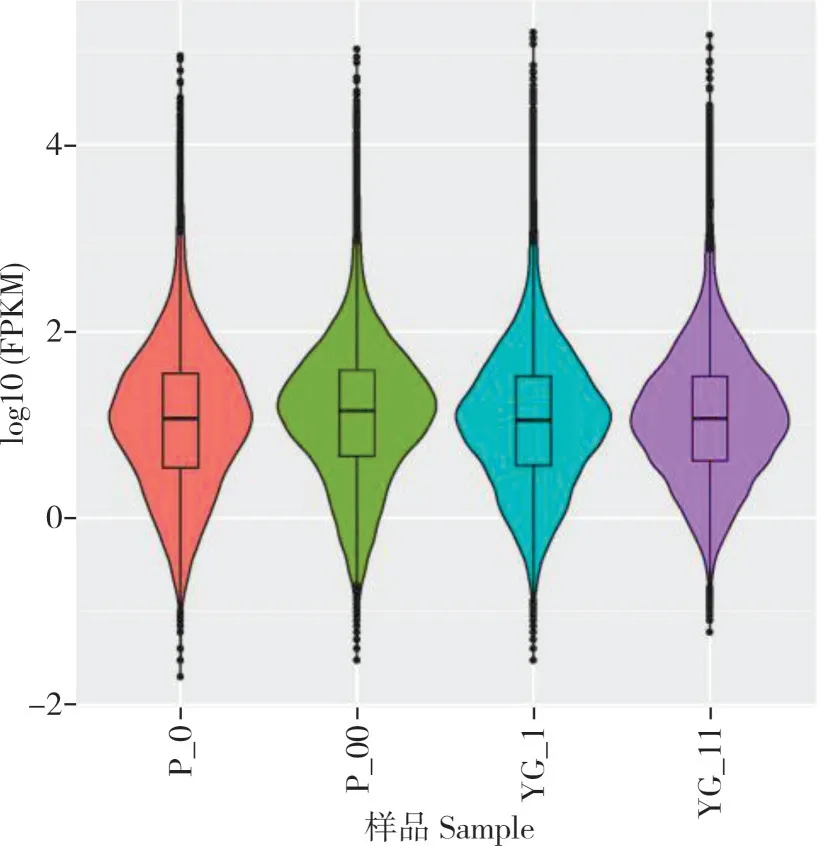
图2 FPKM 密度分布Fig.2 FPKM density distribution
2.4 差异表达分析
采用R 语言ggplots2 软件包绘制差异表达基因的火山图[16,17],结果如图3所示。火山图展示的是基因分布情况、基因的表达倍数差异和显著性结果,2倍表达差异,P-value=0.05为阈值。与自养组比较,混合培养组上调基因总共1081 个,下调基因总共575个,非显著差异表达基因为23 331个。
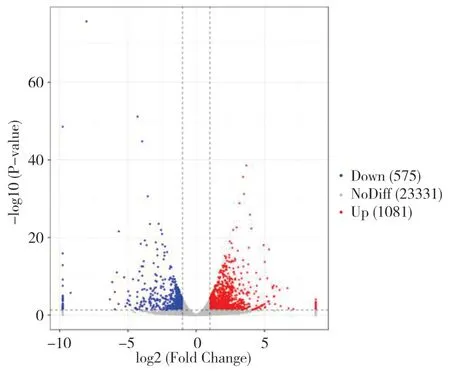
图3 差异表达基因的火山图Fig.3 Volcano map of differentially expressed genes
2.5 差异基因富集分析
根据差异表达的基因的GO 和KEGG 富集分析结果,图4(A)按照细胞组分(Cellular Component)、分子功能(Molecular Function)和生物过程(Biological Process)进行GO 分类,即富集最显著的前20 条KEGG pathway 结果见图4(B)。由图中可知,差异基因GO 富集主要在光合作用的分子功能、生物过程和细胞组分上,如类囊体、光系统I、叶绿素结合、光合电子传输、光反应和暗反应等。KEGG pathway 富集最显著的代谢通路是光合作用,其次还有脂肪酸伸长、内质网中的蛋白质加工以及淀粉和蔗糖合成。
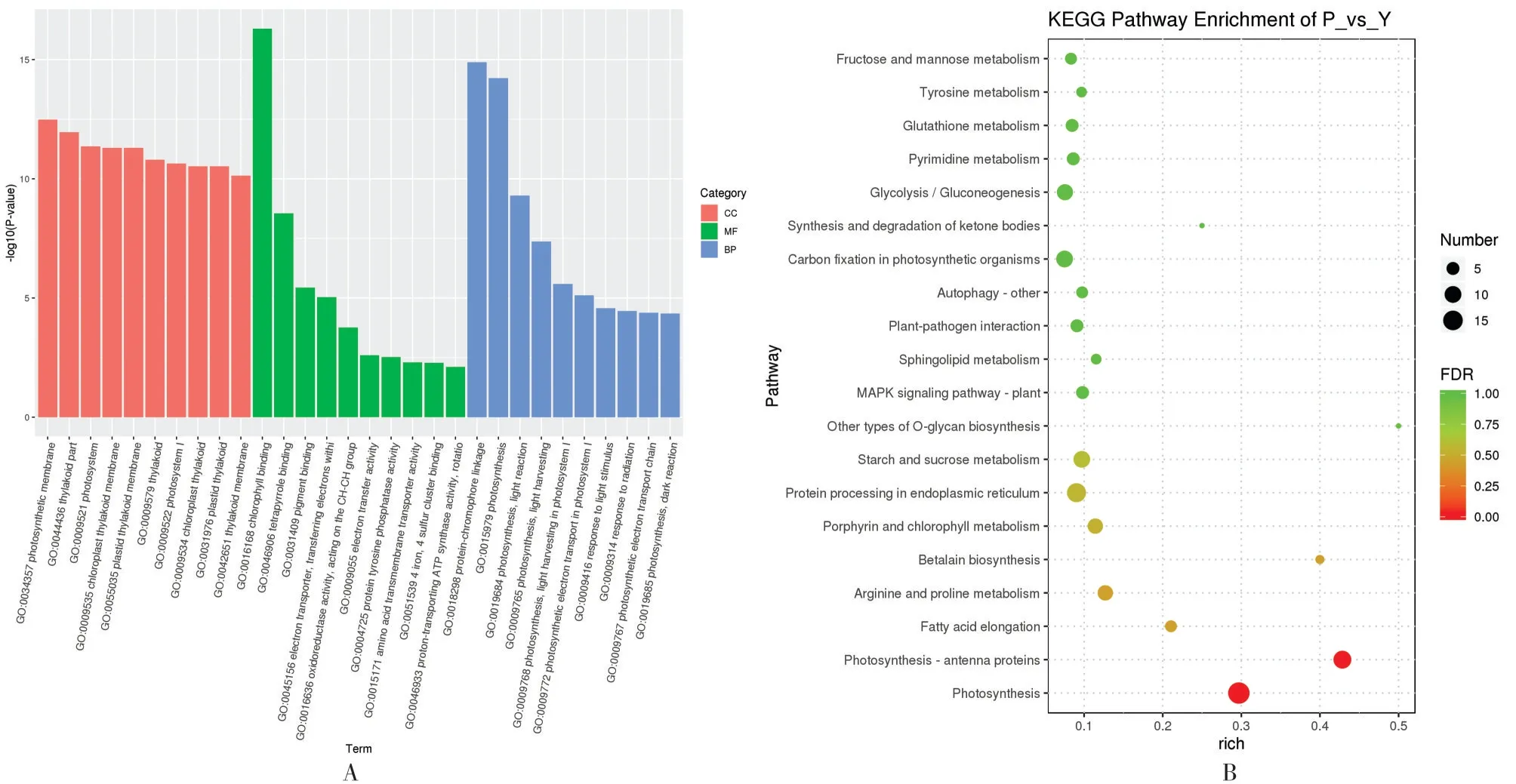
图4 差异基因的GO富集(A)和KEGG富集(B)Fig.4 Differential gene enrichment analysis of GO(A)and KEGG(B)
2.6 代谢途径分析
2.6.1 光合作用 在光合作用固碳过程中,磷酸甘油酸激酶(PGK)的作用下生成1,3-双磷酸甘油酸酯,进而磷酸化形成3-磷酸甘油醛,在果糖二磷酸醛缩酶(ALDO)的作用下生成果糖二磷酸,在这个过程中编码PGK、GAPA 和ALDO 的基因数量分别为5、6、9 个,表达倍数均在2 倍以上,表达水平上调。编码GAPDH 的基因表达倍数为0.06,表达水平下调,固碳作用的初始途径增强。4-磷酸赤藓糖在果糖二磷酸醛缩酶的作用下生成1,7-二磷酸七庚酯,进而在七羟庚糖-1,7-双磷酸酶(glpx-SEBP)的作用下生成7-磷酸七庚糖,最后生成核糖形成卡尔文循环。PGK、ALDO、GAPA、glpx-SEBP 表达水平的上调表明卡尔文循环途径增强。在二羧酸循环中,编码丙氨酸转氨酶(GPT)的基因表达倍数为3.31,有4 个基因编码GPT,其表达水平上调使得苹果酸到丙酮酸、丙氨酸进而生成丙酮酸途径增强。这些都使得光合作用碳固定过程得到增强,为细胞提供更多的甘油三磷酸用于糖酵解和其他细胞过程。
光合作用系统由ATP合酶、光系统I(PSI)、细胞色素b6/f 复合物和光系统II(PSII)组成,从图5 可以看出,PSII 系统P680 反应中心编码D1和D2蛋白、CP43、CP47 叶绿素载体蛋白的基因表达水平下调,表达倍数分别均小于0.5,表明混合培养降低了PSII 对光能的需求。编码放氧增强蛋白1 的基因表达倍数为103,具有显著的表达差异性,表达水平上调表明混合培养提高了氧气的释放。PSI 系统中编码PsaA 和PsaB 的基因分别为2 个和1 个,其表达倍数分别为0.17、0.21、0.13,表达水平显著下调。已有的研究表明,PsbA、PsbC 和PsaB 等与光合作用相关的基因,在混合培养下被下调,从而导致光合色素水平降低[18]。编码蛋白亚基X、PsaN 和PsaO 的基因数量分别为2、1、1,表达倍数均大于3,表达水平上调。放氧蛋白表达水平的提高,PsaA和PsaB结合形成异二聚体的PSI 反应中心,其表达水平的下调表明捕光和光保护能力有所下降。光合电子传输中质体蓝蛋白(PetE)、铁氧还蛋白(PetF)、细胞色素(PetJ)的表达水平均上调,在混合培养条件下,光合电子传输增强,电子传递链从PSII 出发,会裂解水释放氧气,产生ATP 和NADPH,电子传递作用增强表明产生更多的ATP 和NADPH 用于光合作用的碳固定和其他代谢活动。捕光叶绿素蛋白复合物(LHC)镶嵌于类囊体膜上,能够捕获光能并把能量迅速传至反应中心引起光化学反应,是一类在光保护机制中起着关键作用的跨膜蛋白[19]。Lhca1-5 编码的蛋白呈一个半月牙型紧靠在PSI 的一侧,这种二聚体形式能够更好地帮助LHC 行使能量转移及色素识别功能[20]。绿藻具有调节PSI-LHCI 复合物的组成与结构的能力,从而应对环境条件的变化。其中编码Lhca3、Lhca4、Lhca5 的基因表达水平上调,表明混合培养条件下光保护机制、光捕获和光传递能力增强。所有结合叶绿素a/b 的生物体都具有LHCII 同系物,其蛋白均具有一个三聚体结构,LHCII 的三聚体结构较单体在结构上更为稳定,并且能在早期更快地适应光捕获功能[21]。编码Lhcb2和Lhcb5 的基因表达水平上调表明PSII光捕获功能提高。在杜氏盐藻中,铁胁迫可以引起Lhca3 很大的结构变化,从而增大PSI 捕光系统,平衡光系统中的激感现象[22]。
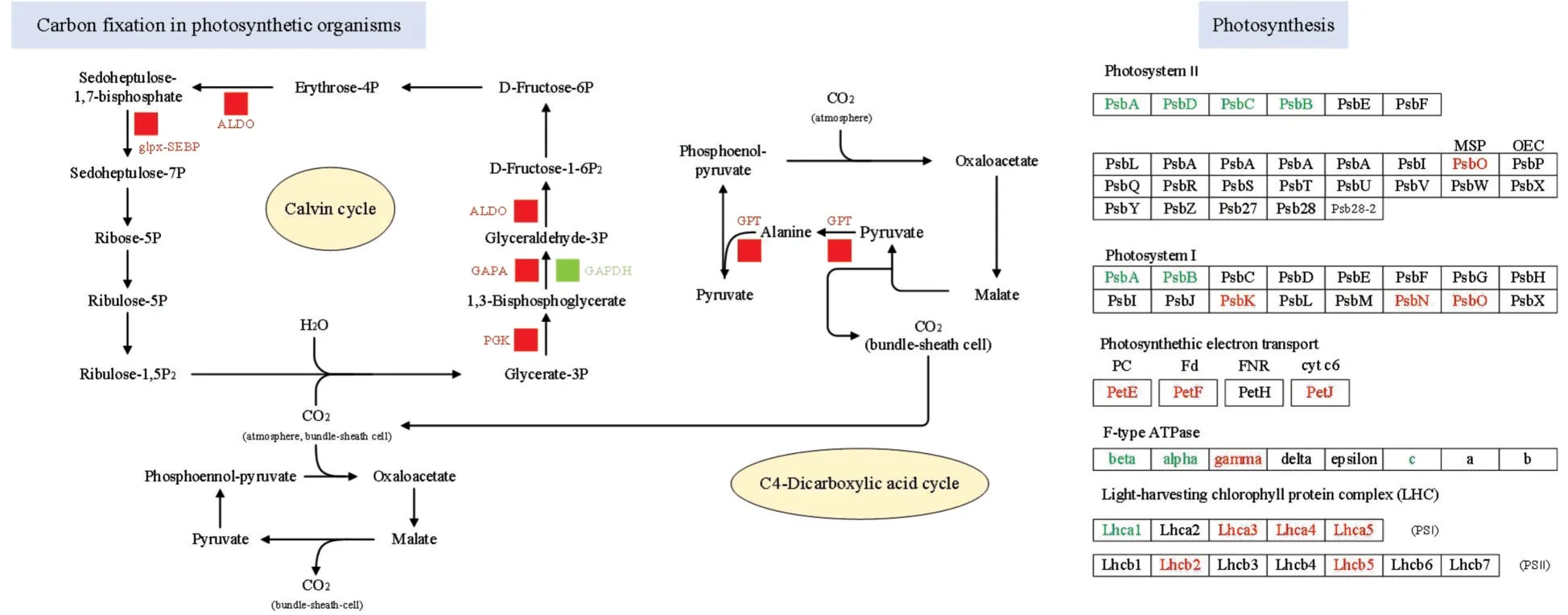
图5 光合作用代谢途径Fig.5 Photosynthesis metabolic pathway

表3 Desmodesmus sp.在自养和混合培养条件下的差异表达基因Table 3 Differentially expressed genes of Desmodesmus sp.under autotrophic and mixotrophic conditions
2.6.2 脂质代谢 与自养条件相比,编码糖酵解途径的限速酶丙酮酸激酶(PK)的基因表达倍数为2.06,其表达水平上调,表明在混合培养条件下糖酵解作用增强,从而为细胞提供了更多的碳骨架、ATP 和NADPH,为磷酸戊糖途径和脂肪酸生物合成提供了更多的底物。据报道(图6),在混合培养条件下,细胞可能处于能量供需之间的不平衡状态(ATP/NADPH 不平衡),从而激发过大能量产生过量的NADPH[23]。在磷酸戊糖途径中,6-磷酸葡萄糖脱氢为6-磷酸葡萄糖酸酯,进而氧化为3-磷酸甘油醛,在果糖二磷酸醛缩酶(ALDO)的催化下生成6-二磷酸果糖,编码ALDO 的基因有3 个,表达倍数均大于2,其表达水平显著上调,进而产生更多的丙酮酸用于糖酵解产生乙酰辅酶A(Acetyl-CoA),乙酰辅酶A 为脂肪酸生物合成提供底物导致脂质生产增强。脂肪酸合成从乙酰辅酶A 转化成丙二酰辅酶A,也是脂肪酸合成的关键,生成乙酰辅酶A 酶水平的提高,促进了脂肪酸生成。脂肪酰基ACP 硫酯酶(FATB)将长链酰基催化为长链脂肪酸,长链酰基辅酶A 合成酶(ACSL)再将长链脂肪酸转化为长链酰基辅酶,其中编码FATB的基因表达水平上调,编码ACSL 的基因表达倍数小于0.5,表达水平下调,使得生成更多的长链脂肪酸,导致脂质生产水平提高。
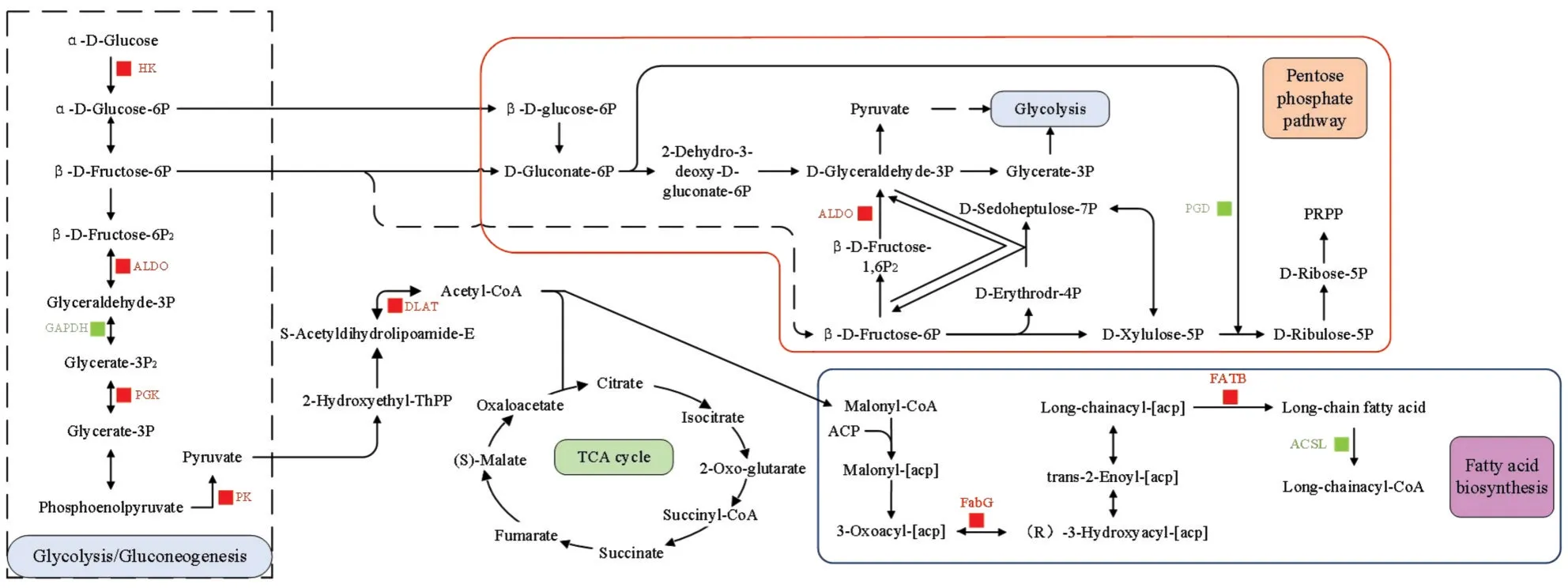
图6 脂肪酸的生物合成及相关的代谢途径Fig.6 Fatty acid biosynthesis and related metabolic pathways
3 结论
Desmodesmussp. 混合培养条件下光保护机制、光捕获和光传递能力增强,光合电子传输增强,裂解水释放氧气,产生更多的ATP 和NADPH用于光合作用的碳固定,为细胞提供更多的甘油三磷酸用于糖酵解和其他细胞过程。在混合培养条件下糖酵解作用增强会导致磷酸戊糖途径产生更多的丙酮酸用于糖酵解产生乙酰辅酶A(Acetyl-CoA),乙酰辅酶A 为脂肪酸生物合成提供底物导致脂质生产增强。转录组学分析为调控污水混合培养条件下微藻油脂产量提供了分子生物学基础。

