面向高光谱显微图像血细胞分类的空-谱可分离卷积神经网络
时 旭,李 远,黄 鸿
(重庆大学 光电技术与系统教育部重点实验室,重庆 400044)
1 引 言
红细胞、白细胞是人体血细胞的主要组成部分。它们在血液中的数量与人体的生理状态息息相关。如红细胞数量过少会导致贫血,白细胞数量过少会引发免疫力缺失[1-3]。血细胞的计数和分类是病灶临床检查的常用方法之一,也是血常规检验的基本内容[4]。对血细胞进行分类,有助于血液学家诊断疾病,如白血病、血液癌症等[5]。因此,在临床诊断中,如何对红、白细胞进行计数具有十分重要的价值。早期通过显微镜计数以及利用血细胞的电阻率来尝试血细胞自动计数,但其精度和效率受限。随着信息技术的蓬勃发展,计算机自动计数以其高效、快速的特点广泛应用于血液细胞分类。
传统计算机自动计数方法可分为几何计数法和统计计数法。其中,典型几何方法包括距离分类器和线性分类器等,但这些方法对具有非线性分布的显微图像效果不佳。统计分析法包括参数估计法、梯度法、贝叶斯准则法、最大似然法和支持向量机(Support Vector Machine,SVM)等。这些方法在样本数较多的情况下具有较好分类能力,但对结构复杂的数据需要先获取样本统计分布得到先验概率和类分布概率密度函数等,才能取得较好的效果,且SVM方法存在核函数和参数选择问题[6]。
深度神经网络在图像分类中的优异表现给医学图像处理带来巨大机遇。卷积神经网络(Convolutional Neural Network,CNN)尤为突出,其使用局部连接有效提取特征,并通过共享权值显著减小参数量,为在医学图像分类任务中奠定了基础。郑婷月等[7]将一种多尺度全卷积神经网络应用在视网膜血管分割任务上,在两个公开眼底数据集上均可达到96%左右的准确率。Raunak等[8]使用双流神经网络,对肺结节CT图像进行良恶性分类,取得了90.04%的分类精度。Khashman等[9]对比了三种不同的神经网络方法,在RGB图像血细胞亚型识别任务中,取得99.17%的分类效果。
但是,上述方法都是针对传统显微光学图像,只包含红、绿、蓝三通道信息,不能有效反映血细胞内在复杂的生化性质。与此同时,高光谱成像技术拥有无接触、非电离、无伤害和光谱信息丰富等优点,且具有“图谱合一”特性。因此,物质间的细微差异可更好地由其连续的光谱曲线表达,在医学图像处理领域,被用于脑癌[10]、乳腺癌[11]、舌癌[12]检测 等,并取得了较好效果。Huang等[13]提出一种调Gabor滤波卷积神经网络(MGCNN)模型,将调制Gabor滤波与卷积神经网络相结合,取得了比传统神经网络更好的血细胞分类结果。Wei等[14]开发了一种双通道CNN,将典型CNN特征和EtoE-Net特征相融合,取得了更好的分类表现。然而这些方法训练时间较长,在实际应用中对硬件条件要求颇高。此外,上述模型需要较多数量的标记样本以保证模型分类性能,且高光谱医学图像标记需要大量人工成本。
基于此,本文提出了一种空-谱可分离卷积神经网络(Spatial-Spectral Separable Convolution Neural Network,S3CNN),该网络通过有效利用高光谱图像中的空间-光谱特征信息来提升分类效果,并利用可分离卷积结构对模型复杂度进行优化,减少训练时间。S3CNN主要思路是利用空-谱联合距离(spatial-spectral combined distance,SSCD)得到训练集中各像素点的空-谱近邻,并对这些近邻点赋予与相应中心像素点相同的标签,进行样本扩充,然后用可分离卷积优化经典卷积,减少训练时间。在Bloodcells1-3和Bloodcells2-2高光谱数据集上的实验结果表明,本文方法能够有效改善高光谱显微图像的细胞分类性能,且在模型训练时间上具有优势。
2 空-谱可分离卷积神经网络(S3CNN)
为表述方便,文中高光谱医学数据集表示为X={x1,x2,…,xN}∈RN×b,其中N为样本数,b为波段数,li∈{1,2,…,c}为标签,c为类别数。
本文所提出空-谱可分离卷积神经网络的算法流程图如图1所示,其主要包括空-谱联合训练集构建和可分离卷积神经网络,下面对其进行一一介绍。
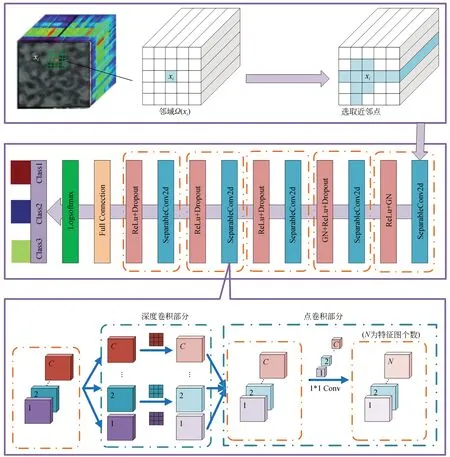
图1 空-谱可分离卷积神经网络流程图Fig.1 Flow chart of spatial-spectral separable convolution neural network
2.1 空-谱联合训练集构建
在高光谱血细胞图像中,各个像素在空间分布上具有一定的相关性[15-16],在同一个空间邻域内的像素点通常属于同一类别[17-20]。基于此,本文利用邻域空间像素中的空-谱联合信息来衡量像素间的相似性,并提出一种空-谱联合距离(SSCD)来更好地选择空-谱近邻点,以扩充训练样本,具体如图2所示。
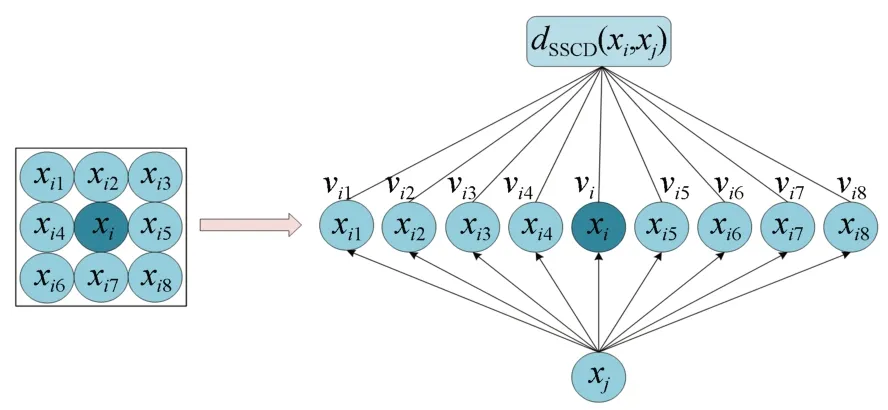
图2 计算空-谱联合距离过程Fig.2 Process of calculating the spatial-spectral combined distance
对于血细胞高光谱显微图像中的像素点xi和xj,Ω(xi)和Ω(xj)分别为xi和xj的近邻空间,空-谱联合距离SSCD可以定义为:
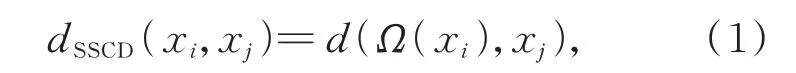
其中,d(Ω(xi),xj)为xj和近邻空间Ω(xi)之间的距离,它可以表示为:
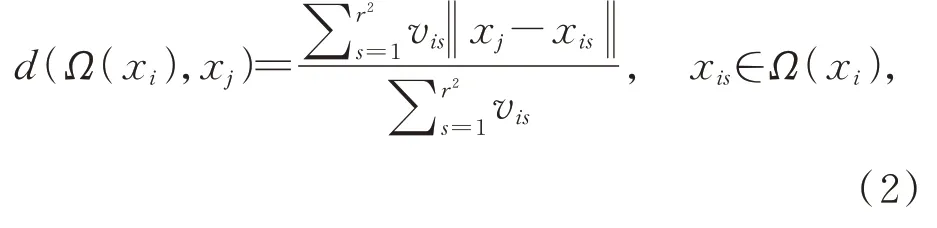
式中vis是xis的权重,可通过核函数来计算:
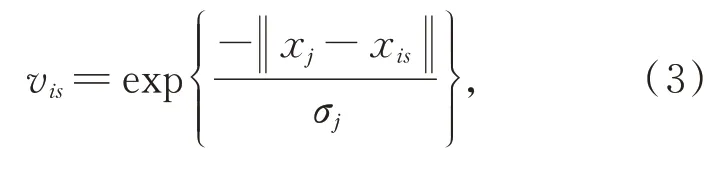
其中,σj为‖xj-xis‖的均值,可定义为:
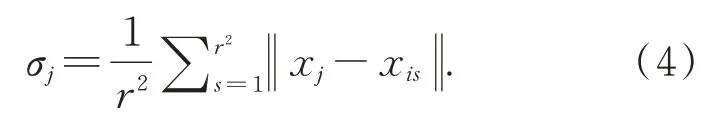
对于有标签的高光谱图像数据,在其周围r×r的方形邻域内根据空-谱联合距离选择离其最近的k个像素点,可认为它们与中心像素的标签相同,扩充到训练样本集,通过利用少量标记样本和一定数量近邻样本进行深度神经网络模型训练。
2.2 可分离卷积神经网络
通过空-谱近邻得到空-谱近邻样本训练集后,如何高效的进行训练是一个关键问题。神经网络大多是由不同尺度的卷积核堆叠而成,伴随着模型精度的不断提高,神经网络的深度和模型参数量也在不断增加[21]。然而,过多的参数给模型的训练带来极大的挑战。
可分离卷积是解决这一问题的有效方法[22],如图3(b)所示。其主要思想是将标准卷积转化为一个深度卷积和一个1×1的点卷积。在正向传播过程中,可分离卷积首先在每个图像通道上进行二维卷积,其次对输出的特征图进行点卷积。
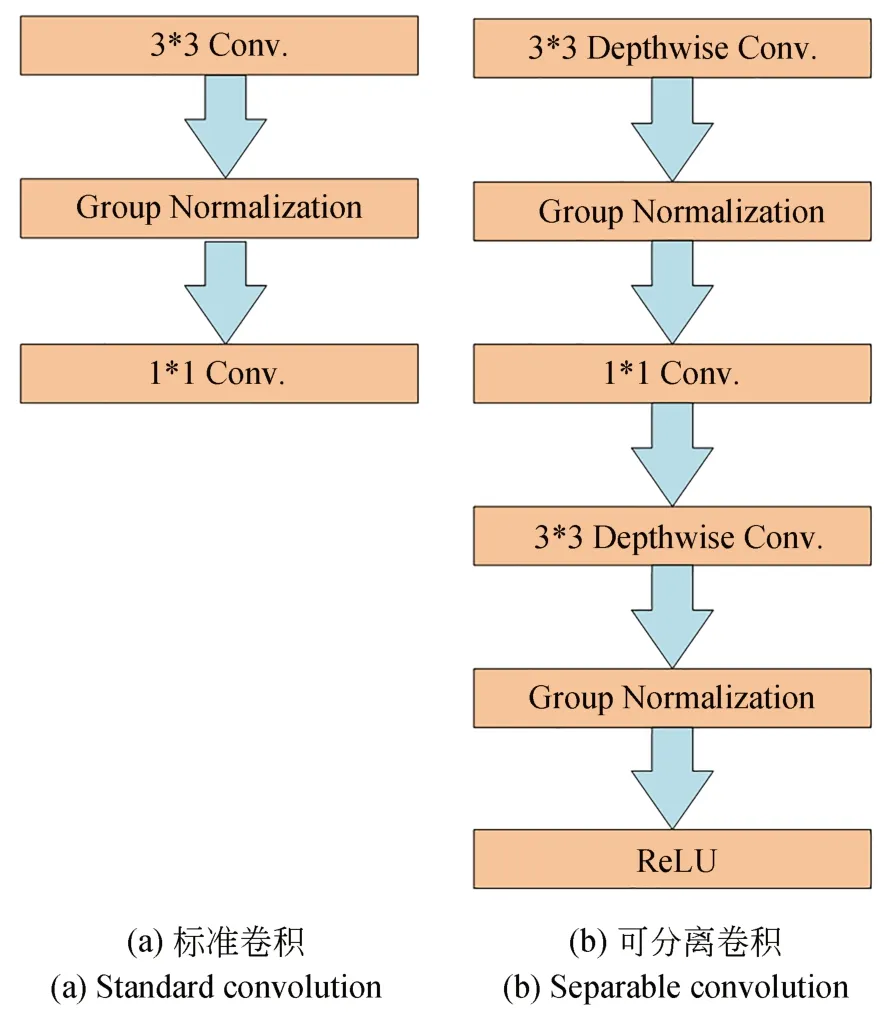
图3 标准卷积与可分离卷积结构对比Fig.3 Comparison of standard convolution and separable convolution
在可分离卷积提取特征后,采用平均池化操作以有效降低空间维度,减少计算量。然后采用Group Normalization(GN)归一化,最后采用如下形式的ReLU激活函数:
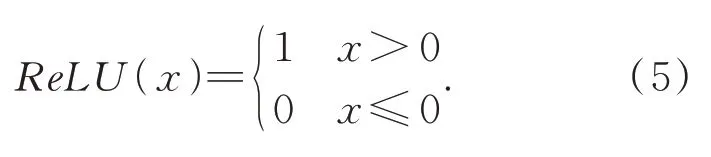
ReLU激活函数有单侧抑制的作用,使得模型在训练过程中的梯度值和收敛状态处于稳定,有助于解决梯度消失的问题。经计算得,最终网络模型参数量params约32M,计算量flops约0.047 G。
3 实验分析
3.1 实验数据集介绍
本文算法主要通过两个不同场景下的高光谱血细胞数据集Bloodcells1-3和Bloodcells2-2进行实验验证。该数据由显微镜和硅电荷耦合器件(CCD)组成VariSpec液晶可调谐滤波器收集,波长范围为400~720 nm,共包含33个波段,其中Bloodcells1-3数据集像素大小为973×699,Bloodcells2-2数据集像素大小为462×451。图4(a)为两个数据集第10个波段的图像,图4(b)为两个数据集的真值图。在真值图中,红色代表白细胞,蓝色代表红细胞,绿色代表背景。

图4 高光谱显微图像Fig.4 Hyperspectral microscopy image
3.2 实验设置
实验的硬件环境采用PANYAO 7048G服务器,其搭载6张TITAN RTX以及256G内存;软件环境是基于Ubuntu 18.04系统的Py Torch深度学习框架(PyTorch 1.6版本),编程语言为Python 3.7。
在实验中,首先按波段对高光谱血细胞数据进行归一化处理。在每次实验中,高光谱血细胞数据集被随机划分为训练样本和测试样本,利用训练数据得到空-谱联合训练集后,将其送入可分离卷积网络进行训练。训练过程中,优化器为Adam,batchsize为1 000,初始学习率设置为0.001。采取“Early Stopping”策略终止训练,即在训练集上若连续10个epoch损失函数的值都不减小就自动停止训练。训练结束后,将测试集送入网络以评估模型。对于分类结果,采用总体分类精度(Overall Accuracy,OA)系数进行评价。为了保证实验结果的可靠性,每种条件下的实验均重复进行10次,并取10次结果的平均值作为最终实验结果。
为了证明本文方法在高光谱显微图像分类上有效性,实验中选取Nearest Neighbor(1-NN)、SVM、1D CNN、2DCNN、3D CNN、Separable Convolution Network(SCN)作为对比方法。其中,1D、2D、3D分别代表卷积方式,SVM核函数采用径向基函数(Radial Basis Function,RBF)。
3.3 Bloodcells1-3实验结果
为研究窗口大小r和近邻数k对算法性能的影响,首先选择Bloodcells1-3数据集进行实验。在红细胞、白细胞、背景三个类别中,每类随机选取40个样本用于训练,2 000个样本用于测试。窗口大小r的取值范围为{1,3,5,7,9,11,15},空-谱近邻数k的取值范围为{2,4,6,8,10,14,18,20}。图5为算法在不同窗口大小和空-谱近邻数下的分类精度。
由图5可知,分类精度随着窗口r的增大先有所提升后下降,这是由于当空间窗口包含了更多的空间近邻时,可利用的空间信息更加丰富,因而能够更好地区分红、白血细胞以及背景,提高分类精度。同时,近邻数k增加会选择更多无标记空-谱近邻样本参与训练,提高分类性能,但k过大时导致近邻点中可能包含较多来自于其他类别的像素点,进而影响模型训练效果。基于上述因素,本文选取k=10,r=9。
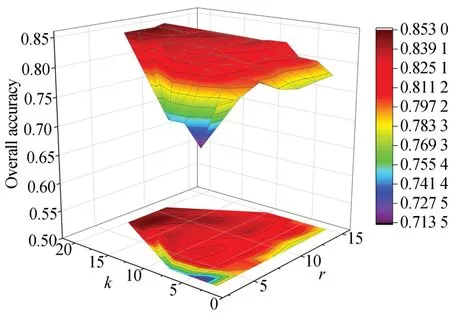
图5 Bloodcells1-3数据集中S3CNN在不同r和k下的分类结果Fig.5 Classification results of S3 CNN with different r and k on Bloodcells1-3 dataset.
为评估不同算法在不同数目训练样本下的分类性能,从Bloodcells1-3数据集中的每类中分别随机选取5、10、20、40、60、80个样本用于训练,选取2 000个样本作为测试,实验结果如表1所示。
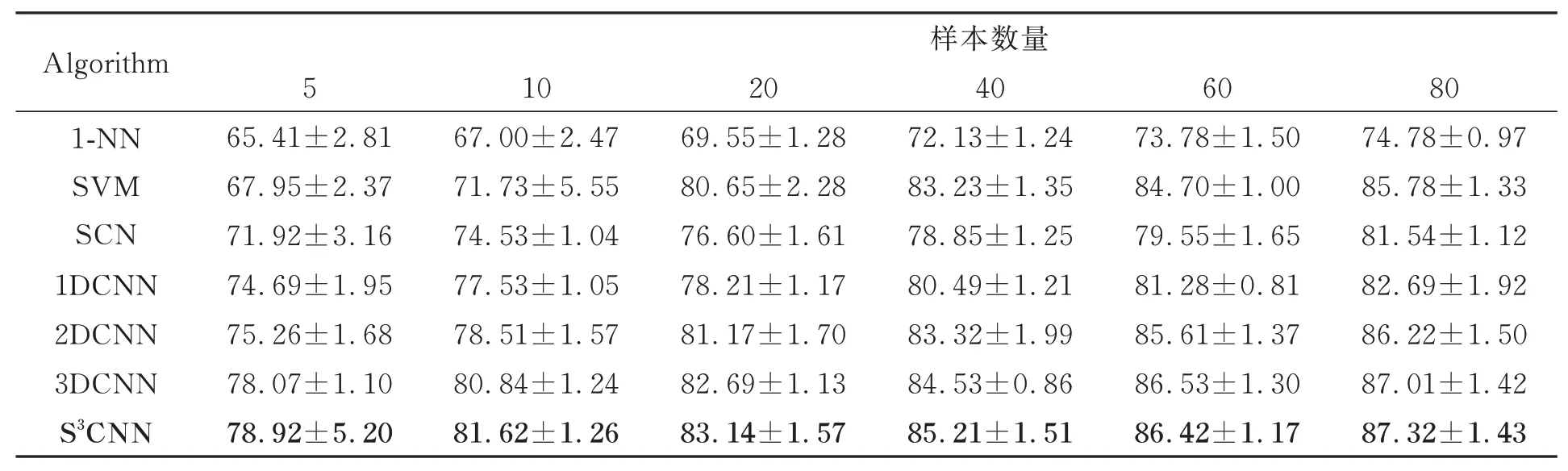
表1 Bloodcells1-3数据集上不同算法的分类结果(总体分类精度±标准差)Tab.1 Classification results of different algorithms on Bloodcells1-3 dataset(overall accuracy±STD) (%)
由表1可知,对于每种分类算法,随着训练样本数量的增加,其分类精度随之提升。这是由于更多的训练样本包含了更丰富的先验信息,更有利于模型提取信息。相比1-NN、SVM等传统方法,所有的深度学习方法都取得了更好的分类结果,这是因为深度学习方法可以更好提取高光谱数据高层特征,有利于分类。在CNN方法中,3DCNN方法的分类性能要优于1DCNN和2DCNN方法,这是因1DCNN只提取光谱特征,而2DCNN提取空间特征但未能充分利用光谱信息,而3DCNN同时利用利用光谱-空间信息,因此分类性能更好。本文提出的S3CNN在多数训练情况下都取得了最好的分类结果,仅在训练样本为60时精度略低于3DCNN约0.001 1,这是因为该网络通过利用空-谱联合距离选择近邻点参与训练,从而有更多的训练样本参与模型训练,可更好的表征不同血液细胞的高层特征,提高模型的鲁棒性,提升了模型分类效果。
图6为各算法的分类结果图。由图可知,本文算法的分类结果图更加平滑,因为相比其他算法,该算法能够更好提取不同血液细胞的空-谱信息,具有更好的分类性能。

图6 各算法在Bloodcells1-3数据集上的分类结果图Fig.6 Classification maps for different methods on Bloodcells1-3 dataset.

为比较S3CNN与3DCNN的差异性,论文引入McNemar统计检验。McNemar检验是一种基于统计学原理的显著性评价方法[23],算法1以及用于比较的算法2的检验统计量可定义为:其中,f12表示被算法2分错而被算法1正确分类的样本数,f21表示被算法1分错而被算法2分对的样本数。一般设置显著性阈值为0.05,Z大于该值时表示算法1优于算法2的性能,且当Z大于2.58和1.96时,分别表示两种算法在99%和95%置信水平下具有统计显著性。对于S3CNN与3DCNN两种算法,计算得Z值为6.68,表明两种算法在统计学意义下具有显著差异性。
3.4 Bloodcells2-2实验结果
实验中,分别从Bloodcells2-2数据集每类中随机选取200个样本用于训练,2000个样本用于测试。对窗口大小r和空-谱近邻数k进行实验,其取值范围与Bloodcells1-3参数实验一致。由图7的实验结果可知,其结果与图5相似,S3CNN方法的分类精度随着r和k增加而增加,而后逐渐稳定,综合考虑分类精度以及算法的运行效率,k设置为10,r值取7。
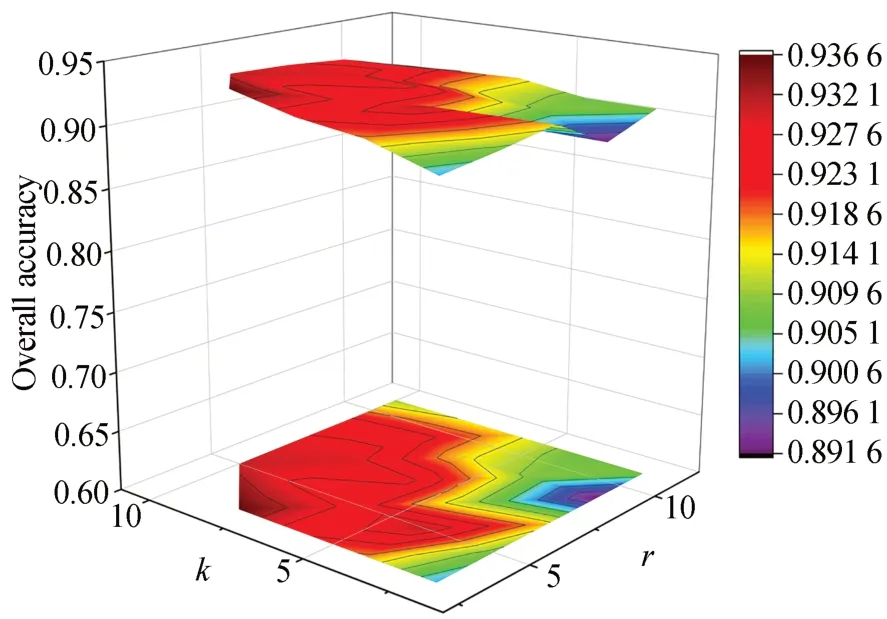
图7 Bloodcells2-2数据集中S3CNN在不同r和k下分类结果Fig.7 Classification result of S3CNN with different r and k on Bloodcells2-2 dataset.
为评估不同数目训练样本下不同算法的分类性能,从Bloodcells2-2数据集中的每类中分别随机选取5、10、20、40、60、80个样本用于训练,选取2000个样本作为测试,实验结果如表2所示。
从表2可以看出,对于每种分类算法,其分类精度随着训练样本个数的增加而递增。与此同时,本文算法始终取得了最好的分类效果。在标记样本数较小时,S3CNN比其他对比方法分类优势更明显。这是由于1DCNN、2DCNN、3DCNN在训练样本不足时,模型训练效果受限,导致分类效果不理想,而S3CNN充分利用高光谱图像的空间一致性原则,有效选取标记样本点的无标记空-谱近邻样本,大大扩充了训练样本,可以满足网络模型对于训练样本的需求,从而可更好的提取不同血液细胞的高层特征,取得更高的分类精度。
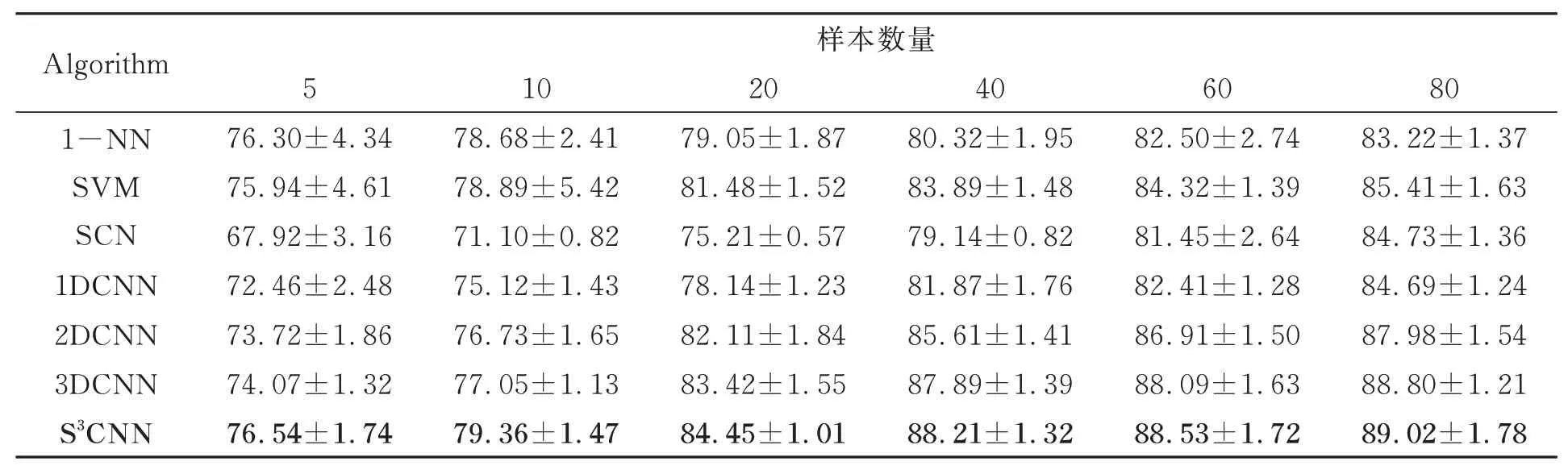
表2 Bloodcells2-2数据集上不同算法的分类结果(总体分类精度±标准差)Tab.2 Classification results of different algorithms on Bloodcells2-2 dataset(overall accuracy±STD) (%)
图8为各算法的分类结果图。由图可知,各算法中S3CNN效果更好,这是因为S3CNN有效地重构了训练样本的空-谱近邻点,充分挖掘了空-谱信息,因此整体准确率更高,具有更好的分类效果。
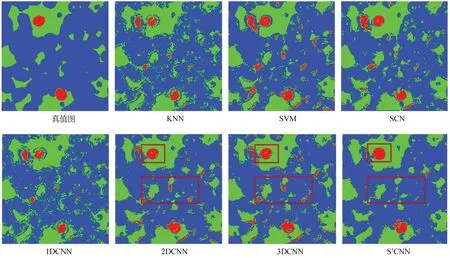
图8 各算法在Bloodcells2-2数据集上的分类结果图Fig.8 Classification maps for different methods on Bloodcells2-2 dataset.
为了探究S3CNN算法的时间复杂度,在Bloodcells2-2数据集上对所采用的可分离卷积与经典卷积算法进行训练时间对比实验。实验中选取6 000个样本点进行训练,训练时间结果如表3所示。
由表3可知,可分离卷积模型和经典卷积相比,可分离卷积模型减少27%训练时间,这是因为可分离卷积使用了一个深度卷积和一个点卷积优化经典卷积,使得参数减少,降低了模型复杂度,更有利于实际应用。

表3 不同卷积方式的训练时间对比Tab.3 Comparison of training time of different convolution methods on Bloodcells2-2 dataset (min)
4 结 论
在高光谱血细胞分类计数任务中,传统深度学习方法需要大量不易获取的标记数据,也未考虑高光谱图像内部空间结构,对高光谱像素特征提取不够充分。本文基于可分离卷积方法和空间一致性原则,提出一种空-谱可分离卷积神经网络(S3CNN)。该方法能有效提取高光谱图像中的空间-光谱信息,通过空-谱联合距离选择各像素点的空-谱近邻,并对这些近邻点赋予中心像素点相同的标签作为训练集的扩充。此外,考虑到传统深度学习网络参数量巨大、对运算硬件要求颇高,采用可分离卷积代替经典卷积,通过降低卷积核参数数量,优化模型的训练时间。在Bloodcells1-3和Bloodcells2-2高光谱数据集上的实验结果表明,本文算法可有效挖掘高光谱血细胞显微图像中各类成分的内蕴空-谱信息,改善了分类性能,同时减少了训练时间。然而,S3CNN只考虑高光谱图像局部空-谱联合信息,未能有效探索高光谱图像非线性复杂结构。因此下一步研究工作考虑将图卷积神经网络用于高光谱血细胞图像分类上,构建空-谱联合图神经网络模型,以进一步提升分类效果。

