青藏高原东部22株栽培与野生羊肚菌的分子进化分析
杜军华 姜东伯 张津京 余建平 宋长新 刘甜 陈辉 陈群 吴晓明






摘要:为了更好地区分不同羊肚菌菌株,确定其生物多样性,对10个栽培羊肚菌和12个野生羊肚菌进行分析。通过对5个基因区域进行DNA测序,借助数据库检索、多序列比对和分子进化等方法进行分析,发现10种栽培样本属于3种已知物种:六妹羊肚菌( Morchella sextelata )、梯棱羊肚菌( M. importuna )、七妹羊肚菌( M. septimelata )。同GenBank核苷酸序列数据库中已测序样本相比,这些样本都含有一定程度的序列差异,体现在栽培的样本也具有遺传多样性。12个野生型样本同六妹羊肚菌、小海绵羊肚菌( M. spongiola )、 Morchella prava 物种更接近,并显示出较大的序列差异,其中1株同所有已知羊肚菌品种均有较大的遗传差异。考虑到这些野生羊肚菌的生长地域同其他羊肚菌生长地域的差异,推断由于独特的地理环境,使野生羊肚菌演化出了特有的基因序列。本研究获得了青海一些地区羊肚菌的分类、分子演化和多样性特征,有助于鉴定、选育适合栽培的羊肚菌菌株。
关键词:子囊菌类;羊肚菌;分子进化;多序列比对;青藏高原;遗传多样性
中图分类号: S646.702 文献标志码: A
文章编号:1002-1302(2022)07-0028-07
收稿日期:2021-06-15
基金项目:青海省应用基础研究(编号:2018-ZG-790)。
作者简介:杜军华(1965—),男,河北故城人,副教授,主要从事生物技术及应用研究。E-mail:djh319@126.com。
通信作者:杜军华,副教授,主要从事生物技术及应用研究,E-mail:djh319@126.com;吴晓明,博士,副教授,主要从事生物信息研究,E-mail:wxm@mail.xjtu.edu.cn。
羊肚菌是公认的最重要的野生食用、药用真菌之一,分布于世界各地,特别是在北温带地区[1-2]。其口感脆嫩,风味独特,富含维生素、人体必需氨基酸和特殊的多糖成分,具有极高的营养价值和药用价值[3-5]。目前我国已报道的羊肚菌种类较多,其中常见的有圆锥羊肚菌( Morchella conica)、小顶羊肚菌(M. angusticeps)、羊肚菌(M. esculenta)、褐色羊肚菌(M. umbrina ) 等[6]。近年来,人们对其营养价值、生物学特性和物种多样性研究取得了重大进展,一些羊肚菌品种也已被广泛栽培,显示出良好的经济效益[7]。
目前,仅有13种羊肚菌品系完成全基因组测序(https://www.ncbi.nlm.nih.gov/assembly/organism/5193/),而已知羊肚菌属(NCBI数据库taxid编号:5193)包含多达30个物种,每个物种都有不同的菌株。目前Index Fungorum数据库(http://www.indexfungorum.org/Names/Names.asp) 也收录了多达353种羊肚菌(截至2021年12月)。许多菌株在形态上相似,难以区分,利用基因序列准确鉴定羊肚菌的种类和菌株尤为重要。
羊肚菌属的分类研究已有多年,其子囊体的外观常随环境、气候等外界条件的变化而变化,在形状和大小上具有高度的多态性。虽然子实体的微观结构相对保守,但从属类群的定义非常困难,菌株之间的定义也不明确,影响了该类群资源的合理利用和开发[8-9]。对于分类学家和野外真菌学家来说,羊肚菌属于一个非常复杂的属。由于形态差异不明显,同一种类的羊肚菌由于生存环境和营养条件的差异往往表现出形态多样性,根据外观和形态对羊肚菌进行分类往往存在一定偏差。
基于多基因位点进行分析已经成为推断羊肚菌物种的新方法。使用ITS-RPB1-RPB2-EF1-α组合数据集进行基因分析,人们已经将羊肚菌分为黑羊肚菌分支( Elata clade )、黄羊肚菌分支( Esculenta clade )、变红羊肚菌分支( Rufobrunnea clade )[10-11]。拉丁二项命名法也已用于羊肚菌命名,即用 Mes(E. clade)或Mel(E. clade) 跟上一个阿拉伯数字表示,自2012年以来也被广泛使用。迄今为止,已经利用系统发生分析的方法,确定了68个羊肚菌品种的关系,包括38个属于黑羊肚菌、28个属于黄羊肚菌、2个变红羊肚菌[3,12-13]。基于这些已知物种的命名,有助于准确地进行其他羊肚菌品种的鉴定。
在青海地区高纬度和寒冷的气候条件下,动植物已经进化出了独特的生长模式。该地区的土壤特性和生态系统,也成为野生羊肚菌的良好生长环境。作为一种重要的经济作物,适合栽培的羊肚菌新品种也需要不断被筛选出来,以促进农业经济的发展。为了更好地了解青海地区羊肚菌的菌株类型、分布特点、分子标记和进化特征,笔者对22个样本进行分类相关基因的测序,通过序列比较的方法,以确定菌株特点及其遗传特征。
1 材料与方法
1.1 样品收集
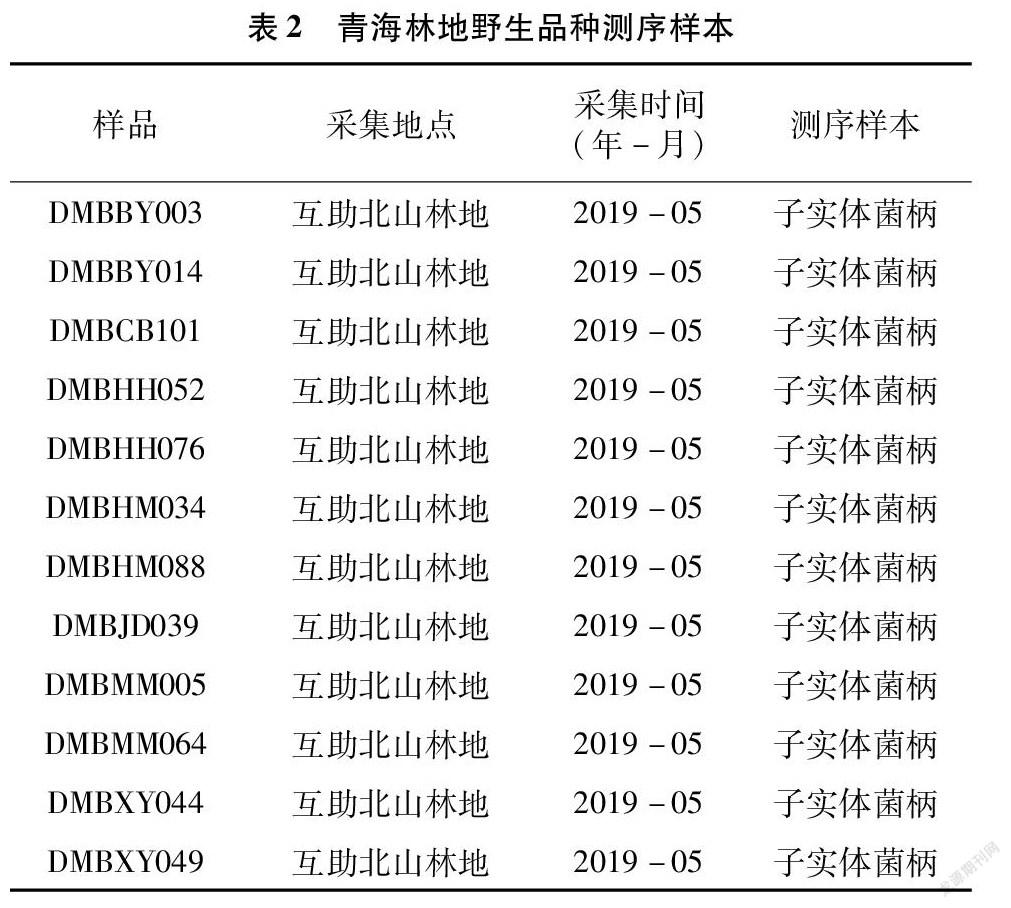
本研究于2020年5月,在大巴山东部地区北山农场共收集了107个样本。经过基于形态的初步筛选,选取其中有代表性的12个野生菌株,继续对其进行分子水平的分析和鉴定。同时,笔者也从青海省西宁市周围的羊肚菌栽培基地,选取了10个栽培品种,进行菌株的亚种鉴定。样本信息见表1、表2。
1.2 DNA测序区域的选择
为对10个栽培品种和12个野生品种进行进化分析和分子鉴定,本研究对5个突变率较高的基因区域设计引物并进行了测序。这些区域是rDNA内部转录间隔区its1~its4 (ITS)、编码RNA聚合酶Ⅱ的最大亚基(RPB1)、编码RNA聚合酶Ⅱ的第二大亚基(RPB2)、翻译延伸因子 (EF1-α)、核糖体大亚基(LSU)。对每个样品进行DNA提取分离后,将凝胶中的DNA样品切割纯化,回收目的片段,用相应的引物和合适的条件进行聚合酶链式反应(PCR)扩增(表3、表4),扩增程序根据引物退火与片段大小做适当调整,扩增后的分子进行测序,最终共获得122条序列。
1.3 生物信息学分析
为了获取样本测序序列在NCBI数据库中的相似序列,本研究利用基于局部比对算法的搜索工具(BLAST)进行同源序列检索,利用MUSCLE多序列比对程序确定各条序列的位点对应关系,利用MEGA等软件分析样品序列的亲缘关系。
1.3.1 数据库同源序列获取
为了检测已测序片段与已知序列的相似性,并确定其同源序列,使用序列相似性搜索程序BLASTN[14],设置查询参数,从GenBank数据库中提取同源序列。对搜索结果中同一样本的不同片段进行合并以剔除重复结果,只保留物种属于羊肚菌属的序列进行进化分析。对于 ITS 基因,从SD样本和DM样本共检索到422个不同物种的序列。另外3个基因RPB1、RPB2、EF1-α检索到的同源序列分别为142、156、127条。
1.3.2 基于索引号的羊肚菌测序序列获取
一些研究人员从事羊肚菌相关研究,其样本的测序序列也已被上传到GenBank数据库[4,15-18],总共获得162个序列ID。笔者使用shell脚本编程,并借助查询工具E-utilities (https://www.ncbi.nlm.nih.gov/home/tools/)下载相关的序列,过滤后,共得到110个样本,包含所有4个基因序列。
1.3.3 多序列比对
使用MUSCLE[19]来进行多序列比对;使用BioEdit 7.2.6进行序列编辑操作;使用Gblock(http://phylogeny.lirmm.fr/phylo_cgi/)进行位点选择[3]。
1.3.4 序列替代模型选择
用IQ-tree中的ModelFinder[20]来找到最佳核苷酸替换模型。该模型基于Bayesian信息准则(BIC)进行模型参数的筛选。ITS-rpb1-rpb2-ef1α的最佳DNA替换模型为K2P+G4,表示碱基的转换、颠换有不同的频率。进化树的构建和可视化采用MEGA软件[21]。
1.3.5 系统发生树的构建
采用基于Tamura-Nei模型[22]的最大似然方法进行分子系统发育分析。算法根据一个初始树进行启发式搜索,保留似然性较好的系统发生树作为最终结果。根据每个位置的替换次数得到树中的分支长度,并用1 000次bootstrap重复估计系统发生树中每个节点的一致性。
2 结果与分析
2.1 基于RPB1基因的羊肚菌亲缘关系
对22个样品进行PCR扩增和DNA测序,成功获得ITS、RPB1、RPB2、EF1-α、LSU序列。对低质量测序結果过滤后,共得到122个片段,总长度为76 kb。
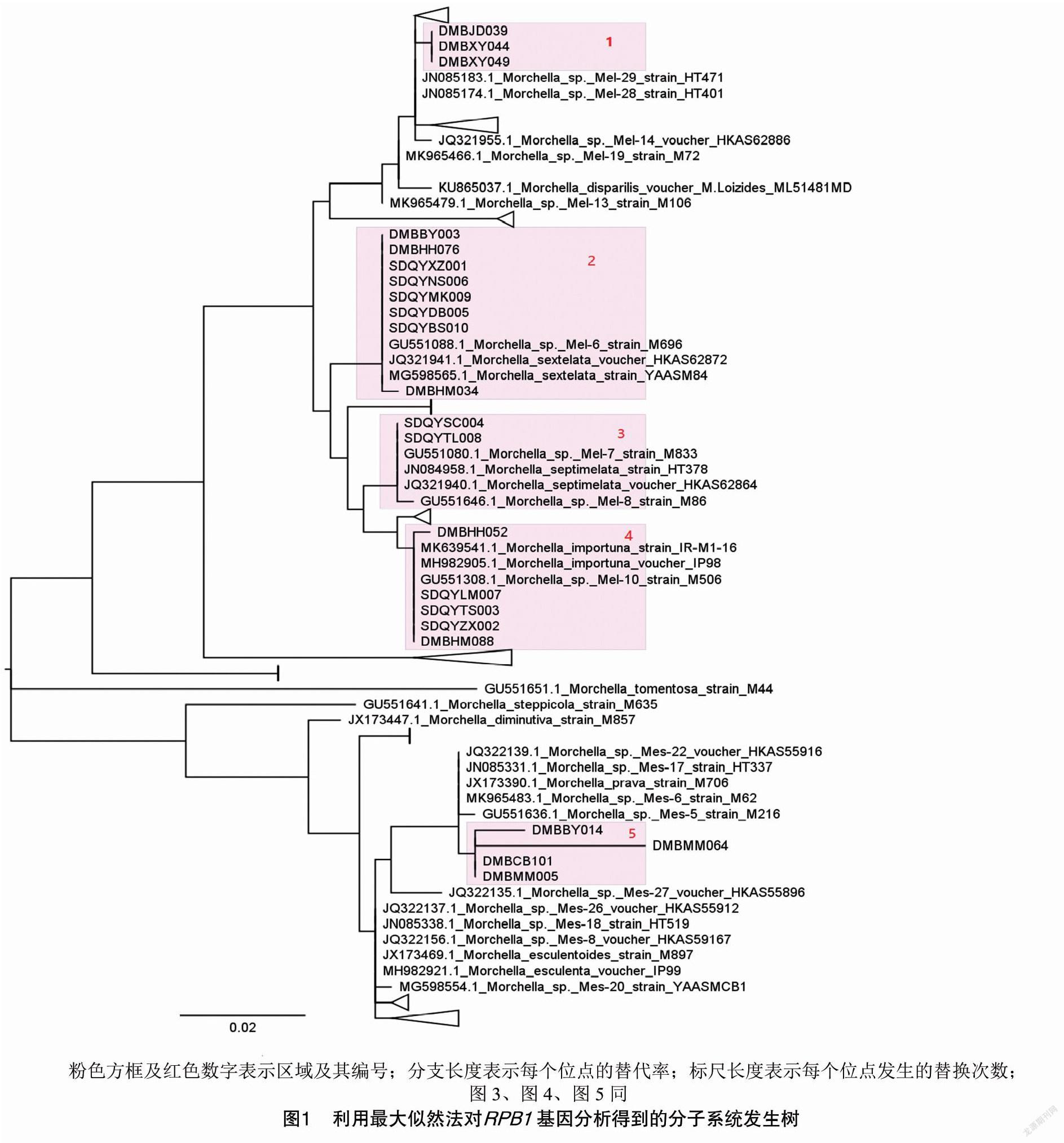
RPB1基因在每个样品中都被成功测序,用它查询NCBI数据库,得到多个羊肚菌属的同源序列。经过筛选,并对于每个羊肚菌品系保留1~2条序列,然后用MUSCLE程序构建多序列比对。利用MEGA和最大似然法进行进化分析,最终得到了1棵包含22个样本和111个已知物种或菌株同源序列的羊肚菌系统发生树。初步确定了样品与已知羊肚菌品种之间的进化关系,并使用FigTree 1.4.4 (http://tree.bio.ed.ac.uk/software/figtree/)来显示进化树,体现品种之间的类别关系(图1)。
如图1所示,在区域1中可以发现样本DMBJD039、DMBXY044和DMBXY049具有非常高的同源性关系,与sp._Mel-29和sp._Mel-28非常接近。从形态上,这3个品种区分为尖顶羊肚菌和小羊肚菌。
在区域2中,含有DMBBY003、DMBHH076、DMBHM034的分支被鉴定为六妹羊肚菌。栽培株SDQYXZ001(图2)、SDQYNS006、SDQYMK009、SDQYDB005和SDQYBS010也存在于该分支。这些栽培株来自西藏林芝、湖北恩施、青海班玛、甘肃迭部、青海互助等地,表明此品种羊肚菌具有较高的适应性。
在区域3中,栽培菌株SDQYSC004和SDQYTL008分别来自四川金堂和青海西宁。它在进化上与同七妹羊肚菌相似,具有较高的同源性。
在区域4中,3个栽培株SDQYLM007、SDQYTS003、SDQYZX002和2个野生菌株DMBHH052、DMBHM088与梯棱羊肚菌具有较高的同源性。SDQYLM007、SDQYTS003、SDQYZX002分别来自青海西宁、甘肃天水、甘肃卓尼。这表明梯棱羊肚菌也适宜在2 000 m的高海拔地区栽培。
区域5中,DMBBY014、DMBMM064、DMBCB101和DMBMM005与sp._Mes-5和sp._Mes-6最接近,但分支较长,表现出种间的差异。从形态上,4个品种有所区别,被认为是半圆羊肚菌、羊肚菌、粗柄羊肚菌等,因此,有必要进一步确定其种类信息。
由于RPB1基因在多数样本中具有较高同源性,一些样本不能很好地区分,需借助更多基因序列进行鉴定。
2.2 基于多基因序列组合的样本亲缘关系分析
本研究对SD样本(表1)和DM样本(表2)均进行多基因测序,以此进行多基因综合分析。对所有样本先进行多序列比对(multiple sequence alignment,简称MSA),再将单个基因MSA结果合并成多基因MSA结果,以确定样本之间的关系。
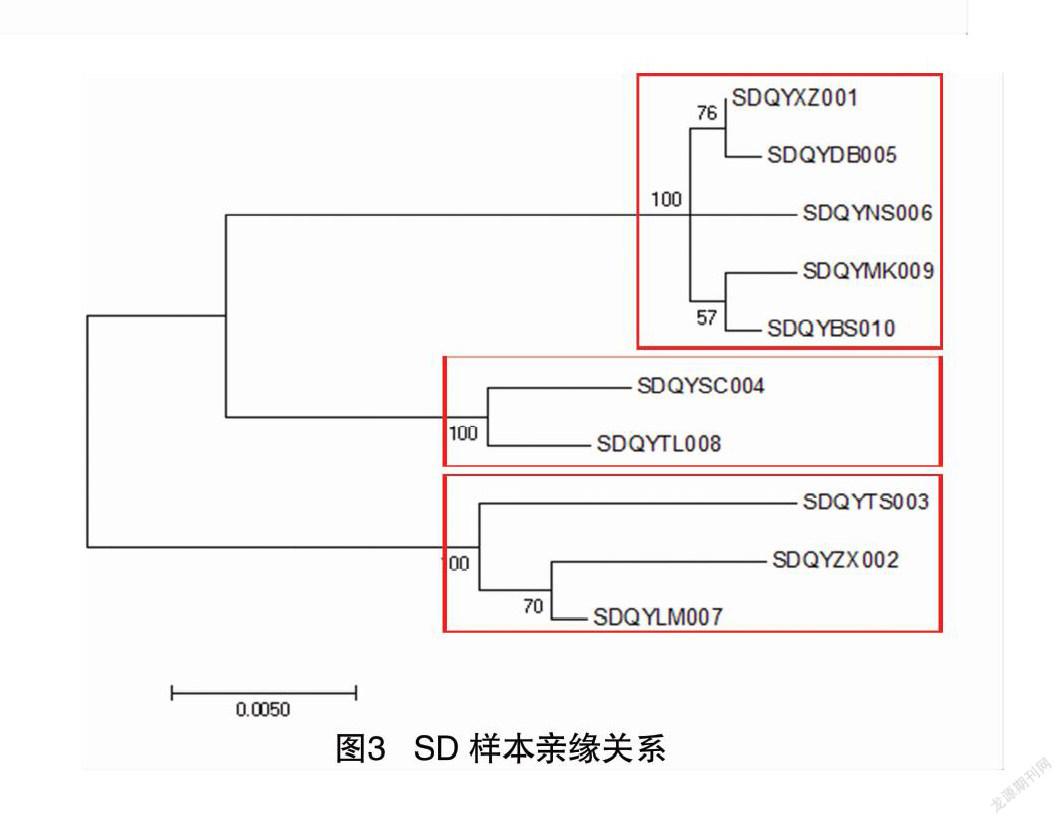
10个SD样本扩增出RPB1和 EF1-α 基因,并获得了测序结果。因此,笔者构建了包含这2个基因的MSA,并选择Tamura-Nei模型作为DNA替代模型构建进化树,结果如图3所示。
单基因分析提供的分辨率不高,很难区分物种之间的详细差异,而通过双基因进化分析,可以清楚地看到样本之间存在差异。在图3中,这棵树可以分为3个分支,分别属于3个不同的物种。各分枝间样品间也存在一定的进化距离,说明在青海成功培植的羊肚菌种也有种间差异。
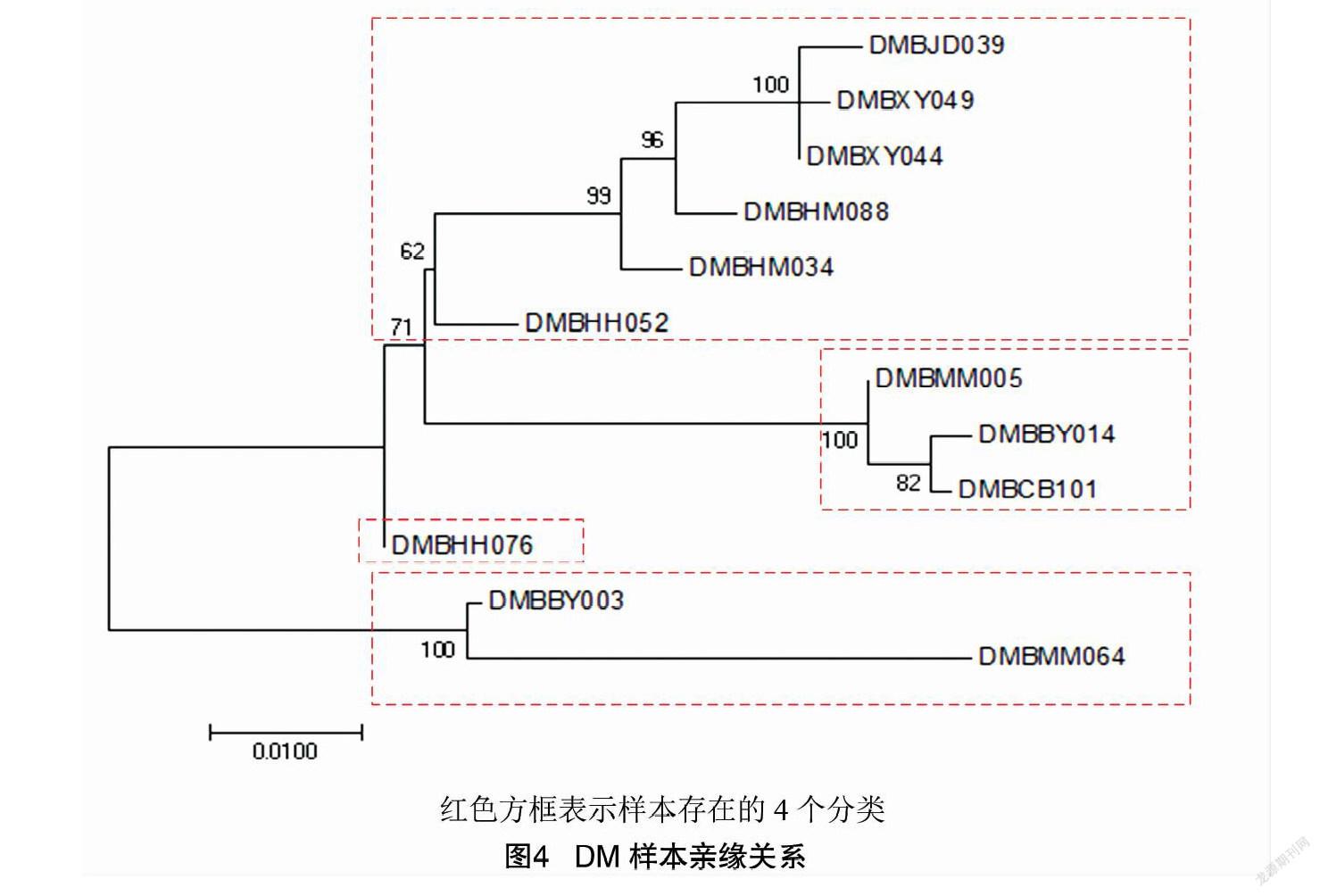
对于野生型DM样本,所有樣本中成功测序的基因有3个,分别是RPB1、RPB2、LSU。利用这些基因的MSA构建进化树,如图4所示。
12个DM样品在形态上有很大差异,说明它们属于不同的物种。从进化树中可以看到4个分支,每个分支与其他分支之间的距离都很大,所以笔者认为其中有超过4个不同的物种。特别是,样本DMBMM064有一个更长的分支,这意味着该样本与其他样本差异更大。
2.3 基于多基因数据库搜索的样本品系推测
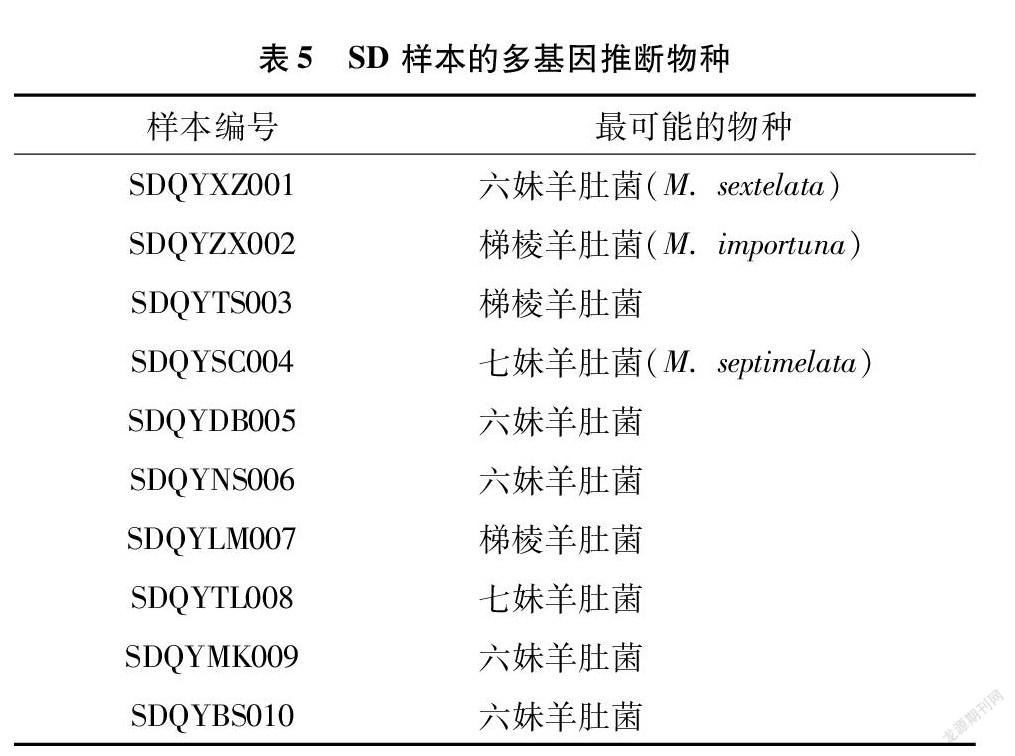
通过在数据库中搜索单个基因序列来识别物种,将会由于序列相似性而检索出多个不同结果,这给确定样本的种类带来了巨大困难。因此,本研究结合了各样本中多个基因的序列相似性搜索结果以及形态学特征进行了样本推断。使用BLAST来进行同源序列搜索,在查询参数中,设置物种限定为羊肚菌属( Morchella ),然后提取每个查询结果的物种信息,并根据每个样本的多基因搜索结果确定羊肚菌品种,获得最接近的物种(表5、表6)。
2.4 同已文献报道羊肚菌品系的关系
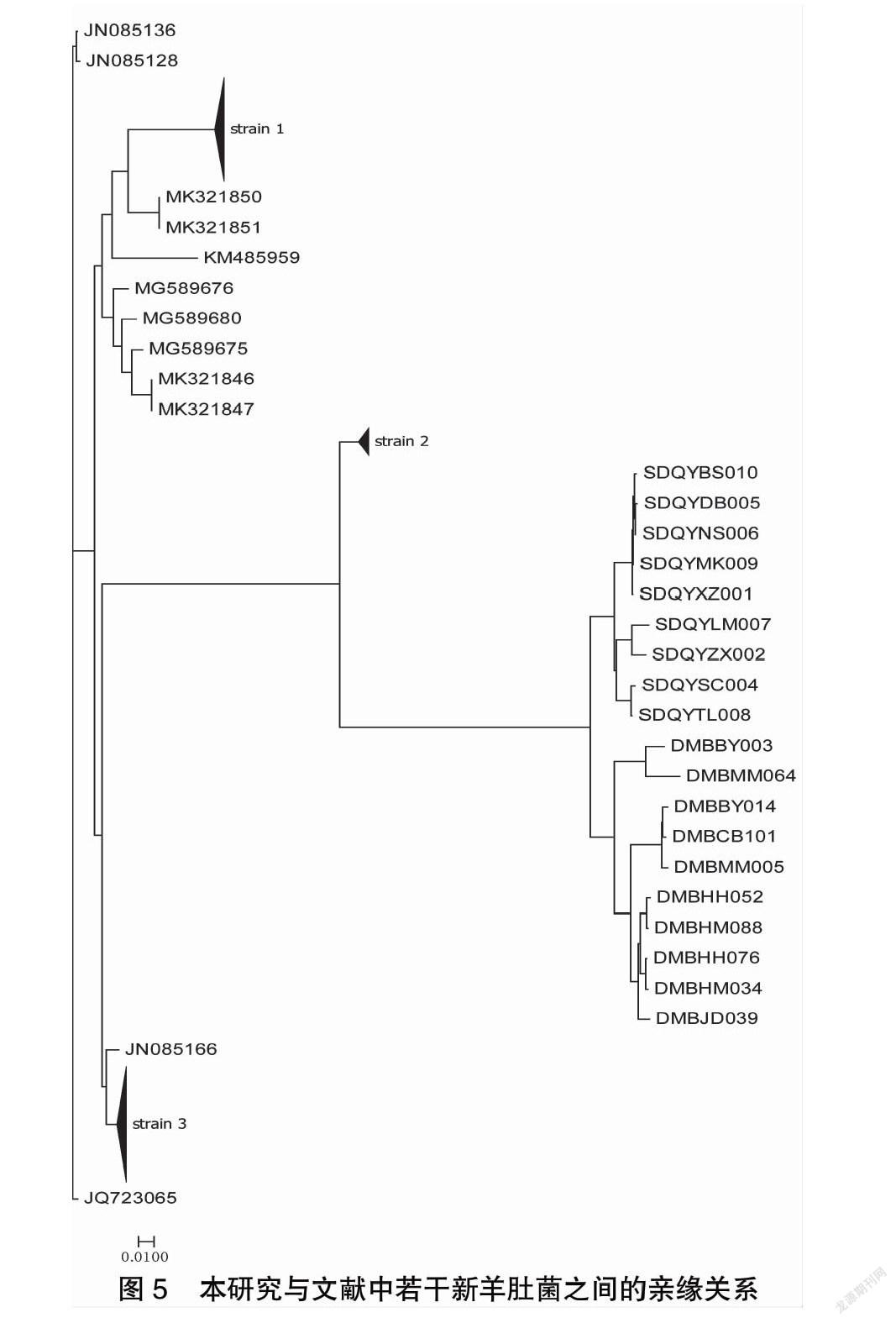
为了研究青海羊肚菌与其它报道品种之间的关系,从文献检索到序列的索引号,并从GenBank提取了162个样本的基因序列。在本研究的样本中,有20个包含同文献匹配的4个基因序列区域。经过序列比对和分子进化分析,得到进化树(图5)。
可以看出,本研究的样品与报道的羊肚菌的遗传距离较大。这也说明青海互助县北山野生羊肚菌具有相对独立的进化系统,且存在种群隔离的趋势。DM样品是野外采集的野生样品,可以看出DM样品之间的差异大于SD样品。
2.5 典型的序列差异
为了识别样本序列和现有已知羊肚菌品种的序列差异,笔者进行了序列比较。对于RPB1基因,合并了133个羊肚菌同源序列,它们是SD样本、DM样本和从数据库检索得到的同源序列。MSA比较结果发现,DMBMM064与其他序列有相对较大的遗传差异。将此序列与最接近的样本序列JQ322139.1进行了比较,获得点阵图分析结果如图6所示,红圈中的线是不连续的,这意味2个样本中的RPB1基因序列不同,表明2个样本有很大的进化距离。
如图7所示,该样本序列同数据库序列的比对结果,是匹配度最高的结果,尚且存在一些差异,显示出本样本独特,其功能和特征还需进一步的探索。
3.6 形态和地理分布
本研究发现,青海野生羊肚菌有较高的序列多样性,与其他已知的羊肚菌品种相比,包含DMBBY014、DMBMM064和DMBMM005的分支,有更高的序列差异和较长的进化距离,可能包含新的品系和菌株,较高的品系多样性,为羊肚菌的栽培和推广提供较好的遗传资源。
3 讨论与结论
在本研究收集了22个羊肚菌样本,并为每个样本进行了5个基因区域的测序。根据样本的测序结果,结合从GenBank数据库下载的同源序列,并进行进化分析,探索它们之间的差异。结果表明,栽培样本包括3个主要品种,分别是六妹羊肚菌、七妹羊肚菌、梯棱羊肚菌,这些品种均属于黑色羊肚菌支系,是我国食用菌栽培行业发展迅速的一类真菌,具有重要的经济和科研价值[23]。栽培样本的DNA序列同数据库已有条目相比,存在一定程度的序列变异,表明具有一定序列变异的品种,已能够很好地适应当地种植环境,某些位点的变化不会影响羊肚菌生长的适应性。
对于野生羊肚菌样本,它们同已知的物种有更高的序列位点差异,在野生品种之间也有较大的遗传距离,这表明它可能含有新的羊肚菌菌株或新的羊肚菌品种。与此同时,这些野生样本收集自较小范围的特定地理区域,该区域同其他羊肚菌的生长区域相隔很远,这也体现出特定的环境有助于进化出这些新的羊肚菌菌株。
序列比较也发现了样本序列和数据库条目之间的一些差异。例如,DMBMM064与所有已知物种的遗传距离相对较大,这表明它经历了较长的分化时期,可能是新的羊肚菌品种。多方面的全面分析将非常有必要。全基因组测序和基因功能研究将有助于发现新品种及其功能特征,这对培养野生的新菌株也非常重要。
本研究初步鉴定了22个适合在中国北方生长的羊肚菌样本,扩大了笔者对羊肚菌属的多样性和分布特点的了解,这对于选育和栽培羊肚菌新品种,获得经济效益,更深入、全面的分子水平分析将非常有帮助。
参考文献:
[1]Du X H,Zhao Q,O Donnell K,et al. Multigene molecular phylogenetics reveals true morels ( Morchella ) are especially species-rich in China[J]. Fungal Genetics and Biology,2012,49(6):455-469.
[2]刘 伟,张倩倩,舒 芳,等. 梯棱羊肚菌全基因组SNP/Indel分析及基于Indel标记的遗传连锁图谱构建[J]. 菌物学报,2019,38(12):2195-2204. .
[3]Chai H M,Chen W M,Zhang X L,et al. Structural variation and phylogenetic analysis of the mating-type locus in the genus Morchella [J]. Mycologia,2019,111(4):551-562.
[4]Cai Z N,Li W,Mehmood S,et al. Structural characterization, in vitro and in vivo antioxidant activities of a heteropolysaccharide from the fruiting bodies of Morchella esculenta [J]. Carbohydr Polym,2018,195:29-38.
[5]王 龙,周庆平,刘建明. 基于rDNA-ITS序列的甘肃甘南野生羊肚菌遗传多样性分析[J]. 江苏农业科学,2020,48(17):81-84.
[6]Zhu D. Research progress on artificial cultivation of Morchella [J]. Edible Fungi of China,2008,27(4):3-5.
[7]刘春丽,刘绍雄,李建英,等. 羊肚菌新品种“中菌羊肚菌1号”选育[J]. 中国食用菌,2020,39(10):22-26,36.
[8]Kuo M,Dewsbury D R,O Donnell K,et al. Taxonomic revision of true morels ( Morchella ) in Canada and the United States[J]. Mycologia,2012,104(5):1159-1177.
[9]Clowez P,Les Morilles. Une nouvelle approche mondiale du genre Morchella [J]. Bulletin de la Société Mycologique de France,2012,126:199-376.
[10]O Donnell K,Rooney A P,Mills G L,et al. Phylogeny and historical biogeography of true morels ( Morchella ) reveals an Early Cretaceous origin and high continental endemism and provincialism in the Holarctic[J]. Fungal Genetics and Biology,2011,48(3):252-265.
[11]Du X H,Zhao Q,Yang Z L,et al. How well do ITS rDNA sequences differentiate species of true morels ( Morchella )?[J]. Mycologia,2012,104(6):1351-1368.
[12]Baroni T J,Beug M W,Cantrell S A,et al. Four new species of Morchella from the Americas[J]. Mycologia,2018,110(6):1205-1221.
[13]Voitk A,Beug M W,O Donnell K,et al. Two new species of true morels from Newfoundland and Labrador:cosmopolitan Morchella eohespera and parochial M. laurentiana [J]. Mycologia,2016,108(1):31-37.
[14]Altschul S F,Madden T L,Schffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):3389-3402.
[15]Pildain M B,Visnovsky S B,Barroetavea C. Phylogenetic diversity of true morels ( Morchella ),the main edible non-timber product from native Patagonian forests of Argentina [J]. Fungal Biology,2014,118(9/10):755-763.
[16]Phanpadith P,Yu Z D,Li T. High diversity of Morchella and a novel lineage of the Esculenta clade from the north Qinling Mountains revealed by GCPSR-based study[J]. Scientific Reports,2019,9:19856.
[17]Richard F,Bellanger J M,Clowez P,et al. True morels ( Morchella ,Pezizales) of Europe and North America:evolutionary relationships inferred from multilocus data and a unified taxonomy[J]. Mycologia,2015,107(2):359-382.
[18]Du X H,Wu D M,He G Q,et al. Six new species and two new records of Morchella in China using phylogenetic and morphological analyses[J]. Mycologia,2019,111(5):857-870.
[19]Edgar R C. MUSCLE:multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research,2004,32(5):1792-1797.
[20]Minh B Q,Schmidt H,Chernomor O,et al. IQ-TREE 2:new models and efficient methods for phylogenetic inference in the genomic era[J]. Molecular Biology & Evolution,2020,37(5):1530-1534.
[21]Kumar S,Stecher G,Li M,et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology & Evolution,2018,35(6):1547-1549.
[22]Tamura K,Nei M.Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees[J]. Molecular Biology & Evolution,1993,10(3):512-526.
[23]杜習慧. 黑色羊肚菌支系的物种资源、生殖模式和遗传多样性研究进展[J]. 菌物研究,2019,17(4):240-251.

