紫肉甘薯及其突变体花青素积累差异的比较转录组分析
后猛 李臣 宋炜涵 张允刚 李强


摘要:紫肉甘薯因其塊根中富含紫色花青素而得名。本研究以2组栽培种紫肉甘薯及其淡黄肉突变体为材料,基于甘薯转录组测序(RNA-seq)数据进行生物信息学分析。以|log2 FC|≥1且错误发现率(FDR)<0.01作为筛选标准,鉴定差异表达基因(DEGs),对DEGs进行GO功能分类和KEGG代谢通路分析,并采用实时荧光定量PCR(qRT-PCR)方法对测序结果进行验证。样品经转录组测序后,均获得超过8 Gb的高质量数据,拼接组装共得到164 427条Unigenes。在2组甘薯材料及其突变体中分别检测到2 262个和1 546个DEGs,其中有244个DEGs是2组甘薯材料共有的。GO功能分析发现,DEGs主要富集于苯丙氨酸解氨酶活性、着色、类黄酮生物合成过程、含花青素化合物生物合成过程等。KEGG通路分析发现,DEGs主要分布在苯丙素类生物合成、苯丙氨酸代谢、类黄酮生物合成和花青素生物合成等代谢通路。随机选择27个与花青素生物代谢相关的候选基因进行实时荧光定量PCR验证,表达模式与测序结果一致。将2组材料中候选基因的表达水平与花青素含量进行相关性分析,结果表明,花青素生物合成相关的候选基因PAL-1、CHS、ANS、CCoAOMT、GST-2、ABC-1、bHLH和WRKY-2的相对表达量与花青素积累量的相关性达到显著或极显著水平。表明,2个甘薯突变体材料块根薯肉紫色的缺失是由花青素的合成、转运和相关转录因子表达水平的降低共同引起的。通过转录组测序可以从中挖掘一些与花青素代谢相关的候选基因,为研究其在生物合成途径中的调控机制奠定基础。
关键词:紫肉甘薯;转录组测序;花青素代谢;差异表达基因
中图分类号:S531文献标识码:A文章编号:1000-4440(2022)02-0313-13
Comparative transcriptome analysis on anthocyanin accumulation differences in purple-fleshed sweetpotatoes and their mutants
KOU Meng LI Chen ,SONG Wei-han ZHANG Yun-gang LI Qiang
Abstract:Purple-fleshed sweetpotato (PESP) is famous for the rich storage of purple anthocyanins in its storage root. In this study, two groups of PFSP cultivated species and their mutants with light yellow flesh were used as materials, and bioinformatics analysis was conducted based on transcriptome sequencing (RNA-seq) data of sweetpotatoes.|log2FC |≥1 and FDR (False discovery rate)<0.01 were used as screening standards to identify differentially expressed genes (DEGs). Gene ontology (GO) functional classification and KEGG metabolic pathway analysis of DEGs were performed, and real-time quantitative PCR (qRT-PCR) method was used to verify the sequencing results. After transcriptome sequencing of the samples, more than eight Gb data with high quality were obtained for each sample, and a total of 164 427 Unigenes were obtained by splicing and assembling. 2 262 and 1 546 DEGs were detected respectively in two groups of sweetpotato materials and their mutants, among which 244 DEGs were shared between two groups of sweetpotato materials. Results of GO functional analysis showed that, DEGs mainly enriched in phenylalanine ammonia-lyase activity, pigmentation, flavonoid biosynthesis process and anthocyanin-containing compound biosynthesis process. Results of KEGG pathway analysis showed that, DEGs mainly distributed in metabolic pathways such as phenylpropanoid biosynthesis, phenylalanine metabolism, flavonoid biosynthesis and anthocyanin biosynthesis. In addition, 27 candidate genes related to anthocyanin biological metabolism were selected randomly for real-time quantitative PCR verification, and their expression patterns were found to be consistent with the sequencing results. Results of correlation analysis on gene expression level and anthocyanin content revealed that, relative expression levels of anthocyanin biosynthesis related candidate genes such as PAL-1, CHS, ANS, CCoAOMT, GST-2, ABC-1, bHLH and WRKY-2 were in significant or extremely significant correlation with anthocyanin accumulation. The results indicated that, the deficiency of purple color in the storage root flesh of two sweetpotato mutant materials was caused by the decrease of expression level of genes involved in anthocyanin synthesis, transport and related transcription factors. Some candidate genes related to anthocyanin metabolism can be found by transcriptome sequencing, which lays a foundation for study on their regulatory mechanism in biosynthesis pathways.
Key words:purple-fleshed sweetpotato;transcriptome sequencing;anthocyanin metabolism;differentially expressed genes
江苏农业学报2022年第38卷第2期
后猛等:紫肉甘薯及其突变体花青素积累差异的比较转录组分析
紫肉甘薯是一类特异的甘薯类型,除含有多种对人体有益的微量元素和矿物质外,还富含具有抗氧化能力、防癌抗癌等生理保健功效的天然花青素,因而受到越来越多消费者的欢迎[1-3]。与其他同类色素相比,从紫肉甘薯中提取出的花青素具有更为稳定的理化性质[1],是一种优质的天然食用色素,具有多种营养保健用途和药理功能[2-3]。因此,探讨花青素在紫肉甘薯块根中代谢相关基因的差异表达及调控机制具有重要意义。植物体内的色素主要包括类黄酮、甜菜碱类色素和类胡萝卜素等[4],其中花青素是属于类黄酮的一类天然水溶性色素,由特有的C6-C3-C6碳骨架结构和不同的取代基团构成,因其取代基数目和位置的不同而呈现出红、橙、粉和蓝等各种颜色[4-5]。花青素的生物合成是被广泛研究的植物次生代谢途径之一,花青素的生物合成与代谢过程一般由多种结构基因编码的关键酶、相关转运蛋白质以及起调控作用的转录因子等多个成员参与,目前已在拟南芥、金鱼草和玉米等植物中被深入研究[4-6]。甘薯是中国重要的粮食作物,也是一种重要的能源和保健作物。随着人们保健意识的增强和消费水平的提高,人们对自身健康问题越来越关注,日本、美国等发达国家已把甘薯视为营养全面而平衡的保健食品[7]。尽管已有较多关于紫肉甘薯花青素合成和积累的研究,但这些研究多集中在合成关键酶编码基因方面,调控花青素代谢的分子机制仍不够清楚。本研究以2个紫肉甘薯品系及其淡黄肉自然突变体为材料,进行转录组分析,为进一步挖掘花青素代谢相关基因及功能研究提供依据。
1材料与方法
1.1试验材料及RNA提取
本课题组在育种过程中发现2个甘薯肉色自然突变体,徐1604(紫皮紫肉)的突变体徐1604M(淡红皮淡黄肉),徐0571(紫皮紫肉)的突变体徐0571M(淡红皮淡黄肉)。经过多年田间鉴定,这2个突变体薯肉颜色等性状可以稳定遗传。将这两组栽培种及其突变体材料栽插在江苏徐淮地区徐州农业科学研究所试验田,于栽插后130 d收获薯块(图1)。每个品种随机选取12个中等大小的薯块,将其中4个薯块作为一个生物学重复,共计3个重复,分别为:A1、A2和A3,B1、B2和B3,C1、C2和C3,D1、D2和D3。每个重复清洗擦干后去皮,切碎,混匀,锡箔纸包裹后迅速置于液氮中速冻,然后一部分样品放于超低温冰箱中-80 ℃保存,用于后续试验。另一部分样品提取总RNA后,使用Nanodrop-1000检测RNA的纯度(OD260/OD280)和浓度,并用Agilent 2100生物分析仪(安捷伦科技有限公司产品)检测RNA的完整性。采用高闰飞[8]的方法测定花青素含量。
1.2cDNA文库构建及转录组测序
样品检测合格后,构建cDNA文库,随后检测文库质量。检测结果合格后测序,并对原始数据(Raw data)进行数据过滤。获得高质量的测序数据后,使用Trinity软件将Clean Data进行序列组装,最终得到Unigene库。
1.3Unigene功能注释与分类
采用BLAST软件将本研究获得的甘薯Unigene序列与NR(Non-redundant protein sequences in NCBI)、GO(Gene ontology)、Swiss-Prot(A manually annotated and reviewed protein sequence database)、KOG(eu Karyotic ortholog groups)、KEGG(Kyoto encyclopedia of genes and genomes)等数据库对比,以获得Unigene的注释信息。
1.4花青素代谢途径差异表达基因筛选
基因表达具有一定的时空特异性,不同条件下表达水平具有显著差异的基因或转录本,被称为差异表达基因(DEG),或差异表达转录本(DET)。根据样品间表达水平高低,DEG(或DET)可以划分成上调基因和下调基因。通过比对2个甘薯材料的Unigene序列,我们筛选出与花青素代谢相关的DEG(或DET)。
1.5花青素代谢途径差异表达基因验证
1.5.1甘薯总RNA提取采用多糖多酚植物RNA提取试剂盒(北京华越洋生物科技有限公司产品)提取徐1604、徐1604M、徐0571、徐0571M的块根总RNA,具体操作参考试剂盒说明书进行。使用1.2%甲醛变性琼脂糖凝胶电泳验证提取的RNA的完整性,用NanoDrop-1000微量分光光度计测定RNA的浓度和OD260/OD280。
1.5.2cDNA第一条链合成cDNA合成使用ReverTra Ace qPCR RT Master Mix with gDNA Remover(TOYOBO公司产品)试剂盒,操作按照试剂盒说明书进行。
1.5.3实时荧光定量PCR(qRT-PCR)验证根据转录组测序数据,采用Primer3Plus在线软件(http://www.primer3plus.com/cgi-bin/dev/primer3plus.cgi)设计引物,引物序列见表1。参照2×Q3 SYBR qPCR Master Mix(TOLOBIO公司产品)试剂盒说明书,使用QuantStudio 6 Flex荧光定量PCR儀进行qRT-PCR检测,反应体系为:2×Q3 SYBR qPCR Master Mix 10 μl,模板2 μl,引物-F(10 mmol/L)和引物-R(10 mmol/L)各1 μl,ddH2O补足至20 μl。
反应程序为:95 ℃ 变性3 min;95 ℃ 10 s,60 ℃ 30 s, 40个循环。溶解曲线为:95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s,并检测荧光信号值。以IbARF为内参基因[9],用2-△△Ct法[10]计算相对表达量,每1个样品均设置3个重复。
1.6数据处理
采用Microsoft Excel进行数据的方差分析和相关性分析。
2结果与分析
2.1不同甘薯材料总花青素含量的差异
由图2可以看出,每100 g紫肉甘薯材料徐1604和徐0571花青素含量分别为(52.75±0.08) mg和(19.91±0.10) mg,而淡黄肉突变体材料徐1604M和徐0571M未检测出花青素。结果表明,2组自然突变体与其紫肉栽培种薯块肉色的差异主要表现在花青素含量不同。
2.2转录组测序与组装
采用Illumina HiSeqTM2000高通量测序技术对2个甘薯材料及其突变体块根进行转录组测序,从表2中可以看出,经过测序质量控制,徐1604、徐1604M、徐0571、徐0571M分别获得超过8.32 Gb、10.05 Gb、8.67 Gb和9.73 Gb clean data,Q30(碱基识别出错概率)分别达到92.77%、90.56%、92.86%和93.27%以上,所有样品G+C含量均不低于46.42%,说明本研究获得的甘薯转录组序列数据充足且质量较高,能够满足后续试验的需要。
为避免低表达基因的测序量对构建完整转录本的影响,对低表达的转录本进行过滤,最终得到可靠的转录本。利用Trinity软件对测序序列进行拼接(表3),获得183 440个转录本,总长度达91 725 016 bp,平均长度为500 bp,N50为604 bp;进一步组装共得到164 427条Unigenes,总长度达77 679 292 bp,平均长度为472 bp,N50为544 bp,最大长度为16 835 bp,最小长度为201 bp,结果表明,该甘薯转录组测序和组装结果良好,可用于后续生物信息学研究。
2.3Unigene的功能注释
通过BLAST程序将甘薯Unigene序列与GO、KEGG、KOG、Pfam、Swiss-Prot 和NR等公共数据库进行比对,结果(表4)显示,注释到GO、KEGG、KOG、Pfam、Swiss-Prot、NR数据库中的Unigene分别为56 024个(占57.83%)、25 101个(占25.91%)、47 358个(占48.88%)、48 209个(占49.76%)、65 598个(占67.71%)、87 047个(占89.85%)。因此,在NR数据库中找到相似序列最多,其中300 bp≤长度<1 000 bp的Unigene有40 859个(占46.94%),长度≥1 000 bp的Unigene有12 874个(占14.79%);在KEGG数据库中找到相似序列最少,其中300 bp≤长度<1 000 bp的Unigene有11 632个(占46.34%),长度≥1 000 bp的Unigene有3 915个(占15.60%)。
2.4差异表达基因的比较分析
以∣log2FC∣≥1且FDR<0.01作为筛选标准,我们对2个甘薯材料及其突变体间的差异表达基因进行聚类分析。其中差异倍数(FC)表示两样品(组)间表达量的比值。错误发现率(FDR)是通过对差异显著性P值进行校正得到的。在徐1604 vs 徐1604M和徐0571 vs 徐0571M 2个比较组中分别检测到2 262个和1 546个差异表达基因。其中,在徐1604 vs 徐1604M对比组中,上调表达基因有836个,下调表达基因有1 426个;在徐0571 vs徐0571M对比组中,上调表达基因有453个,下调表达基因有1 093个;在徐1604 vs徐1604M和徐0571 vs 徐0571M 2个组之间的比较中,共有244个差异表达基因,其中上调表达基因12个,下调表达基因232个(图3)。
此外,我们还对2个对比组的DEGs进行了GO分类和KEGG路径分析。根据序列同源性,将DEGs分为生物学过程、细胞成分和分子功能3类。苯丙氨酸解氨酶活性是2个对比组中富集程度较高的GO项,其中与着色相关的5个基因在徐1604 vs 徐1604M中表达差异尤为明显;与类黄酮生物合成过程相关的22个基因以及与含花青素化合物生物合成过程相关的8个基因在徐0571 vs 徐0571M中表达差异尤为明显。选取排名前20位的差异表达基因的KEGG通路,确定了4条与块根肉色相关的通路,即苯丙素类生物合成、苯丙氨酸代谢、类黄酮生物合成和花青素生物合成。
2.5花青素代谢途径差异表达基因筛选及验证
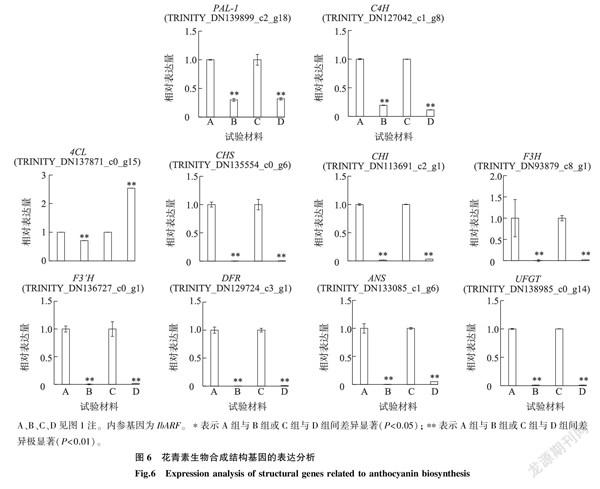
本研究共筛选出11个编码花青素生物合成途径相关的10种酶的Unigenes(图4)。这些假定的结构基因包括PAL(2 Unigenes)、C4H(1 Unigene)、4CL(1 Unigene)、CHS(1 Unigene)、CHI(1 Unigene)、F3H(1 Unigene)、F3′H(1 Unigene)、DFR(1 Unigene)、ANS(1 Unigene)、UFGT(1 Unigene)。这11个Unigenes在2組甘薯材料及其突变体(徐1604 vs 徐1604M、徐0571 vs 徐0571M)中呈现出相似的表达模式。在淡黄肉突变体材料徐1604M和徐0571M的块根中,所有编码花青素生物合成的DEGs都比其紫肉栽培种甘薯材料(徐1604和徐0571)中的表达水平明显降低。尤其是4CL(Unigene编号:TRINITY_DN137871_c0_g15 )、CHS(Unigene编号:TRINITY_DN135554_c0_g6)、CHI(Unigene编号:TRINITY_DN113691_c2_g1)、F3H(Unigene编号:TRINITY_DN93879_c8_g1)、F3′H(Unigene编号:TRINITY_DN136727_c0_g1)、DFR(Unigene编号:TRINITY_DN129724_c3_g1)、ANS(Unigene编号:TRINITY_DN133085_c1_g6)和UFGT(Unigene编号:TRINITY_DN138985_c0_g14),在徐1604 vs 徐1604M和徐0571 vs 徐0571M中的表达量下降幅度更大。除此之外,在突变体材料徐1604M和徐0571M的块根中,还发现1个与木质素生物合成相关的基因,咖啡酰辅酶A-O-甲基转移酶(CCoAOMT)(TRINITY_DN124453_c7_g2)编码基因的表达水平显著降低(图4)。
为了筛选调控花青素生物合成的转录因子,从2组材料共有的244个DEGs中筛选到104个编码转录因子(TFs)的候选DEGs。由图5可知,筛选得到的转录因子中以NAC、MYB、C2H2、bHLH家族成员为主,其次是AP2/ERF、B3、LOB等转录因子家族成员。在104个TFs中,通过转录组注释分析,发现有11个TFs被注释为可能参与花青素生物代谢(图4)。MYB(Unigene编号:TRINITY_DN78368_c0_g1)在栽培种徐1604和徐0571中的转录水平,分别是突变体徐1604M和徐0571M的453.68倍和35.24倍,而bHLH(Unigene编号:TRINITY_DN140405_c1_g7)在栽培种徐1604和徐0571中的转录水平,分别是突变体徐1604M和徐0571M的1 682.92倍和33.71倍。此外,注释为NAC、WRKY、ERF和LOB的TFs在徐1604 vs 徐1604M和徐0571 vs 徐0571M 2个比较组中的表达也发生了显著下調。
本研究鉴定到5个编码花青素转运的基因(图4),分别为2个GST、1个MATE和2个ABC,在紫肉甘薯材料徐1604和徐0571中的表达水平都分别高于其淡黄肉突变体徐1604M和徐0571M,尤其是其中的1个GST(Unigene编号:TRINITY_DN128107_c8_g3)、1个MATE(Unigene编号:TRINITY_DN118813_c0_g1)和1个ABC(Unigene编号:TRINITY_DN135734_c2_g2),在徐1604 vs 徐1604M和徐0571 vs 徐0571M中的表达量下降幅度均较大。
以上结果表明,2组甘薯材料及其突变体块根薯肉颜色表型的差异是由花青素的合成、转运和转录因子调控共同引起的。为了验证转录组数据的可靠性,本研究选择了27个与花青素生物合成相关的候选DEGs进行qRT-PCR检测,结果表明,除4CL和MATE外,所选择的DEGs表达水平与转录组测序数据基本一致,包括10个花青素合成相关的结构基因(图6),1个木质素合成相关基因(图7),5个花青素合成转运蛋白质编码基因(图8),以及11个花生素生物合成转录因子编码基因(图9)。因此,本研究中的转录组测序数据是可信的,而qRT-PCR与RNA-seq之间的相关性也较高(R2=0.624 4)(图10)。此外,将这27个候选基因的相对表达量与块根花青素含量进行相关性分析。结果显示,除MATE外,其他与花青素生物合成相关的候选基因的相对表达量均与花青素含量呈正相关,其中PAL-1、CHS、ANS、CCoAOMT、GST-2、ABC-1、bHLH和WRKY-2的相对表达量与花青素含量的相关性达到显著或极显著水平(表5)。本研究结果证实了测序数据是可靠的,另一方面还可以从中挖掘一些新的与花青素代谢相关的候选基因,尤其是与花青素转运和积累相关的基因,如ABC-1和GST-2等。
3讨论
3.1甘薯转录组测序及Unigene功能注释分析
甘薯是世界上最重要的粮食作物之一[11-13]。由于其含有淀粉、膳食纤维、胡萝卜素等对人体有益的成分,因此被联合国粮农组织推荐为最健康的食物。随着保健意识的增强和消费水平的提高,人们对自身的健康问题越来越关注,紫肉甘薯因其块根富含花青素等具有生理保健功效的物质[14-16],受到越来越多消费者的喜爱。
近些年来,伴随着高通量测序技术的不断改进和运用,植物基因组学研究得到迅猛发展。而转录组测序技术因其测序准确度高、通量大、速度快、运行成本较低以及不需要任何基因信息等优点,已在多种无参考序列物种中得到广泛应用[17-19],尤其是在一些植物的颜色变化研究方面[20-22]。本研究采用Illumina高通量测序技术对不同类型甘薯进行转录组测序,分别获得超过8 G的原始数据,经组装后得到164 427个Unigenes,数量高于前人的测序结果[23-25]。Q30(>90%)、G+C含量(>46%)、N50(544 bp)等测序数据表明,测序结果较为可靠,可以满足后续转录组分析的要求。将本研究获取的甘薯Unigene与GO、KEGG、KOG、Pfam、Swiss-Prot、NR等数据库进行比对,分析可知共有96 882个(58.92%)Unigenes能够获得注释信息,但是仍有67 545个(41.08%)Unigenes未能得到注释,这可能与相关数据库基因功能注释信息匮乏、所得Unigene序列长度较短、甘薯中存在特有新基因等因素有关。
通过对2个甘薯材料的DEGs进行GO分类分析,其功能主要分为生物学过程、细胞成分和分子功能3类,尤其是在苯丙氨酸解氨酶活性、类黄酮生物合成过程、与含花青素化合物生物合成过程相关等方面差异表达基因富集程度较高。甘薯块根花青素合成代谢涉及很多生化过程,本研究中244个差异表达基因参与多个KEGG代谢通路,主要包括苯丙素类生物合成、苯丙氨酸代谢、类黄酮生物合成、花青素生物合成、植物激素信号转导、淀粉和蔗糖代谢、植物-病原互作等。而参与到次生代谢生物合成通路的DEGs主要是与苯丙烷、类黄酮和花青素等生物合成相关的酶编码基因,本研究的测序结果将为发掘更多与花青素等黄酮类物质代谢相关的基因提供数据支撑。
3.2花青素合成相关差异表达基因的筛选及分析
了解块根花青素生物合成途径的分子机制,有助于辅助紫肉甘薯育种。前期研究结果表明,花青素含量与许多植物中花青素生物合成相关基因的表达有关,如山药[20]、马铃薯[26]、梨[27]和甜樱桃[28]。比较RNA-seq分析发现,与栽培种紫肉甘薯相比,突变体材料中有11个花青素合成相关结构基因(PAL 2个,C4H、4CL、CHS、CHI、F3H、F3′H、DFR、ANS和UFGT各1个)下调表达,这些基因的表达模式与此前报道的结果相似。例如,在5个不同甘薯品种中,IbANS和IbF3′H的表达水平与花青素的积累模式高度匹配[29-30];IbCHI和其他花青素生物合成相关基因(IbCHS、IbF3H和IbDFR)的表达模式与不同品种甘薯成熟块根中的花青素积累有关[31-32]。有趣的是,我们发现了不止1个注释为PAL的Unigenes与花青素生物合成相关,表明这些Unigenes代表了1个基因家族的不同成员或单个转录本的不同片段[33]。另外,我们还发现4CL在徐0571 vs 徐0571M中的表达水平与转录组测序结果不同,这可能是由于4CL是植物类黄酮类化合物等生物合成途径的一种关键酶,除参与花青素合成外,还可能在木质素等多种次生代谢物合成方面发挥重要作用。在苯丙烷类生物合成途径中,除花青素合成相关基因外,还有与木质素合成相关的关键基因。根据测序结果,本研究鉴定出1个参与木质化的Unigene TRINITY_DN124453_c7_g2(注释为CCoAOMT),它在突变体块根中表达水平较低,其具体功能需要进一步研究。
3.3花青素代谢调控转录因子差异表达基因的筛选及分析
花青素代谢途经是由MYB-bHLH-WD40(MBW)转录因子复合物控制的[34-35]。本研究中,我们从DEG数据中发现1个候选MYB基因,它在非紫肉突变体材料中明显下调表达,与已报道的R2R3 MYB转录因子编码基因IbMYB1最为相似。研究结果表明,在拟南芥[36]、烟草[37]和甘薯[38]中过表达IbMYB1,诱导了花青素结构基因的表达,进而增强了转基因植物的花青素色素沉积。 此外,在紫肉甘薯块根中,发现1个bHLH上调表达,此基因表达量与花青素含量呈极显著正相关。然而,本试验中没有发现任何WD40的转录水平在紫肉甘薯品种及其突变体之间存在明显差异。
除MBW复合体外,其他TFs也被认为在控制花青素积累方面发挥了重要作用,如猕猴桃的SVP3(一种MADS结构域TF的编码基因)[39]、桃的BL (一种NAC TF的编码基因)[40]、苹果的MdHB1[41] (一种HD-Zip TF的编码基因)和MdERF1B[42](一种AP2/ERF TF的编码基因)、番茄的SlHY5[43](一种bZip TF的编码基因)。最后我们从244个DEGs中选出3个NACs、2个WRKYs、3个ERFs和1个LOB,这些转录因子均在突变体材料中相对于紫肉甘薯下调表达,尤其是WRKY-2编码基因的表达水平与花青素的积累量呈显著正相关。因此,这些TFs可能被认为是调控花青素合成的新的候选基因,其功能值得进一步研究。
3.4花青素转运差异表达基因的筛选及分析
花青素转运的分子机制已在多种植物中有所报道。在众多花青素转运体编码基因中,GSTs在花青素运输和积累过程中发挥着关键作用,如荔枝LcGST4[44]、草莓FvRAP[45]、苹果MdGSTF6[46]、猕桃AcGST1[47]、桃PpRiant1[48]。前人从葡萄中分离出5个GST编码基因(VviGST1~VviGST5)[49],结果顯示,VviGST4与花青素和原花青素(PAs)的积累均相关,而VviGST3仅参与PAs的转运[50]。研究结果表明,与突变体相比,紫肉甘薯徐1604和徐0571中2个GST编码基因的转录水平上调,相关性分析结果也表明花青素含量与其中1个GST编码基因(Unigene号:TRINITY_DN134429_c6_g32)的表达水平相关性极显著。此外,MATE和ABC转运体也与花青素的转运有关[51-52]。本研究还从DEGs中鉴定出1个MATE和2个ABC,它们在紫肉品种中表达水平显著上调,尤其是ABC-1(Unigene号:TRINITY_DN135734_c2_g2)的表达量与花青素含量呈极显著正相关。我们还发现MATE编码基因在徐0571 vs 徐0571M中的表达模式与转录组数据有所不同,这可能是由于MATE是植物中一个重要的转运蛋白质,其除了转运花青素等类黄酮类化合物外,还在铁转运、铝脱毒和生物碱等次生代谢物转运方面起重要作用[53]。目前仅有1个花青素相关转运蛋白IbGSTF4被报道[54],因此,还需要进一步研究来揭示甘薯中花青素转运体及其功能。
参考文献:
[1]王玲,邓敏姬. 紫甘薯天然红色素的提取及其稳定性研究[J]. 食品科技, 2011, 36(4): 179-183.
[2]SHAN Q, ZHENG Y L, LU J, et al. Purple sweet potato color ameliorates kidney damage via inhibiting oxidative stress mediated NLRP3 inflammasome activation in high fat diet mice[J]. Food and Chemical Toxicology, 2014, 69:339-346.
[3]SUN H N, MU T H, LIU X L, et al. Purple sweet potato (Ipomoea batatas L.) anthocyanins: preventive effect on acute and subacute alcoholic liver damage and dealcoholic effect[J]. Journal of Agricultural and Food Chemistry, 2014, 62:2364-2373.
[4]TANAKA Y, SASAKI N, OHMIYA A. Biosynthesis of plant pigments: anthocyanins, betalains and carotenoids[J]. The Plant Journal, 2008, 54(4): 733-749.
[5]PETRUSSA E, BRAIDOT E, ZANCANI M, et al. Plant flavonoids-biosynthesis, transport and involvement in stress responses[J]. International Journal of Molecular Sciences, 2013, 14(7):14950-14973.
[6]宫硖,薛静,张晓东. 植物花青素合成途径中的调控基因研究进展[J]. 生物技术进展, 2011, 1(6):381-390.
[7]兰岚,周婵媛,唐乐,等. 甘薯营养价值及综合开发利用[J]. 现代化农业, 2016 (12): 27-30.
[8]高闰飞. 利用形态学和SSR标记分析中国紫心甘薯育成品种遗传多样性[D]. 北京:中国农业科学院,2019: 11-12.
[9]PARK S C, KIM Y H, JI C Y, et al. Stable internal reference genes for the normalization of real-time PCR in different sweetpotato cultivars subjected to abiotic stress conditions[J]. PLoS One, 2012, 7(12): e51502.
[10]LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct-method[J]. Methods, 2001, 25(4): 402-408.
[11]FU Y F, MA L L, QIU F, et al. A prenyltransferase gene confirmed to be a carotenogenic CRTE gene from sweet potato[J]. Journal of Genetics and Genomics, 2014, 41(11): 613-616.
[12]李臣,后猛,馬猛,等. 甘薯块根熟化过程中麦芽糖变化及其影响因素研究进展[J].江苏农业学报,2021,37(2):539-544.
[13]周志林,唐君,曹清河,等. 淀粉专用型甘薯品质形成规律及其与主要农艺性状的相关性[J]. 江苏农业学报,2020,36(2):277-283.
[14]HU Y J, DENG L Q, CHEN J W, et al. An analytical pipeline to compare and characterise the anthocyanin antioxidant activities of purple sweet potato cultivars[J]. Food Chemistry, 2016, 194: 46-54.
[15]吴洁,沈学善,屈会娟,等.紫肉食用型甘薯品种川紫薯2号优化栽培技术[J].江苏农业科学,2020,48(16):120-125.
[16]王辉,牟琴,聂廷,等.基于相关性与主成分分析法综合评价不同品种甘薯脆片加工适宜性[J].江苏农业科学,2020,48(6):173-179.
[17]GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652.
[18]韦丽君,俞奔驰,宋恩亮,等. 基于转录组测序的木薯性别决定相关基因挖掘[J].南方农业学报,2020,51(8):1785-1796.
[19]鞠烨,江建平,尹增芳,等. 孝顺竹笋箨全长转录组测序分析[J]. 南京林业大学学报(自然科学版), 2020,44(6):175-183.
[20]WU Z G, JIANG W, MANTRI N, et al. Transciptome analysis reveals flavonoid biosynthesis regulation and simple sequence repeats in yam (Dioscorea alata L.) tubers[J]. BMC Genomics, 2015, 16: 346.
[21]GONZLEZ M, SALAZAR E, CASTILLO J, et al. Genetic structure based on EST-SSR: a putative tool for fruit color selection in Japanese plum (Prunus salicina L.) breeding programs[J]. Molecular Breeding, 2016, 36(6): 68.
[22]SHANGGUAN L F, MU Q, FANG X, et al. RNA-Sequencing reveals biological networks during table grapevine (‘Fujiminori′) fruit development[J]. PLoS One, 2017, 12(1): e0170571.
[23]WANG Z Y, FANG B P, CHEN J Y, et al. De novo assembly and characterization of root transcriptome using Illumina paired-end sequencing and development of cSSR markers in sweetpotato (Ipomoea batatas) [J]. BMC Genomics, 2010, 11(1):726-740.
[24]XIE F L, BURKLEW C E, YANG Y F, et al. De novo sequencing and a comprehensive analysis of purple sweet potato (Ipomoea batatas L.) transcriptome[J]. Planta, 2012, 236(1): 101-113.
[25]LI R J, HONG Z, CHEN K, et al. De novo transcriptome sequencing of the orange-fleshed sweet potato and analysis of differentially expressed genes related to carotenoid biosynthesis[J]. International Journal of Genomics, 2015, 2015(13): 1-10.
[26]LIU Y H, LIN-WANG K, DENG C, et al. Comparative transcriptome analysis of white and purple potato to identify genes involved in anthocyanin biosynthesis[J]. PLoS One, 2015, 10: e0129148.
[27]YANG Y N, YAO G F, ZHENG D, et al. Expression differences of anthocyanin biosynthesis genes reveal regulation patterns for red pear coloration[J]. Plant Cell Reports, 2015, 34:189-198.
[28]WEI H R, CHEN X, ZONG X J, et al. Comparative transcriptome analysis of genes involved in anthocyanin biosynthesis in the red and yellow fruits of sweet cherry (Prunusavium L.) [J]. PLoS One, 2015, 10: e0121164.
[29]ZHOU W, GONG Y F, LU X, et al. Molecular cloning and characterization of a flavonoid 3′-hydroxylase gene from purple-fleshed sweet potato (Ipomoea batatas) [J]. Molecular Biology Reports, 2012, 39(1):295-302.
[30]ZHOU W, HUANG C T, GONG Y F, et al. Molecular cloning and expression analysis of an ANS gene encoding anthocyanidin synthase from purple-fleshed sweet potato [Ipomoea batatas (L.) Lam][J]. Plant Molecular Biology Reporter, 2010, 28(1): 112-121.
[31]GUO J Y, ZHOU W, LU Z L, et al. Isolation and functional analysis of chalcone isomerase gene from purple-fleshed sweet potato[J]. Plant Molecular Biology Reporter, 2015, 33(5):1451-1463.
[32]WANG H X, FAN W J, LI H, et al. Functional characterization of dihydroflavonol-4-reductase in anthocyanin biosynthesis of purple sweet potato underlies the direct evidence of anthocyanins function against abiotic stresses[J]. PLoS One, 2013, 8(11): e78484.
[33]SUN C, LI Y, WU Q, et al. De novo sequencing and analysis of the American ginseng root transcriptome using a GS FLX Titanium platform to discover putative genes involved in ginsenoside biosynthesis[J]. BMC Genomic, 2010, 11:262.
[34]FELLER A, MACHEMER K, BRAUN E L, et al. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J]. The Plant Journal, 2011, 66:94-116.
[35]ZHAO L, GAO L P, WANG H X, et al. The R2R3-MYB, bHLH, WD40, and related transcription factors in flavonoid biosynthesis[J]. Functional and Integrative Genomics, 2013, 13:75-98.
[36]CHU H, JEONG J C, KIM W J, et al. Expression of the sweetpotato R2R3-type IbMYB1a gene induces anthocyanin accumulation in Arabidopsis[J]. Physiologia Plantarum, 2013, 148: 189-199.
[37]AN C H, LEE K W, LEE S H, et al. Heterologous expression of IbMYB1a by different promoters exhibits different patterns of anthocyanin accumulation in tobacco[J]. Plant Physiology and Biochemistry, 2015, 89: 1-10.
[38]PARK S C, KIM Y H, KIM S H, et al. Overexpression of the IbMYB1 gene in an orange-fleshed sweet potato cultivar produces a dual-pigmented transgenic sweet potato with improved antioxidant activity[J]. Physiologia Plantarum, 2015, 153(4): 525-537.
[39]WU R M, WANG T C, MCGIE T, et al. Overexpression of the kiwifruit SVP3 gene affects reproductive development and suppresses anthocyanin biosynthesis in petals, but has no effect on vegetative growth, dormancy, or flowering time[J]. Journal of Experimental Botany, 2014, 65:4985-4995.
[40]ZHOU H, LIN-WANG K, WANG H L, et al. Molecular genetics of blood-fleshed peach reveals activation of anthocyanin biosynthesis by NAC transcription factors[J]. The Plant Journal, 2015, 82:105-121.
[41]JIANG Y H, LIU C H, YAN D, et al. MdHB1 down-regulation activates anthocyanin biosynthesis in the white-fleshed apple cultivar ‘Granny Smith[J]. Journal of Experimental Botany, 2017, 68:1055-1069.
[42]ZHANG J, XU H F, WANG N, et al. The ethylene response factor MdERF1B regulates anthocyanin and proanthocyanidin biosynthesis in apple[J]. Plant Molecular Biology, 2018, 98:205-218.
[43]LIU C C, CHI C, JIN L J, et al. The bZip transcription factor HY5 mediates CRY1a-induced anthocyanin biosynthesis in tomato[J]. Plant Cell and Environment, 2018, 41:1762-1775.
[44]HU B, ZHAO J T, LAI B, et al. LcGST4 is an anthocyanin-related glutathione S-transferase gene in Litchi chinensis Sonn[J]. Plant Cell Reports, 2016, 35(4): 831-843.
[45]LUO H F, DAI C, LI Y P, et al. Reduced anthocyanins in petioles codes for a GST anthocyanin transporter that is essential for the foliage and fruit coloration in strawberry[J]. Journal of Experimental Botany, 2018, 69:2595-2608.
[46]JIANG S H, CHEN M, HE N B, et al. MdGSTF6, activated by MdMYB1, plays an essential role in anthocyanin accumulation in apple[J]. Horticulture Research, 2019, 6:40.
[47]LIU Y F, QI Y W, ZHANG A L, et al. Molecular cloning and functional characterization of AcGST1, an anthocyanin-related glutathione S-transferase gene in kiwifruit (Actinidia chinensis) [J]. Plant Molecular Biology, 2019, 100:451-465.
[48]ZHAO Y, DONG W Q, ZHU Y C, et al. PpGST1, an anthocyanin-related glutathione S-transferase gene, is essential for fruit coloration in peach[J]. Plant Biotechnology Journal, 2020, 18:1-12.
[49]CONN S, CURTIN C, BZIER A, et al. Purification, molecular cloning, and characterization of glutathione S-transferases (GSTs) from pigmented Vitis vinifera L. cell suspension cultures as putative anthocyanin transport proteins[J]. Journal of Experimental Botany, 2008, 59(13): 3621-3634.
[50]PREZ-DAZ R, MADRID-ESPINOZA J, SALINAS-CORNEJO J, et al. Differential roles for VviGST1, VviGST3, and VviGST4 in proanthocyanidin and anthocyanin transport in Vitis vinifera[J]. Frontiers in Plant Science, 2016, 7:1166.
[51]FRANCISCO R M, REGALADO A, AGEORGES A, et al. ABCC1, an ATP binding cassette protein from grape berry, transports anthocyanidin 3-O-glucosides[J]. The Plant Cell, 2013, 25(5): 1840-1854.
[52]GOMEZ C, TERRIER N, TORREGROSA L, et al. Grapevine MATE-type proteins act as vacuolar H+-dependent acylated anthocyanin transporters[J]. Plant Physiology, 2009, 150(1): 402-415.
[53]吴远双,宋毅豪,吴宝尧,等. 植物多藥和有毒化合物排出转运蛋白研究进展[J]. 广西植物, 2018, 38(11):128-138.
[54]KOU MENG, LIU Y J, LI Z Y, et al. A novel glutathione S-transferase gene from sweetpotato, IbGSTF4, is involved in anthocyanin sequestration[J]. Plant Physiology and Biochemistry, 2019, 135: 395-403.
(责任编辑:陈海霞)
收稿日期:2021-08-03
基金项目:国家重点研发计划项目(2019YFD1001300、2019YFD1001304);国家现代农业产业技术体系项目(CARS-10)
作者简介:后猛(1981-),男,山东曹县人,博士,副研究员,主要从事甘薯遗传育种研究。(E-mail)koumeng2113@163.com
通讯作者:李强,(E-mail)instrong@ 163.com

