质粒介导的印第安纳沙门氏菌耐药机制及分子溯源研究
王 天,徐红红,张林吉,王学锋,张小荣,吴艳涛,巢国祥,4
沙门氏菌是重要的人兽共患病原菌,沙门氏菌的防控是个令人关注的公共卫生问题[1]。近年来动物源印第安纳沙门氏菌泛耐药克隆(ST17克隆)的出现和传播,导致人类沙门氏菌病例增加,成为公共卫生存在的新威胁[2-3]。
沙门氏菌拥有的血清型数量繁多,不同血清型的抗生素耐药性以及致病性之间均存在差异[5]。耐药基因既可以存在于细菌染色体上,也可以存在于细菌质粒上。质粒是在多数G-细菌中广泛存在的一种染色体外的遗传因子,质粒特性使细菌更易获得抗生素耐药性、增强细菌的致病性、代谢能力等多种特性[4],并具增强细菌耐药基因传播能力。耐药I类整合子、转座子Tn3、TnsA、插入序列IS26、IS91作为细菌中存在的重要可移动元件,可对外源基因进行捕获和表达(尤其是存在于质粒上的整合子及移动元件)[5-6]。盛焕精等[7]通过质粒接合的实验说明菌株携带的质粒类型和相应的耐药表型存在关联,并且耐药基因可通过接合作用在不同种属细菌间传播,使受体菌获得耐药性。质粒消除试验可确认相应耐药基因在质粒上的存在,并可检测其在质粒上的稳定性[8]。JI XM等通过质粒的消除试验证实质粒在细菌耐药中的重要作用[9]。
本研究通过对泛耐药的ST17型动物源性印第安纳沙门氏菌进行全基因组测序并结合NCBI基因库(https://www.ncbi.nlm.nih.gov/)发布的动物源性泛耐药印第安纳沙门氏菌全基因序列的质粒结构、耐药I类整合子及移动元件、耐药基因。 通过质粒消除试验分析多株多重耐药沙门氏菌耐药性表型、耐药基因携带前后的变化并结合本实验室质粒接合试验结果,以进一步证实质粒在沙门氏菌尤其是印第安纳沙门氏菌泛耐药性的存在和传播中所发挥的作用。 同时与基因库中其它肠杆菌科质粒通过BLAST进行同源性分析,进一步探索印第安纳沙门氏菌泛耐药克隆的起源。
1 材料与方法
1.1 菌株信息 来源于前期实验室研究8株沙门氏菌菌株,其中动物源性印第安纳沙门氏菌ST17克隆菌株3株(编号分别为15、38、222),肠炎沙门氏菌1株(编号为17)、鼠伤寒沙门氏菌1株(编号为32)、德尔卑沙门氏菌1株(编号为163)[3]。
1.2 方 法
1.2.1 全基因组测序、组装 利用TAKARA DNA提取试剂盒提取菌株DNA,使用PacBio○RRS II System进行全基因组测序(NGS)。序列拼接使用SPAdes-3.5.0软件对预处理后的IllumIna 数据和PacBio数据进行拼接组装。本实验室进行的全基因NGS测序菌株为印第安纳沙门氏菌株15(GenBank:CP092259.1),222(GenBank:CP031190)。
1.2.2 质粒同源性和结构分析 选择3株来源于国内动物源性印第安纳沙门氏菌研究的菌株D90(GenBank: CP022450.1)[10]、 C629(GenBank:CP015724.1)[11]、SI67(GenBank:CP050783.1)[12]进行结构比对分析。通过NCBI以菌株15 plasmid-1全序列进行BLAST比对,选择同源性最高的菌株(Query Cover>85%、Per.ident>99%)溯源分析。耐药基因使用CARD (https://card.mcmaster.ca/anaiyze) 数据库注释。SNP分析使用CSI Phylogeny 1.4(https://cge.cbs.dtu.dk/services/CSIPhylogeny)[13]。基因组圈图绘制使用Blast Ring Image Generator绘制[14]。
1.2.3 质粒消除 采用高温SDS质粒消除的方法[8]: 菌株复苏后接种于5 mL的无抗LB液体培养基中,37 ℃、220 r/min摇床过夜培养。取50 μL接种于2 mL的5.00% SDS LB液体培养基中,摇床内37 ℃、220 r/min振荡培养12 h。取50 μL接种于2 mL的无抗LB液体培养基中,摇床内43 ℃、220 r/min振荡培养12 h,此为一轮质粒消除结束,重复上述步骤进行多轮消除。与5 mL的无抗LB液体培养基中,并在摇床内37 ℃、220 r/min过夜振荡培养,于甘油管-80 ℃内保存。
1.2.4 耐药基因的PCR检测 使用PCR扩增TEM-1、CTX-M、bla-OXA、sul1、aacC4、aac6-1b、dfrA17、floR、tet(A)、tet(G)、qnrB、qnrS等12种耐药基因。
1.2.5 药敏实验 运用自动化微量肉汤稀释法,使用药敏板进行药敏实验:头孢噻肟(CTX)、头孢他啶(CAZ)、亚胺硫霉素(IPM)、四环素(TE)、头孢唑啉(CFZ)头孢西丁(FOX)、庆大霉素(CN)、氨苄西林(AMP)、氯霉素(C)、环丙沙星(CIP)、萘啶酸(NA)、阿奇霉素(AZM)、阿米卡星(AK)、卡那霉素(K)共 14 种。根据美国临床实验室标准化委员会(CLSI)标准(CLSI 2017) 判断结果。
2 结 果
2.1 全基因测序的结果分析 本研究进行的全基因NGS测序菌株222和15的结果显示菌株222全基因组中含有1个染色体(GenBank:CP031189)和2个质粒。质粒222-scf71810(GenBank:CP031190)长308622 bp携带有27种耐药基因、2个耐药I类整合子。染色体上仅有emrR、gols和aac6-Iy3个耐药基因和一些非特异性的外排泵类抗生素抗性相关基因,另一个质粒携带有一个耐药基因CTX-M。菌株15全基因组中含有1个染色体和4个质粒,染色体长4 740 845 bp,其中一个大质粒15-plasmid1长214 299 bp,携带有22种耐药基因、1个耐药I类整合子。菌株15 的染色体上仅有gols、aac6-Iy两个耐药基因和一些非特异性的外排泵类抗生素抗性相关基因,其他质粒上并无耐药基因存在。3株来源于国内动物源性印第安纳沙门氏菌研究的质粒pD90-1(GenBank:CP022451.1)全长3 222 470 bp含有23种耐药基因和1个耐药I类整合子;pC629(GenBank:CP015725.1)全长210 106 bp,携带有24种耐药基因和2个耐药I类整合子;pSI67-1(GenBank:CP050784.1)全长255 307 bp,携带有19种耐药基因和2个耐药I类整合子。同时5个质粒基因结构十分相似且均大于210 000 bp(图1、图2)。以上质粒都携带了大量的转移酶、Tn、IS等可移动元件并分布于耐药基因两侧,数量最多的可移动元件是插入序列IS26,其次是IS6和IS91。质粒15 plamisd1、pC629、pD90-1、pSI67-1分别携带2-3种转座子,质粒222-scf71810携带了近10种转座子(图1)。

注:红色方块表示抗生素耐药基因。蓝色方块表示插入序列、转座酶和转座酶相关的结构蛋白。绿色方块表示整合酶(图中方块大小不代表序列长度)。图1 质粒pSI67-1(GenBank:CP050784.1)、222-scf71810(GenBank:CP031190)、pD90-1(GenBank:CP022451.1)、pC629(GenBank:CP015725.1)和质粒15-plasmid1的序列结构特征Fig.1 Sequence structural features of plasmids pSI67-1 (GenBank:CP050784.1), 222-scf71810 (GenBank:CP031190.1), pD90-1 (GenBank:CP022451.1), pC629 (GenBank:CP015725.1) and plasmid 15-plasmid1
2.2 质粒同源性溯源结果 基于NCBI数据库进行质粒15-plasmid1的 Blastn结果显示其他的肠杆菌中也同样存在与本研究中相似的大质粒,其中相似度最高的32株菌株(Query Cover>85%、Per.Ident>99%)中包含了10株大肠杆菌、10株印第安纳沙门氏菌、9株鼠伤寒沙门氏菌、3株肠炎沙门氏菌、1株志贺菌。33株质粒均拥有高度相似的结构(图2),其中最大质粒是来自中国浙江的大肠杆菌携带的质粒pHZ003(GenBank:MN476094.1)全长320 681 bp;最小的是来自中国浙江的印第安纳沙门氏菌携带的质粒pTB501(GenBank:CP034820.1)全长188 527 bp。本文3株印第安纳沙门氏菌携带质粒也包含在其中。15-plasmid1和pD90-1同源性最高、222-scf71810和SI67同源性最高(图3)。33株肠杆菌携带的耐药基因主要包括β-内酰胺类耐药基因:TEM-1和OXA-1、磺胺类耐药基因:sul1、sul2氨基糖苷类耐药基因:Aph(6)-Id、Aph(3″)-Ib、Aph(4)-Ia、aacC4、aac(6)-Ib、氯霉素类耐药基因catB3、季铵类耐药基因QacEΔ1,其中有28个质粒都携带有耐药I类整合子(图3)。
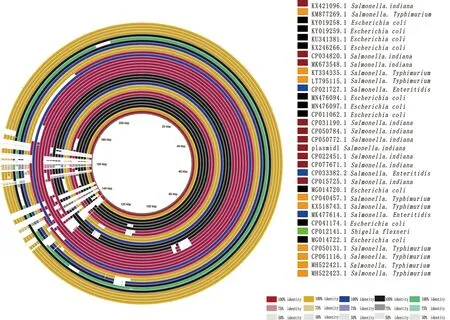
注:基于质粒15 plasmid1全序列BLAST结果中同源性最高的前32株(Query Cover>85%、Per.Ident>99%)使用Blast Ring Image Generator绘制的基因组圈图(红色:印第安纳沙门氏菌、黄色:鼠伤寒沙门氏菌、蓝色:肠炎沙门氏菌、黑色:大肠杆菌、绿色:志贺菌)。图2 33株肠杆菌质粒BRIG(Blast Ring Image Generator)比对结果FIg.2 Blast Ring Image Generator results of 33 strains of Enterobacteriaceae plasmids

基于质粒15 plasmid1全序列BLAST结果中同源性最高的前32株(Query Cover>85%、Per.Ident>99%)绘制的NJ进化树,菌株编号标灰的部分是前文中的5个质粒pSI67-1、222-scf71810、pD90-1、pC629(GenBank:CP015725)和15-plasmid1。图3 33株肠杆菌质粒SNP的Neighbor-Joining (NJ)进化树与耐药基因携带情况Fig.3 Neighbor-Joining (NJ) tree of 33 Enterobacteriaceae plasmid SNPs and resistance-genes carriage
2.3 质粒消除前后耐药基因变化 经质粒消除试验后,菌株15不再携带TEM-1、OXA-1、sul1、aacC4、dfrA17、floR、qnrB等7种耐药基因,菌株38不再携带TEM-1、bla-CTX-M、OXA-1、sul1、aacC4、dfrA17、floR,菌株222不再携带TEM-1、OXA-1、sul1、aacC4、aac6-1b。而菌株17携带的TEM-1,菌株32携带的floR和tet(G),菌株163携带的sul1等耐药基因被消除(表1)。

表1 质粒消除前后耐药基因携带情况Tab.1 Carriage of resistant genes before and after plasmid elimination
2.4 质粒消除前后耐药性变化 在我们检测的14种抗生素中质粒消除前印第安纳沙门氏菌均对其中的9种以上的抗生素表现出耐药性。质粒消除试验后,菌株15 和菌株38对TE、CN、CIP、AZM、AK等抗生素恢复敏感,菌株222对CTX、CN、CIP、NA、K等抗生素恢复敏感。而其他的3种血清型的沙门氏菌耐药性改变较小,其中菌株17 和菌株32的耐药性没有改变(表2)。
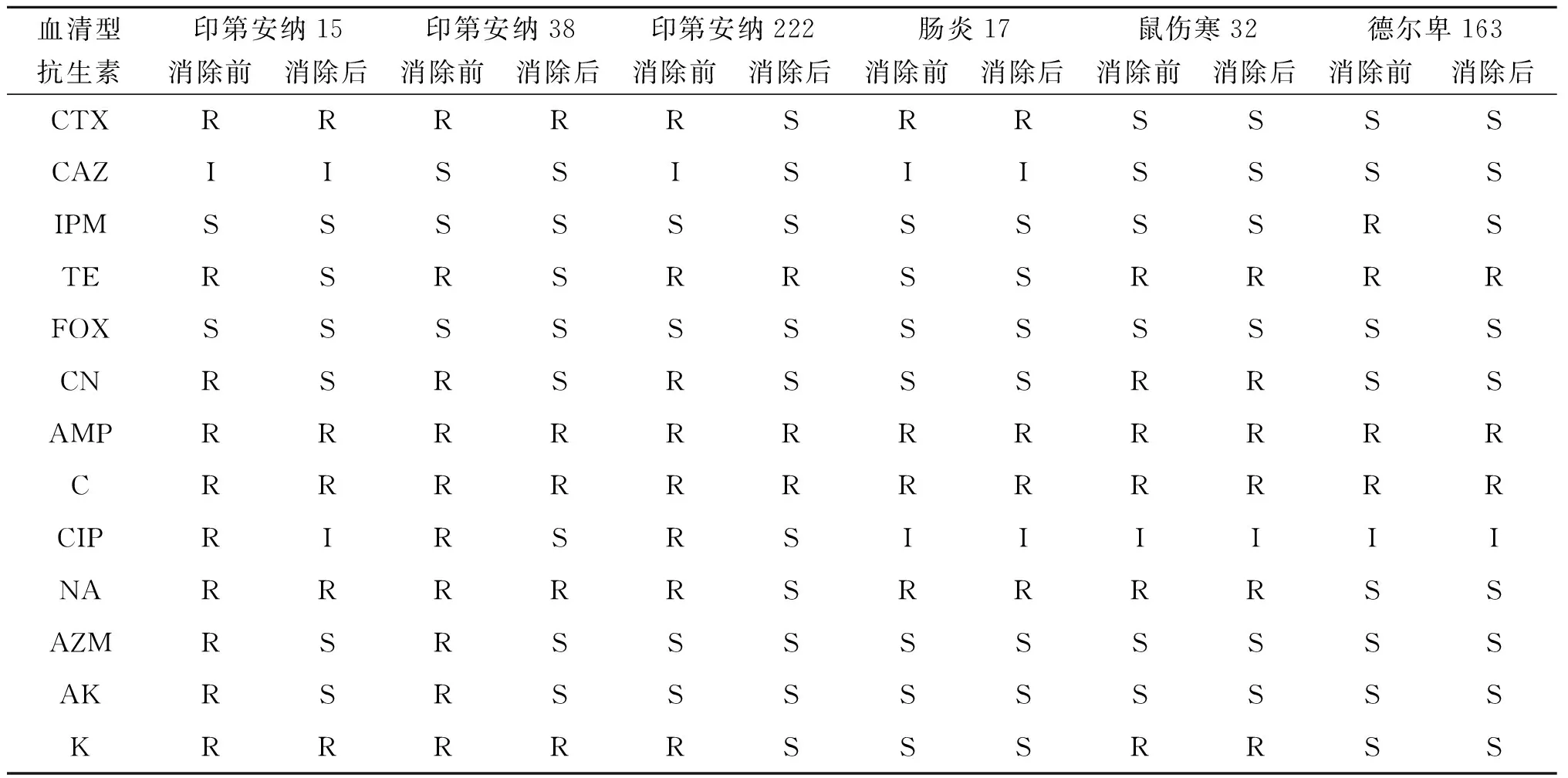
表2 6株沙门氏菌质粒消除前后耐药性情况Tab.2 Phenotypic resistance profiles of 6 strains of Salmonella before and after plasmid elimination
3 讨 论
印第安纳沙门氏菌是近年来发现的泛耐药克隆[3]。本研究结果表明印第安纳沙门氏菌携带有200 000 bp左右的大质粒,这些质粒高度相似同源。该质粒携带有1个以上的耐药I类整合子、整合酶及多个转座子Tn及多个插入序列IS。质粒上的耐药基因大部分都位于耐药I类整合子上,在所有的耐药基因和耐药基因簇的两侧都分布了Tn3、IS26、IS6等转座酶和插入序列。耐药I类整合子有强大的从其它细菌或环境中获得耐药基因能力,而整合酶、转座子、插入序列更进一步加强了质粒对耐药基因的捕获能力。本实验室前期的研究表明,印第安纳沙门氏菌耐药I类整合子远远高于其它如肠炎、鼠伤寒等沙门氏菌[15,17],而本实验室质粒接合转移实验结果进一步表明携带耐药I类整合子的质粒接合转移频率显著高于没有耐药I类整合子的质粒,且含有耐药I类整合子的质粒更容易在菌株之间水平传播进而获得耐药基因[18]。质粒本身除了在耐药基因的获得中发挥作用,通过接合转移等作用增加了细菌耐药性的传播。正是这种大质粒的存在并被印第安纳沙门氏菌获得而导致印第安纳沙门氏菌泛耐药克隆的形成。
本研究中质粒存在许多相似的遗传结构:如由sul1—qacEΔ1—Arr-3—CatB3—bla-OXA-1—AAC(6′)-Ib-cr6—aacC4-Aph(4)-Ia组成的耐药基因簇;I类整合酶附近的IS26-aadA5-dfrA17-IntI1;由IS26-oqxA-oqxB-IS26组成的操纵子oqxRAB;而这些结构在其他的菌株如大肠杆菌质粒pAH01-4(GenBank:CP055255.1)、奇异变形杆菌MPE0734(CP053615.1)均有多个高度相似的序列,且耐药基因的位置分布也具有相似的特点。肠杆菌质粒的NJ进化树(图3)结果表明这种质粒与肠杆菌中存在的相似质粒高度同源。大多存在于大肠杆菌和沙门氏菌中,推测这种质粒来源于大肠杆菌。且这种质粒有向其他肠杆菌水平传播的可能与趋势。
高温SDS的消除机制为破坏细胞膜完整性,改变质粒在细胞膜上的结合位点,导致耐药质粒因不能完成正常的复制及分配到子代细胞[16]不会影响染色体上的耐药基因。质粒消除后菌株耐药性的改变说明在泛耐药印第安纳沙门氏菌中对CTX、TE、CN、CIP、AZM、AK、K等多种抗生素耐药性发挥决定作用的是存在其质粒上耐药基因。印第安纳沙门氏菌多数耐药基因被消除,消除效果较高,说明这类位于核苷酸上的基因不稳定,进一步说明这类耐药基因最大可能是位于质粒上。本研究中同样的耐药基因在不同的菌株中的消除情况并不相同。消除剂的浓度以及初始的菌液浓度等因素均会对质粒消除的效果产生影响[8]。其他血清型的沙门氏菌的耐药性并没有随着质粒消除发生明显的抗生素恢复敏感现象。这可能是由于这类菌株中耐药基因大部分存在于染色体上,而导致其携带的耐药基因以及耐药性不容易随着质粒的消除而消除。
综上所述,携带有耐药I类整合子大质粒的印第安纳沙门氏菌,更易从环境或其它细菌中获得耐药基因,是形成印第安纳沙门氏菌耐药克隆的主要原因。此质粒是其在环境进化过程中从其它肠杆菌科细菌获得。通过质粒消除及结合试验进一步证明这一点。
利益冲突:无
引用本文格式:王天,徐红红,张林吉,等. 质粒介导的印第安纳沙门氏菌耐药机制及分子溯源研究[J].中国人兽共患病学报,2022,38(7):619-625. DOI:10.3969/j.issn.1002-2694.2022.00.076
——一道江苏高考题的奥秘解读和拓展

