宏基因组测序分析奶牛运动场土壤抗生素抗性基因
胡 平,关文怡,雷莉辉,蔡泽川
北京农业职业学院,北京 102442
抗生素的使用在养殖业中不可避免,研究表明,真正参与动物新陈代谢而被利用的抗生素仅占用药总量很少的比例,而大部分抗生素则随着动物粪便和尿液直接排出体外,随粪尿排出体外的还包括在动物肠道内诱导产生的带有抗性基因的菌株[1-4]。动物粪便中含有抗性基因的菌株成为了土壤和地下水中重要的抗性基因污染源[5]。研究表明,抗性基因除了可以在动物的肠道菌群之间传播,还会在进入土壤后与土壤中的微生物之间发生传播[6]。通过基因的横向转移,抗性基因在土壤、水源、空气等环境介质中迁移转化,并在生存选择中逐渐增加丰度,当这些抗性基因随着食物链进入人体时,就增加了人体的抗生素耐药性[7]。为了减少奶牛蹄病发生,奶牛运动场地面通常使用三合土或部分铺设透水砖,随着冲洗用水和降雨等冲刷作用,粪便与尿液中残留的抗生素与微生物逐渐渗透进入土壤并向周围逐渐扩散,造成养殖场粪便中抗生素抗性基因在环境中的富集与传播。因此,研究土壤中抗性基因分布情况对减少抗生素的滥用和耐药菌株的产生具有重要意义。本试验通过采集北京市某区规模化奶牛运动场土壤对抗性基因进行宏基因组分析,旨在找出奶牛场抗性基因分布情况,并指导抗生素使用。
1 材料与方法
1.1 样本采集与处理
试验样本采集自北京市某规模化奶牛运动场内,共计12 份土壤样本。按照采样部位不同分为3组,CTN(1-4)为运动场中心位置土壤样本,CBH(1-4)为运动场围栏外0.5 m 土壤样本,CB2M(1-4)为运动场外2 m 土壤样本。所有土壤样本均采集自距地表0~20 cm 深度的松软土壤,采用灭菌铲子取样,封装于灭菌样本管中。所有样本均置于干冰盒中迅速转运到实验室-80 ℃冰箱中保存待检。
1.2 总基因组DNA 提取与建库测序
将土壤样本按照PowerSoil DNA Isolation Kit(MoBio Laboratories,Carlsbad,CA)试剂盒说明书进行微生物组DNA 提取。然后用1%琼脂糖凝胶电泳和分光光度法检测DNA 质量和浓度。质检合格的DNA 样品用Covaris 超声波破碎仪将基因组DNA 随机打断成长度约300 bp 的小片段,经纯化、PCR 扩增等步骤完成文库制备。使用Qubit 初步定量构建完成的文库,然后使用Agilent 2100 检测文库的插入片段,符合预期后使用Q-PCR 法对文库的有效浓度进行准确定量(文库有效浓度>3 nmol/L)。最后把不同文库按照有效浓度及目标下机数据量的需求进行pooling,在Illumina HiSeq4000 平台上进行测序。
1.3 数据分析处理
原始数据使用Trimmomatic 软件进行系列质控,将质控后得到的高质量序列clean reads 使用DIAMOND BLASTX 算法进行比对和物种注释。用组装软件MEGAHIT(v1.0.6)对测序数据进行组装,过滤组装结果中500 bp 以下的片段。采用Prodigal 软件对组装得到的contig 序列进行ORF(open reading frame)预测,使用CD-HIT 软件对预测的结果去冗余,从而得到非冗余基因集。采用Bowtie 软件将测序数据与构建的非冗余基因集进行比对,并统计单个基因在不同样本的丰度信息。将预测得到的非冗余基因集与功能注释数据库nr、Swiss-Prot、Kegg、Cog/Kog、eggNOG、GO、Pfam、ARDB/CARD、CAZyme 进行比对和注释。
1.4 前期研究基础
本试验前期通过宏基因组测序对该奶牛运动场土壤和粪便微生物群落结构进行分析,测序结果表明变形菌门和放线菌门在土壤中占据绝对优势;子囊菌门、担子菌门为土壤中的优势真菌,奇古菌门和广古菌门为优势古菌群落。在新鲜粪便中厚壁菌门和拟杆菌门为优势菌;优势真菌群落为担子菌门、毛霉门和壶菌门。土壤中微生物丰度见表1。

表1 宏基因组测序分析土壤中微生物丰度排名
2 结果与分析
2.1 测序数据组装与分析
通过宏基因组测序,3 个组12 个样本中,总计获得343 850 519 条clean reads,每个样本的reads数范围在16 632 548~25 716 812 条,组装后的总contigs 长度超过1 000 bp 的为778 998,N50 为893 bp,L50 为989 937 bp,表明组装的结果较好,序列长度可用。
2.2 运动场土壤样本中ARGs 多样性
通过宏基因组检测及CARD 数据库注释,在3个分组的12 个样本中,占比前10 的抗性基因为ARO:3000501、ARO:3004480、ARO:3000804、ARO:3000444、ARO:3002983、ARO:3003318、ARO:3004144、ARO:3003031、ARO:3003923、ARO:003010。其 中,ARO:3000501 在CB2M 组中占比为29.67%,在CBH组占比为28.29%,在CTN 组占比为32.67%;ARO:3004480 在CB2M 组占比为16.49%,在CBH 组占比为28.96%,在CTN 组占比为19.87%;ARO:3000804 在CB2M 中占比为9.69%,在CBH 中占比为4.84,在CTN 中占比为13.46%;ARO:3000444 在CB2M 组占比为5.67%,在CBH 组占比为4.61%,在CTN 占比为6.80%;ARO:3002983 在CB2M 占比为5.08%,在CBH 占比为7.22%,在CTN 占比为2.13%;ARO:3004144 在CB2M 占比为4.78%,在CBH 占比为3.81%,在CTN 占比为7.24%;其余抗性基因在各组占比都低于5%(图1、图2)。
2.3 CRAD 注释的前13 位抗性基因
通过分析抗生素抗性基因检出率发现,土壤中抗性基因检出率最高的是rpoB2 和rpoB,在CRAD注释为利福霉素抗性基因,耐药菌为诺卡氏菌属和双歧杆菌属。其次是MexF和MexW,注释为苯丙醇类、二氨基嘧啶、氟喹诺酮、四环素类、吖啶染料、大环内酯类抗生素,显示了多重耐药特点,耐药菌株主要为铜绿假单胞菌。此外值得注意的是抗性基因mexQ、oqxB、MuxC和MexK都显示为多重耐药性,主要耐药菌株为肺炎克雷伯菌、铜绿假单胞菌和伯克氏菌等。此外rphA和oleB分别显示了多重耐药性,但是在CARD 注释中未有报告,显示了上述2个抗性基因可能是少见但容易被忽略的多重耐药基因,其中携带oleB的耐药菌株可能导致大部分G+球菌发生广泛耐药(表2)。
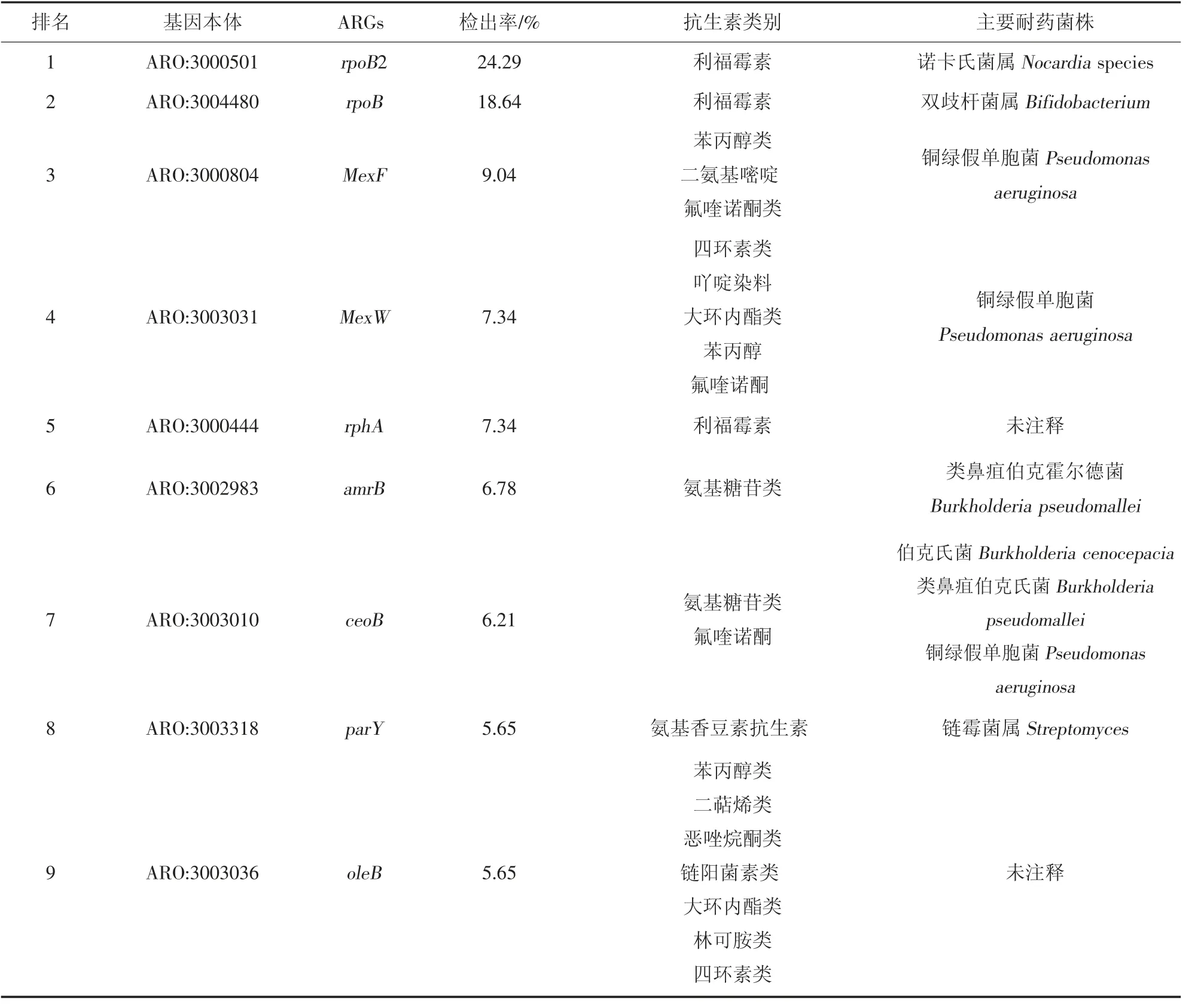
表2 运动场土壤抗性基因与抗生素类别

续表2
2.4 Lefse 分析组间差异抗性基因
通过LDA 阈值3 筛选3 组样本之间的差异基因,结果显示,在CB2M 组中显著高于其他组的抗性基因主要有以下12 个:parY、oqxB、MexW、MexI(ARO:3000808)、MexK、aadA11(ARO:3002611)、cmlR(ARO:3002690)、mexQ、mexX(ARO:3003034)、smeR(ARO:3003066)、aadA6(ARO:3002606)、CpxR(ARO:3004054)。
在CBH 组中显著高于其他组的抗性基因主要有以下10 个:利福霉素耐药双歧杆菌rpoB突变株(ARO:3004480)、amrB、oleB、vanRO(ARO:3002930)、MexB(ARO:3000378)、ceoB、NovA(ARO:3002522)、MuxC(ARO:3004075)、tlrC(ARO:3002827)、muxB(ARO:3004074)。
在CTN 组中显著高于其他组的抗性基因主要有 以 下5 个:MexF(ARO:3000804)、rphA、AxyY、MexD(ARO:3000801)、oleC(ARO:3003748)(图3)。
2.5 抗性基因circlize 分析
基因抗性基因的注释结果,选取Top10 的抗性基因进行circlize 分析(图4):图4 上半圆显示的是样本名称,下半圆为各个样本对应的抗性基因,从图4 可以看出不同抗性基因在每个样本中的丰度情况。
3 讨 论
3.1 运动场不同位置土壤中抗性基因分布情况
通过对奶牛运动场3 个不同位置CB2M、CBH和CTN 的土壤宏基因组分析可见,从运动场中心向运动场外的土壤中,随着与运动场中心点半径增大,其相应的土壤中抗性基因数量也显著增多。其中,CB2M 组检出率最高的抗性基因是parY,CBH组检出率最高的抗性基因是rpoB,CTN 组检出率最高的抗性基因是MexF。分析该结果可能的原因是,奶牛场排泄物中携带的抗生素残留物以及奶牛消化道中携带抗性基因的耐药菌随着粪便和尿液逐渐渗透进入运动场的土壤中,并随着渗透与冲刷向外扩散。因为土壤和沉积物是固体物质,密度较大、流动较慢,容易发生富集作用,造成抗生素更易在土壤中富集和持久传播,从而胁迫土壤中原有的土著微生物产生耐药性[8]。
有报道称施用过奶牛和猪粪的土壤中,以低G+C 革兰氏阳性硬壁菌和高G+C 革兰氏阳性放线菌占主导地位,尤其是链霉菌属占主导地位[9]。这与本次试验中统计的CB2M 检出率最高的parY抗性基因相一致。不同的国家和地区施用过奶牛粪便的土壤中优势菌种也有区别[10-12],但从各项研究均可看出粪便污染的土壤中都含有大量抗生素抗性细菌和抗性基因,对环境中的耐药菌株产生都有重要的意义。
3.2 奶牛运动场土壤中抗性基因菌群分析
由图4 观察可见,3 组土壤样本中排名前10 位的 抗 性 基 因 为rpoB2、rpoB、MexF、rphA、amrB、AxyY、streptomyces、mexW、oqxB、ceoB,对应的耐药菌株分别为诺卡氏菌属、双歧杆菌属、铜绿假单胞菌、不溶性无色杆菌、链霉菌属、肺炎克雷伯菌等。
rpoB是来源于双歧杆菌的抗性基因。双歧杆菌是耐抗生素的益生菌,是肠道的有益菌群,然而肠道微生物之间的水平基因转移可能产生有害的耐药菌株,如结核分枝杆菌。Dhanashree 等[13]分析了双歧杆菌抗结核药物耐药和rpoB突变,研究发现动物双歧杆菌、长双歧杆菌和青少年双歧杆菌对吡嗪酰胺、异烟肼和链霉素表现出相当大的耐药性,而青少年双歧杆菌在利福霉素袋和袋外区域都有突变,并且对利福霉素也表现出相当大的耐药性。由于基因重复,诺卡氏菌的基因组包括对利福平敏感的RNA 聚合酶β 亚单位(rpoB)和对利福平耐药的RNA 聚合酶β 亚单位(rpoB)基因,2 种基因产物之间约有88%的相似性。rpoB变体的表达导致利福平敏感性被利福平耐药性取代[14]。
MexF 是MexEF-OprN 复合物的多药内膜转运体。mexF对应于铜绿假单胞菌PAO1 中的2 个位点(基因名称:mexF/mexB)和铜绿假单胞菌LESB58 中的4 个位点(基因名称:mexD/mexB)。MexW 是外排复合物MexEF-OprN 的RND 型膜蛋白,具有广泛的耐药性。携带该突变基因的铜绿假单胞菌对多种抗生素产生多重耐药,如苯丙醇类、二氨基嘧啶、氟喹诺酮类、四环素类、吖啶染料、大环内酯类[15]。
amrB 是AmrAB-OprM 多药外排复合物的膜融合蛋白,具有该抗性基因的类鼻疽伯克氏菌,铜绿假单胞菌具有抗生素外排泵,从而对氨基糖苷类抗生素发生耐药。
AxyY 是无色杆菌AxyXY-OprZ 外排泵系统的周质衔接蛋白,具有该抗性基因的不溶色杆菌、木糖氧化酶无色杆菌、洋葱伯克霍尔德菌、阪崎克龙杆菌、肺炎克雷伯菌通过抗生素外排泵,对多种抗生素如大环内酯类、头孢菌素、氨基糖苷类和氟喹诺酮产生多重耐药性。
oqxB是对氟喹诺酮耐药的RND 外排泵,具有该抗性基因的弗氏柠檬酸杆菌、霍玛肠杆菌、大肠杆菌、肺炎克雷伯菌、肠沙门氏菌、宋内志贺氏菌等菌株对多种抗生素如四环素类、甘氨酰环素、硝基呋喃类、二氨基嘧啶类和氟喹诺酮类产生多重耐药性[16]。
3.3 奶牛场耐药情况分析
通过本试验分析发现,该奶牛运动场土壤中存在大量抗性基因分布,耐药菌包括诺卡式菌属、链霉菌属、铜绿假单胞菌、双歧杆菌属、肺炎克雷伯菌等。其中,诺卡氏菌属、铜绿假单胞菌感染常与严重的奶牛乳房炎有关[17-19]。铜绿假单胞菌又称绿脓杆菌,是引起奶牛乳房炎的重要致病菌之一。在本试验的检测中发现奶牛场绿脓杆菌多个抗性基因,具有多重耐药菌株出现。此外,本奶牛场出现对利福霉素显著耐药的抗性基因,利福霉素是治疗奶牛子宫内膜炎常用的抗生素种类,随着利福霉素类抗生素使用量的增加,该奶牛场土壤中抗利福霉素的抗性基因逐渐富集[20]。
4 结 论
通过分析发现,该奶牛场土壤中利福霉素、苯丙醇类、氟喹诺酮类、四环素类、大环内酯类、氨基糖苷类抗生素抗性基因数量多,说明该牛场对这几类抗菌药物已经产生了较明显的耐药性,建议减少上述抗菌药物的使用,并应根据抗性基因情况选择其他有效替代药物,从而减少抗生素滥用,降低对环境中微生物的胁迫,减少超级耐药菌株的产生几率。

