花生InDel标记开发及其在含油量QTL定位中的应用
陶顺玉 吴 贝 刘 念 罗怀勇 黄 莉 周小静 陈伟刚 郭建斌 喻博伦 雷 永 廖伯寿 姜慧芳
花生InDel标记开发及其在含油量QTL定位中的应用
陶顺玉 吴 贝 刘 念 罗怀勇 黄 莉 周小静 陈伟刚 郭建斌 喻博伦 雷 永 廖伯寿 姜慧芳*
中国农业科学院油料作物研究所 / 农业农村部遗传育种重点实验室, 湖北武汉 430062
培育高含油量品种是花生重要的育种目标, 开发稳定高效的分子标记对于标记辅助选择花生高含油量品种具有重要价值。本研究针对2个含油量差异显著的亲本徐花13 (高含油量)和中花6号(低含油量), 利用PacBio三代测序技术检测基因组结构变异, 分别从徐花13和中花6号中检测到35,794个和74,703个结构变异。根据双亲结构变异信息, 针对前期定位的含油量区间开发InDel (插入/缺失)标记84个, 其中9个InDel标记的扩增片段证实在双亲间具有多态性。基于RIL (重组自交系)群体中的杂合残余获得的NIL (近等基因系)构建的一个包含1160个单株的F2群体, 使用9个多态性InDel标记鉴定其基因型, 构建了总长度为149.84 cM的局部遗传连锁图谱。结合F2群体的含油量表型数据, 将该含油量QTL定位在标记M23至M11之间, 位于A08染色体1.2 Mb区段内。本试验证实了InDel标记开展花生含油量QTL定位的可行性, 其定位的含油量位点及其紧密连锁的InDel标记能够为花生分子标记辅助育种提供理论和技术指导。
花生; 基因组测序; InDel; 含油量
花生(L.)作为植物油脂和蛋白的重要来源, 是全球重要的油料、经济作物。我国是世界上花生总产量和消费量最大的国家, 花生单产量和总产量均居我国油料作物首位。在所有油料作物中花生单位产油量最高, 是仅次于油菜的第二大植物油来源[1], 具有很高的经济价值, 对我国国民经济发展和食用油供给安全具有重要意义。目前我国食用油的自给率低, 对外依存度大, 约70%的食用油供给依赖进口[2]。为保障食用油供给安全, 提高花生含油量成为花生育种工作者的首要目标。
研究花生含油量的遗传规律对促进花生基因型改良的发展至关重要, 花生含油量变异丰富, 范围为31.07%~60.26%[3-4], 遗传改良潜力较大。目前花生育种主要根据多个环境下鉴定的表型数据对高含油量花生品种进行选择, 培育周期长[5], 利用分子标记辅助选择目标性状能够提高选择效率, 加快育种进程。然而异源四倍体栽培花生基因组大且结构复杂[6], 全基因多态性分子标记开发难度大。目前报道的花生含油量稳定主效QTL位点不多, Shasidhar等[7]利用F2群体鉴定到8个与含油量相关的QTL, 其中2个主效QTL (和), 分别位于标记Ah5507-Ah5719和Ah3864-Ah2573之间; Wilson等[8]利用SSR标记定位到3个稳定的含油量QTL,获得与含油量紧密相关的标记2个; Guo等[9]在不同环境下鉴定到2个稳定主效的QTL (和), 证实了紧密连锁的标记Ai06B29452和AGGS2133-1与花生含油量相关。这些含油量QTL主效位点定位区间较大, 可利用的信息少, 难以指导含油量分子标记辅助选择技术的开发。因此有必要加强含油量分子标记开发研究, 为深化花生分子遗传改良提供技术支撑。
InDel作为基因组主要的变异形式之一, 是遗传标记的丰富来源。InDel标记属于共显性标记[10], 广泛分布于全基因组, 可根据目标区域设计分子标记[11],其检测手段简单且扩增效率高。近年来, 由于高通量测序成本低、周期短和效率高的特点, 利用全基因组测序对亲本进行重测序, 与参考基因组序列进行比对, 检测序列差异, 再根据基因组间结构差异开发InDel多态性标记[12], 对目标性状进行定位的研究已在玉米[13]、大白菜[14]、甜椒[15]、陆地棉[16]等报道。目前花生上已有InDel标记开发的报道, 但仅应用于抗病[17]和产量[18]相关研究, 在花生含油量性状的应用尚未见报道。
本课题组前期利用2个含油量差异明显的高含油量徐花13和低含油量中花6号杂交构建RIL群体, 对群体的含油量进行检测, 结果显示群体内含油量性状连续变异且出现超亲分离, 连续2年鉴定到花生含油量QTL (), 能够解释9.76%~22.00%的表型变异, 初步定位在A08染色体的1.8 Mb区间内, 标记区间为TClE5–AHZ851[19]。本研究在此基础上对亲本徐花13和中花6号进行测序, 挖掘基因组结构变异, 与参考基因组进行比对, 开发在目标区段内与含油量相关的InDel标记, 在双亲中筛选出具有多态性的InDel标记, 将鉴定出的多态性标记对F2群体进行基因分型, 同时结合表型数据定位花生含油量QTL (), 旨在为提高花生含油量及分子标记育种提供参考。
1 材料与方法
1.1 材料
在前期通过RIL群体“徐花13 (含油量52.21%)×中花6号(含油量48.96%)”获得含油量QTL位点基础上, 筛选目标位点为杂合基因型的单株自交获得NIL, 通过NIL杂交构建F2次级分离群体并种植于试验农场, 每行12株, 行长2 m, 行距0.33 m, 常规田间管理。
1.2 方法
1.2.1 InDel标记开发及多态性筛选 选取亲本苗期健康幼嫩的叶片, 利用DNA提取试剂盒(天根生化科技(北京)有限公司, DP305)提取花生基因组DNA, 送至北京贝瑞和康生物技术有限公司进行PacBio三代测序, 利用Bwa-0.7.12软件将测序数据与参考基因组进行比对, 挖掘序列变异, 筛选在亲本间具有差异的InDel位点, 根据比对结果找出亲本在A08染色体上QTL ()目标区段内存在的InDel差异位点。利用Primer 5.0软件将这些差异位点设计成引物, 引物长度范围为151~610 bp, 提取InDel上下游各500 bp的序列, 并根据上下游序列设计引物, 退火温度范围为50~62℃, 引物由北京擎科生物科技(武汉)有限公司合成, 将开发的InDel标记在亲本中进行多态性筛选。
1.2.2 DNA的提取、PCR扩增以及琼脂糖凝胶电泳检测 选取花生苗期健康幼嫩的叶片3~4片, 利用改良的CTAB法[19]提取基因组DNA, 分别采用1%琼脂糖凝胶电泳和NanoDrop (Thermo Fisher Scientific, 美国)检测DNA的质量和含量。利用开发的InDel引物进行PCR扩增, 反应体系10 μL, 包括全式金2× EcoPCR SuperMix (+dye) 5 μL, 基因组DNA模板l μL (20~30 ng μL–1), 引物对0.4 µmol L–1, ddH2O补至10 μL。PCR反应程序为: 95℃预变性3 min; 然后95℃变性30 s、50~59℃复性30 s、72℃延伸1 min kb–1, 共40个循环; 最后72℃延伸5 min, 4℃保存。使用琼脂糖凝胶电泳检测PCR反应扩增产物, 凝胶成像系统下照相记录, 统计分析条带类型, 利用Microsoft Excel整理数据并分析对应基因型。
1.2.3 表型数据测定 收获后的种子自然条件下风干(含水率控制在10%以下), 取3~4 g完整饱满的种子采用核磁共振分析仪(PQ001)快速测定种子含油量, 为确保表型结果的准确性, 测定结果取3次重复的平均值, 利用Microsoft Excel进行数据统计与计算。
1.2.4 构建局部遗传连锁图谱及QTL定位 利用筛选出的InDel多态性标记对F2群体(=1160)进行基因型分析, 对于共显性InDel标记, 与徐花13带型相同的记为“1”, 与中花6号带型相同的记为“2”, 具有双亲杂合带型的记为“3”; 对于显性InDel标记, 与徐花13带型相同的记为“1”, 与中花6号带型相同的记为“2”, 带型缺失或模糊的样品基因型记为“-”。结合F2群体单株表型数据和基因型分析数据, 利用QTL IciMapping V4.0软件构建局部遗传连锁图谱, 映射参数设置为1.0 cM步长, 概率0.001, LOD阈值为3, 采用ICIM-ADD (完备区间作图法)[20]对花生含油量进行QTL定位。
2 结果与分析
2.1 PacBio测序鉴定基因组结构变异
利用PacBio三代测序技术对亲本徐花13和中花6号进行测序, 所得的测序数据经过质控(表1), 将双亲过滤后的数据比对到花生参考基因组, 在基因组中的比对率分别为98.03% (徐花13)和97.58% (中花6号), 覆盖度均高于96.59%, 表明测序数据质量良好, 可以用于后续分析。结构变异类型包括基因组水平上大片段的插入、缺失、倒位、易位和重复, 将鉴定到的结构变异进行统计, 徐花13共鉴定到35,794个结构变异(表1), 其中插入片段12,293个, 缺失片段4496个, 倒位片段14个, 易位片段16,305个, 重复片段2868个; 中花6号共鉴定到74,703个结构变异, 插入片段26,833个, 缺失片段12,906个, 倒位片段24个, 易位片段31,412个, 重复片段3528个。
对InDel的长度分布进行统计分析, 发现其长度分布较广, 插入突变的片段长度主要分布在0~100 bp, 其次是100~1000 bp, 而超过1000 bp的很少; 缺失突变的片段长度主要在100~1000 bp, 其次是0~100 bp (图1)。根据参考基因组, 使用ANNOVAR软件对DEL、INS、INV进行变异注释。根据基因组注释, 将InDel注释到基因上游或下游(转录起始位点上游/转录末端位点下游1 kb区域)、外显子、内含子、基因间区、剪切位点(内含子中靠近外显子/内含子边界的2 bp)、5′非编码区和3′非编码区。分析InDel注释结果, 发现识别到的InDel多态性位点大部分位于基因间区, 徐花13和中花6号分别检测到30,158个(占84.25%)和60,637个(占81.17%), 而发生在基因外显子的InDel多态性位点分别为901个(徐花13)和1595个(中花6号) (图2)。
2.2 InDel标记的开发及筛选
针对初定位的含油量QTL (), 该目标区间位于A08染色体1.8 Mb (45,605,931~47,465,778)区间内, 基于亲本结构变异信息, 在目标区间内根据变异位点的物理位置, 将这些变异位点全部设计成引物, 共开发出84个InDel标记, 将开发的InDel标记以亲本徐花13和中花6号的基因组DNA为模板进行PCR扩增, 经过PCR和产物检测发现共9个InDel标记在亲本中均能扩增出差异条带(图3), 扩增条带的大小与预测产物大小吻合, 多态性频率为10.7%, 与目标性状连锁的InDel标记相关信息见表2。
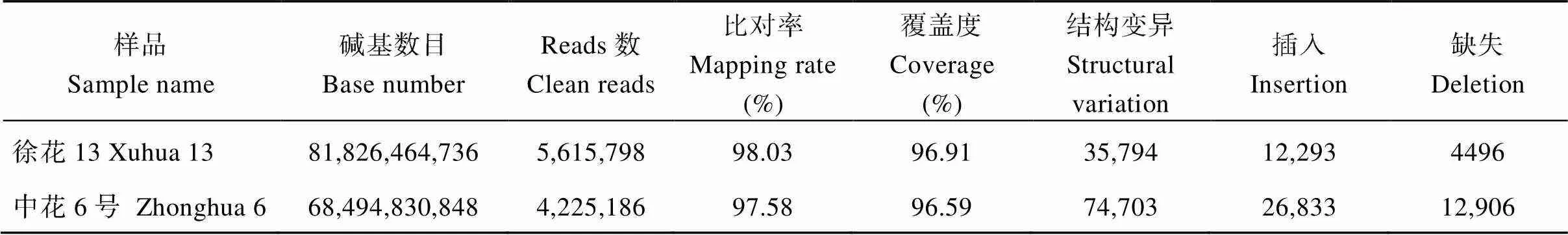
表1 亲本测序信息
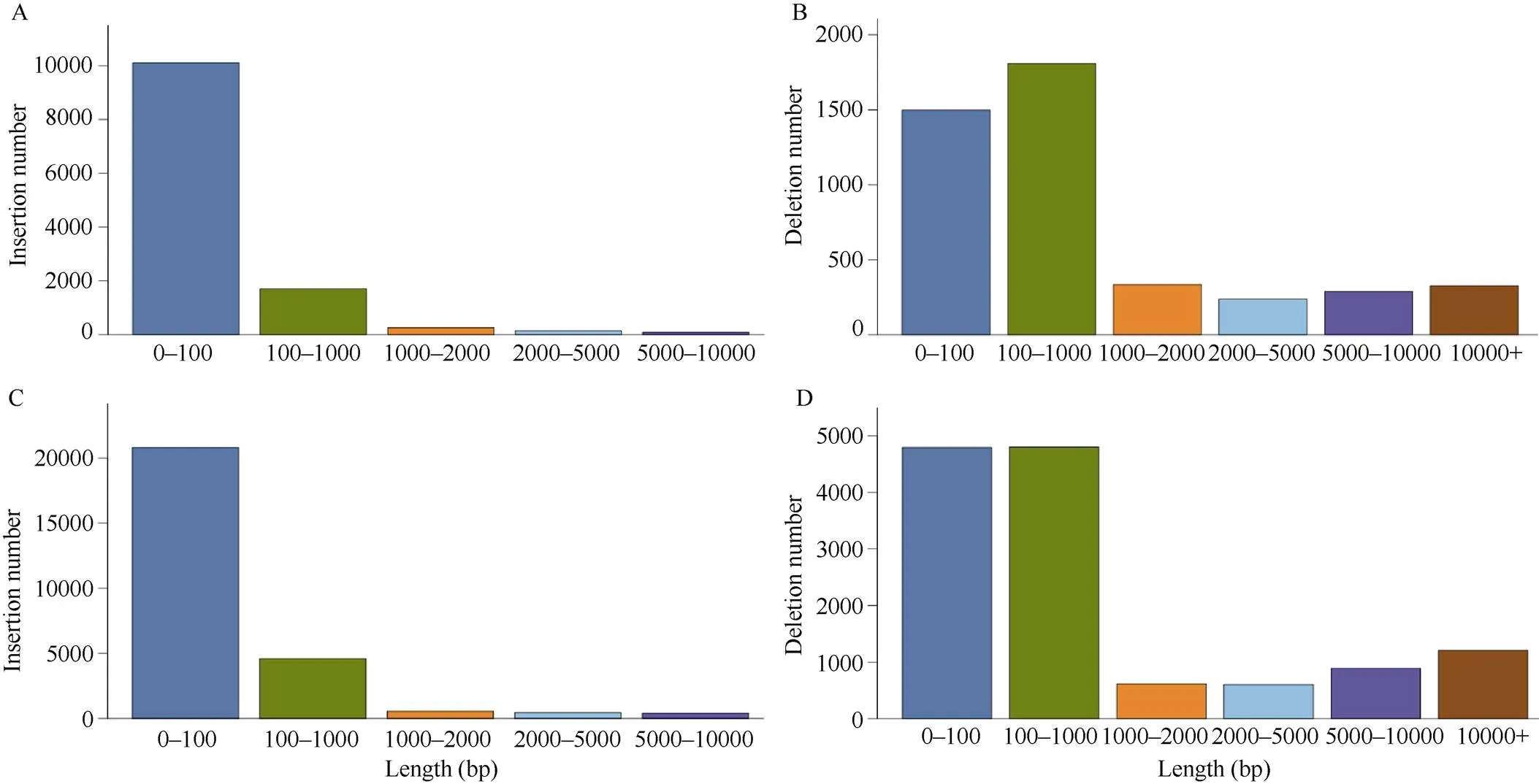
图1 亲本中插入、缺失长度分布
A: 徐花13插入变异的长度分布; B: 徐花13缺失变异的长度分布; C: 中花6号插入变异的长度分布; D: 中花6号缺失变异的长度分布。
A: length distribution of insertion variation in Xuhua 13; B: length distribution of deletion variation in Xuhua 13; C: length distribution of insertion variation in Zhonghua 6; D: length distribution of deletion variation in Zhonghua 6.
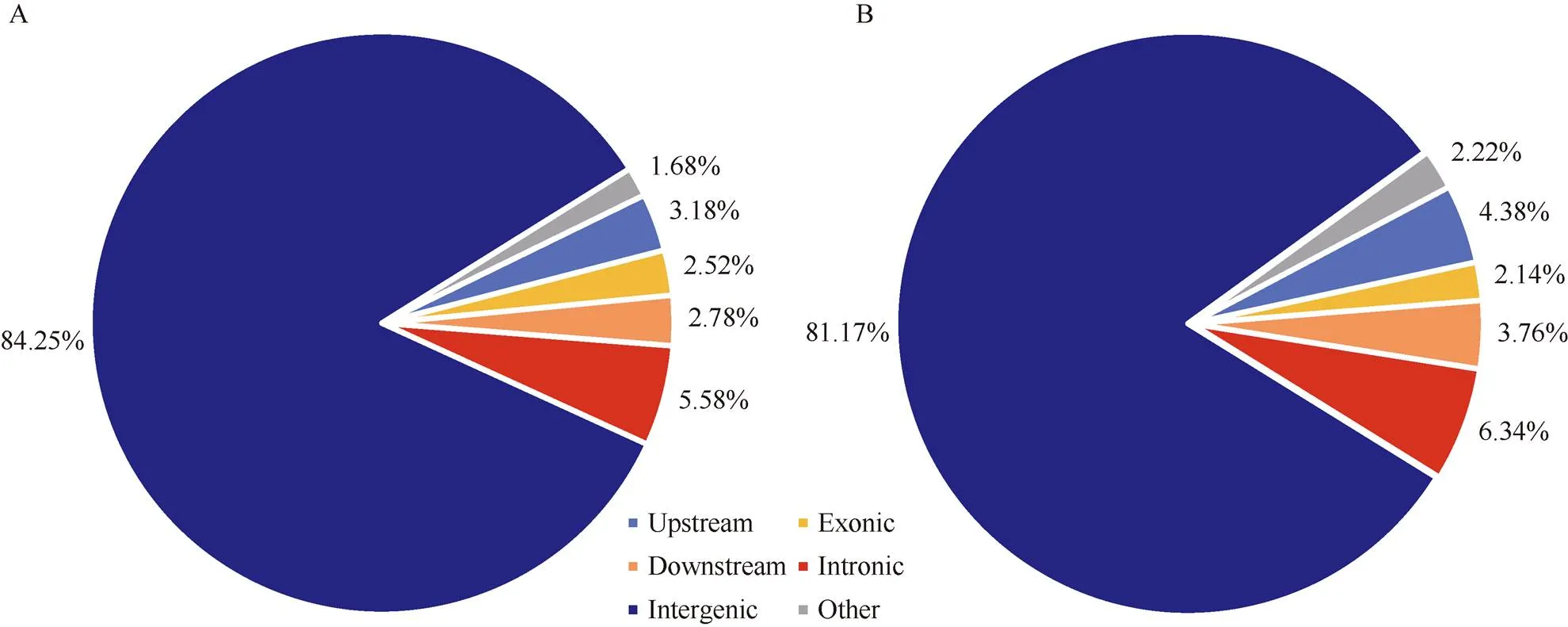
图2 结构变异发生在基因组不同区域的比例
A: 徐花13的结构变异在基因组不同区域的比例; B: 中花6号的结构变异在基因组不同区域的比例。
A: the ratio of different genomic region with structural variations in Xuhua 13; B: the ratio of different genomic region with structural variations in Zhonghua 6.
2.3 含油量表型测定与分析
从徐花13和中花6号杂交组合的RIL群体中(F8), 以目标QTL侧翼的SSR分子标记作为前景选择, 筛选出QTL ()位点为杂合基因型的单株。挑选出均匀分布在各条染色体上的分子标记作为背景选择, 结合前景选择从该单株自交后代筛选获得NIL。利用NIL杂交后代构建1个包含1160个单株的F2次级分离群体用于含油量QTL的定位。测定F2群体花生植株的含油量表型, 统计分析后发现含油量变化范围为43.52%~57.96%, 平均值为50.42%±1.56%, 绘制含油量表型频率分布直方图(图4)。含油量范围分布符合正态分布, 表现为数量性状的遗传特点, 表明该含油量表型数据可靠, 可用于QTL定位研究。
2.4 遗传连锁图谱构建及含油量QTL定位分析
利用筛选获得的9个多态性InDel标记结合F2群体基因型数据分析, 其中共显性标记8个, 分别为M9、M21、M6、M22、M23、M11、M14、M8, M13为显性标记, 去除1个不稳定的共显性标记M14, 获得共包含8个多态性InDel标记, 图谱总长度为149.84 cM的遗传图谱。根据构建的遗传连锁图, 结合F2群体1160个单株的表型数据和基因型分析数据, 对花生含油量QTL ()进行定位, 最终将该含油量QTL定位在标记M23至M11之间, 位于A08染色体1.2 Mb区段内(图5), LOD值为25.21, 能够解释11.21%的表型变异, 加性效应为0.77, 表明与含油量相关的基因来自高含油量亲本徐花13。

图3 亲本中9个多态性InDel标记PCR扩增结果
M1: 2000 bp; M2: 5000 bp. M9: InDel-A08-37791529; M21: InDel-A08-42578036; M6: InDel-A08-44213248; M22: InDel-A08-44339712; M23: InDel-A08-44988232; M11: InDel-A08-46212241; M13: InDel-A08-46400655; M14: InDel-A08-46555343; M8: InDel-A08-49660135.
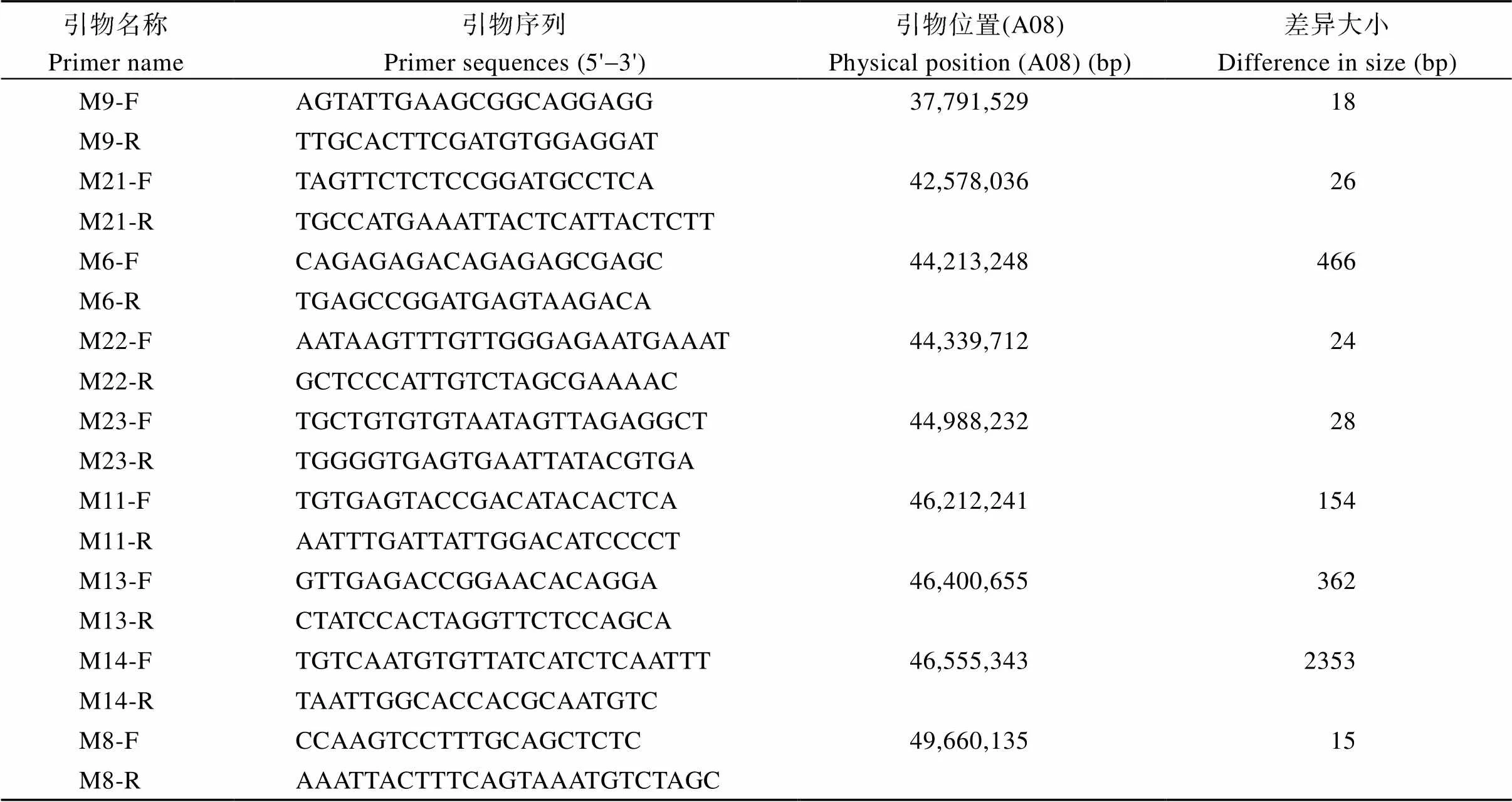
表2 亲本中具有多态性的InDel标记信息

图4 F2群体含油量表型的频率分布
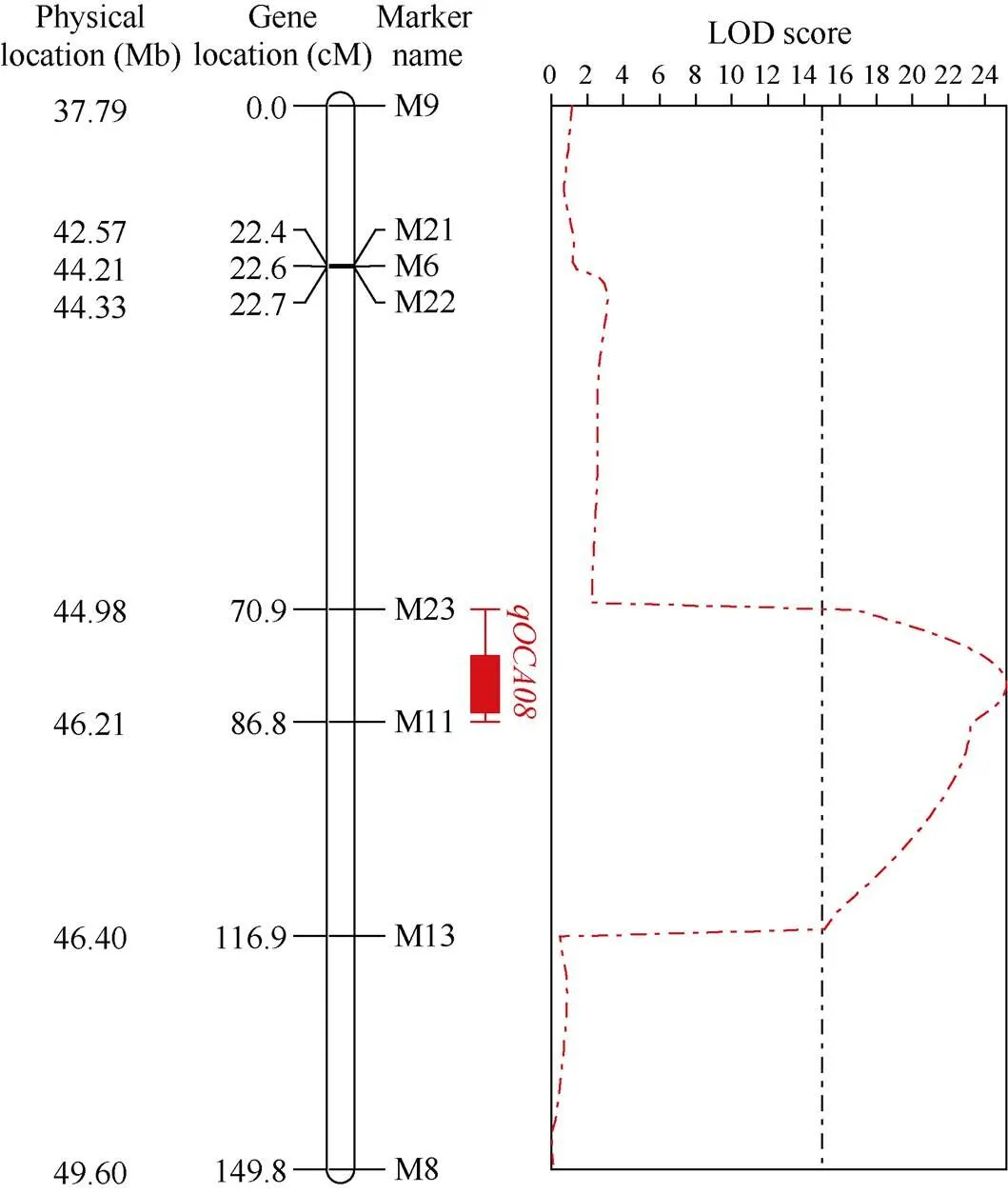
图5 F2群体定位结果
通过对QTL峰值处的标记M11在F2群体中进行分析, 挑出该标记在F2群体中的与亲本基因型相一致的单株, 同时将对应株系的表型数据进行统计, 具有徐花13基因型(AA)的后代有298株, 含油量变化范围为46.87%~56.00%, 平均值为52.60%±1.55%; 具有中花6号基因型(aa)的后代有206株, 含油量变化范围为46.41%~53.83%, 平均值为49.71%±1.41%; 不同基因型间株系含油量相差2.89个百分点, 且在0.01的水平下差异极显著(图6), 表明该InDel标记可用于花生含油量分子标记辅助育种。

图6 M11位点等位基因型含油量的表型效应
**代表在0.01概率水平著差异(检验)。
**represents significant difference at the 0.01 probability level by-test.
3 讨论
随着分子生物学和生物信息学的发展, 分子标记辅助花生育种的应用越来越多。InDel标记在基因组中分布广, 密度大, 是丰富性仅次于SNP、远高于SSR的一种标记类型, 作为重要的分子标记来源, 因其多态性高、稳定性好、不受环境影响, 分型系统简单[21]而得到广泛应用。与SNP标记相比, 虽然InDel标记在基因组中分布密度和多态性频率低于SNP标记, 但其引物设计简单, 多态性检测便捷高效, 只需要普通PCR扩增、琼脂糖凝胶电泳等简单步骤即可达到基因分型的目的。此外InDel标记比需要聚丙烯凝胶电泳检测的SSR标记更节省人力和物力。前人在选择InDel差异位点时, 通常优先选择多态性差异为5~20 bp差异的InDel位点[22-24], 很少关注大片段InDel标记, 扩增产物大多数需要通过聚丙烯酰胺凝胶电泳检测, 操作步骤繁琐。本研究中基于三代测序鉴定到的长片段InDel位点开发引物, 扩增后的产物条带差异效果明显, 在电泳检测中便于区分, 基因分型清晰简单, 用琼脂糖凝胶电泳即可检测。
前人研究中, 通常基于二代重测序技术开发相关性状的分子标记, 而本研究利用PacBio三代测序技术构建超长片段文库, 得到超长的读长, 从而提高大片段变异位点的检出率, 能够解决二代测序中无法跨越高重复和低复杂度区域等问题。通过对序列的结构变异分析可以为InDel分子标记的开发奠定基础, 本研究基于测序数据, 在位点区间内根据InDel差异位点开发出84个InDel标记, 将开发的InDel标记在亲本中进行多态性筛选, 筛选获得具有多态性的InDel标记9个, 多态性频率为10.71%, 扩增产物特异性好且条带清晰。在F2群体中进行基因分析时, 扩增产物带型在株系之间的条带差异效果明显, 便于记录和分析。虽然本研究开发出84个InDel标记, 但只有9个InDel标记具有多态性, 其余标记没有扩增出目的片段或者在亲本间没有表现出多态性, 可能是因为栽培种花生是异源四倍体, 基因组庞大, A亚组和B亚组间同源性程度较高[6], 在检测插入和缺失突变时会受到亚组之间差异的影响, 导致序列同基因组序列比对时可能将来自不同亚组的序列比对到同一位置, 造成假阳性, 因而检测到标记的多态性较低。
由于含油量遗传研究基础薄弱, 使得我国花生高含油量育种进展缓慢。通过检测与含油量相关的QTL位点可以快速有效地指导育种工作, 采用含油量差异明显的亲本杂交组合, 借助分子标记辅助选择技术可以实现多基因聚合, 在早期世代进行选择, 加快育种效率[25]。目前报道的与花生含油量稳定主效QTL位点紧密连锁的标记数量有限, 黄莉等[26]利用远杂9102和中花5号为亲本构建RIL群体, 获得2个与花生含油量相关的SSR标记, 其中标记2A5- 240与高含油量相关, 标记2A5-250与低含油量相关; Wilson等[8]利用野生种衍生的双二倍体TxAG-6与栽培基因型Florunner杂交构建高级回交群体, 获得2个紧密连锁的标记PM36和TC7A02能够提高花生含油量; 郭建斌等[5]利用多态性SSR标记对60份花生材料的含油量进行相关性分析, 获得与含油量极显著相关的标记11个, 其中7个标记与高含油量相关; 刘念等[27]对292份中国花生品种组成的自然群体进行了含油量的表型鉴定, 运用SSR标记鉴定出12个关联标记, 证实标记AGGS1014_2和AHGS0798可以提高RIL群体的含油量; 徐平等[28]对49个花生栽培品种进行转录组分析获得的InDel位点, 通过关联分析发现29个InDel位点与花生含油量相关, 开发出1个可用于分子标记辅助高含油量育种的InDel标记。本研究利用开发的多态性InDel标记对F2群体含油量进行相关性分析, 发现标记M11的InDel位点与含油量极显著相关, 表明该位点可用于高含油量聚合杂交育种。
另外, 为了降低或消除遗传背景对定位结果的影响, 本研究利用目标位点的侧翼标记作为前景选择, 筛选出群体(F8)中杂合基因型的单株, 从其自交后代获得背景一致且在位点等位纯合的NIL。利用NILs杂交后代构建F2群体, 基于测序开发InDel标记对花生含油量复杂性状进行遗传分析, 屏蔽了非重组交换单株的影响, 定位结果更加准确。利用获得的9个InDel多态性标记, 对F2群体的1160个单株进行基因型分析, 获得总长度为149.84 cM的局部遗传连锁图谱, 结合F2群体的表型数据进行QTL定位, 可以解释11.21%的表型变异,表明位点是一个主效的QTL位点, 具有研究和利用的潜力, 这与前人的研究一致[19,29], 最终将QTL ()缩小至A08染色体1.2 Mb区段内。因此, 在本研究基础上进一步开展对含油量主效位点QTL ()的精细定位和相关基因的挖掘将是以后研究的一个重点方向。
4 结论
本研究利用PacBio三代测序技术检测基因组大片段的InDel变异, 在目标区间QTL ()内基于其紧密连锁标记开发了1个可用于分子标记辅助育种与种质评价的InDel标记, 并在F2群体中证实其具有提高花生含油量的作用, 可为花生含油量分子标记辅助育种提供理论依据和技术支撑, 最终将QTL ()缩小至A08染色体1.2 Mb区段内。
[1] 廖伯寿. 我国花生生产发展现状与潜力分析. 中国油料作物学报, 2020, 42: 161–166.
Liao B S. A review on progress and prospects of peanut industry in China., 2020, 42: 161–166 (in Chinese with English abstract).
[2] 张婧妤, 许本波, 郑家喜. 我国食用植物油消费变化分析及改革对策. 中国油脂, 2022, 47(3): 5–10.
Zhang J Y, Xu B B, Zheng J X. Analysis on consumption changes and reform countermeasures of edible vegetable oil in China., 2022, 47(3): 5–10 (in Chinese with English abstract).
[3] 姜慧芳, 任小平. 我国栽培种花生资源农艺和品质性状的遗传多样性. 中国油料作物学报, 2006, 28: 421–426.
Jiang H F, Ren X P. Genetic diversity of agronomic and quality traits in cultivated peanut resources in China., 2006, 28: 421–426 (in Chinese with English abstract).
[4] Yol E, Ustun R, Golukcu M, Uzun B. Oil content, oil yield and fatty acid profile of groundnut germplasm in Mediterranean climates., 2017, 94: 787–804.
[5] 郭建斌, 吴贝, 陈伟刚, 贾朝阳, 荆建国, 陈四龙, 刘念, 陈玉宁, 周小静, 罗怀勇, 任小平, 姜慧芳, 黄莉. 基于核磁共振法的花生品种含油量遗传变异分析. 中国油料作物学报, 2017, 39: 326–333.
Guo J B, Wu B, Chen W G, Jia C Y, Jing J G, Chen S L, Liu N, Chen Y N, Zhou X J, Luo H Y, Ren X P, Jiang H F, Huang L. Variation of oil content in peanut varieties based on nuclear magnetic resonance technology., 2017, 39: 326–333 (in Chinese with English abstract).
[6] Bertioli D J, Cannon S B, Froenicke L, Huang G D, Farmer A D, Cannon E K S, Liu X, Gao D Y, Clevenger J, Dash S, Ren L H, Moretzsohn M C, Shirasawa K, Huang W, Vidigal B, Abernathy B, Chu Y, Niederhuth C E, Umale P, Araújo A C G, Kozik A, Kim K D, Burow M D, Varshney R K, Wang X J, Zhang X Y, Barkley N, Guimarães P M, Isobe S, Guo B Z, Liao B S, Stalker H T, Schmitz R J, Scheffler B E, Leal-Bertioli S C M, Xun X, Jackson S A, Michelmore R, Ozias-Akins P. The genome sequences ofand, the diploid ancestors of cultivated peanut., 2016, 48: 438–446.
[7] Shasidhar Y, Vishwakarma M K, Pandey M K, Janila P, Variath M T, Manohar S S, Nigam S N, Guo B Z, Varshney R K. Molecular mapping of oil content and fatty acids using dense genetic maps in groundnut (L.)., 2017, 8: 794–807.
[8] Wilson J N, Chopra R, Baring M R, Selvaraj M G, Simpson C E, Chagoya J, Burow M D. Advanced backcross quantitative trait loci (QTL) analysis of oil concentration and oil quality traits in peanut (L.)., 2017, 10: 512.
[9] Guo J B, Liu N, Li W T, Wu B, Chen H W, Huang L, Chen W G, Luo H Y, Zhou X J, Jiang H F. Identification of two major loci and linked marker for oil content in peanut (L.)., 2021, 217: 29.
[10] Väli U, Brandström M, Johansson M, Ellegren H. Insertion- deletion polymorphisms (indels) as genetic markers in natural populations., 2008, 9: 8.
[11] Li X M, Gao W H, Guo H L, Zhang X L, Fang D D, Lin Z X. Development of EST-based SNP and InDel markers and their utilization in tetraploid cotton genetic mapping., 2014, 15: 1046.
[12] Weber J L, David D, Heil J, Fan Y, Zhao C F, Marth G. Human diallelic insertion/deletion polymorphisms science direct., 2002, 71: 854–862.
[13] Liu J, Qu J T, Yang C, Tang D G, Li J W, Lan H, Rong T Z. Development of genome-wide insertion and deletion markers for maize, based on next-generation sequencing data., 2015, 16: 601.
[14] 刘栓桃, 张志刚, 司立英, 王荣花, 李巧云, 王立华, 赵智中, 梁水美, 张全芳, 步迅. 基于InDels标记的大白菜育种材料的亲缘关系鉴定. 植物遗传资源学报, 2018, 19: 657–667.
Liu S T, Zhang Z G, Si L Y, Wang R H, Li Q Y, Wang L H, Zhao Z Z, Liang S M, Zhang Q F, Bu X. Identification of genetic relationships of Chinese cabbage inbred lines using InDels markers., 2018, 19: 657–667 (in Chinese with English abstract).
[15] 孟雅宁, 严立斌, 田玉, 范妍芹. 利用重测序InDel位点开发甜椒隐性核不育分子标记. 分子植物育种, 2019, 17: 6041–6046.
Meng Y N, Yan L B, Tian Y, Fan Y Q. Development of recessive genic male sterile molecular markers in sweet pepper using resequencing InDel sites., 2019, 17: 6041–6046 (in Chinese with English abstract).
[16] 季高翔, 何守朴, 潘兆娥, 龚文芳, 贾银华, 王立如, 王朋朋, 耿晓丽, 杜雄明. 基于重测序开发的InDel标记定位陆地棉矮化突变体. 棉花学报, 2018, 30: 448–454.
Ji G X, He S P, Pan Z E, Gong W F, Jia Y H, Wang L R, Wang P P, Geng X L, Du X M. Localization of a dwarfing mutation in upland cotton using InDel markers based on genome re-sequencing data., 2018, 30: 448–454 (in Chinese with English abstract).
[17] Liu L F, Dang P M, Chen C Y. Development and utilization of InDel markers to identify peanut (L.) disease resistance., 2015, 6: 988–998.
[18] Meng S, Yang X L, Dang P M, Cui S L, Mu G J, Chen C, Liu L F. Evaluation of insertion-deletion markers suitable for genetic diversity studies and marker-trait correlation analyses in cultivated peanut (L.)., 2016, 15: gmr.15038207.
[19] 郭建斌. 花生含油量及脂肪酸组成的QTL分析. 华中农业大学硕士学位论文, 湖北武汉, 2016.
Guo J B. QTL Analysis for Oil Content and Fatty Acid Traits in Peanut (L.). MS Thesis of Huazhong Agricultural University, Wuhan, Hubei, China, 2016 (in Chinese with English abstract).
[20] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations., 2015, 3: 269–283.
[21] Jander G, Norris S R, Rounsley S D, Bush D F, Levin I M.map-based cloning in the post-genome era., 2002, 129: 440–450.
[22] Mithra S V A, Kar M K, Mohapatra T, Robin S, Sarla N, Seshashayee M, Singh K, Singh A K, Singh N K, Sharma R P. DBT propelled national effort in creating mutant resource for functional genomics in rice., 2016, 110: 543–548.
[23] 刘阳杰, 何美敬, 崔顺立, 杨鑫雷, 穆国俊, Chen C Y, 刘立峰. 美国花生种质资源果腐病抗性分子标记的筛选及分析. 农业生物技术学报, 2019, 27: 743–751.
Liu Y J, He M J, Cui S L, Yang X L, Mu G J, Chen C Y, Liu L F. Screening and analysis of molecular markers for pod rot resistance in American peanut (L.) germplasm resources., 2019, 27: 743–751 (in Chinese with English abstract).
[24] 郭青青, 周蓉, 陈雪, 陈蕾, 李加纳, 王瑞. 甘蓝型油菜桔红花显性基因候选区域的NGS定位及InDel标记开发. 作物学报, 2021, 47: 2163–2172.
Guo Q Q, Zhou R, Chen X, Chen L, Li J N, Wang R. Location and InDel markers for candidate interval of the orange petal gene inL. by next generation sequencing., 2021, 47: 2163–2172 (in Chinese with English abstract).
[25] 张胜忠, 焦坤, 胡晓辉, 苗华荣, 陈静. 花生百仁质量和含油量的遗传分析. 花生学报, 2018, 47(4): 7–12.
Zhang S Z, Jiao K, Hu X H, Miao H R, Chen J. Genetic analysis for seed mass and oil content of peanuts., 2018, 47(4): 7–12 (in Chinese with English abstract).
[26] 黄莉, 赵新燕, 张文华, 樊志明, 任小平, 廖伯寿, 姜慧芳, 陈玉宁. 利用RIL群体和自然群体检测与花生含油量相关的SSR标记. 作物学报, 2011, 37: 1967–1974.
Huang L, Zhao X Y, Zhang W H, Fan Z M, Ren X P, Liao B S, Jiang H F, Chen Y N. Identification of SSR markers linked to oil content in peanut (L.) through RIL population and natural population., 2011, 37: 967–1974 (in Chinese with English abstract).
[27] Liu N, Huang L, Chen W G, Wu B, Pandey M K, Luo H Y, Zhou X J, Guo J B, Chen H W, Huai D X , Chen Y N, Lei Y, Liao B S, Ren X P, Varshney R K, Jiang H F. Dissection of the genetic basis of oil content in Chinese peanut cultivars through association mapping., 2020, 21: 60.
[28] 徐平, 尹亮, 石延茂, 任艳, 王效华, 李双铃, 袁美. 一个与花生含油量相关的InDel标记的开发. 花生学报, 2017, 46(4): 1–6.
Xu P, Yin L, Shi Y M, Ren Y, Wang X H, Li S L, Yuan M. Identifying of InDel marker associated with oil content in peanut., 2017, 46(4): 1–6 (in Chinese with English abstract).
[29] Liu N, Guo J B, Zhou X J, Wu B, Huang L, Luo H Y, Chen Y N, Chen W G, Lei Y, Huang Y, Liao B S, Jiang H F. High-resolution mapping of a major and consensus quantitative trait locus for oil content to a ~0.8-Mb region on chromosome A08 in peanut (L.)., 2020, 133: 37–49.
Development and employment of InDel marker in peanut QTL mapping of oil content
TAO Shun-Yu, WU Bei, LIU Nian, LUO Huai-Yong, HUANG Li, ZHOU Xiao-Jing, CHEN Wei-Gang, GUO Jian-Bin, YU Bo-Lun, LEI Yong, LIAO Bo-Shou, and JIANG Hui-Fang*
Oil Crops Research Institute, Chinese Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture and Rural Affairs, Wuhan 430062, Hubei, China
The developing of stable and efficient molecular markers is of great value for marker-assisted selection of peanut varieties with highoil content. In this study, two parents with significant differences in oil content, Xuhua 13 (high oil content) and Zhonghua 6 (low oil content), were used to detect genomic structural variation by PacBio third-generation sequencing technology, and 35,794 and 74,703 structural variations were detected in Xuhua 13 andZhonghua 6, respectively. 84 InDel markers in the target region were detected based on the parents’ structural variations and the previous QTL mapping information. And 9 InDel markers were found to be polymorphic between the parents. Meanwhile, F2population with 1160 individual plants was constructed based on near isogenic lines (NILs) derived from heterozygous residuals in the recombined inbred line (RIL) population. The polymorphic markers were used to genotype the population and to construct the genetic linkage map with 149.84 cM. Combining with phenotype data of population for oil content, QTL () was fine mapped between marker M23 and marker M11, which was located on 1.2 Mb interval of chromosome A08. This study demonstrated the feasibility of InDel marker for peanut QTL mapping. The locus of oil content and closely linked InDel markers can provide the theoretical and technical guidance for peanut molecular marker-assisted breeding.
peanut; genome resequencing; InDel; oil content
10.3724/SP.J.1006.2023.24140
本研究由广东省重点领域研发计划项目(2020B020219003), 国家自然科学基金项目(31801403, 31871666), 农作物种质资源保护项目(2017NWB033), 国家农作物种质资源共享服务平台(NICGR2017-36), 财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-13-花生种质资源评价)和中国农业科学院创新工程项目(2022-2060299-089-031)资助。
This study was supported by the Key Area Research and Development Program of Guangdong Province (2020B020219003), the National Natural Science Foundation of China (31801403, 31871666), the Crop Germplasm Resources Protection Project (2017NWB033), the Plant Germplasm Resources Sharing Platform (NICGR2017-36), the China Agriculture Research System of MOF and MARA (CARS13), and the Innovation Project of Chinese Academy of Agricultural Sciences (2022-2060299-089-031).
姜慧芳, E-mail: peanutlab@oilcrops.cn
E-mail: ynautsy@163.com
2022-06-10;
2022-07-21;
2022-09-23.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220922.1605.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

