海南稻作区稻瘟病菌AvrPik等位基因的变异监测与多样性分析
危艺可 王倩 吴伟怀 陆英 贺春萍 梁艳琼 易克贤


摘要 海南省地處热带。由于具有国内其他地区无可比拟的光温资源优势,每年有大量的水稻材料在南繁区繁育与种植,给稻瘟病菌的变异及传播带来了极大的便利。自20世纪70年代以来,Pik等位基因已被广泛地用作水稻抗稻瘟病育种的主要抗源。为了解海南稻瘟病菌AvrPik等位基因的变异及多样性,本研究从海南稻作区采集穗茎瘟病样,通过单孢分离获得100株稻瘟病菌株。利用AvrPik基因特异性引物扩增病原菌DNA,所得PCR产物经克隆、测序后与参考序列进行比对分析。结果从100株菌株中鉴定出AvrPik_A、AvrPik_B、AvrPik_D、AvrPik_E和AvrPik_F共5种等位基因类型。其中AvrPik_D类型最多,出现频率为50.00%;其次为AvrPik_E类型,出现频率为32.08%;第三为AvrPik_B类型,出现频率为8.49%。在AvrPik等位基因核苷酸多样性方面,非信号肽区域多样性要明显高于信号肽区域。就群体受到的选择压而言,南繁种植区与常规种植区的稻瘟病菌群体的Ka/Ks值均大于1,分别为2.703 1和1.236 6,表明,无论是南繁区还是常规种植区,AvrPik等位基因均受到了强烈的正选择压,且南繁区群体受到的选择压更强。上述5种不同基因型的代表菌株中菌株20MG48 (AvrPik_F)长势最快,其产孢量却最少。本研究初步明确了海南稻作区AvrPik等位基因的群体结构,为本稻作区抗性品种的布局以及抗瘟育种计划提供参考。
关键词 稻瘟病菌; AvrPik等位基因; 多样性
中图分类号: S 435.111.41
文献标识码: A
DOI: 10.16688/j.zwbh.2021682
Abstract Hainan province is located in the tropical region. Because of the incomparable advantages of light and temperature resources in Hainan over the other regions in China, a large number of rice varieties are bred and planted in off-season breeding areas in Hainan every year, which brings great convenience to the variation and transmission of Magnaporthe oryzae. The major resistant gene Pik for rice blast and its alleles have been extensively exploited as sources of genetic resistance in rice breeding programs since 1970s. In order to understand the variation and diversity of AvrPik alleles in Magnaporthe oryzae in rice-growing areas of Hainan, 100 M. oryzae strains were isolated from the rice growing areas of Hainan. The AvrPik genes were cloned from the genomic DNA, sequenced and then compared with the reference sequences. Finally, five main allele genotypes including AvrPik_A, AvrPik_B, AvrPik_D, AvrPik_E and AvrPik_F were identified. The frequencies of AvrPik_D, AvrPik_E and AvrPik_B were 50.00%, 32.08%, 8.49%, respectively. In terms of the nucleotide diversity of AvrPik alleles, the nucleotide diversity of non-signal peptide regions was obviously higher than that of signal peptide regions. In terms of selection pressure on populations, the Ka/Ks ratios of M.oryzae populations from off-season breeding area and the conventional cultivation area were all greater than 1 (2.703 1 and 1.236 6, respectively). These results indicated that AvrPik alleles in both growing areas were subjected to strong positive selection pressure, and the pressure on AvrPik alleles in off-season breeding area were stronger than that in the conventional area. The hypha of strain 20MG48 (AvrPik_F) grew fastest, but its spore production was the least among the five representative strains. The population structure of Avrpik alleles was preliminarily clarified in rice-growing areas of Hainan province, which provides a reference for the planting of resistant varieties and the breeding program for blast resistance in the rice growing area.
Key words Magnaporthe oryzae; AvrPik alleles; diversity
海南省地处热带,素有“天然大温室”之美称,具有国内其他地区无可比拟的光温资源优势,是我国著名的南繁基地。几十年来,“南繁”在加速水稻品种改良、原原种扩繁和制种方面为国家农业发展做出了巨大贡献。我国育成的杂交水稻新组合中80%以上经过了南繁加代选育。生产上使用品种的更新周期由过去的10年以上缩短到目前的5~7年[1]。南繁已经成为新品种选育的“孵化器”和“加速器”,而南繁基地则被誉为中国种业的“硅谷”[2]。与此同时,南繁成为“全国和世界危险性有害生物的汇集地及中转站”的风险也逐渐加大。生物安全将成为南繁最大的威胁,如控制不力,海南将成为有害生物的大“染缸”。就水稻稻瘟病而言,作为水稻新品种培育、种子生产和质量鉴定的南繁基地,每年都有大批的种子、种苗频繁出入基地,给稻瘟病菌的变异及传播带来了极大的便利。
稻瘟病是全球水稻生产上最具毁灭性的病害之一[3]。种植抗病品种是防治该病最经济、环保的策略。然而,水稻寄主特异抗性的表达不仅取决于该品种本身的抗病基因型,还取决于稻瘟病菌的无毒基因型[4-5]。因此,准确地了解稻瘟病菌无毒基因的群体结构及其变异规律,是实现抗性品种合理培育与利用,以及病情预测预报所面临的课题。
Pik位点是位于水稻第11号染色体长臂近端粒处的一个主效抗稻瘟病位点。由于该位点具有Pi-k、Pi-kp、Pi-km、Pi-kh等多个等位基因[6-12],且均对水稻稻瘟病具有较高的抗性,因此,自20世纪70年代以来,这些基因被广泛地用作水稻抗稻瘟病育种的主要抗源[6, 10, 13-14]。目前该位点的合理持续应用仍有很大的挖掘潜力[6, 13, 15-17]。与Pik等位基因对应的无毒基因为AvrPi-k/-kp/-km/-kh (后续简写为AvrPik)[16, 18-20]。
无毒基因AvrPik从稻瘟病菌Magnaporthe oryzae日本菌株Ina168中发现,编码由113个氨基酸组成的分泌蛋白,含有2个保守结构域,该蛋白能被水稻抗性基因Pik编码的蛋白识别,并引起防御响应[18]。田间稻瘟病菌群体中AvrPik等位基因既有基因存在/缺失,又有单碱基突变,Yoshida等检测了日本田间稻瘟病菌菌株的AvrPik等位基因型,并将不同点突变类型命名为AvrPik_A、AvrPik_B、AvrPik_C、AvrPik_D、AvrPik_E等[18]。Wu等分析了来自我国广东、湖南、黑龙江、辽宁的240株稻瘟病菌菌株的AvrPik等位基因,发现在编码区共存在16个SNP (single nucleotide polymorphism, SNP),其中4个SNP在所测试的菌株中广泛存在,且与Pik等位基因之间存在阶梯性“军备竞赛”关系[19]。Zhang等利用Avr基因特异性引物对1975年-2009年从美国南部商业农田采集的258株稻瘟病菌基因组DNA进行PCR扩增及测序,检测出AvrPik_F、AvrPik_G、AvrPik_H、AvrPik_I、AvrPik_J、AvrPik_K等新类型[21]。然而,海南作为水稻的南繁区以及重要生产区,其稻瘟病菌无毒基因AvrPik的群体结构及其变异仍不清楚。为此本文开展了海南稻作区无毒基因群体结构的研究,研究结果将有助于抗瘟材料的创制及应用,对保障国家粮食安全和种业发展安全具有不可替代的作用。
1 材料与方法
1.1 病样采集与单孢分离
在水稻生长季节,从海南三亚、陵水、乐东、保亭、东方、琼中、白沙、屯昌、琼海、定安、文昌等地水稻田采集穗茎瘟病样。获得的稻瘟病样品于室内常温保湿,24 h后于超净台上进行单孢分离。用于本研究的100个菌株来源信息见表1。其中61个菌株来自三亚、陵水、乐东、保亭和东方的南繁育种区,39个菌株来自琼中、白沙、屯昌、琼海、定安和文昌的常规种植区。所获菌株保存于中国热带农业科学院环境与植物保护研究所。
1.2 菌丝体的培养及DNA提取
将单孢菌株从PDA试管转移至PDA平板培养,5 d后挑取2~3个菌丝块于液体培养基(1%葡萄糖,0.3%酵母粉)中,28℃,180 r/min振荡培养4~5 d。用灭菌纱布和滤纸过滤收集菌丝体,冻干后于-20℃冰箱中保存备用。病原菌基因组总DNA提取参照OMEGA真菌DNA小量提取试剂盒说明书进行。
1.3 PCR扩增与AvrPik基因克隆测序
参照GenBank登录号AB498875~AB498879的序列设计AvrPik等位基因的特异性引物AvrPikF (5′-GAAGGTCACGGTTTGAAGAGG-3′)和AvrPikR (5′-ATTATCTTATGAGCCGTCAACCA-3′)。引物由英濰捷基(广州)贸易有限公司合成。以该引物扩增各菌株的总DNA,预期大小为615 bp。PCR反应总体系为20 μL:包含10×PCR buffer 2 μL,10× dNTPs 1.6 μL,rTaq酶 0.2 μL,10 μmol/L正、反向引物各0.5 μL,模板DNA 1.0 μL,ddH2O 14.2 μL。PCR扩增条件为:94℃预变性3 min;94℃变性30 s,60℃退火15 s,72℃延伸50 s,34个循环;72℃延伸5 min,最后于4℃保存。PCR产物经1.2%琼脂糖凝胶电泳检测,根据扩增产物的纯度直接或回收后与克隆载体pEASYTM连接。连接产物转化大肠杆菌感受态细胞,经蓝白斑筛选后选取3个阳性克隆送英潍捷基(广州)贸易有限公司测序。
1.4 AvrPik等位基因的FNP分型
每个克隆的序列去除载体序列后,以AB498875~AB498879标准序列[18]作为参考,对编码区第136、139、143、200、233、234等6个功能性核苷酸多态性位点(functional nucleotide polymorphism,FNP)进行比对。上述位点碱基如与AB498876一致则该序列(菌株)记为AvrPik_A型;如与AB498877一致则该序列(菌株)记为AvrPik_B型;如与AB498878一致则该序列(菌株)记为AvrPik_C型;如与AB498875一致则该序列(菌株)记为AvrPik_D型;如与AB498879一致则该序列(菌株)记为AvrPik_E型。其余类型则参照Zhang等[21]。
1.5 DNA核苷酸多态性与选择压分析
核苷酸多态性主要由Tajimas π指标[22]来反映。π值通过软件DnaSP version 5.0[23]计算。选择压通过Ka/Ks值来反映。利用DnaSP version 5.0分别计算Ka (非同义替换率, non-synonymous substitution rate)与Ks (同义替换率, synonymous substitution rate),进而计算Ka/Ks值。如果Ka/Ks>1,则认为存在正选择效应;如果Ka/Ks=1,则认为存在中性选择效应;如果Ka/Ks<1,则认为存在负选择效应。
1.6 具有不同AvrPik等位基因类型菌株培养观察
经序列比对筛选,选取具有不同AvrPik 等位基因类型的菌株,于PDA平板上培养,10 d后测量菌落直径。于超净台中,加少量灭菌蒸馏水用药勺将气生菌丝刮洗干净,置于自制产孢箱28℃培养2~4 d,加1 mL灭菌双蒸水(ddH2O)配制孢子悬浮液,于显微镜下观察分生孢子形态,通过血球计数板计算产孢量。
2 结果与分析
2.1 AvrPik等位基因关键FNP位点分型
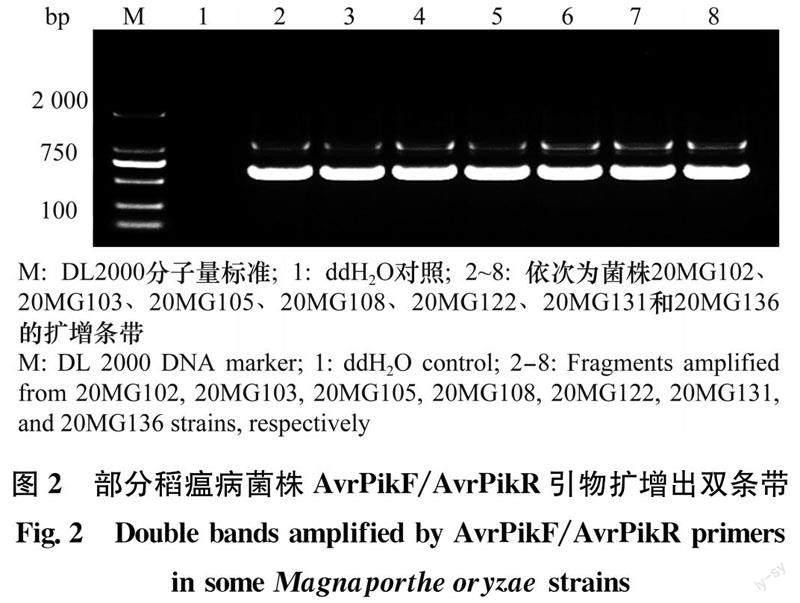
利用AvrPikF/AvrPikR引物扩增菌株DNA,100个菌株DNA中有93个菌株DNA扩增出预期大小(615 bp)的特异条带(图1),其余7个菌株扩增出双条带,其中一条与目标条带大小一致(615 bp),另外一条略大于目标条带(1 115 bp) (图2)。
93个条带单一菌株的PCR产物克隆后经测序比对发现,其中6个菌株(20MG158、20MG167、20MG201、20MG204、20MG206、MG42)存在两种不同类型的AvrPik等位基因,因此共得到99条DNA序列。而7个双条带的菌株(20MG102、20MG103、20MG105、20MG108、20MG122、20MG131、20MG136),经胶回收测序后序列比对表明,同一样品的两个条带均含有完整且相同的AvrPik等位基因,只是比目标条带大的条带(1 115 bp)在编码区终止密码子后有一个500 bp的序列插入,故均只保留目标条带序列用于后续分析。
最终,从供试的100个菌株中共获得了106条AvrPik等位基因序列。其中来自南繁区菌株序列63条,常规种植区菌株序列43条。经与已报道的参考序列进行比对,在342 bp的编码区中存在6个FNP位点(#136、#139、#143、#200、#233、#234)。最终鉴定出AvrPik_A、AvrPik_B、AvrPik_D、AvrPik_E、AvrPik_F共5种AvrPik等位基因型(表2)。其中AvrPik_D检出最多,其检出频率为50.00%,为优势类型;其次为AvrPik_E,其检出频率为32.08%。然而,在这106条菌株序列中没有检测到AvrPik_C类型。
2.2 AvrPik等位基因核苷酸多样性与选择压分析
将测序获得的106条序列输入MEGA软件进行比对,发现AvrPik等位基因的编码区中共存在62个SNP位点,其中9个同义突变(#43、#87、#96、#111、#150、#162、#246、#249、#279),53个非同义突变。非同义突变中C136A、C139G、G143A、C200A、T233A、G234A位点普遍存在,即关键的6个FNP位点。
进一步从结构域与群体水平方面分析AvrPik等位基因核苷酸多样性。在结构域水平上,整個编码区共检测到62个SNP位点,其中7个来自信号肽区,55个来自非信号肽区。非信号肽区的核苷酸多样性(0.008 92)明显高于信号肽区(0.002 97)。表明AvrPik等位基因核苷酸多样性在这两个结构域之间存在明显差异(表3)。在群体方面,南繁区与常规种植区群体检测出变异位点数分别为43个和24个,其对应的核苷酸多样性也是南繁区(0.009 26)高于常规种植区(0.005 02) (表3)。
结构域与群体的Ka/Ks检验表明,Ka/Ks值在非信号肽区与信号肽区分别为2.054 8与3.223 8。可见AvrPik等位基因受到正选择压作用(表3)。在群体方面,南繁区(2.703 1)的Ka/Ks值明显高于常规种植区(1.236 6)。表明,南繁区的AvrPik等位基因受到了强烈的正选择压,且强于常规种植区受到的选择压(表3)。
2.3 不同AvrPik等位基因的菌株生长特性比较
经序列比对确认后,选取只在6个FNP位点存在差异(其余位点均一致)的菌株,即20MG56 (AvrPik_A)、20MG189 (AvrPik_B)、20MG108 (AvrPik_D)、20MG27 (AvrPik_E)、20MG48 (AvrPik_F)共5个菌株进行表型观察。培养10 d后,5个菌株的菌落形态均为圆形;除20MG27 (AvrPik_E)菌丝体为黑灰色外,其余菌株菌丝均为灰白色(图3)。在生长速率方面,菌株20MG48 (AvrPik_F)生长最快,其平均菌落直径为65.8 mm;菌株20MG189 (AvrPik_B)生长最慢,其平均菌落直径为46.9 mm (表4)。显微镜观察显示,各个菌株的孢子形态无差异。在产孢方面,菌株20MG189 (AvrPik_B)和20MG108 (AvrPik_D)的产孢量较大,平均产孢数分别为8.75×107个/皿和8.55×107个/皿;菌株20MG48 (AvrPik_F)的产孢量最少,平均产孢数为9.0×106个/皿(表4)。
3 结论与讨论
海南省虽然不是中国的稻米生产大省,但得天独厚的气候优势使其成为全国杂交水稻种子生产大省。海南杂交水稻种子生产优势区域规划面积约为1.27万 hm2,主要集中在北部的临高,南部的乐东、三亚、陵水及周边地区[1]。与南繁基地形成鲜明对比的是,常规水稻生产分布于全岛,多为小农户生产模式,管理粗放,部分稻田稻瘟病、白叶枯病、稻飞虱、稻纵卷叶螟等病虫害发生严重[2]。由于水稻生产在海南尚未形成一个高效产业,故在水稻病虫害防控及科研投入方面还比较薄弱。针对海南稻瘟病菌的研究主要侧重于水稻品种对稻瘟病的抗性调查、鉴定与评价方面[2, 24]。
然而,无论是南繁育种区还是常规水稻种植区,稻瘟病均是水稻生产所面临的障碍。因此,系统、准确地了解海南稻瘟病菌的群体遗传结构及其变异规律是一个不可回避的课题。朱名海等对6个无毒基因(ACE1、AvrPia、AvrPik、AvrPita、AvrPiz-t和PWL2)在南繁核心区和非核心区60个稻瘟病菌菌株中的分布情况进行了PCR检测,初步明确了这6个无毒基因在南繁区稻瘟病菌菌株中的分布规律[25]。本研究利用AvrPik基因特异性引物对海南稻作区分离的100株穗茎瘟菌株进行DNA扩增并对PCR产物进行克隆和序列分析。结果鉴定出AvrPik_A、AvrPik_B、AvrPik_D、AvrPik_E和AvrPik_F共5种主要等位基因类型,其中AvrPik_D类型最多,出现频率为50.00%。2012年Kanzaki等[26]对来自欧洲(3个)、美洲(6个)、非洲(7个)、亚洲(23个)共计39个稻瘟病菌菌株的AvrPik等位基因型进行了分析,其中AvrPik_D为主要类型,本研究结果与其一致。不同的是Kanzaki等没有检测出AvrPik_B类型,而本研究未检测出AvrPik_C类型。Li等对我国云南省东南、西南、东北以及西北稻作区的366个稻瘟病菌株进行检测,发现AvrPik等位基因检出率为66.7%~90.3%,并且除AvrPik_A、B、C、D、E类型之外,还鉴定出5个不同的等位基因类型[27]。由此可见,AvrPik等位基因存在地理分化,在不同水稻生产区出现了不同的变异,因此在生产应用过程中,种植含有Pik等位基因抗性品种时,应掌握目标区域AvrPik等位基因类型的组成和分布情况,科学地进行水稻稻瘟病抗性品种布局,有的放矢地开展抗瘟育种工作。
对南繁区与常规种植区病原菌群体核苷酸多样性及其所承受的选择压分析表明,南繁区病原菌AvrPik等位基因的核苷酸多样性与选择压均高于常规种植区。其原因可能是两个区域稻种资源的抗性水平存在差异。与常规种植区相比,南繁区稻种资源更丰富,每年有大量包括Pik等位基因在内的抗源材料或抗性品种进入南繁区选育、加代种植等。而在稻瘟病菌与水稻长期协同进化过程中,AvrPik等位基因受到了水稻抗性基因Pik的定向选择,与此同时,AvrPik等位基因为了逃避Pik等位基因的识别产生了更加丰富的变异。
本研究观察了5个含有不同AvrPik等位基因型的代表菌株的生长特性和产孢能力。结果表明,5个菌株在菌落生长以及产孢能力方面存在一定差异。这一结果与郭敬玮等对云南罗平120个田间稻瘟病菌株性状研究结果一致[28]。本研究中5个菌株的差异是否与AvrPik等位基因型相关联,还有待选取更多的田间菌株进行培养比较,以及进一步通过克隆与遗传转化,获得单个基因的阳性克隆子进行深入研究。
参考文献
[1] 钟兆飞. 海南省杂交水稻种子产销情况分析[J]. 杂交水稻, 2014, 29(3): 35-37.
[2] 唐清杰, 严小微, 孟卫东. 海南水稻生产现状分析及发展对策[J]. 杂交水稻, 2015, 30(1): 1-5.
[3] ZHANG Shijie, XU Jinrong. Effectors and effector delivery in Magnaporthe oryzae [J/OL]. PLoS Pathogens, 2014, 10(1): e1003826. DOI: 10.1371/journal.ppat.1003826.
[4] ZHANG Shulin, WANG Ling, WU Weihuai, et al. Function and evolution of Magnaporthe oryzae avirulence gene AvrPib responding to the rice blast resistance gene Pib [J/OL]. Scientific Reports, 2015, 5(1): 11642. DOI: 10.1038/srep11642.
[5] ORTIZ D, GUILLEN K D, CESARI S, et al. Recognition of the Magnaporthe oryzae effector AVR-Pia by the decoy domain of the rice NLR immune receptor RGA5 [J]. Plant Cell, 2017, 29(1): 156-168.
[6] 陳子强, 田大刚, 梁廷敏, 等. 229份水稻品种及重要育种材料抗稻瘟病Pik位点基因型鉴定[J]. 福建农业学报, 2016, 31(6): 553-559.
[7] SHARMA T R, MADHAV M S, SINGH B K, et al. High-resolution mapping, cloning and molecular characterization of the Pi-kh gene of rice, which confers resistance to Magnaporthe grisea [J]. Molecular Genetics and Genomics, 2006, 274(6): 569-578.
[8] LI Luoye, WANG Ling, JING Jinxue, et al. The Pikm gene, conferring stable resistance to isolates of Magnaporthe oryzae, was finely mapped in a crossover-cold region on rice chromosome 11 [J]. Molecular Breeding, 2007, 20(2): 179-188.
[9] XU Xin, HAYASHI N, WANG Chuntai, et al. Efficient authentic fine mapping of the rice blast resistance gene Pik-h in the Pik cluster, using new Pik-h-differentiating isolates [J]. Molecular Breeding, 2008, 22(2): 289-299.
[10]WANG Ling, XU Xiaoke, LIN Fei, et al. Characterization of rice blast resistance genes in the Pik cluster and fine mapping of the Pik-p locus [J]. Phytopathology, 2009, 99(8): 900-905.
[11]YUAN Bin, ZHAI Chun, WANG Wenjuan, et al. The Pik-p resistance to Magnaporthe oryzae in rice is mediated by a pair of closely linked CC-NBS-LRR genes [J]. Theoretical & Applied Genetics, 2011, 122(5): 1017-1028.
[12]ZHAI Chun, ZHANG Yu, YAO Nan, et al. Function and interaction of the coupled genes responsible for Pik-h encoded rice blast resistance [J/OL]. PLoS ONE, 2014, 9(6): e98067. DOI: 10.1371/journal.pone.0098067.
[13]张羽, 冯志峰, 崔明珠, 等. 稻瘟病抗性基因Pi-km 在陕西省稻种资源中的分布状况[J]. 分子植物育种, 2013, 11(3): 311-316.
[14]ZHAI Chun, LIN Fei, DONG Zhongqiu, et al. The isolation and characterization of Pik, a rice blast resistance gene which emerged after rice domestication [J]. New Phytologist, 2011, 189(1): 321-334.
[15]刘辉, 孟德龙, 査日扬, 等. 江苏水稻品种稻瘟病主效抗性基因鉴定及应用评价[J]. 福建农业学报, 2015, 30(5): 452-458.
[16]ZDRZAEK R, KAMOUN S, TERAUCHI R, et al. The rice NLR pair Pikp-1/Pikp-2 initiates cell death through receptor cooperation rather than negative regulation [J/OL]. PLoS ONE, 2020, 15(9): e0238616. DOI: 10.1371/journal.pone.0238616.
[17]PENG Zhirong, LI Ling, WU Shenghai, et al. Frequencies and variations of Magnaporthe oryzae avirulence genes in Hunan province, China [J]. Plant Disease, 2021, 105(12): 3829-3834.
[18]YOSHIDA K, SAITOH H, FUJISAWA S, et al. Association genetics reveals three novel avirulence genes from the rice blast fungal pathogen Magnaporthe oryzae [J]. Plant Cell, 2009, 21(5): 1573-1591.
[19]WU Weihuai, WANG Ling, ZHANG Shu, et al. Stepwise arms race between AvrPik and Pik alleles in the rice blast pathosystem [J]. Molecular Plant Microbe Interactions, 2014, 27(8): 759-769.
[20]MAIDMENT J H R, FRANCESCHETTI M, MAQBOOL A, et al. Multiple variants of the fungal effector AVR-Pik bind the HMA domain of the rice protein OsHIPP19, providing a foundation to engineer plant defense [J/OL]. Journal of Biological Chemistry, 2021, 296: 100371. DOI: 10. 1016/j. jbc. 2021.100371.
[21]ZHANG Zhen, JIA Yulin, WANG Yanli, et al. A rapid survey of avirulence genes in field isolates of Magnaporthe oryzae [J]. Plant Disease, 2020, 104(3): 717-723.
[22]TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism [J]. Genetics, 1989, 123(3): 585-595.
[23]LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 2009, 25(11): 1451-1452.
[24]唐清杰, 王效宁, 云勇, 等. 海南普通野生稻稻瘟病的抗性鉴定与评价[J]. 中国野生植物资源, 2010, 29(6): 8-10.
[25]朱名海, 赵美, 舒灿伟, 等. 南繁区稻瘟病菌无毒基因的检测[J]. 华中农业大学学报, 2017, 36(4): 21-25.
[26]KANZAKI H, YOSHIDA K, SAITOH H, et al. Arms race co-evolution of Magnaporthe oryzae AVR-Pik and rice Pik genes driven by their physical interactions [J]. The Plant Journal, 2012, 72(6): 894-907.
[27]LI Jinbin, WANG Qun, LI Chengyun, et al. Novel haplotypes and networks of AVR-Pik alleles in Magnaporthe oryzae [J/OL]. BMC Plant Biology, 2019, 19(1): 204. DOI: 10.1186/s12870-019-1817-8.
[28]郭敬瑋, 肖倩, 李怡然, 等. 云南罗平田间稻瘟菌株性状及无毒基因研究[J]. 植物病理学报, 2021, 51(2): 235-247.
(责任编辑:杨明丽)

