微生物组生物合成基因簇发掘方法及应用前景
赖奇龙,姚帅,查毓国,白虹,宁康
(华中科技大学生命科学与技术学院,分子生物物理教育部重点实验室,生物信息与分子成像湖北省重点实验室,人工智能生物学研究中心,生物信息与系统生物学系,湖北 武汉 430074)
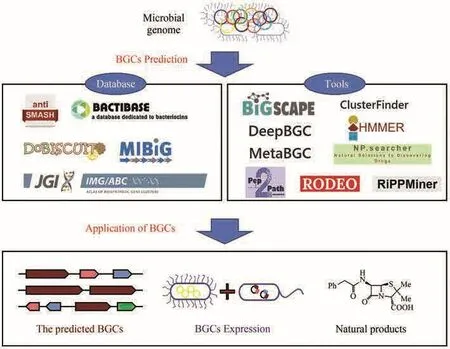
1 生物合成基因簇:序列与功能
天然产物(natural product, NP)是指生物体内的组成成分或其代谢产物,具有广泛的应用价值[1],其中源自微生物的次级代谢产物,在生物医学、工业和农业应用中具有重要意义[2]。然而,由于大量环境微生物无法培养[3],因此挖掘生物合成基因簇(biosynthetic gene cluster, BGC)以检验并生产新型NP当前仍十分困难[4]。在过去的数十年里,随着高通量测序技术和生物大数据处理工具的快速发展,直接从宏基因组(metagenome)中探索BGC的策略已经越来越成熟[5],这极大地加快了从不可培养微生物(包括极端微生物和稀有微生物等)中发掘新型BGC的进度[6]。
生物合成基因簇是一类非常重要的基因集合(gene set)类型。一个BGC通常包含数个到上百个功能基因,共同产生一个或者若干个小分子代谢物[7]。例如,合成青霉素的一系列基因,就共同组成了一个BGC[8]。从现有实验验证过的BGC来看,BGC在序列上和功能上均有鲜明的特征:
从序列上来说,一般情况下,一个BGC所囊括的基因,即参与代谢途径中生物合成酶的基因在染色体上成簇排列[9]。例如,青霉素的合成由三个基因控制,分别是pcbAB、pcbC和penDE,这三个基因位于同一条染色体上[10][图1(a)]。
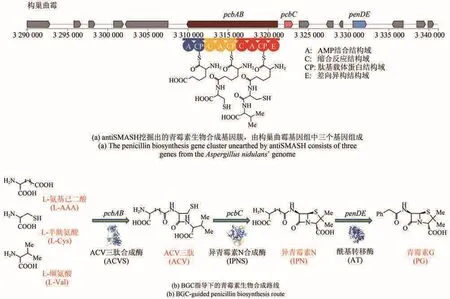
图1 BGC在序列和功能上的特征示意图(以青霉素的生物合成为例)Fig.1 Schematic diagram for sequences and functions of BGC (with penicillin biosynthesis as an example)
从功能上来说,一个BGC所囊括的基因,通常共同产生一个或者若干个小分子化合物[11][图1(b)]。次生代谢产物(secondary metabolites,SM)是BGC合成的主要产物[12],大部分具有生物活性,通常是低分子量的化合物,在生长和发育的特定阶段产生,这类分子最知名的临床应用包括抗生素(如青霉素)、免疫抑制剂(如环孢菌素)等[13]。又例如,翻译后修饰核糖体多肽(ribosomally synthesized posttranslationally modified peptide, RiPP),是由核糖体合成,经由翻译后修饰得到的一大类天然产物,具有广泛的结构和生物活性多样性[14]。由于其化学结构比其他天然产物更具基因组学数据上的可预测性,因此可以通过识别编码RiPP的BGC,在宏基因组中发现新型的RiPP[15]。
现有数据库中的BGC通常是通过湿实验确定的。例如,MIBiG数据库[16]详细记录了来自于上千个微生物物种的上千个经实验验证的非冗余BGC。实验验证的工作流程包括新型天然产物的发现和生物合成,这种手段极大地促进了丰富但尚未开发微生物BGC的挖掘[17]。在来自世界各地科学家的共同贡献下,MIBiG数据库于近期又有更新,包括2019年新增的851个条目[18],以及2022年对现有条目的重新注释与661个新条目的大规模验证[19],目前该数据库收录了2502条已验证的BGC信息。
然而,基于湿实验确定BGC非常复杂且费时,因此一些BGC数据库和计算机比对方法应运而生,如基于局部比对算法的搜索工具(basic local alignment search tool, BLAST)[20]与隐马尔可夫模型(hidden Markov model, HMM)[21]。通过数据库的搜索,能够较为便捷地在基因组中发掘跟已知BGC同源的BGC。例如,antiSMASH数据库[22](https://antismash.secondarymetabolites.org/)中包含了所有NCBIGenBank数据库上公布(截止至2022年11月17日)的可用细菌基因组信息(25 802生物物种的82 855条信息)。antiSMASH数据库为研究者提供了一个使用方便、注释了生物合成基因簇的最新集合,以及配套的进行生物合成基因簇搜索分析的方法。然而,针对已知BGC的远源BGC,当前基于数据库的同源搜索尚不能完全胜任。近年来,基于机器学习和深度学习的方法以预测核糖体合成和翻译后修饰肽(RiPP)为重点的方法迅猛增加[23]。下文将通过详细的实例阐明机器学习方法的特点以及其在BGC挖掘中的应用,如metaBGC[24]和DeepBGC[25]等。
2 基于微生物组的生物合成基因簇挖掘与转化研究
许多微生物的次级代谢产物具有抗真菌、抗细菌、抗肿瘤等生物活性,是微生物药物开发和新药创制的重要来源[26]。目前,放线菌和黏细菌等是细菌次级代谢调控和天然产物发掘的重要研究对象[27]。但是,目前对于细菌能合成多少种次级代谢产物、不同类群的细菌在合成次级代谢产物能力方面的差异以及次级代谢产物生物合成基因簇(以下简称次级代谢基因簇)如何进化等问题,尚存在很多未知规律和模式,仍有待研究[28]。
当前,由于BGC转化应用具有广泛的应用价值,重要的BGC通常通过干湿实验共同确定[29]。例如,2022年武汉大学药学院刘天罡课题组[30]开发了“基因簇功能元件理性可控重组”策略,实现了萜类沉默基因簇的批量挖掘及高效合成。这一工作展示了以“基因簇功能元件理性可控重组”策略为指导,从微生物基因组数据出发,进行新化合物挖掘、筛选并实现目标产物高效合成的巨大优势。该项工作详细介绍了从基因组挖掘到萜类化合物生物合成与鉴定的全套流程,为利用人工智能方法(antiSMASH)加速发现微生物组中新型天然产物提供了良好的示范。
目前,有相当多的基于微生物组BGC挖掘和转化的研究项目已经或正在开展[24,31-44]。例如,针对海洋微生物群落进行挖掘,发现了一类全新的海洋细菌(Candidatuseudoremicrobiaceae),并预测了近4万种潜在的生物合成基因簇[32]。又比如,针对肠道微生物群落的挖掘,发现了肠道菌群能产生大量不同结构和生物活性的次生代谢产物,与肠道菌分泌的抑菌肽小菌素类似,这些次生代谢产物在药物研发与临床上有很广泛的应用前景[41]。再比如,针对土壤微生物群落进行挖掘,通过对生长在抑病土壤中的甜菜幼苗根进行宏基因组测序分析,区分出哪些BGC在感染过程中表达增加,并通过位点定向诱变分析检验其重要程度,发现抑病土壤中的植物益生菌通过增强真菌细胞壁降解相关酶的活性,为植物提供额外保护[38]。此外,针对特定的微生物,BGC挖掘结果揭示了放线菌基因组具有巨大的天然产物合成潜力[36],其生产的抗生素在临床中应用前景光明。
3 BGC的分析和比对
BGC的分析和比对,主要是建立在BGC数据库基础之上。大多数BGC数据库提供网页端入口,提交目标序列之后,服务器会根据同源性比对或隐马尔可夫预测等方法展示出最为相似的现有数据,通过解读结果的注释信息即可辅助BGC的分析与预测(图2)。
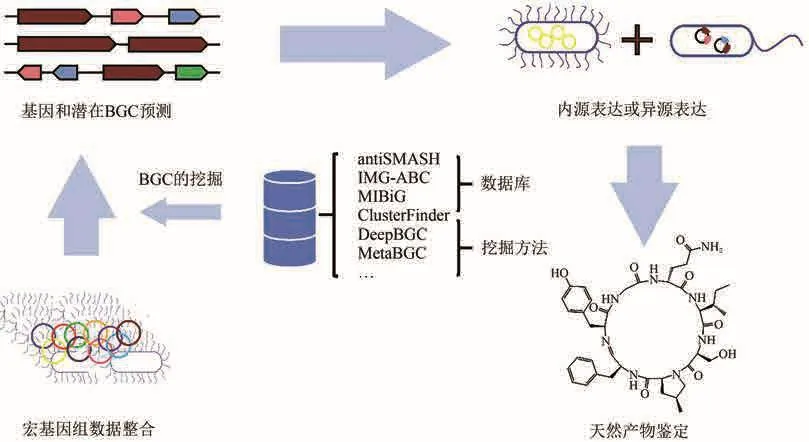
图2 BGC挖掘的整体过程(该过程包括:宏基因组数据的整合,基因和潜在BGC的预测,内源表达或异源表达、天然产物的鉴定等。本图中选用的案例是诺糖环肽A2,是从地衣Nostoc属ATCC53789中提取分离的天然产物,可作为20S蛋白酶体的抑制剂,具有抗癌活性[45])Fig.2 Overall process for BGC mining(This process includes the integration of metagenomic data, prediction of genes and potential BGC, endogenous or heterologous expression,identification of natural products, etc.The case chosen in this figure is Nostocyclopeptide A2, which is extracted from Nostoc sp.ATCC53789 isolated from lichen.It can be used as an inhibitor of 20S proteasome and exhibits anticancer activity[45].)
在BGC数据资源方面,当前服务于不同目的的BGC数据库都有较为广泛和频繁的访问和应用(表1)。
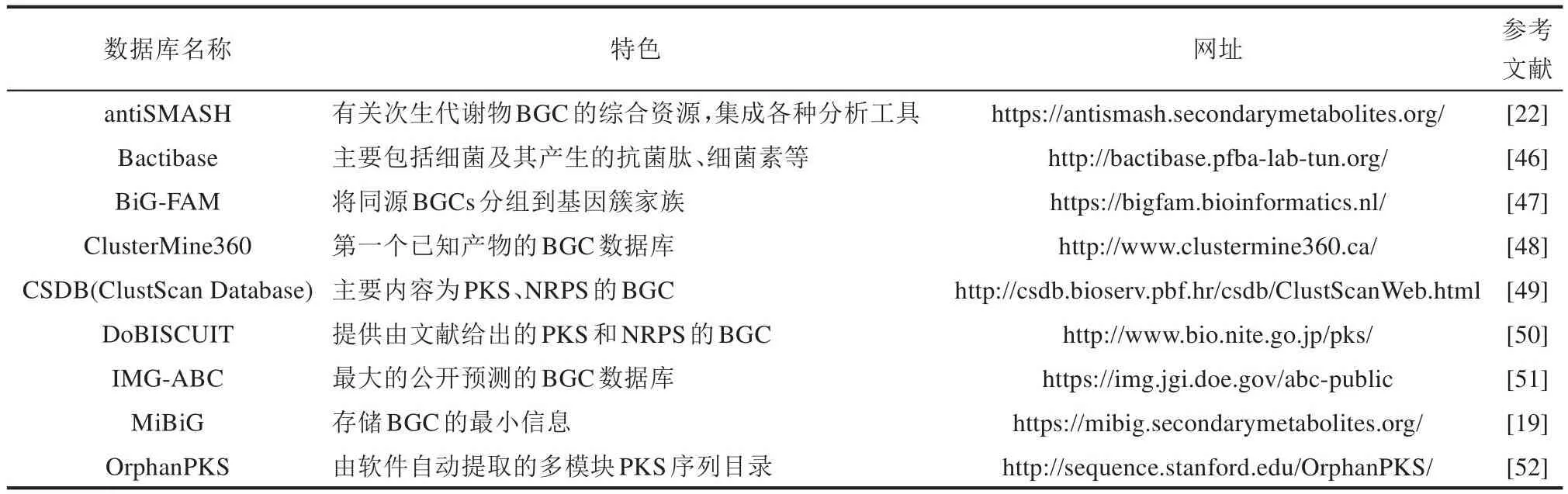
表1 代表性BGC数据库介绍Table 1 Summary for representative BGC databases
例如,BiG-FAM数据库[47]从公开来源获取了1 225 071个BGC,并使用BiG-SLiCE[53]软件将其聚类为29 955个基因簇家族模型。又例如,IMGABC数据库[51]包含了411 412个预测BGC,其中1332个BGC已得到实验验证,14 985个BGC是从高质量的宏基因组数据中预测得到(截止到2022年12月)。特定类型的BGC数据库如Bactibase[46],则覆盖了由206种革兰氏阳性菌和19种革兰氏阴性菌产生的230种抗菌肽或细菌素的BGC信息。
在BGC比对方法方面,主要包括序列比对和特征比对,多数BGC数据库通常都提供了这两种方法进行比对(图3)。
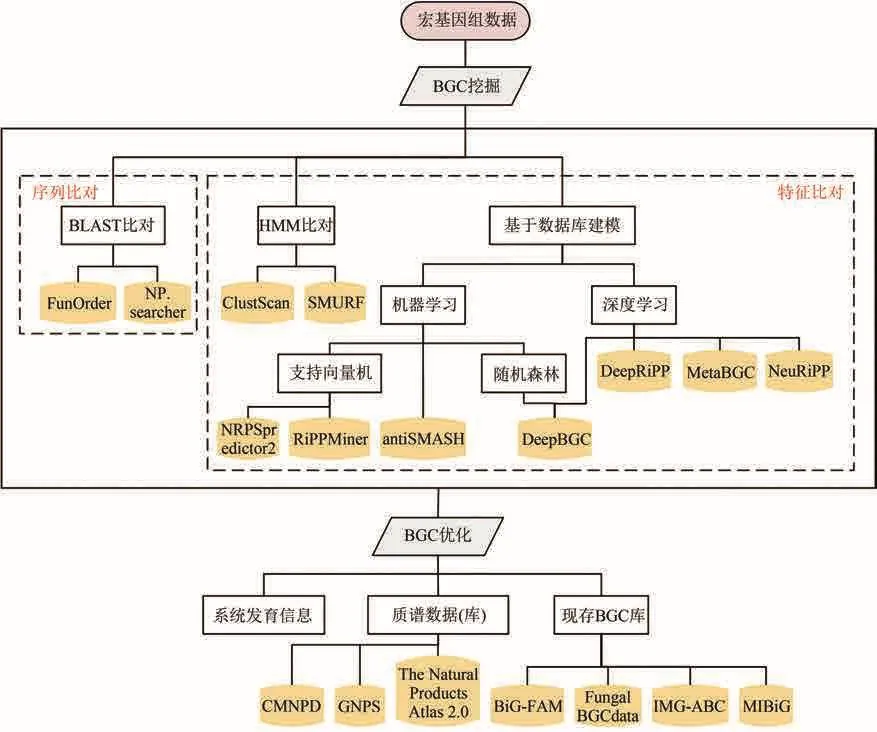
图3 BGC挖掘的一般分析流程及相关方法[从宏基因组数据中挖掘BGC,主要包括:BGC的挖掘方法(序列比对、特征比对等)和BGC的优化方法(数据库搜索、进化分析等)。其中BGC的挖掘方法主要有序列比对和特征比对两大类:序列比对主要是BLAST等方法,特征比对既包括隐马尔科夫模型(HMM)比对等传统方法,也包括基于数据模型的深度学习等方法。其中BGC的优化方法主要有数据库搜索、进化分析等:数据库搜索包括BGC序列数据库的搜索,以及BGC相关小分子质谱数据库的搜索,而进化分析的主要目标是分析BGC的演化和变异模式[54]]Fig.3 Overall flow for BGC analysis and mining[It mainly includes: BGC mining methods (sequence alignment, feature characterization, etc.) and BGC optimization methods (database searching,evolutionary analysis, etc.).Among them, the mining methods of BGC mainly include sequence alignment and feature characterization.Sequence alignment mainly uses BLAST and other methods, while feature characterization employs both traditional methods such as hidden Markov model(HMM) alignment and deep learning based on data model.The optimization methods of BGC mainly include database searching, evolutionary analysis, etc.Database searching includes the searching of BGC sequence database and BGC related small molecule mass spectrometry database, and the main purpose of evolutionary analysis is to analyze the evolution and variation patterns of BGC[54].]
例如,antiSMASH数据库[55]中提供基于BLAST的ClusterBlast工具,能将目的基因簇与数据库中的其他基因簇进行序列比对,展示相似性得分最高的多个结果,辅助判断BGC的功能与进化上的联系。antiSMASH数据库还提供了HMMer3工具[56],可以由基于群落画像(community profile)的隐马尔可夫模型(profile hidden Markov model, pHMM)[57]刻画特征,与目的序列进行特征比对,检测目的序列中多个特定蛋白质结构域存在的可能性,从而判断出BGC。
次生代谢产物是BGC合成的主要产物,因此构建序列比对和特征比对方法,将次生代谢产物与其对应BGC联系起来也是计算分析中非常重要的一部分内容(图4)。

图4 建立BGC和次生代谢产物关联性的分析方法[58](a)逆生物合成:从已知化合物开始,预测生产该化合物所需的活性酶(主干酶和裁剪酶),并从这些预测中找到与基因组中需求匹配的假定簇。本图中选用的案例为青霉素G[59]。(b)同源搜索:从物种1产生的已知化合物和物种2产生的相同或相似的化合物开始,使用来自物种2的已知基因集群在物种1的基因组中搜索相似的基因集群,从而确定感兴趣的基因集群。(c)比较基因组学:从一组生物开始,其中一些生物产生目标化合物,而另一些生物则不产生,有可能在生产中识别同源基因簇,并在非生产中没有同源基因的基础上进行筛选,从而识别候选基因簇Fig.4 Analytical methods for establishing correlation between BGC and the production of secondary metabolites[58](a) Retro-biosynthesis: starting with a known compound but no related gene clusters identified, it is possible for predicting enzyme(s) to catalyze the synthesis of such a compound (backbone and tailoring enzymes), and with these predictions putative gene clusters matching the requirements can be found in the genome.The selected case in this figure is penicillin G[59].(b) Homology searching: starting with a known compound produced by organism 1 and the same or similar compound produced by organism 2 with gene cluster identified, it is possible to use the known gene cluster from organism 2 to search for a similar gene cluster in the genome of organism 1, and thereby identify the gene cluster of interest.(c) Comparative genomics: starting with a group of organisms, some of which produce compounds of interest and some of which do not, it is possible to identify homologous gene clusters in the species that produce them and to screen on the basis of the absence of homologous genes in the species that does not produce them, thereby identifying candidate gene clusters.
当在某个物种中发现了未知的次生代谢产物时,可以先找到与其结构相似且基因簇已被确定的化合物,再根据已知的基因簇通过构建序列比对或特征比对等同源搜索的方式,确定出产生该未知次生代谢产物的候选基因簇。而从BGC确定其次生代谢产物的验证过程,则要利用如异源表达、激活沉默基因等基因工程的手段合成一系列次生代谢产物,其验证方法本文暂不拓展。
4 BGC挖掘的人工智能方法
BGC本质上是基因组编码的遗传信息集合,主要是通过序列数据的分析方法进行分析。因此序列分析的人工智能方法,在很大程度上涵盖了挖掘BGC的人工智能方法,其中成熟的方法对BGC的人工智能挖掘具有较高的借鉴与参考价值。
4.1 序列分析的人工智能方法
随着生物大数据规模的不断提高,针对生物大数据分析的人工智能(artificial intelligence,AI)方法层出不穷[60]。目前,AI技术在生物医药领域应用主要包括药物研发、医学影像、辅助诊疗和基因分析四个子领域。其中,国外借助先进的药品研发技术和人工智能技术起步更早,以AI药物研发为主[61];我国则借助海量大数据的优势,以AI医学影像为主[62]。大数据可以减少临床研究中的试错成本、大大加快临床实验的成功,也可以集成患者的信息,生成无数生物数据模型,帮助人类理解生命奥秘,实现疾病的精准判断与精准治疗。人工智能可能用人类无法实现的方式整合或解开复杂的基因组数据或是帮助研究者寻找纷繁复杂实验数据中的规律、理解疾病在组学层面的时空动态模式,将为新药研发、临床研究、治疗模式等各方面带来翻天覆地的变革[63]。
序列分析的人工智能方法[64],是人工智能在生物序列分析特定场景下的方法,包括PICS[65]、DeepCell[66]等图像识别方法,Enformer[67]、DeepLinc[68]等基因表达分析方法,以及AlphaFold2[69]等结构功能预测方法。基因二代测序技术产生了大量的测序数据,AI在基因大数据的分析上亦表现出良好的和不断扩展的应用趋势(图5),即在分子层面的基因组学、转录组学、蛋白质组学、代谢组学等层面,预测各种变异和调控规律;在宏观层面的细胞和表型组学层面,通过图像识别等方法进行各类样本分类[70]。随着计算机性能的不断提升,超级计算机强大的数据处理能力可以对TB级的海量基因组数据进行处理和挖掘,从而极大地缩短基因检测的时间,提高基因检测效率。将人工智能方法应用于海量的基因组数据,可以带来传统医疗向精准医疗的范式转变,人工智能方法能使医生和研究人员更准确地预测出预防与治疗方法在哪些人群中更起作用[71]。
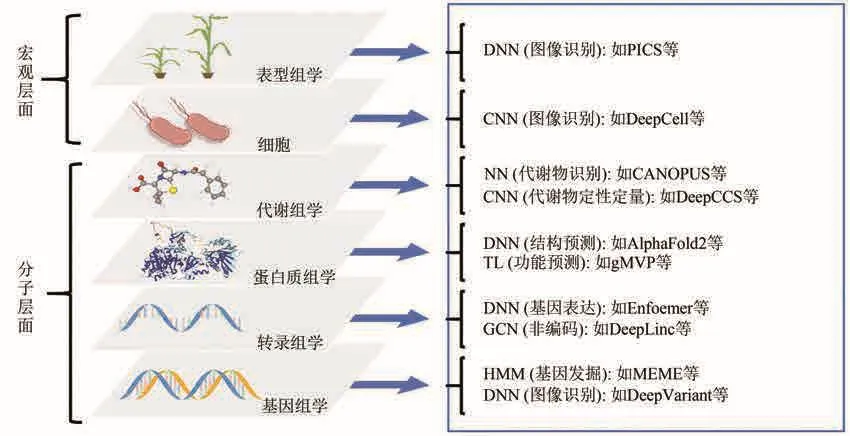
图5 序列数据的类型,以及相应的人工智能分析方法DNN—深度神经网络;CNN—卷积神经网络;NN—神经网络;TL—迁移学习;GCN—图卷及网络;HMM—隐马尔科夫模型Fig.5 Types of sequence data and corresponding AI analysis methods DNN— deep neural network; CNN— convolutional neural network; NN— neural network; TL— transfer learning;GCN— graph convolutional network; HMM— hidden markov model
4.2 BGC挖掘的人工智能方法:经典方法和发展趋势
伴随着生物序列人工智能分析方法能力的不断提高,BGC挖掘的方法也在不断更新换代。其中antiSMASH[22]、ClusterFinder[72]、MetaBGC[24]、DeepBGC[25]是成功应用于各领域的经典人工智能数据挖掘方法(图6)。
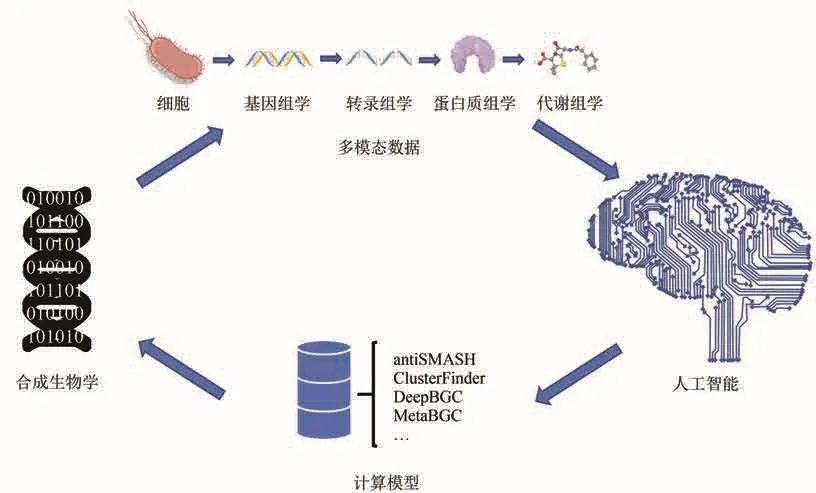
图6 利用人工智能进行BGC挖掘的现状和趋势(从数据出发,通过人工智能方法进行数据挖掘和模型构建,进而服务于合成生物学的转化研究,产生更多的多模态数据,形成良性循环)Fig.6 Status quo and trend of BGC mining using artificial intelligence(Starting from the data, data mining and model construction are carried out with artificial intelligence methods, thus serving the transformation research of synthetic biology, generating more multimodal data and forming a virtuous cycle.)
(1)antiSMASH工具集[22]antiSMASH在数据库基础上提供了一系列基于人工智能的计算工具,是目前寻找代谢基因簇最常用的软件之一。其主体功能主要基于的原理是:参与代谢途径中生物合成酶的基因在染色体上一般成簇排列,基于指定类型的模型,可以准确鉴定所有已知的次级代谢基因簇。在antiSMASH中,将次级代谢基因簇分为了数十类,然后通过序列比对等方法进行BGC的同源比对和发掘[73]。通过分析与目的基因相似的BGC结果,可以大致解读出目的基因的功能[74]。除此之外还提供了一些独立的工具,如由质谱引导的肽挖掘工具Pep2Path[75]、抗生素耐药性靶标搜寻器ARTS[76]和sgRNA设计工具CRISPy-web[77]等。
(2)ClusterFinder[72]ClusterFinder基于隐马尔可夫模型(hidden Markov models, HMM),它将BGC的核苷酸序列转换为一串连续的Pfam结构域,因为仅基于Pfam域频率,ClusterFinder能更精准地识别新型BGC。且有别于在此之前的算法只能识别少数BGC类别,ClusterFinder基于手动汇总的732个BGC训练集可以检测数种特征明确的基因簇类别,提供基因簇识别问题更通用的解决方案。将该算法应用到人类相关的微生物组中,鉴定出3118个小分子BGC,在临床试验中发现一类硫肽抗生素的BGC,随后通过实验确定了硫肽抗生素lactocillin的结构,并证明其对革兰氏阳性阴道病原体具有一定的抗菌活性[44]。
(3)MetaBGC[24]MetaBGC方法是一种基于“读段”(reads)的算法,能够从人类微生物组中发掘之前从未被报道过的BGC。在不需要分离培养细菌或测序的情况下,该算法允许直接在人类微生物组衍生的宏基因组测序数据中识别BGC:通过构建基于群落画像的隐马尔可夫模型,可在单一的宏基因组读取水平上识别、定量和聚集微生物组衍生的BGC。研究人员使用MetaBGC的算法在口腔、肠道和皮肤这三个部位的宏基因组样本发现了多种新型酶的BGC,即Ⅱ型聚酮化合物合酶BGC,简称为TⅡ-PKS BGC[78-79],并运用合成生物学策略将两种BGC进行异源表达,纯化与确定了产物的结构,发现其具有抗菌活性,这一结果揭示了人类微生物组产生先导化合物的能力。
(4)DeepBGC[25]DeepBGC使用深度学习来检测细菌和真菌基因组中的BGC。DeepBGC使用了双向长期短期记忆递归神经网络[80]和类似word2vec[81]的Pfam蛋白域嵌入,并使用随机森林分类器[82]预测产品类别和检测到的BGC的活性。将DeepBGC应用到实际的细菌基因组中,能预测出具有编码抗生素活性分子的全新BGC候选物。
发掘全新的BGC个例和BGC类型是微生物组研究中比较重要的数据挖掘目标[83],然而现有的数据挖掘方法难以发掘新型BGC[84]。基于更大的BGC数据集构建更加智能的挖掘模型,有可能发掘新型BGC[53]。在BGC数据集方面,BiG-SLiCE方法[53]能将BGC投射到欧几里得空间,以便使用时间复杂度为近线性的分区聚类算法,有助于大型BGC数据集的分析。此外,Medema等[85]提出的基于网络的计算框架(biosynthetic gene similarity clustering and prospecting engine, BiG-SCAPE)可用于BGC的聚类,以便更好地分析大数据集上微生物群落的生物合成潜力。在BGC挖掘模型方面,基于自然语言处理(natural language processing,NLP)技术的深度学习方法Genomic-NLP已经被成功地用于解码未知微生物基因的功能[86]。在未来的研究中,开发基于NLP技术的人工智能模型有可能发掘出与现有数据库中已知的BGC不存在任何同源性,然而在代谢产物方面又有一定关系的新型BGC。
5 新型BGC的挖掘与功能验证案例
新型BGC的功能验证,通常是通过培养实验来完成的[84]。人工智能数据挖掘(artificial intelligence data mining)和培养组学(culturomics)各自都有明显的优缺点,并且它们之间具有极强的互补性[87](图7)。高通量测序方法能短时间内产生大量数据,再由人工智能方法迅速挖掘出有用信息;而来自于测序的数据挖掘方法,也需要由培养组学来补充未知细菌的生长条件等信息[88]。
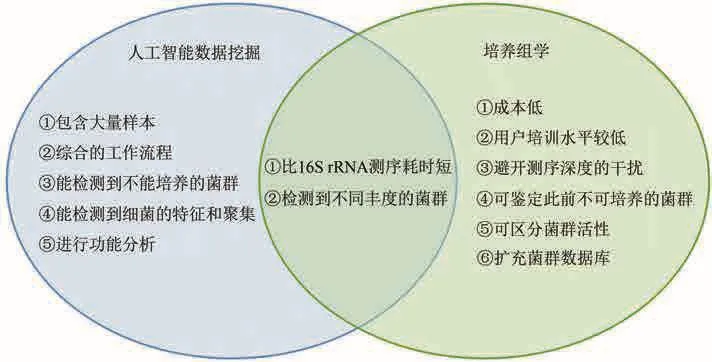
图7 人工智能数据挖掘和培养组学的各自优缺点和互补性(相关方法优缺点的罗列,是基于互相比较和与传统分子生物学方法比较的结果)Fig.7 Advantages, disadvantages and complementarities of artificial intelligence data mining and culturomics(The list of advantages and disadvantages of the relevant methods is based on the results of comparison with each other and with traditional molecular biological methods as well.)
新型BGC转化的应用范围很广,在临床、环境和生物制造方面均有非常迫切的需求[43]。目前有害生物对抗生素、癌症化疗药物和杀虫剂的耐药性上升,这一现象是现代医学与农业的主要威胁,而微生物次级代谢产物是解决这一问题的主要有效方法之一[89],即通过发掘新型BGC合成新型次级代谢产物,从而开发出新型产品消除或减缓有害生物对人类及农作物的危害。
5.1 肠道微生物BGC的挖掘和分析研究
2019年,一项人类肠道微生物宏基因组挖掘工作揭示了未培养的细菌基因组编码数百种新的生物合成基因簇,并具有独特的功能[90]。课题组通过从11 850个人类肠道微生物群中重建92 143个宏基因组组装基因组(metagenome assembled genome,MAG),鉴定了1952个未培养的候选细菌物种。这些未经培养的细菌物种及其基因组大大扩展了人类肠道微生物群的已知物种库,将目前的系统发育多样性增加了281%。这些候选物种编码数百个新的生物合成基因簇,并在铁-硫和离子结合等代谢方面具有独特的功能,揭示了未培养肠道细菌的多样性,为肠道微生物群的分类和功能特征提供了前所未有的解决方案[91]。
5.2 土壤环境微生物BGC的挖掘和分析研究
2018年,研究人员基于草原土壤的宏基因组数据,重建了上千个基因组,其中几百个近乎完整(near-complete),并鉴定出先前未被研究过的微生物(一类酸杆菌),这些微生物能编码多种聚酮化合物和非核糖体肽合成的基因组簇[92]。具体而言,研究者鉴定出了两个来自不同谱系类群的酸杆菌(Acidobacteria)基因组,每个基因组都拥有一个异常庞大的生物合成基因库,并且含有多达15个大型聚酮化合物和非核糖体肽生物合成基因位点。为了追踪土壤中聚酮化合物合成酶和非核糖体肽合成酶基因的表达,研究者设计了一个微观操作实验(microcosm manipulation experiment),采集了120个时间点的样品,使用转录组学的手段,发现基因簇对不同环境扰动的响应情况并不相同。通过对微生物的转录共表达网络分析,发现生物合成基因的表达与双组分系统、转录激活、假定抗微生物剂抗性和铁调节模块的基因相关,这一结果将代谢物生物合成与环境感知和生态竞争过程联系起来。作者因此判断,土壤微生物的生物合成潜力以前被大大低估了,而这些微生物代表了一种天然产物来源,能够进行转化研究以满足人们对新型抗生素和其他先导化合物的需求。据文献报道,上述聚酮化合物和非核糖体肽生物合成基因簇来自于Acidobacteria、Verrucomicobia和Gemmatimonadetes以及候选门Rokubacteria的微生物。这些微生物类群在土壤中非常丰富,但过往研究并没有把次生代谢产物与基因组信息联系起来[93-95]。
5.3 水体环境微生物BGC的挖掘和实验验证
2022年,瑞士苏黎世联邦理工学院的研究团队借助基因组学技术和大数据挖掘方法,发现了多种海洋细菌生物合成基因簇,相关成果在Nature发表[32]。研究团队首先获取了全球215个采样点不同深度层共1038个海水样本的基因测序数据,构建了26 293种海洋微生物基因组,其中2790种来自新发现的细菌。结合已公布的基因组数据,研究人员创建了海洋微生物组学数据库(ocean microbiomics database, OMD),发现了39 055个生物合成基因簇,参与约6873种化合物的生物合成过程。进一步实验验证两类与任何已知BGC不相似的RiPP生物合成簇能产生新的代谢物,表明了部分基因簇在亚磷酸盐等化合物的生物合成中起着关键作用。该研究通过基因组学方法发现了新型海洋细菌和生物合成基因簇,并对部分BGC进行了实验验证,其研究成果对海洋生态、生物进化和天然产物等领域的研究具有重要意义[96]。
5.4 重要天然产物的发掘和再利用
硒(Se)是一种天然的非金属元素,主要存在于硒蛋白和硒酸生物聚合物中。由于硒具有营养学和毒理学的双重作用,因此在医学和生物学领域广受关注[97]。2022年,发表在Nature上的一项新研究确定了第一条将硒引入微生物的小分子生物合成途径[98]。首先,由于SelD基因编码了细胞内所有已知硒代谢过程的第一步,因此科研人员利用无假设的方式从美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库中搜索了SelD的遗传背景,具体而言即通过量化了一个或多个碱基对与selD开放阅读框重叠的基因丰度,识别与其共定位的基因。结果表明,前5个selD的重叠基因包括SelA、SelU、yedF和duf3343,后两个基因被认为在硒的还原和/或转运中发挥尚未确定的作用。其次,对SelD-tigr04348遗传背景的深入研究揭示了第三种常见的共定位基因egtB的同源物,其编码麦角硫因生物合成中C—S键形成酶[99]。之后,科研人员通过代谢组学和生化方法表征上述生物合成途径,发现含有SelD-egtB-tigr04348基因簇的放线菌Amycolatopsis palatopharyngis和争论贪噬菌Variovorax paradoxus可以产生麦角硫因及其硒酮类似物。进一步分析揭示硒酮实际上是新基因簇的产物。该团队将其命名为“Sen”,SenA、SenB和SenC分别编码egtB同源物、一个假定的糖基转移酶和一个SelD同源物。这些发现证明SenB是一类新的硒糖合酶,继SelA和SelU之后成为迄今为止第三种C—Se键形成酶。这标志着硒元素首次在天然产物中被发现,并为硒生物学研究开辟了更广阔的前景。
5.5 天然药物资源的发掘和再利用
2022年,Nature Catalysis发表了丝状真菌来源萜类生物合成基因簇的高效挖掘研究工作[30]。该研究基于antiSMASH开发了“基因簇功能元件理性可控重组”策略,实现了萜类沉默基因簇的批量挖掘及高效合成,有效解决了困扰该研究领域的“三低”(研究通量低、产物集中度低、产量低)研究瓶颈[100],显著提高了活性新产物的合成效率。该研究借助自动化平台实现了丝状真菌来源萜类基因簇及其产物的高通量挖掘,并开发了真菌高效萜类前体供给底盘,实现了产物的高效合成。在丝状真菌米曲霉(Aspergillus oryzae)底盘中,通过模块化组合重构了5种真菌来源的39个Ⅰ型萜类生物合成基因簇,随后借助抗炎活性高通量筛选模型快速锁定高活性产物及其对应的突变株,紧接着回溯突变株对应的基因簇,解析了具有显著抗炎活性的二倍半萜化合物mangicol类(酯萜多元醇)[101]的生物合成机理。
以上应用案例表明:针对微生物组的BGC挖掘和解读,能够极大地提高天然产物的发掘效率,促进生物工程与合成生物学的发展,并在多领域取得明显的成效。
6 结论和展望
本文通过对BGC相关微生物组大数据以及相关数据挖掘方法的介绍,配合详实的案例,描绘了BGC挖掘和转化研究方面的全景图。首先,较全面地回顾了BGC挖掘的意义和瓶颈问题,指出当前实验验证的BGC数据不够全面,而基于序列比对的BGC挖掘难以发现新类型的BGC资源。其次,系统性地总结了当前BGC发掘中的数据资源和挖掘方法,尤其是人工智能方法,指出了其巨大的潜力。同时,通过回顾当前培养组学和合成生物学方面的技术进展,指出了干湿结合方法对于验证新发掘的BGC的重要价值。最后,通过展示到表型的新发掘BGC案例,指出BGC挖掘被应用于不同的研究领域,且取得了较好的研究成果。
6.1 BGC挖掘的研究是合成生物学与人工智能交叉研究方向上非常重要的一环
BGC挖掘的研究,是合成生物学与人工智能交叉研究方向上非常重要的一个环节,其重要性体现在方法上代表着人工智能生物数据挖掘的趋势,在转化应用上也具有非常高的价值。
首先,BGC挖掘的研究,是合成生物学中重要的一个部分。合成生物学(synthetic biology)是一门汇集生物学、基因组学、工程学和信息学等多种学科的交叉学科,其实现的技术路径是运用系统生物学和工程学原理,以基因组和生化分子合成为基础,综合生物化学、生物物理和生物信息等技术,旨在设计、改造、重建生物分子、生物元件和生物分化过程,以构建具有生命活性的生物系统[102]。将新型BGC作为原件,对已有的底盘生物进行理性设计,是合成生物学的典型应用场景[103],而利用生物信息学分析和计算工具,能挖掘大量未知的BGC,再将这些BGC通过上述合成生物学手段进行验证,即完成BGC的挖掘研究,这一流程将极大地加快天然产物的开发与利用。
其次,BGC挖掘的研究,在方法上代表着较为高级的生物大数据挖掘趋势[84]:BGC在序列和功能上的特征决定了针对其挖掘的人工智能手段必须比传统的单个基因挖掘方法要复杂,这种需要上下文感知的人工智能挖掘手段,是生物大数据挖掘趋势[86]。人工智能与合成生物学的结合,可以实现更为智能化、数字化、工程化的合理设计和优化,这也是BGC挖掘研究的重点和难点[84]。另外需要指出的是:人工智能与合成生物学的结合,干湿实验的结合,都指向更为高通量的“发掘-验证”流程,而高通量的“发掘-验证”流程,能够更为快速地发掘潜在新类型的BGC并加以验证,具有明显的工程属性,同时也能够极大地提高BGC发掘、验证和转化的效率。
最后,BGC挖掘的研究,在转化应用上具有非常高的价值[9]:通过人工智能挖掘的元件和模块,可以直接结合合成生物学的研究进行验证[104],并快速进行转化应用,尤其是在精准医学等转化领域日益精进的今天,通过有效开展BGC及其相关化合物的转化研究,快速有效地实现从实验室到临床(from bench to bedside)的转化,具有非常高的经济价值和社会价值[105]。
6.2 BGC挖掘的研究需要重视干湿实验等方面全方位结合
此外,BGC挖掘研究的成功,十分依赖于BGC数据库和相关基因实体库相结合,依赖于人工智能挖掘和培养实验验证相结合。只有在干湿实验等方面全方位结合,才能更有效地实现BGC挖掘、验证以及转化等方面的研究。
BGC数据库和相关基因实体库相结合,能够更好地推动BGC挖掘研究和转化应用的开展[6],是保证BGC挖掘研究和转化应用顺利开展的基本材料和数据条件[106]。基于数据库的不断更新,配合相关序列和结构等规律的发掘,为更全面的BGC发掘打下了数据基础。同时实体库能较为便捷地进行BGC验证实验,也为发掘新型BGC提供了保障。因此,作为数据基础的数据库和实体库相结合,能够更好地推动BGC挖掘研究和转化应用的开展。
人工智能挖掘和培养实验验证相结合,是保证BGC挖掘研究和转化应用顺利开展的基本技术条件[25]。传统的基于序列比对的BGC挖掘难以发现新类型的BGC资源,而利用人工智能技术,基于已有的BGC及其同源序列集合进行大数据建模,将有望批量发掘新型BGC。另一方面,培养组学等实验技术,将能够快速有效地验证新发掘BGC的有效性。因此,人工智能挖掘和培养实验验证技术作为关键引擎,是保证BGC挖掘研究和转化应用顺利开展的基本技术条件。
由上述讨论可知,BGC在系统生物学与合成生物学中具有核心地位(图8):不但在数据上打通了数据库和实体库,而且在技术上打通了人工智能挖掘和培养实验验证。因此BGC的研究能够紧密连接系统生物学与合成生物学,实现从数据到模型,从验证到应用的无缝转化。
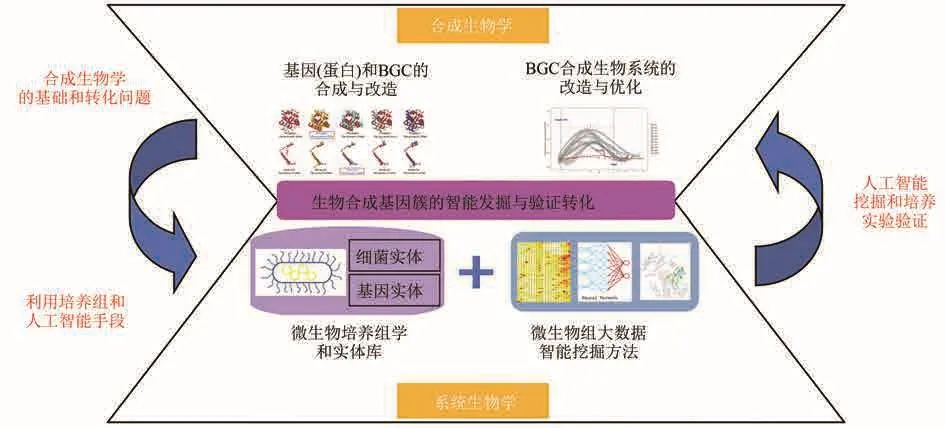
图8 BGC在系统生物学与合成生物学中的核心地位(生物合成基因簇的智能发掘与验证转化的研究,不但在数据上打通了数据库和实体库,而且在技术上打通了人工智能挖掘和培养实验验证。生物合成基因簇的智能发掘与验证转化的研究,能够紧密连接系统生物学与合成生物学,实现从数据到模型、从验证到应用的无缝转化)Fig.8 BGC’s central role in systems biology and synthetic biology(Research on intelligent mining and verification transformation of biosynthetic gene clusters not only connects BGC database with entity database,but also connects artificial intelligence mining and culture experiment verification.Research on intelligent discovery and transformation verification for biosynthetic gene clusters can closely link systems biology and synthetic biology, and realize seamless transformation from data to model and from verification to application.)
系统生物学和合成生物学协同发展的趋势,尤其是作为在系统生物学与合成生物学中具有核心地位之一的BGC挖掘与转化研究快速发展的趋势,会更为突出地显示出来。而多组学技术和人工智能分析方法,将会极大地助力这一方向的快速进步。我们乐观地展望,在BGC被充分挖掘和认识之后,系统生物学与合成生物学的结合将会深刻地改变世界:从科学探索方面来说,新发掘的BGC能够快速地被研究并转化于实际应用,高效实现各类小分子化合物从“实验到临床”(from bench to bedside);从健康和环境领域等方面来说,从需求端倒推BGC资源的特征,能够快速地实现转化研究领域中需要的特定功能小分子合成系统的“即插即用”(plug-and-play)。从而在技术上较为高效、准确、完整、安全地实现针对BGC合成生物系统从理解到创造(from understanding to creation)的过程。

