基于网络药理学和分子对接探析槲皮素对抑郁的作用机制
杨翼驰,刘 芳,聂家奇,冯倩倩,李小松,王素青
(1.武汉大学 公共卫生学院,武汉 430000;2.武汉大学 护理学院,武汉 430000)
抑郁是一种情绪障碍,以心理愉悦感消失、情绪低落超过2个星期、明显躯体不适和功能缺陷为特征,临床表现为失眠、意志减退、食欲异常、精神颓废等,严重者有自杀倾向[1]。世界卫生组织(WHO)表示,截至2017年,全球患有抑郁症的人数约为3.22亿,而2005—2015这十年间,抑郁症的患病率提升了18.4%[2]。目前,临床上大部分抗抑郁药治疗周期长,有严重的副反应,且费用高昂,给抑郁症患者及家庭造成了巨大的压力[3]。因此,研究分子机制及靶点,研制出安全且高效的药物对抑郁症的治疗极为重要。
槲皮素(3,3’,4’,5,7-五羟基黄酮)是一种普遍存在于人类膳食中的类黄酮物质[4]。广泛存在于桑树、山茶花、菖蒲、辣木、金丝桃、葱、苜蓿、刺槐、葡萄和银杏叶中[5]。槲皮素具有许多生物活性,例如抗氧化剂、抗炎、神经保护和脑保护等[6]。一项膳食类黄酮摄入量与认知功能的队列表明,较高的类黄酮膳食摄入量可能与成年个体更好的认知健康有关,并且在单个多酚中,只有槲皮素与认知健康相关[7]。 FANG Ke等[8]发现,槲皮素可以缓解大鼠腹膜内注射脂多糖(LPS)诱导的抑郁症。Samad等[9]也证明,槲皮素可以防止压力引起的焦虑和抑郁样行为,并改善雄性小鼠的记忆力。但截至目前,槲皮素改善抑郁的具体作用机制并未得到充分解释 。
网络药理学是一门从系统生物学和网络生物学中衍生而来的新学科[10]。建立 成分、靶点、疾病间的生物网络关系是其常用方法,以研究药物对机体的整体影响以及具体作用机制。本文拟采用网络药理学及分子对接技术,探讨槲皮素对抑郁的作用机制,为临床应用提供依据,以期开发出新的药物用于抑郁症的治疗。
1 资料与方法
1.1 药物靶点的收集
在Pubchem (https://pubchem.ncbi.nlm.nih.gov/)数据库中收集槲皮素(Quercetin)名称、结构等相关信息,下载其二维结构并以SDF格式储存。将该文件导入SwissTargetPrediction(http://www.swisstargetprediction.ch/)平台草图绘制器,蛋白质靶标种类选择“智人”,对靶点进行预测。SwissTargetPrediction平台可以鉴定具有与查询分子高度相似的已知配体的蛋白质,进而预测分子的靶标。进行预测时,3D 相似性值的阈值设置为 0.75,2D 相似性值的阈值设置为 0.45[11]。低于这些阈值,配体与查询分子的相似性非常低,不显示在结果中。接下来在Superpred(https://prediction.charite.de/)数据库中收集槲皮素的靶点。Superpred生物物种默认为“智人”,置信度为大于等于0.7,采用逻辑回归机器学习模型对靶点进行预测。将收集到的靶点置于同一Excel文档中,进行去重整合,校正为人源靶点。
1.2 疾病靶点收集
以“Depression”为关键词,分别在OMIM(https://omim.org)和Genecards(http://Genecards. org)两个数据库中获取抑郁疾病靶点信息。在OMIM中选择具有已知序列的基因进行搜索。在Genecards中,若靶点数目过多,则以大于等于“Relevance score”的2倍中位数为筛选条件。将靶点信息置于同一Excel文档中,整合抑郁相关靶点。
1.3 蛋白质互作网络(PPI)构建及核心靶点筛选
通过Jvenn(http://jvenn.toulouse.inra.fr/app/index.html)对槲皮素和抑郁靶点求交集,获得槲皮素-抑郁的共同靶点,以CSV文件形式导出。将交集靶点文件上传至STRING平台(https://cn.string-db.org/)进行PPI构建,蛋白质类型设置为智人,置信度设置为中等(0.400)。再将该网络数据导入到Cytoscape3.9.1应用软件 ,并使用CytoHubba插件中的MCC算法筛选出度值排行前10的基因为hub基因。CytoHubba插件是基于应用程序编程接口在Java中实现的。PPI网络中每个节点定义为v,度值定义为F(v)。MCC计算后的度值定义为MCC(v) = ∑C∈S(v)(|C| - 1)!,其中S(v)是包含v的最大集团的集合,(|C|-1)!是所有小于|C|的正整数的乘积[12]。
1.4 GO和KEGG富集分析
在David平台输入槲皮素-抑郁的共同靶点进行富集分析,物种选择“Homo sapiens”,最小基因数目为2,评分阈值<0.1。利用Excel软件计算并筛选富集分析中校正后P<0.05的项,并对GO 生物过程、GO 分子功能以及 GO 细胞成分分析的前15项进行可视化展示。KEGG 通路按校正后P值大小排序,利用R软件,以图片形式对前15条通路进行展示。
1.5 分子对接
在RCSB PDB(https://www.rcsb.org)下载度值前五的蛋白分子对应的复合体晶体结构,删除配体部分,去除水分子,利用Discovery Studio2019中Macromolecules/Prepare Protein/Clean Protein功能对蛋白分子进行预处理。接着利用LibDock依次进行分子对接,并需要LibDockScore以确定靶点蛋白与槲皮素之间的结合力,得分较高的组分被视为与槲皮素相互作用的活性组分。
2 结果
2.1 药物及疾病靶点的收集
在SwissTargetPrediction数据库收集到100个槲皮素靶点,在Superpred数据库收集到111个靶点。取并集后共获得槲皮素人源靶点198个。在Genecards平台共查询到3 675个抑郁靶点,在OMIM平台共查询到188个靶点,去重合并后共获得疾病相关靶点3 718个。
2.2 PPI网络以及核心靶点分析
Jvenn中共获得103个槲皮素-抑郁共同靶点,用韦恩图表示(见图1)。利用STRING平台绘制槲皮素和抑郁交集靶点的PPI网络图,包括103个节点,536条边。将其结果信息上传至Cytoscape3.9.1软件进行简化(见图2)。经MCC算法计算后,度值排名前10的hub基因依次为:SRC、MTOR、EGFR、AKT1、PTK2、CXCR4、KDR、IGF-1R、PIK3R1、PTPN11。依据MCC分数对hub基因进行可视化(见图3),颜色由红转黄,MCC得分越高,其基因颜色越趋近于红色。

图1 槲皮素靶点与抑郁靶点的Venny图Fig.1 Venny diagram of quercetin targets and depression targets

图2 槲皮素和抑郁共同靶点的PPI网络图Fig.2 PPI network diagram of common targets of quercetin and depression
2.3 GO与KEGG通路分析
应用David平台进行GO、KEGG富集分析。GO是基因本体(Gene ontology)的简称,用于注释与其相关的生物功能、通路、以及其他的基因产品。经校正后P<0.05 的富集条目筛选,获得了162条GO富集分析结果,其中包含88项生物过程条目,40项细胞成分条目,34项分子功能条目。图4展示了各部分校正后P值最高的15条通路。各个部分颜色不同。横向坐标代表校正后的P值,校正后P值越高,说明基因在通路富集程度越大。其中,最具有代表性的是生物进程条目,涉及凋亡过程的负向调节(Negative regulation of apoptotic process)、信号转导(Signal transduction)和蛋白质磷酸化(Protein phosphorylation)等生物学过程。

图3 排名前10的hub基因Fig.3 Top 10 hub genes

图4 槲皮素作用靶点的GO富集分析Fig4 GO enrichment analysis of quercetin targets
经校正P<0.05筛选后,共得到107条KEGG信号通路结果。校正后P值排名前15的通路结果如图5所示,主要有:缺氧诱导因子-1信号通路(HIF-1 signaling pathway)、ErbB信号通路(ErbB signaling pathway)、磷脂酶D信号通路(Phospholipase D signaling pathway)、神经营养素信号通路(Neurotrophin signaling pathway)等。横轴表示Gene ratio ,表示 KEGG通路中所含的基因数目占所有基因的百分比;纵坐标表示路径名称;气泡大小代表着富集的基因数目;纵轴展示通路名称;气泡尺寸表示富集基因的数量,其大小与富集基因数呈整正相关;以不同色彩代表P值,气泡颜色随P值依次渐变。其中,ErbB信号通路高度值hub基因富集程度最高,可能是最重要的信号通路(见图6)。
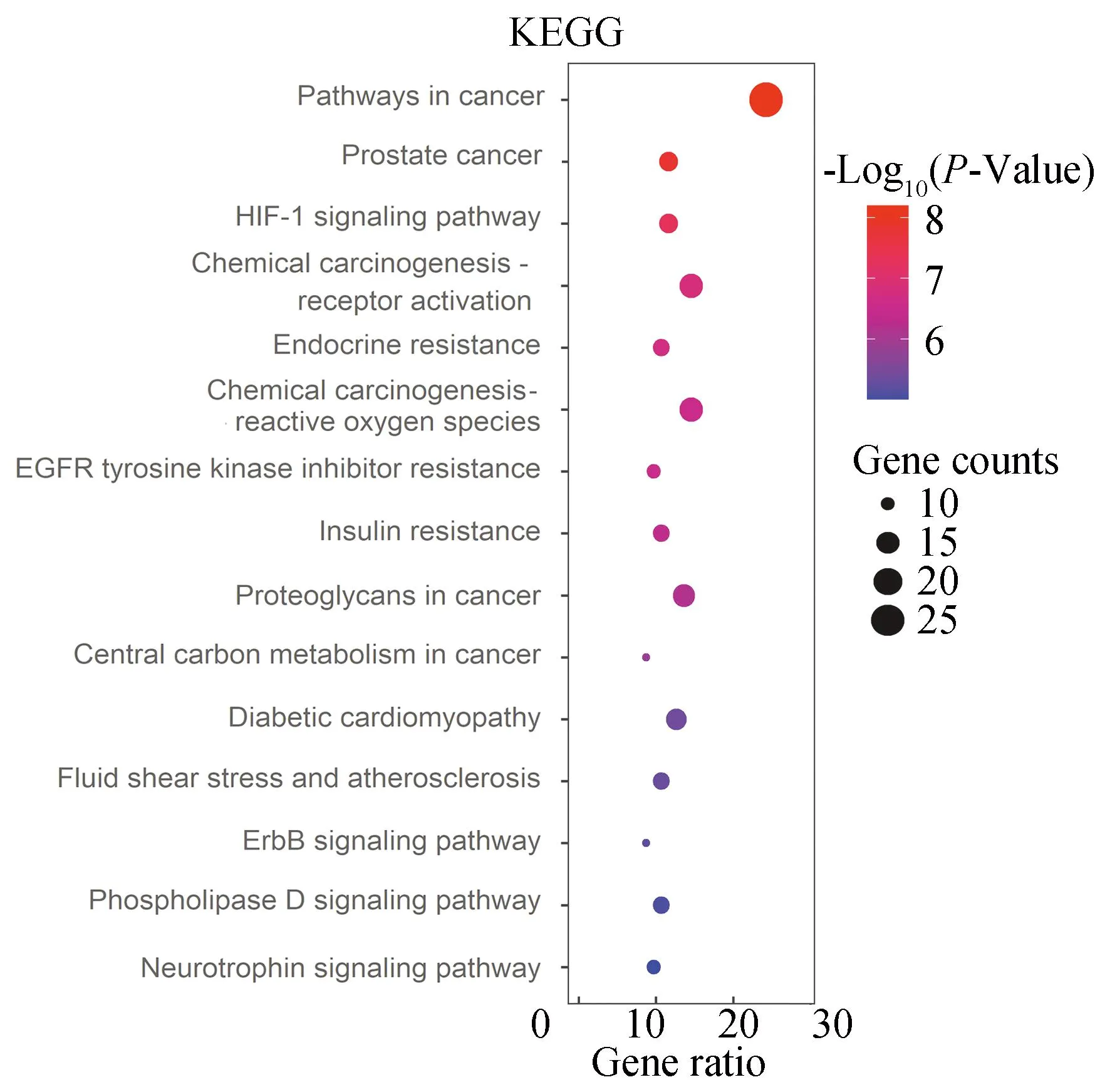
图5 槲皮素作用靶点的KEGG 通路富集分析Fig 5 KEGG pathway enrichment analysis of quercetintargets Molecular docking results
2.4 分子对接结果
分子对接结果显示(见图7),槲皮素与SRC 、MTOR、EGFR、AKT1、PTK2 分子对接LibDockSccore值依次为 95.718 8、97.583 3、77.993 5、114.16、93.314 5,对接评分均较高,说明槲皮素与这些靶标具有良好的结合能力。
3 讨 论
本研究的主要目的是探析槲皮素治疗抑郁的潜在机制。网络药理学结果表明,SRC、AKT1和mTOR可能是槲皮素改善抑郁症的关键作用靶点。以往研究表明,LPS注射可导致小鼠神经炎症和抑郁样行为。LPS注射组小鼠脑组织中SRC-1表达降低,提示SRC-1在炎症和抑郁中的作用[13]。 pCREB影响海马突触可塑性和空间记忆功能,它的表达减少与阿尔茨海默症的学习和记忆受损有关。体外实验中,SRC-1敲低显著降低了pCREB表达水平[14]。神经炎症可以诱导神经元再生,使神经营养因子释放减少,导致神经活动失调,并有助于抑郁症的发展[15]。有研究证实,PI3K / Akt通路的激活诱导NF-κB的磷酸化,这导致大脑区域的神经炎症,并有助于啮齿动物抑郁样行为的发展[16]。PI3K / Akt / NF-κB通路也能在新生儿母体剥夺和慢性轻度应激的抑郁模型中被激活[17]。mTOR作为一种丝氨酸/苏氨酸激酶[18],可通过p70S6激酶的磷酸化和哺乳动物eIF4E结合蛋白(4EBP)的抑制来调节神经发生、树突状脊柱生长和蛋白质翻译启动,从而合成神经元突触所需的蛋白质[18-19]。这与目前抗抑郁药的作用机制一致,即通过促使神经突触再生,从而达到抗抑郁的目的。这些发现与我们的结果一致。因此我们推测槲皮素可能通过作用于SRC、AKT1和mTOR产生抗抑郁效应。

图6 ErbB信号通路图Fig 6 ErbB signaling pathway map

图7 分子对接结果Fig 7 Molecular docking results
KEGG通路富集分析结果显示,槲皮素治疗抑郁可能与 HIF-1信号通路、ErbB信号通路等多条通路密切相关。HIF-1是一种由α、β两种亚基构成的转录因子,能对缺氧作出反应。已有研究表明,HIF-1通路激活的显着增加导致脑源性神经营养因子(BDNF)的持续表达[20]。BDNF广泛存在于中枢神经系统,在海马、皮质中大量表达,并参与神经元功能的维持、发育以及神经元受损后的修复,从而产生抗抑郁药样效应[21]。以上结果提示激活HIF-1信号传导可在槲皮素治疗抑郁中发挥重要作用。神经调节素-1(NRG1)是精神分裂症,双相情感障碍等多种精神疾病的易感基因,对皮质神经元、运动神经元、多巴胺能神经元、耳蜗感觉神经元和PC12细胞具有神经保护作用[22]。NRG1相关通路的异常改变与抑郁症的发生密切相关。Bi等人发现,阻断NRG1-ErbB4信号传导增强了焦虑样行为[23]。NRG/ErbB信号传导改变后,转基因小鼠出现异常行为以及学习记忆缺陷。因此,综合既往文献及本研究结果,推测槲皮素可能通过调控HIF-1、NRG/ErbB等多条信号通路来维持神经元功能和发育,从而发产生抗抑郁效应。
目前已有研究仅说明槲皮素可改善抑郁样行为,并未对其具体机制进行研究。本研究利用网络药理学方法,发现了槲皮素治疗抑郁的潜在靶点及通路,为设计和开发新的有效抗抑郁药物奠定基础。但本研究也存在一定局限性。首先,研究所使用的数据库数据覆盖范围有限,不包含未经证实和未记录的化合物或靶标。其次,分子对接本质上属于计算机预测技术,无法完全代替动物实验。因此,研究结果需在动物模型中进行进一步验证。
4 结 论
研究以网络药理学方法为指导,利用分子对接技术,对槲皮素-抑郁的关键靶点进行验证,发现槲皮素对抑郁的作用主要涉及SRC、AKT1、mTOR等靶点,HIF-1、NRG/ErbB等信号通路,提示槲皮素的抗抑郁机制可能是促进神经突触再生、改善脑肠轴功能以维持神经系统正常功能。