弧菌16S rRNA基因特异引物设计及海洋弧菌菌群结构分析
孙 颖,刘 泳,王华磊,陈吉祥,杨官品
(中国海洋大学海洋生命学院,山东青岛266003)
弧菌16S rRNA基因特异引物设计及海洋弧菌菌群结构分析
孙 颖,刘 泳,王华磊,陈吉祥,杨官品**
(中国海洋大学海洋生命学院,山东青岛266003)
弧菌是海水中最常见的细菌类群之一。弧菌属的一些种是致病菌,能引起海洋生物弧菌病,给养殖业造成损失。根据已有弧菌16S核糖体RNA基因(16S rDNA)序列,设计了3条弧菌属特异引物VF169、VR744和VR1150,与细菌16S rDNA通用引物VF27一起,组成VF169/VR744、VF169/VR1150和VF27/VR744 3引物对。从海水中直接提取浮游生物总DNA,用VF169/VR744,VF169/VR1150和VF169/VR744(巢式)以及VF27/VR744引物分别扩增弧菌16S rDNA片段并克隆,分别构建了弧菌16S rDNA片段变异类型文库。从各文库中分别随机选取一定数量的克隆测序,共获得72条序列。系统学分析表明所有72个序列都与已知的弧菌属细菌的序列聚类,具有很高的相似性,证明所设计的引物能用于海水样品弧菌菌群分析。另外,VF27/VR744既具有对海洋弧菌的高度特异性,也具有恰当的海洋弧菌覆盖面,是1对选择扩增海水中弧菌16S rDNA的较理想引物。从获得序列的分布情况看,胶州湾存在较高的弧菌多样性,其中,类灿烂弧菌菌群(Vibrio splendidus-related group)是优势类群。
弧菌;16S rRNA基因;弧菌属特异引物;弧菌菌群结构
弧菌是一类直的或略为弯曲的革兰氏阴性菌,极生单鞭毛或多鞭毛,可运动。弧菌属属于弧菌科, Bergey氏细菌系统学手册第二版(2005)中收录的弧菌已达63种。弧菌广泛分布于近岸海水、河口、海产品及海洋生物体表和肠道中。正常情况下,大多数弧菌属于海水、动物体表和肠道的正常菌群。但当外界环境恶化时,Vibrio anguillarum,V.cholera,V.parahaemolyticus,V.vulnif icus,V.harveyi,V.ordalii和V.damsela等可引起海水鱼类、甲壳类和贝类等的弧菌病。目前,弧菌病已成为鱼类、对虾等海水经济动物养殖业发展的主要病害之一,造成巨大经济损失[1-3]。英国健康保护署(Health Protection Agency)已公布V.parahaemolyticus,V.cholerae,V.alginolyticus, V.f urnissi,V.carchariae,V.metschnikovii,V. cincinnatiensis,V.mimicus,V.f luvialis和V. vulnif icus等10种弧菌可引起人类腹泻、菌血症以及严重外伤感染。在全球范围内,至今已有过7次霍乱大流行,导致上千万人死亡[4],其病原菌就是霍乱弧菌(V.cholerae)。另据报道,创伤弧菌V.vulnif icus可迅速致死,致死率高达60%[5-6]。因此,监测水产品生产、加工和消费链中的弧菌菌群动态变化对养殖安全和食品安全都具有极其重要的意义[7]。
传统的细菌研究都以分离的纯培养菌株为基础,结合一系列形态学、生理生化反应特征以及免疫学特性进行鉴定。但是,绝大多数微生物不能在现有人工培养条件下存活。人工培养基可能人为引入选择压。原核生物表型特征分析很大程度上依赖研究者经验。因此,基于纯培养的方法具有一定的局限性。自1977年Woese和Fox[8]用16S核糖体RNA基因序列分析细菌菌群结构以来,多种分子生物学技术被用于细菌研究,如分子杂交、PCR、SSCP、DGGE、TGGE、PFGE、RAPD、RFLP、PNA以及寡核苷酸芯片等。
PCR技术作为1种快速、高效、敏感的检测手段,已被用于检测V.vulnif icus[9]和V.cholerae[10]。这种检测手段的前提是将代表细菌的目标基因片段从复杂的环境或临床样品中特异性富集并根据不同菌种序列的特异性予以鉴别。到目前为止,已设计出多套不同菌群的特异引物,但尚未有弧菌属特异引物的发表。本研究旨在描述海洋环境中弧菌菌群结构,同时为弧菌高通量检测技术建立标记基因片段的富集方法。我们设计了1套弧菌特异的PCR引物,从海水样品DNA中扩增弧菌16S rDNA片段。经过数据库数据分析和实际海水样品应用结果比较,最终确定了1对弧菌属特异性和属内覆盖面都比较理想的引物,用于环境样品弧菌菌群结构分析。
1 材料与方法
1.1 实验材料
于2003年10月分别用无菌取样瓶取胶州湾各生态观测站位(35°38′N~36°18′N,120°04′E~120°23′E)[11]表层海水1 L并混合,当天(从取第一个站位的水样到实验室过滤完海水约需要15 h)带回实验室,进行水样过滤,收集浮游生物样品。DNA提取在当天进行或者将收集的样品-70℃保存备用。
1.2 实验方法
1.2.1 引物设计 设计弧菌属特异引物要兼顾特异性和覆盖面2个方面。海水中,细菌种类复杂多样, PCR体系比较容易被污染。同时,16S rDNA序列比较保守,细菌种间变异小。因此,设计特定细菌类群特异性高、覆盖面宽的引物是检测、鉴定特定细菌类群、分析其菌群结构、多样性及其与环境因子相关的基础。在Olsen等[12]原核生物系统树中,γ-变形菌纲是海洋中复杂度最高、丰度最大的细菌类群。绝大多数已分离鉴定的海洋细菌都属于这一类群,如交替单胞菌属(A lteromonas)、海洋螺菌属(Oceanospirillum)和海杆菌属(Marinobacter)等。肠杆菌科(Enterobacteriaceae)与弧菌科(Vibrionaceae)系统进化关系较近。弧菌科主要包括海水中大量存在的弧菌属(Vibrio)、发光杆菌属(Photobacterium)、格瑞蒙特菌属(Grimontia)、肠道弧菌属(Enterovibrio)和盐弧菌属(S alinivibrio)细菌。同时,巴斯德氏菌科(Pasteurellaceae)也是水环境中常见的菌群。
据此,从GenBank下载全部弧菌16S rDNA序列(截至2004年3月),在覆盖尽可能多的弧菌种和已知致病弧菌的前提下,选取了118个全长或近全长序列(不少于1300 bp);同时下载了Photobacterium, Pseudoalteromonas,Proteobacterium,S almonella, Escherichia,Yersinia等与弧菌属系统关系较近的序列,用DAMBE(version 1.19,http:∥aixl.uottawa. ca/)进行多序列对位分析,根据弧菌属序列的保守区筛选其不同于其它菌群的变异位点设计引物,用Primer 5分析引物特征,再用BLAST分析检验其特异性。
本文筛选到VF169(5’-GGA TAA CC/TA TTG GAA ACG ATG-3’),VR744(5’-CAT CTG A GT GTC AGT G/ATC TG-3’)和VR1150(5’-TCA CCG GCA GTC TCC CTG-3’)3条弧菌属特异引物,与细菌通用引物VF27(5’-A GA GTT TGA TCC/A TGG CTC A G-3’)一起,组成VF169/VR744、VF169/ VR1150和VF27/VR744 3引物对,用于分析海洋弧菌菌群结构。引物中V表示弧菌,F,正方向引物,R,反方向引物,引物后面的数字是各引物5’起点在大肠杆菌16S rRNA基因核苷酸序列上的位置。
1.2.2 DNA的提取 用0.22μm混合纤维素膜过滤混合海水样品约15 L,富集海洋浮游生物,根据Polen-Fuller[13]描述的方法抽提DNA,酚、氯仿抽提纯化。提取缓冲液组成为100 mmol/L Tris-HCl,p H= 8.0;1.4 mol/L NaCl;20 mmol/L EDTA,p H8.0; 2%(质量浓度)CTAB;0.1%(质量浓度)PVPP。
1.2.3 PCR扩增 用VF169/VR744,VF169/VR1150和VF169/VR744(巢式)以及VF27/VR744 3引物组合分别进行PCR扩增,以确定特异性最优和属内复杂性最大的组合。以VF169和VR1150为外侧引物对,进行巢式PCR第一轮扩增。反应体系体积为25μL,包含20~25 ng模板DNA,2 mmol/L Mg2+,dNTP各0.2 mmol/L,正反向引物各0.2μmol/L,1单位TaqDNA聚合酶和1倍缓冲液。循环条件为95℃预变性4 min,94℃变性1 min,55℃退火1 min,72℃延伸1 min 30个循环,最后在72℃延伸10 min。以VF169和VR744作为内侧引物对,将第一轮扩增产物稀释100倍,取1μL进行第二轮扩增。除以1μL第一轮产物代替DNA模板。将退火温度下调至53℃,反应体系及扩增循环条件与第一轮基本相同。用VF169/ VR744引物对以及VF27/VR744引物对的扩增条件与巢式PCR第二轮相同。
1.2.4 PCR扩增片段克隆、测序 PCR扩增片段经苯酚、氯仿纯化,连接到pMD18-T载体上,转化感受态E.coliJM109,随机挑选重组子进行测序。针对3种引物组合,分别选取29、33和10个克隆进行测序。这些序列在GenBank中的提取号为AY729028、AY729029、AY702244-AY702274、Y848846-AY848855。
1.2.5 数据处理 用Chromas软件(version 1.62,测序服务商提供)从测序峰谱图中提取序列并剪除引物区,用DAMBE软件包进行多序列对位分析(Multiple alignment)拼接序列,对所得序列进行BLAST分析,初步确定其分类归属。在此基础上,利用MEGA软件包(version 3.0)按照gamma分布模型,以Jukes-Cantor法计算序列相对遗传距离和序列相似性;以neighbor-joining法构建系统树,重复1000次计算bootstrap值。
2 结果
2.1 巢式PCR扩增结果
结果表明(见图1),测序得到的29个序列全部与已知的弧菌聚类。同时,与γ-变形菌纲的其它菌属相距较远。将这29个序列分别进行BLAST(数据未提供)分析,所有序列的最相似序列均为弧菌属已鉴定种。其中,21个序列与已知种的相似性在99%以上,7个序列与已知种相似性在98%以上,V TP25与V. mytili的相似性为93.8%。表明用3个特异引物进行巢式PCR,可以将弧菌属与其它菌群区分开,使弧菌得到选择性扩增。
2.2 VF169/VR744引物对的扩增结果
结果表明(见图2),用VF169/VR744进行PCR扩增,得到的33个序列全部与已知弧菌聚类,并与其他菌群明显分开。BLAST分析表明,33个序列的最相似序列均为弧菌属成员,除VDP57与Vibriosp._ strain Mel 13的相似性为95.2%,VDP59与Vibrio chagasii相似性为97.1%,VDP65与Vibrio f ortis相似性为95%外,其余30个序列与已知种的相似性均在98%以上,表明该引物对具有较好的弧菌属特异性。

图1 基于巢式PCR扩增16S rRNA基因测序所得序列及γ-变形菌纲中常见类群代表序列,以Neighbour-Joining法构建的系统树。以β-变形细菌为外组群,重复1 000次计算bootstrap值。其中,VTP表示本研究中经巢式PCR扩增得到的序列Fig.1 Phylogenetic tree based on partial sequences obtained by means of nested PCR,showing affiliation of 16S gene clones recovered from the surface water of Jiaozhou Bay.Sequence ofβProteobacteriawas used as outgroup.VTP series sequences indicated sequences obtained in this study.This tree was constructed by the N-J method and the bootstrap values were obtained with 1000 resamplings

图2 基于VF169/VR744引物对扩增16S rRNA基因测序所得序列及γ-变形菌纲中常见类群代表序列构建的系统树。以β-变形细菌为外组群,重复1 000次计算bootstrap值。其中,VDP表示本研究中VF169/VR744扩增得到的序列Fig.2 Phylogenetic tree based on approximately 576 nucleotide positions amplified with VF169/VR744 primer pair,showing affiliation of 16S gene clones recovered from the surface water of Jiaozhou Bay.Sequence ofβProteobacteriawas used as outgroup.VDP series sequences indicated sequences amplified with the primer pair of VF169/VR744.This tree was constructed by the N-J method and the bootstrap values were obtained with 1000 resamplings
2.3 VF27/VR744引物对扩增结果
如图3所示,以细菌通用引物VF27和文中设计的弧菌属特异引物VR744进行PCR扩增,测得的10个序列均与已知弧菌聚类,BLAST分析表明,其中9个序列的最相似序列为弧菌种类,相似性从97.2%到99.9%不等,只有V GP6与1株未鉴定的海洋细菌(AJ002567)最相似,但不能排除其为弧菌的可能性。因此,VR744对弧菌属有很高的特异性,同时,细菌通用引物VF27可保证该引物对能最大限度的覆盖弧菌属。可以确定,VF27/VR744引物对是3对引物组合中最佳的弧菌属特异引物。
2.4 胶州湾表层海水弧菌菌群结构初步分析
根据3个引物组合获得的序列,用MEGA软件包分别构建系统树,初步分析了各取样点覆盖区域表层海水弧菌菌群结构(见图1~3)。结果表明,与类灿烂弧菌群(V.splendidus-related group)相似性最高的弧菌是秋季胶州湾海域优势菌群,其在3组测序结果中分别占到48.3%(14/29),48.5%(16/32)和50%(5/ 10);同时,除优势群外,测得的序列在3株系统树中分布较均匀。因此,从序列(代表不同的物种)覆盖面上看,胶州湾海域弧菌多样性较丰富,且秋季弧菌复杂度较高。虽然可以将序列转换成操作分类单元(Operational taxonomic unit,OTU),计算多样性指数,但没有标准界定种内变异程度,只能根据序列分布初步比较多样性情况。
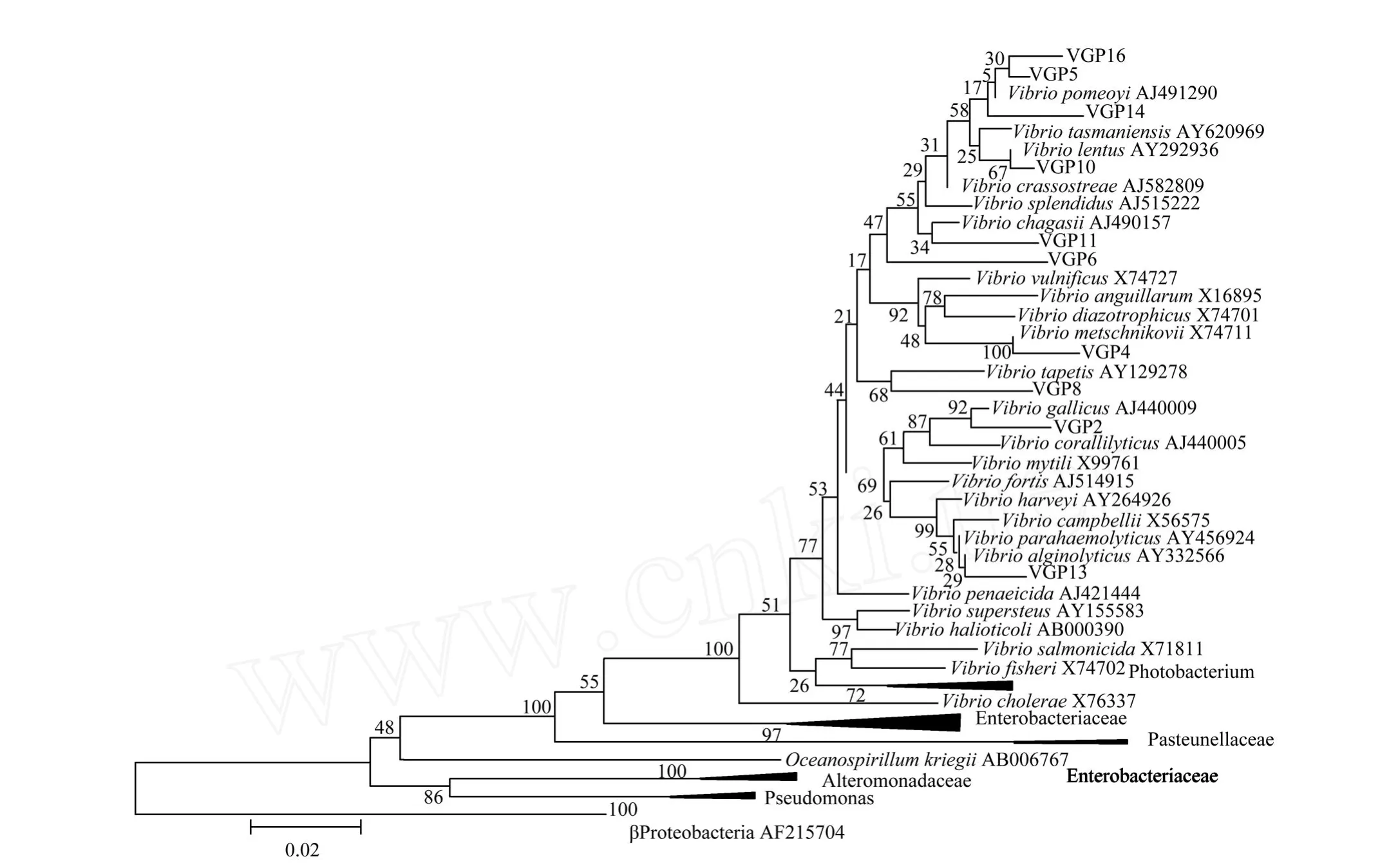
图3 基于VF27/VR744引物对扩增16S rRNA基因测序所得序列及γ-变形菌纲中常见类群代表序列构建的系统树。以β-变形细菌为外组群,重复1000次计算bootstrap值。其中,VGP表示本研究中VF27/VR744扩增得到的序列Fig.3 Phylogenetic tree based on approximately 725 nucleotide positions amplified with VF27/VR744 primer pair,showing affiliation of 16S gene clones recovered from the surface water of Jiaozhou Bay.Sequence ofβProteobacteriawas used as outgroup.VGP series sequences indicated sequences amplified directly by VF27 and VR744 in this study.This tree was constructed by the N-J method and the bootstrap values were obtained with 1000 resamplings
3 讨论
3.1 16S rDNA在细菌分类中的应用
从1970年代开始,分子生物学方法开始用于生物进化研究,16S rDNA序列成为研究细菌系统发生关系的1个重要标记。1994年,Olsen和Woese首次根据16S rDNA基因的差异将原核生物划分成12个大的类群。近年来,16S rDNA逐步在微生物的分类鉴定中发挥作用,然而,分子分类和传统分类之间存在一定的差异[14]。
尽管国际细菌系统学委员会及新版的“Bergey氏细菌鉴定手册”都以16S rDNA基因作为细菌分类的辅助依据,一直以来并未形成一个公认的通用标准,一般倾向于将16S rRNA序列相似性达到97%以上确定为同一个种[15-16]。然而,弧菌属不适用这一标准。Kim等已经证明16S rRNA序列在弧菌种间相似性非常高,作为区分弧菌种的依据不恰当[17-18]。本研究中,作者从GenBank随机选取了10个>1.3kb的V. splendidus和V.lentus序列,计算得到V.lentus种内的平均遗传距离为0.002,而V.lentus和V.splendidus种间最小遗传距离只有0.001,说明弧菌属中种内变异有可能大于种间变异。这与Kim的结论一致。另外,本研究构建的系统树中弧菌属内有些节点bootstrap值很低,作者认为这可能是由于弧菌属16S rRNA序列的变异以近似连续的形式存在,种间差异不显著,导致在进行以序列信息为依据的系统学分析时许多种的弧菌不具有唯一的聚类特征。鉴于以上原因,本研究未能确定一个相似性标准将所测序列划分成可操作分类元,只根据系统树对胶州湾海域弧菌菌群结构进行了初步分析。
3.2 不同引物组合的比较
将设计的引物序列分别在GenBank中做BLAST分析,结果显示(见表1),与VF169完全匹配的全部序列中弧菌序列占73.6%(480/652),与VR744完全匹配的序列中弧菌序列占88.3%(483/547),与VR1150完全匹配的序列中弧菌序列仅占59.8%(404/676),其中,又以VR744对应的弧菌序列绝对数量最大(483个);同时,表中有部分完全匹配序列γ-变形菌纲的未鉴定种,这一部分序列也很可能是弧菌属成员,因此, VR744的弧菌属特异性可达到90%左右。BLAST分析提示,从遗传学角度看,弧菌属的主要背景干扰来自γ-变形菌纲内部,该结果与前人的结论一致。因此,文中设计引物时重点考虑排除来自该菌群内部的干扰。PCR过程中,引物-模版在3’端的3~5个碱基是否严谨配对对DNA能否正常延伸起着至关重要的作用[19-20]。根据这一认识,作者尽可能的把引物的选择位点放在引物的3’端(见图4)。VF169可以排除发光杆菌、肠杆菌和假交替单胞菌,VR1150可以将除发光杆菌外的绝大部分干扰菌排除,而VR744自身可通过3~9个碱基的差异排除个别发光杆菌(GenBank中435个发光杆菌序列只有10个与VR744完全匹配)外的所有干扰菌排除。因此,本文设计的引物序列中, VR744具有最佳的弧菌特异性。

表1 GenBank中与所设计的引物完全匹配的序列所代表的原核生物分类阶元及其丰度Table 1 Abundances of the taxa represented by 16S ribosomal RNA gene sequences that perfectly match the primers designed in this study

表2 与引物序列完全匹配的已鉴定的弧菌种类(来自GenBank)Table 2 Well-identified species of genusvibriowith their 16S rDNAs available in GenBank which have identical matches with designed primers

续表1

图4 本研究中设计的引物与γ-变形菌纲常见菌16S rRNA基因对应片段的对位分析(Multiple alignments)Fig.4 Alignments of primers and their corresponding regions in the 16S ribosomal RNA genes ofvibrioand its close genera.The specificities of primers forvibriowere determined by the mismatches at different nucleotide sites,especially at 3’termini
为了检验引物覆盖弧菌属的程度,作者于2004年7月统计GenBank中已分离鉴定并根据双名法正式命名的弧菌序列66种以及原来归入弧菌属近年来重新归类命名的鳗利斯顿氏菌(L istonella anguillarum)、海利斯顿氏菌(L istonella pelagia)和美人鱼发光杆菌(Photobacterium damsela)[21-22]共69种细菌。将3个特异引物分别在GenBank做BLAST分析,结果显示(见表2),在不考虑菌株间变异的前提下,VF161可以覆盖58种弧菌(84.1%),VR744可以覆盖63种(91.3%),VR1150可覆盖64种(92.8%)。同时,除Vibrio rotif erianus外,VR744可以覆盖VF161的覆盖范围。说明VR744可以较全面的覆盖弧菌属成员。
以上分析表明,VR744具有最佳弧菌属特异性和属内最大的覆盖范围。因此,正向引物应该选择不具有任何选择性的细菌通用引物,即VF27。综上所述,实验结果和数据分析都充分证明,VF27/VR744引物对对海洋环境中的弧菌菌群表现出很高的特异性,同时较广地覆盖了该环境中弧菌的种类。它为从食品、环境样品以及临床样品中迅速检定是否污染弧菌提供了1种快捷的手段,对于海洋环境监测,水产养殖中病害预测和监控有明显的研究意义和应用价值。
[1] Muroga K.Viral and bacterial diseases in larval and juvenile marine fish and shellfish-A review[J].Fish Pathology.19955,30: 71-85.
[2] Actis L A,Tolmasky M E,Crosa J H.Vibriosis[M].Fish Diseases and Disorders.1999,3:523.
[3] Elena A.Numerical taxonomy ofVibrionaceaeisolated from cultured amberjack(Seriola dumerili)and surrounding water[J]. Current Microbiology,2003,46:184-189.
[4] Shangkuan Y,Lin H,Wang T.Diversity of DNA sequences amongVibrio choleraeO1 and non-O1 isolates detected by wholecell repetitive element sequence-based polymerase chain reaction [J].Journal of Applied Microbiology,1997,82:335-344.
[5] Rice,S A,McDougald D,Kjelleberg S.Vibrio vulnif icus:a physiological and genetic approach to the viable but nonculturable response[J].Journal of Infection and Chemotherapy,2000,6: 115-120.
[6] Torres L,Escobar S,L pez A I et al.Wound infection due tovibrio vulnif icusin Spain[J].European Journal of Clinical Microbiology&Infectious Diseases,2002,21:537-538.
[7] Croci L.Microbiological risk associated with seafood consumption [J].Annali dell’Istituto Superiore di Sanita,2003,39:35-45.
[8] Woese C R,Fox G E.Phylogenetic structure of the prokaryotic domain:the primary kingdoms[J].Proceedings of the National Academy of Sciences of the United States of America,1977,74: 5088-5090.
[9] Walter E H,Stacye P K,Mary W T,et al.Polymerase chain reaction identification ofVibrio vulnif icusin artificially contaminated oysters[J].Applied and Environmental Microbiology,1991, 57:707-711.
[10] Bej A K,Ng W Y,Morgan S,Jones D D,Mahbubani M H.Detection of viableVibrio choleraeby reverse-transcriptase polymerase chain reaction(RT-PCR)[J].Molecular Biotechnology, 1996,5:1-10.
[11] 刘瑞玉.胶州湾生态学和生物资源[M].北京:科学出版社,1992.
[12] Olsen GJ,Woese C R,Overbeek R.The winds of(evolutionary)change:Breathing new life into microbiology[J].Journal of Bacteriology,1994,176:1-6.
[13] Polen-Fuller M.A two-hour method for extraction of DNA from seaweeds[J].Phycological Newsletter,1991,23:2.
[14] 张英培.分子分类的若干问题[J].动物学研究.1994,15(1): 1-10
[15] Stackebrandt E,Goebel B M.Taxonomic note:a place for DNADNA reassociation and 16S rRNA sequence analysis in the present species in bacteriology[J].International Journal of Systematic Bacteriology,1994,44:846-849.
[16] Sacchi C T,Whitney A M,Mayer L W et al.Sequencing of 16S rRNA gene:a rapid tool for identification ofBacillus anthracie [J].Emerging Infectious Diseases,2002,8:1117-1123.
[17] Kim YB,Okuda J,Matsumoto C,et al,Nishibuchi M.Identification ofVibrio parahaemolyticusstrains at the species level by PCR targeted to the toxR gene[J].Journal of Clinical Microbiology,1999,37:1173-1177.
[18] Kita-Tsukamoto K,Oyaizu H,Nanba K,et al.Phylogenetic relationships of marine bacteria,mainly members of the family Vibrionaceae,determined on the basis of 16S rRNA sequences [J].International Journal of Systematic Bacteriology,1993,43: 8-19.
[19] Kwok S,Chang S-Y,Sninsky J J,et al.A guide to the design and use of mismatched and degenuste primers[M].PCR Methods and Applications,1994,3:S39-S47.
[20] Sommer R,Tautz D.Minimal homology requirements for PCR primers[J].Nucleic Acids Research,1989,17:6749-6755.
[21] MacDonell M T,Colwell R R.Phylogeny of the Vibrionaceae and recommendation for two genus,Listonella and Shewanella [J].Systematic and Applied Microbiology,1985,6:171-182.
[22] Smith S K,Sutton D C,Fuerst J A,et al.Evaluation of the genus Listonella and reassignment of Listonella damsela(Love et al.)MacDonell and Colwell to the genus Photobacterium as Photobacterium damsela comb.nov.with an emended description [J].International Journal of Systematic Bacteriology,1991,41: 529-534.
[23] 徐怀恕,杨学宋,李筠,等.对虾苗期细菌病害的诊断与控制[J].青岛:青岛海洋大学出版社,1999.
Abstract: Vibriois among the most prevalent microbial groups in marine environment.The pathogenic species of genusVibriocausevibriosis in marine animals and should have to be responsible for severe economic losses in aquaculture worldwide every year.According to the sequences ofVibrio16S rRNA gene (16S rDNA)retrieved from GenBank,we designed threeVibrio-specific primers,VF169,VR744 and VR1150.From these designed primers and VF27(one of the universal primers of eubacteria),three pairs of PCR primers(VF169/VR744,VF169/VR1150 and VF27/VR744)were arranged.SeawaterVibrio16S rDNAs were amplified using VF169/VR744,VF169/VR1150 and VF169/VR744 in nested manner and VF27/VR744,respectively,and clone libraries were constructed accordingly.An amount of clones selected randomly from three libraries were sequenced,and 33,29 and 10 sequences were obtained from these three libraries,respectively.Phylogenetically,all of the sequences show their highest similarities to and cluster with those of diverse knownVibriospecies.Primer pair VF27/VR744 was proven to be fairly specific toVibrio-genus and could cover most species of the genus.This primer pair is useful for the analysis ofVibriocommunity structure with surface seawater samples.We can also infer from the sequences obtained thatVibrioshow marked diversity,andV.splendidusrelated group predominate in Jiaozhou Bay surface seawater in autumn.The method and data obtained in this study make it possible to develop new rapid techniques for detectingVibriospecies specifically.
Key words: Vibrio;16S rRNA gene(rDNA);Vibrio-specific primer;Vibriocommunity structure
责任编辑 于 卫
Designing of Vibrio 16S rDNA Specific Primers and Analysis of Seawater Vibrio Community Structure
SUN Ying,LIU Yong,WANG Hua-Lei,CHEN Ji-Xiang,YANG Guan-Pin
(College of Marine Life Sciences,Ocean University of China,Qingdao 266003,China)
Q939.1;Q178.53
A
1672-5174(2010)09Ⅱ-122-09
国际自然科学基金(瑞典)课题(A/3224-1)资助
2009-02-25;
2010-01-15
孙 颖(1982-),硕士生。E-mail:oucsy@hotmail.com
E-mail:yguanpin@ouc.edu.cn

