碱胁迫应答GsGASA1及GsGASA2基因表达特性研究
李昆仑,柏 锡,卢 姗,袁 悦,李 勇,纪 巍,才 华,季佐军,朱延明
(东北农业大学生命科学学院,哈尔滨 150030)
植物的逆境应答是一个很复杂的生物学过程,当植物受到外界环境胁迫后,会引发体内一系列的生理生化反应,并最终通过信号传导过程诱导或者抑制某些基因的表达,以避免或减少逆境对自身的危害[1]。近年来,通过基因芯片等分子生物学技术,筛选出一批受逆境诱导表达的基因[2]。野生大豆是栽培大豆的近缘野生种,在我国分布广泛,类型丰富。具有抗逆性和适应能力强、产量性状突出等众多优良性状,野生大豆具有很强的抗逆性和适应能力,是利用基因工程手段进行作物抗逆分子育种的重要基因来源供体材料[3]。
GASA(Gibberbllic acid-stimulated inArabidopsis)基因家族是近年来研究较多的一类受GA调控的基因家族[4]。第一个被发现的GASA基因家族成员是由Shi等在赤霉素缺失的番茄突变体gib1中分离得到的,后陆续从番茄[5]、矮牵牛[6]、马铃薯[7]、水稻[8]、非洲菊[9]、拟南芥[4]等中获得了同源基因。已有的研究表明,大多数GASA基因受GA调控。例如,GA诱导番茄中GAST1的表达,而GAST1的另一个同源基因RSI-1的表达则受生长素的诱导[5];赤霉酸(GA3)诱导矮牵牛中分离得到的4个GIP同源基因(GIP1、GIP2、GIP4和GIP5)的表达[6];GA3促进水稻和GA合成缺失突变体中OsGASR1和OsGASR2的表达[8];GA诱导非洲菊(Gerbera hybrida)GEG基因在花冠和心皮中表达[9]。在非生物胁迫方面,研究表明GASA基因可以响应多种逆境胁迫并在其中起到重要作用,如Snakin-2基因在受到机械损伤下显著上调表达,超量表达后可以提高对病原体的抵抗能力[7];拟南芥GASA4基因的表达在热激处理下短时间内迅速积累[10],超量表达后也提高了转基因植株耐热性[10]。
本研究在前期构建的野生大豆碱胁迫芯片表达谱中选取了两个在碱胁迫中显著上调表达并且经生物信息学预测属于GASA基因家族的基因[11],分别命名为GsGASA1、GsGASA2。进一步检测其在不同非生物胁迫条件及激素处理下的表达模式,为研究GsGASA1和GsGASA2基因在植物逆境胁迫应答方面的功能及其作用机制奠定了基础,也为综合改良作物耐盐碱能力提供基因资源。
1 材料与方法
1.1 材料
植物材料为野生大豆(G07256),采自吉林省白城地区盐碱地,由东北农业大学植物生物工程研究室提供。
1.2 野生大豆的处理
用浓硫酸处理10 min以消化泥膜,蒸馏水洗净,播种于由营养土和蛭石按1∶1比例配制成的基质中,培养21 d后,洗净根部,分别用50 mmol·L-1NaHCO3、100 mmol·L-1NaCl、30%PEG(W/V)、4 ℃、100 μmol·L-1GA3、10 μmol·L-1PAC、100 μmol·L-1ABA进行处理,以未处理的野生大豆幼苗作为对照(0 h),取样时间为处理后0.5、1、3、6和12 h。用NaHCO3处理的材料,取幼嫩的根和叶,其他方式处理的材料,取幼嫩的根。
1.3 总RNA的提取与cDNA第一链的合成
采用天根公司RNA提取试剂盒并按照其说明提取总RNA。取RNA 5 μg,采用Invitrogen公司的SuperScript II反转录酶试剂盒合成双链cDNA。
1.4 生物信息学预测
应用在线工具InterProScan(http://www.ebi.ac.uk/Tools/InterProScan/)预测目的基因编码的蛋白质的结构域、信号肽和跨膜区。利用DNAMAN分析蛋白质序列之间的相似性。
1.5 半定量RT-PCR分析
内参采用β-Actin,扩增程序为94℃预变性7 min,94℃变性30 s,60℃退火30 s,72℃延伸20 s,26个循环。GsGASA1、GsGASA2基因的表达分析使用相应数量的cDNA,反应条件与内参的相同,PCR扩增均进行26个循环。用2%的琼脂糖凝胶进行电泳检测。进行两次重复。应用ImageJ软件(http://rsbweb.nih.gov/ij/)计算目的基因的相对吸光值。所用的引物序列如下:
GsGASA1-Sense 5'CACATATCATAGCAATGGC AGCAC 3';
GsGASA1-RTSense 5'ATTTGGGTCCTCCTTCCT TGG 3';
GsGASA2-Sense 5'AATGGCTAAGTTCTTTGCTG CTATG 3';
GsGASA2-RTSense 5'CTCCTTCCTTGGTCTTCC AGTTG 3';
β-Actin-Sense 5'GAAGATGGCAGACGCTGAG GAT 3';
β-Actin-Antisense 5'ACGACCTACAATGCTGG GTAACAC 3'。
2 结果与分析
2.1 GsGASA1、GsGASA2基因的获得
利用实验室前期构建的野生大豆碱胁迫基因芯片表达谱,筛选得到了两个在盐碱胁迫下明显上调的候选基因(probset分别为Gma.15958.1.S1_at;GmaAffx.90343.1.S1_at)。根据芯片杂交结果,这两个基因在栽培大豆基因组数据库中对应同源基因的GlymaID分别为Glyma02g41280.1、Glyma18g49400.1。应用在线工具Interproscan对Glyma02g41280.1、Glyma18g49400.1基因编码的蛋白质序列进行结构域、信号肽和跨膜区预测(见图1)。预测结果表明,这两个基因都具有GASA基因家族共同的结构特征,由100个左右氨基酸组成,在N端均有一可剪切的信号肽,分别由27、24个氨基酸组成,因此可能属于分泌蛋白。在紧接信号肽下游,含有一个亲水区域,由22~23个亲水氨基酸组成。C端含有60个氨基酸的GASA结构域,其中包括12个保守的半胱氨酸残基,跨膜结构预测表明两个蛋白在N端都有一个跨膜区。根据野生大豆和栽培大豆之间序列的高度相似性,推测野生大豆中的这两个基因也属于GASA基因家族,因此分别命名为GsGASA1、GsGASA2基因,并且推测GsGASA1和GsGASA2基因编码的蛋白质也同样属于分泌蛋白,它们各自的信号肽可能将引导其成熟产物定位于细胞壁。另外,由于GsGASA1、GsGASA2基因序列较短,仅编码100个左右的氨基酸,可以推测C端60个氨基酸构成的GASA结构域对基因的功能是非常重要的。利用DNAMAN对Glyma02g41280.1与Glyma18g49400.1基因所编码的蛋白质序列进行比对,发现二者一致性达60%(见图2)。同样根据野生大豆和栽培大豆序列的高度相似性,我们可以推测GsGASA1基因和GsGASA2基因分别所编码的蛋白质序列之间的相似性也在60%左右。
2.2 GsGASA1、GsGASA2基因芯片结果的sqRTPCR验证
野生大豆中NaHCO3处理下GsGASA1、GsGASA2基因的sqRT-PCR验证结果表明(见图3),GsGASA1基因在根中于0.5 h时出现显著上调表达,这种上调趋势维持到1 h,在3 h开始下降,在处理6 h时再次出现上升,随后在12 h恢复至处理前的表达水平。GsGASA2基因在根中也于0.5 h时出现显著上调表达,而后一直到处理12 h都呈现下降的趋势。因此,碱胁迫处理下,GsGASA1、GsGASA2基因在野生大豆根中的sqRT-PCR结果和在基因芯片表达谱中表达模式相比较[11],虽然GsGASA1、GsGASA2基因的表达峰度大小存在一定差异,但是变化趋势一致,均呈现在根中碱胁迫处理短时间内迅速上调表达随后恢复到最初的表达水平的变化模式,进一步验证了基因芯片结果。另外,在叶中,GsGASA1基因的表达量出现先下降再上升的趋势,最终回到胁迫处理前的表达水平。GsGASA1基因在根和叶中表达模式的不同表明在碱胁迫反应中GsGASA1基因在野生大豆不同部位所参与的调控模式不同。而GsGASA2基因可能由于时间延迟的原因在处理3 h才出现明显上调表达,随后下降至处理前的表达水平。以上结果表明,GsGASA1和GsGASA2基因均响应碱胁迫,作为候选基因用于下一步试验。
2.3 非生物胁迫及激素处理下GsGASA1、GsGA SA2基因的表达情况
通过sqRT-PCR来检测野生大豆根中GsGA SA1、GsGASA2基因在另外三种非生物胁迫—盐、干旱、低温处理下的表达情况。图4的结果表明,GsGASA1、GsGASA2基因均可以被NaCl、PEG、4℃诱导表达。然而两个基因在这三种胁迫处理下的表达模式是不同的,GsGASA1基因在三种胁迫处理下,均迅速在0.5 h出现明显上调表达,特别是在干旱处理下,在1 h达到高峰,随后下降。GsGASA2基因在NaCl处理下,也在0.5 h出现上调,但直到12 h才开始下降,而在干旱处理下,表达变化没有GsGASA1基因的显著,但也在短时间内上调,在3 h达到最大值。综上所述,GsGASA1、GsGASA2基因除了响应碱胁迫外,还参与到盐、干旱、低温三种胁迫中,并且在这四种胁迫处理中均呈现在短期内显著上调表达的模式。通过蛋白质序列比对分析发现GsGASA1与GsGASA2基因所编码的蛋白质序列一致性较高(达60%左右),这可能是GsGASA1与GsGASA2基因在碱、盐、干旱、低温四种非生物胁迫以及激素处理下表达模式相似的原因。由此我们推测GsGASA1和GsGASA2基因在植物应答非生物胁迫反应中将起到一定作用。
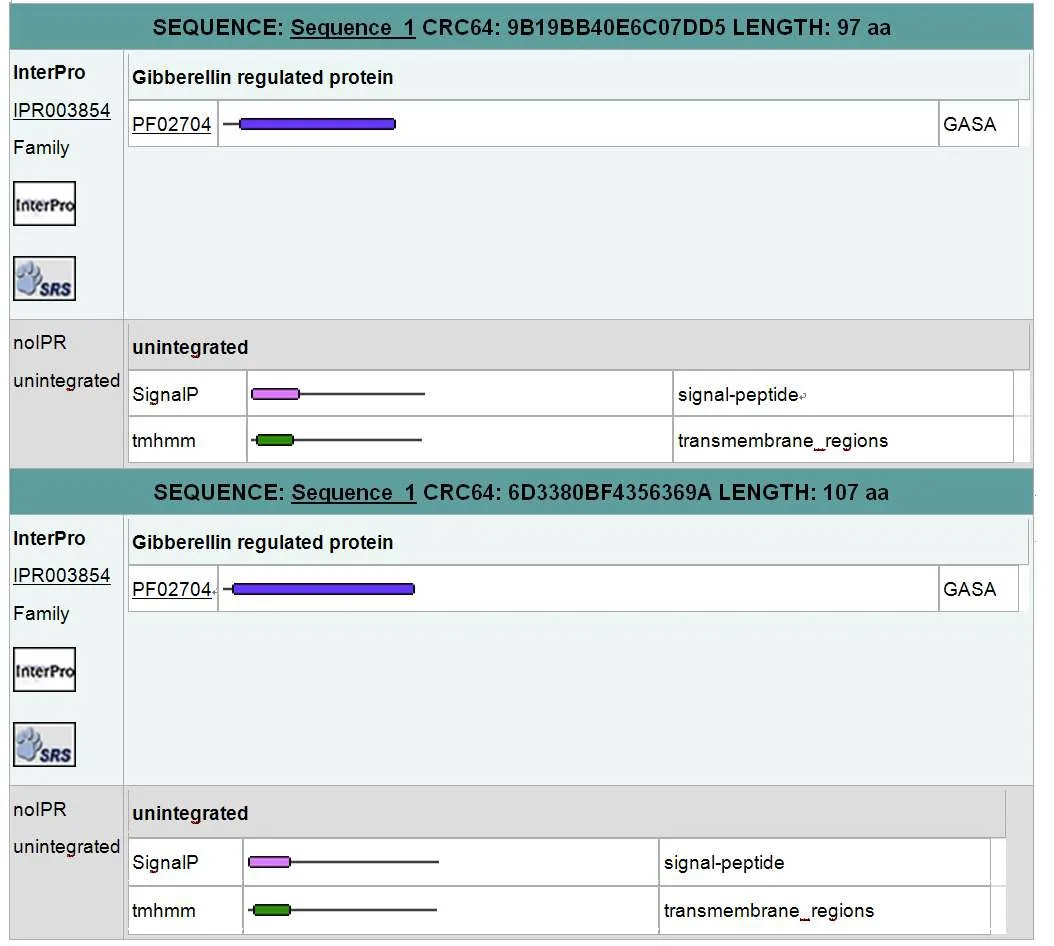
图1 Glyma02g41280.1、Glyma18g49400.1基因InterProScan预测结果Fig.1 Predict result of Glyma02g41280.1,Glyma18g49400.1 by InterProScan

图2 Glyma02g41280.1、Glyma18g49400.1基因编码蛋白质序列比对结果Fig.2 NCBI Blast result of Glyma02g41280.1,Glyma18g49400.1 amino acid sequence
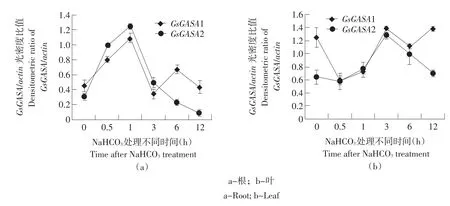
图3 GsGASA1、GsGASA2基因在NaHCO3胁迫的表达特性分析结果Fig.3 Expression patterns of GsGASA1,GsGASA2 gene in response to NaHCO3treatments
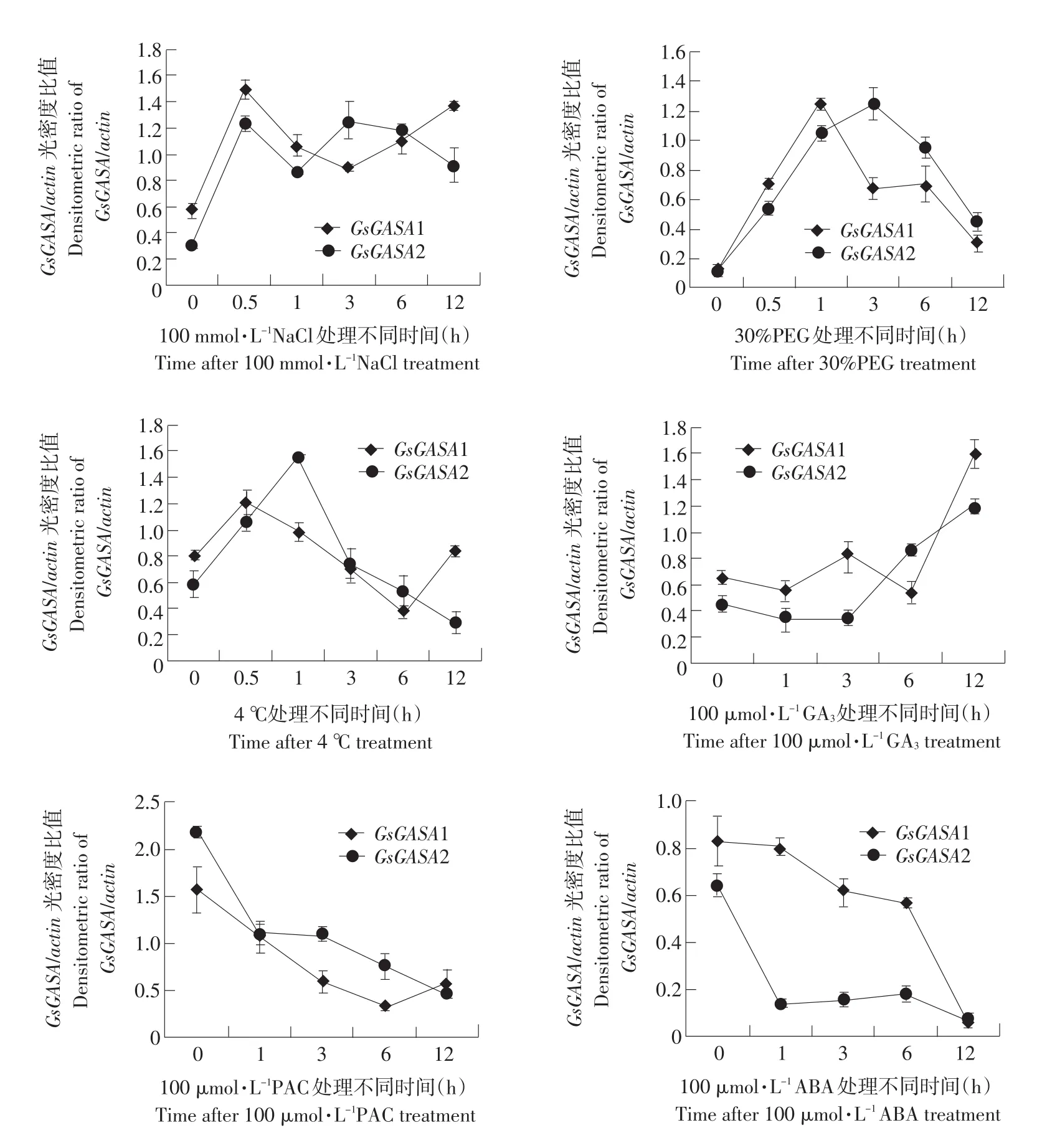
图4 野生大豆根中GsGASA1、GsGASA2基因在不同非生物胁迫、不同激素处理的表达模式Fig.4 Expression patterns of the GsGASA1,GsGASA2 gene in response to various treatments and plant hormones
另外,GA3处理下GsGASA1基因在12 h明显上调表达,GsGASA2基因在处理6 h时就开始上升,但是上升程度没有GsGASA1基因显著,而在GA生物合成抑制剂PAC处理下,GsGASA1、GsGASA2基因的表达均明显受到抑制(见图4)。因此GsGASA1、GsGASA2基因的表达可能受到GA的诱导。而GsGASA1、GsGASA2基因在GA处理下表现出先下调才上调的表达特点,可能与GA信号通路的反馈抑制特点有关。另外,在与GA互为拮抗剂的ABA处理下,GsGASA1、GsGASA2基因均表现出明显下调的趋势。因此,可以推测GsGASA1与GsGASA2基因的表达均受到GA的诱导以及ABA的抑制。
3 讨论与结论
通过野生大豆中GsGASA1与GsGASA2基因在碱、盐、干旱、冷胁迫下能被诱导表达的试验结果,可初步证明GsGASA1与GsGASA2基因均响应多种非生物胁迫,进而可能与野生大豆的耐逆性有关,这为以后进行GsGASA1、GsGASA2基因全长基因的克隆以及在非生物胁迫方面的功能验证奠定了基础。大多数GASA基因的表达都受GAs的正向调控,如GA促进GASA家族中的GIP1[6]、GEG[9]、AtGASA4[4]、OsGASR[8]等的表达。对野生大豆施加外源GA3和ABA处理后,GsGASA1、GsGASA2基因表达分析的试验结果表明(见图4),GsGASA1、GsGASA2基因对外源激素的响应基本一致,都受GA3的诱导,受PAC和ABA的抑制。说明GsGASA1、GsGASA2基因可能受GA和ABA的双重调控。本研究结果表明,GsGASA1、GsGASA2基因可能是GA和ABA这两种重要植物激素信号的整合子,为研究GA和ABA的相互作用提供了重要线索。下一步准备将GsGASA1与GsGASA2基因对模式植物拟南芥超量表达,从而分析其在非生物胁迫处理下的抗性以及进一步探讨功能机理。
本研究在前期构建的野生大豆碱胁迫基因芯片表达谱中选取了两个经生物信息学预测属于GASA家族的两个基因,分别命名为GsGASA1、GsGASA2,通过sqRT-PCR验证了芯片结果。另外,发现GsGASA1与GsGASA2基因除了响应碱胁迫处理外,均还受到盐、干旱以及低温胁迫的诱导表达,并且在这四种非生物胁迫处理下均呈现短期内显著上调表达的模式,推测GsGASA1与GsGASA2基因可能在植物逆境胁迫应答反应中起到作用。另外GsGASA1与GsGASA2基因的表达均受到GA的诱导和ABA的抑制。本研究将为研究GsGASA1和GsGASA2基因在非生物胁迫中的功能以及在GA和ABA信号通路中的作用奠定基础。
[1] 李杰,陈丽华,朱延明.植物抗渗透胁迫基因工程研究进展[J].东北农业大学学报,2005,36(2):241-248.
[2] Kreps J A,Wu Y,Chang H S,et al.Transcriptome changes forArabidopsisin response to salt,osmotic,and cold stress[J].Plant Physiology,2002,130:2129-2141.
[3] Zhang G D.Studies on annual wild soybeans[J].Journal of Northeast Agricultural University:English Edition,1994,1(1):9-19.
[4] Zhang S C,Wang X J.Expression pattern of GASA,downstream genes of DELLA,inArabidopsis[J].Chinese Science Bulletin,2008,53(24):3839-3846.
[5] Taylor B H,Scheuring C F.A molecular marker for lateral root initiation:the RSI-1 gene of tomato(Lycopersicon esculentumMill.)is activated in early lateral root primordia[J].Mol Gen Genet,1994,243:148-157.
[6] Wigoda N,Ben N G,Granot D,et al.The gibberellin-induced,cysteine-rich protein GIP2 fromPetunia hybridaexhibitsin planta antioxidant activity[J].Plant Journal,2006,48:796-805.
[7] Berrocal L M,Segura A,Moreno M,et al.Snakin-2,an antimicrobial peptide from potato whose gene is locally induced by wounding and responds to pathogen infection[J].Plant Physiology,2002,128:951-961.
[8] Furukawa T,Sakaguchi N,Shimada H.Two OsGASR genes,rice GAST homologue genes that are abundant in proliferating tissues,show different expression patterns in developing panicles[J].Genes Genet Syst,2006,81(3):171-180.
[9] Kotilainen M,Helariutta Y,Mehto M,et al.GEG participates in the regulation of cell and organ shape during corolla and carpel development inGerbera hybrida[J].Plant Cell,1999(6):1093-1104.
[10] Ko C B,Woo Y M,Lee D J,et al.Enhanced tolerance to heat stress in transgenic plants expressing theGASA4 gene[J].Plant Physiol Biochem,2006,45:722-728.
[11] Ge Y,Li Y,Zhu Y Y,et al.Global Transcriptome profiling of wild soybean(Glycine soja)roots under NaHCO3treatment[J].BMC Plant Biology,2010(10):153.

