基于mtDNA COⅠ基因的DNA条形码技术鉴定鞘翅目幼虫
刘翠霞,林恭华,苏建平,陈生云,张同作
(1.中国科学院西北高原生物研究所,青海 西宁 81008; 2.中国科学院大学,北京 100049;3.中国科学院寒区旱区环境与工程研究所,甘肃 兰州 730000)
鞘翅目是昆虫纲中最大的类群,在全球范围内都有分布,目前已知有36万多种,是昆虫纲及动物界种类最多、分布最广的第一大目[1]。传统的鞘翅目分类主要依据成虫的形态学结构差异进行分类。然而,相近种类鞘翅目昆虫的幼虫(包括卵、蛹)形态特征非常相似,有些特征在不同的发育时期不稳定,根据非成虫态的虫体形态学特征进行种类鉴定非常困难,对于样本碎片和残缺个体更是无计可施。近年来,随着分子生物学技术的兴起和高速发展,特别是PCR技术的日趋完善,DNA条形码(DNA Barcoding)技术作为新的物种分类方法和手段极大地弥补了传统形态学分类的缺陷,并以其高速、快捷、准确的特点成为广大生物分类学家关注的热点[2-8]。
DNA条形码是一种利用短链DNA片段,对物种进行快速、准确地识别和鉴定的分子生物学技术。细胞色素氧化酶Ⅰ(Cytochrome Oxidase Ⅰ,COⅠ),由于相对容易扩增,较少发现插入、缺失现象,变异速率在种内相对较小,在种间有相对较大变异速率等优势[2],已成为当前DNA条形码在动物界的主要分子标记。许多基于COⅠ作为DNA条形码分子标记的研究都显示了其在物种鉴定方面的巨大作用[9-12]。
DNA条形码技术在昆虫分子鉴定中应用较多,并以鳞翅目研究最为广泛。Hebert等[2]应用COⅠ基因成功鉴定出200多个鳞翅目物种。Hajibabaei等[13]应用DNA条形码技术对多种热带鳞翅目昆虫进行了准确识别。Monaghan等[14]应用DNA序列在马达加斯加热带水生甲虫中发现几个隐存种。但是,目前国内尚未见用DNA条形码技术鉴定鞘翅目昆虫的研究。因此,本研究以线粒体COⅠ基因为标记,利用DNA条形码技术首次对疏勒河上游及青海湖北岸常见鞘翅目幼虫进行种类鉴定,以探讨DNA条形码技术及COⅠ基因在鞘翅目昆虫分类鉴定中的可行性及准确性。
1 材料与方法
1.1试验材料 试验样品于2010 年7-8月采自疏勒河上游及青海湖北岸地区,置于无水乙醇中保存。根据形态特征选取20只鞘翅目幼虫作为DNA样本提取材料。
1.2试验方法
1.2.1基因组DNA的提取 取整只幼虫为材料提取基因组DNA,用蒸馏水浸泡直至幼虫中的酒精去净。加入700 μL细胞裂解液、4 μL蛋白酶K(20 mg·mL-1)及2 μL RNase A(10 mg·mL-1)进行消化,55 ℃恒温作用直至消化完全。苯酚、氯仿/异戊醇 (24∶1)抽提,800 μL无水乙醇沉淀,产物以300 dd H2O溶解,用10 ms·mL-1琼脂糖凝胶电泳检测并估计浓度,于4 ℃下保存备用。
1.2.2PCR扩增与测序 以20只鞘翅目幼虫的总DNA为模板,用引物扩增COⅠ基因的部分序列(约830 bp)。引物由上海生物工程公司(Sangon) 合成,序列是,引物1:5′-CAACATTTATTTTGATTTTTTGG-3′;引物2:5′-TCCAATGCACTAATCTGCCATATTA-3′。
PCR 扩增在ABIVeritiTM 1000型PCR仪上进行,反应体积为25 μL,其中dd H2O 18.7 μL、10×PCR Reaction Buffer 2.5 μL、dNTPs(2.5 mmol·L-1) 1.2 μL(Takala)、Jerry 1.0 μL(10 μmol·L-1)、 Pat 1.0 μL(10 μmol·L-1)、Taq 酶0.2 μL(5 U·μL-1,Takala)、DNA模板 0.4 μL。反应条件为94 ℃ 预变性 5 min,94 ℃变性30 s,52 ℃退火1 min,72 ℃延伸1 min,共34个循环,最后72 ℃延伸10 min。PCR 产物上样至1.0%琼脂糖凝胶电泳,EB染色,用凝胶成像系统观察、照相。检测以Markers (Takala DL2000)作为分子量标记。PCR产物送至上海生工生物技术公司进行测序。
1.2.3序列分析 应用Chromas软件对测序原始结果进行人工校对,去掉两端不整齐的序列,利用MEGA 4.0软件进行序列比对,同源排序,并在GenBank中应用Blast搜索下载相似科COⅠ序列。利用MEGA 4.0对得到的20条幼虫序列和相似性搜索下载的鞘翅目COⅠ序列用Kimura双参数遗传距离法计算遗传距离,基于遗传距离用邻接法(Neighbor-Joining)构建系统树,用Bootstrap检验系统树各分支置信度,1 000次循环,检查序列鉴定的准确性。
2 结果与分析
2.1Blast相似性搜索结果 在GenBank中用Blast搜索相似的基因序列,对比片段信息的可靠性,初步判定这20个样本的科,亚科和属。其中,15号鉴定至科,8、12和16号鉴定至属,其余的鉴定至亚科。1、2、10、13和18号属于金龟子科(Scarabaeidae),其中,1、2号分别属于花金龟亚科(Cetoniinae)的不同属,10、13和18号属于鳃金龟亚科(Melolonthinae);8号属于拟步甲科(Tenbrionidae)的Nyctoporis属;12、16号属于拟步甲科的Dailognatha属;15号属于天牛科(Cerambycidae);3、4、6、14和20号属于步甲科(Carabidae);5、7、11、19号属于象甲科(Curculionidae)的Molytinae亚科,9、17号属于象甲科的Curculioninae亚科。
2.2遗传距离分析 Raupach等[15]对欧洲344个步甲科物种基于COⅠ基因序列用K2P法计算了种内和种间遗传距离,结果显示,同科的平均种间距离为0.126,并且最大距离不大于0.2,而同属的种间距离为0~0.005。郑福山等[16]基于COⅠ基因用K2P法对叶甲科小萤叶甲属10种昆虫进行遗传距离计算,结果显示,叶甲科的种间遗传距离介于0.169~0.198,而同属的种间遗传距离介于0.001~0.134,种间距离与Raupach得出的范围较为一致。
由本研究测得的20条鞘翅目幼虫COⅠ序列与相似性搜索下载的7条鞘翅目COⅠ序列用双参数法计算所得遗传距离可知(表1),1、2、10、13和18 号与金龟子科的遗传距离介于0.160~0.164,与其他科种间的遗传距离均大于0.2,参考Raupach等[15]和郑福山等[16]的同科平均种间距离小于0.2的标准,可判定这几个鞘翅目幼虫属于金龟子科。以此类推,12和16号属于拟步甲科的Dailognatha属,8号属于拟步甲科的Nyctoporis属,15号属于天牛科,5、7、9、11、17和19号属于象甲科,3、4、6、14和20号属于步甲科。7、9、11、17号与象甲科的两个亚科的遗传距离都介于0.138~0.197,说明象甲科的这两个亚科亲缘关系较近,由遗传距离可推断9、17号属于象甲亚科(Curculioninae),而5、7、11、19号属于象甲科的Molytinae亚科。
2.3基于COⅠ条码序列构建的系统树 20个鞘翅目幼虫COⅠ基因序列和7条从GenBank下载的鞘翅目5个科的COⅠ (566 bp)(其中象甲科和拟步甲科各有两个亚科)基因部分序列数据通过NJ法构建系统进化树。此NJ树具有很高的置信度(图1),20个鞘翅目幼虫分为5支,其中1、2、10、13、18号与金龟子科聚为一支;8、12、16号与拟步甲科聚为一支,就科内种间关系来看,8号优先与拟步甲科的Nyctoporis相聚,而12、16号优先与拟步甲科的Dailognatha相聚;3、4、6、14、20号与步甲科聚为一支 ;15号与天牛科聚为一支;5、7、9、11、17、19号与象甲科聚为一支,就科内种间关系来看,9和17号与象甲科的Curculioninae亚科优先相聚,5、7、11、19号与象甲科的Molytinae亚科优先相聚。
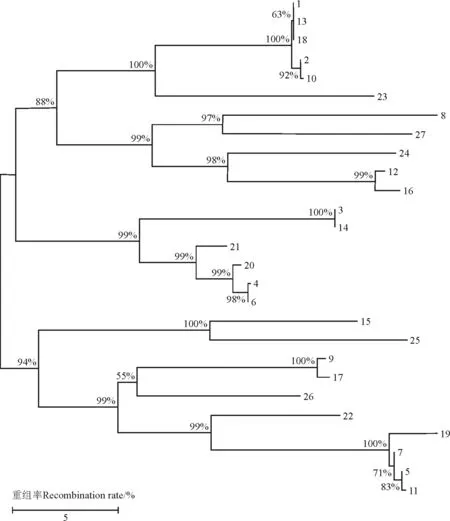
图1 NJ法构建的鞘翅目幼虫分子系统树Fig.1 Molecular phylogenetic tree of Coleoptera constructed by NJ method
3 讨论
DNA条形码开启了物种分类鉴定的新时代,其显著特点是可以及时快速获得分子数据,极大地简化物种分类和鉴定工作。mtDNA上COⅠ序列条形码被广泛应用于物种分类研究。利用DNA条形码进行分类和鉴定,能够发现传统形态学上很难区分的新种和隐存种。在昆虫分类鉴定方面,运用DNA条形码技术进行种类鉴定和新种发现研究工作在鳞翅目[17]、等翅目[18]、鞘翅目[19]及双翅目[20]中已经得到了广泛应用,特别是鳞翅目研究中应用最为普遍。 Hebert等[21]选取COⅠ基因的一段序列对鳞翅目200多个种进行了种类鉴定,结果表明,该特定片段可以100%成功地鉴定每个个体。迄今为止,尚未见COⅠ序列条形码用于鞘翅目幼虫的分类研究报道。本研究通过对鞘翅目幼虫mtDNACOⅠ中566 bp的基因序列进行分析,利用GenBank基因序列信息,运用Blast相似性搜索,用双参数法计算遗传距离,并以NJ法构建系统树,鉴定出20个未知鞘翅目幼虫分别属于5科9属。3个分析方法所得结果基本一致,表明应用DNA条形码技术能够对鞘翅目幼虫进行分类鉴定,mtDNACOⅠ基因用于鞘翅目幼虫分类鉴定具有可行性、方便性和快捷性。NJ无根系统发育树结果进一步表明试验鞘翅目幼虫中科之间的遗传距离较远,容易区分。在科内属的水平,象甲科的Curculioninae属和Molytinae属,拟步甲科的Dailognatha属和Nyctoporis属也能够明确区分。
经过多年的发展,DNA条形码技术得到了很多的支持,用DNA条形码对物种进行鉴定研究越来越普遍,而作为能够用作条形编码的基因,一是必须具有相对的保守性,便于用通用引物扩增;二是必须具备足够的变异能将物种鉴别。本研究中采用COⅠ基因不仅能扩增出鞘翅目的幼虫,甚至能将最初错误判定为鞘翅目的双翅目和膜翅目幼虫的序列扩增出来,说明此基因在昆虫中具有一定的保守性,而通过遗传距离等分析,也表明mtDNA上COⅠ部分序列具有足够的变异,能有效地对鞘翅目幼虫进行科属鉴定。
由于该方法快速、简便和精确,有助于对形态结构相似的幼虫、卵和蛹进行准确地鉴定,解决了鞘翅目幼虫、卵和蛹鉴定的一大难题。但是,对COⅠ中部分基因序列进行分析,虽然可以较精确地鉴定这20个鞘翅目幼虫的科、属,但对个别属间亲缘关系较近的个体(如金龟子科的各属之间)则很难鉴别,必须依靠多个基因片段来共同鉴定,这正是未来DNA条形码技术的重要发展方向。此外,DNA条形码技术用基因差异计算出的遗传距离,目前都缺少精细的区分标准,采用通用的遗传距离范围不太可能解决所有的物种鉴定问题,应该在不同的目、科甚至属之间选用不同的标准遗传距离范围,这样才能够更加精确地对不同物种进行分类鉴定。
[1] Bouchard P,Bousquet Y,Davies A E,etal.Family-group names in Coleoptera (Insecta)[J].ZooKeys,2011,88:1-972.
[2] Hebert P D N,Cywinska A,Ball S L,etal.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society of London Series B:Biological Sciences,2003,270:313-321.
[3] Ahrens D,Monaghan M T,Vogler A P.DNA-based taxonomy for associating adults and larvae in multi-species assemblages of chafers (Coleoptera:Scarabaeidae)[J].Molecular Phylogenetic Evolution,2007,44(1):436-449.
[4] Amaral A R,Sequeka M,Coelho M M.A first approach to the usefulness of cytocllrome coxidase Ⅰ barcodes in the identification of closely related delphinid cetacean species[J].Marine and Freshwater Research,2007,58(6):505-510.
[5] Will K W,Rubinof D.Myth of the molecule:DNA barcodes for species cannot replace morphology for identification and classification[J].Cladistics,2004,20(1):47-55.
[6] Timmermans M J T N,Dodsworth S,Culverwell C L,etal.Why barcode? High-throughput multiplex sequencing of mitochondrial genomes for molecular systematics[J].Nucleic Acids Research,2010,38(21):1-14.
[7] Hunt T,Bergsten J,Levkanicova Z,etal.A comprehensive phylogeny of beetles reveals the evolutionary origins of a superradiation[J].Science,2007,318:1913-1916.
[8] Hebert P D N,Penton E H,Burns J M,etal.Ten species in one:DNA barcoding reveals cryptic species in the neotropical skipper butterfly astraptes fulgerator[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(41):14812-14817.
[9] Ward R D,Zemlak T S,limes B H,etal.DNA barcoding Australia’s fish species[J].Philosophical Transactions of the Royal Society Lond B:Biological Sciences,2005,360(1462):1847-1857.
[10] Smith M A,Fisher B L,Hebert P D N.DNA barcoding for effective biodiversity assessment of a hyperdiverse arthropod group:The ants of Madagascar[J].Philosophical Transactions of the Royal Society Lond B:Biological Sciences,2005,360(1462):1825-1834.
[11] Smith M A,Poyarkov N A,Hebert P D N.COⅠ DNA barcoding amphibians:Take the chance,meet the challenge[J].Molecular Ecology Resources,2008,8:235-236.
[12] Tavares E S,Baker A J.Single mitochondrial gene barcodes reliably identify sister-species in diverse clades of birds[J].BMC Evo1utionary Bio1ogy,2008,8(1):81-81.
[13] Hajibabaei M,Janzen D H,Burns J M,etal.DNA barcodes distinguish species of tropical Lepidoptera[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(4):968-971.
[14] Monaghan M T,Balke M,Gregory T R,etal.DNA-based species delineation in tropical beetles using mitochondrial and nuclear markers[J].Philosophical Transactions of the Royal Society Lond B:Biological Sciences,2005,360(1462):1925-1933.
[15] Raupach M J,Astrin J J,Hannig K,etal.Molecular species identification of Central European ground beetles (Coleoptera:Carabida)using nuclear rDNA expansion segments and DNA barcodes[J].Frontiers in Zoology,2010,7(26):1-15.
[16] 郑福山,杜予州,王志杰,等.基于线粒体COⅠ基因序列的小萤叶甲属部分种类分子系统学研究(鞘翅目:叶甲科:萤叶甲亚科)[J].昆虫学报,2007,50(5):501-507.
[17] John W B,Scotte E M,Marinne H.Studies on New Guinea moths.2.Description of a new species ofXenothictisMeyrick (Lepidoptera:Tortricidae:Archipini)[J].Proceedings of the Entomological Society of Washington,2003,105:1043 -1050.
[18] Foster B T,Cognato A I,Gold R E.DNA-based identification of the eastern subterranean termiteReticulitermesflavipes(Isoptera:Rhinotermitidae)[J].Journal of Economic Entomology,2004,97:95-101.
[19] Cardoso A Vogler A P.DNA taxonomy, phylogeny and Pleistocene diversification of the Cicindela hybrida species group (Coleoptera:Cicindelidae)[J].Molecular Ecology,2005,14:3531-3546.
[20] Pestano J Brown R P,Suarez N M,etal.Diversification of sympatric Sapromyza (Diptera:Lauxaniidae) from Madeira:Six morphological species but only four mtDNA lineages[J].Molecular Phycogenetics and Evolution,2003,27:422-428.
[21] Hebert P D N,Cywinska A,Ball S L,etal.Biological identifications through DNA barcodes[J].Proceedings of Royal Society of London,2003,270:313-321.

