基于位图索引和B+树的BLAST改进算法
1.中山大学 数学与计算科学学院,广州 510275
2.北京师范大学珠海分校 信息技术学院,广东 珠海 519085
1.中山大学 数学与计算科学学院,广州 510275
2.北京师范大学珠海分校 信息技术学院,广东 珠海 519085
1 引言
BLAST是Basic Local Alignment Search Tool的简写。是采用启发式算法的一个生物序列比对的软件工具包,最初由Altschul S在1990年发展起来[1-2]。由于其在序列比对之前进行一次定位,并对定位片段使用得分矩阵进行打分的方式找出高分片段HSP(High Score Pair),因此虽然BLAST软件采用的算法丧失了一些敏感度,可是它也达到了计算速度的极大提高,从而BLAST逐渐成为生物信息学领域的最重要的一种软件,使用BLAST算法进行序列搜索与比对也成为了分子生物学的必备。
虽然BLAST算法速度非常快,可是面对爆炸式增长的生物数据库就显得力不从心。现代生物数据库大小增长速度十分快速,NCBI的GenBank核酸碱基数目大概每14个月就翻一倍。而即使依照摩尔定律,计算机的性能也要18个月才能更新一倍。因此,如何能够更好地使用序列比对算法将是一个重要的问题。
为此,本文设想使用位图索引组织BLAST程序使用的生物数据库,并依靠B+树进行二次索引,使得BLAST在进行序列搜索的时候,能够更快地找到HSP。
2 问题描述
BLAST算法的过程[3]分为3个步骤。分别是播种(Seeding),扩展(Extension),以及评估(Evaluation)。
BLAST算法假定序列比对中查询序列和数据库序列中的重要序列有共同之处,因此可以依靠两者之间的共同点找出应该搜索的区域。根据这个条件,BLAST程序从查询序列中提取出所有长度为一固定值W的子串,并定义这些长度固定的子串为单词(Word),然后BLAST程序利用这些单词建立一个单词表,再根据这张表,对数据库序列进行一次遍历,找出所有与单词表中单词相匹配的片段。如DNA序列S=GCACCTACTA,当W=8时序列S可以生成三个单词:GCACCTAC,CACCTACT,ACCTACTA。BLAST程序首先找出搜索数据库中有哪个位置有这三个单词,然后标记这些单词。这些搜索数据库中找到的共同单词被称为命中(Word Hits),如图1。之后BLAST只考虑在这些命中区域附近搜索。这些命中就像种子一般,这就是BLAST算法的第一个步骤:播种。后面的计算都从这里发展,依靠这样BLAST能够节省大量搜索不必要区域的时间。
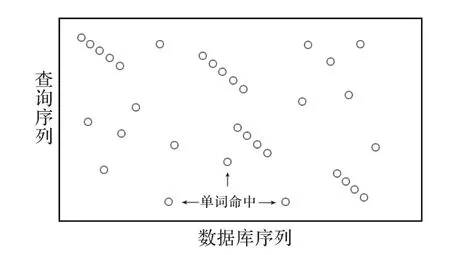
图1 命中
对于蛋白序列,命中的范围除了包括准确的匹配,还包括相似的单词。因为只考虑准确的匹配可能会忽略一些重要的匹配。例如搜索数据库中有段序列T=MGKGD,明显MGR,GRG和RGD都和序列T不匹配。可是在生物学中两个序列是十分相似的。为了避免出现这种情况。BLAST定义单词除了包括准确的匹配之外,还包括依据得分矩阵生成的邻域(Neighborhood)。每个邻域都有自己的得分,BLAST在搜索前会设置一个值T,并搜索时自动生成所有得分大于T的单词。由于蛋白序列搜索与核酸序列搜索有很大相似性,以下主要讨论DNA序列搜索的BLAST算法。
在实际的操作过程中,BLAST程序会先利用查询序列根据用户设定的W值和T值生成一个单词表。然后顺序查询数据库序列,对于每一个长度为W的子序列,BLAST都在单词表中进行查找,如果找到,则说明这是一个命中,否则跳过。假设单词长度为W,序列生成单词数目为v。序列数据库共有n条序列,序列平均长度为l,假定没有长度小于w的序列,则每个序列能够产生的片段数为l-w+1。总共产生片段数为n(l-w+1)。对于每一个片段,BLAST都要从单词库中查找一次,在对单词表排序的情况下,采用二分法查找则使用的平均时间复杂度为O(lnv)。因此整个过程时间复杂度为:

无论比对的序列为蛋白序列还是DNA序列,其W值通常为3~12左右。而数据库中序列的长度通常都是超过100。因此可以忽略上式的w值。可记时间复杂度为:

实际上,n与l的乘积代表序列数据库的总大小,用C代替,上式可化简为:

3 基于位图索引和B+树的BLAST改进算法
由式(1)知上述过程耗时取决于数据库大小C与生成单词数目v。其搜索速度与数据库大小成线性关系。而现代生物数据库发展的速度将会对计算资源提出巨大的要求。因此逐字顺序检索数据库序列是不现实的。而对于一个DNA查询序列,其生成的单词数理论最多有4w个,当W=11时约为4×106个。如果能够找出一种办法,能够加快单词从数据库中找到匹配的时间,则效果可能会好很多。
如果在搜索数据库中对所有可能的单词建立其所在数据库位置的表,并对这个表对单词字段建立位图索引[4-5],则可以极大地加快单词匹配的速度。为了加快单词的检索,可以对单词字段再建立一个表,采用B+树[6-7]存储。如此可以大幅降低单词匹配的耗时。
改进的算法主要在于对序列数据库存储方式的改进。首先把数据库序列进行编号,如果序列长度过大,可能还要分段。在此,规定长度大于L=250的数据库序列都进行分段,使长度大小不超过L。设编号总数为N,即该数据库有N段。然后对于每一个可能的单词,都根据数据库生成一个长度为N的位向量。位向量的生成方法是查找数据库的每一段,如果第i条有该单词的命中,则对应位向量第i个元素为1,否则记0。
以表1的DNA数据库作为例子。这个数据库只包含3条序列,而且每条长度为60,因此不需要对序列进行分段。
首先对数据库按序列进行编号。这里片段总数N=3。然后第1个DNA段第1个长度为W的片段TACTTGCCAAG,其在第1个DNA段有1个命中,因此位向量第1个元素为1。对于第2个DNA段,片段TACTTGCCAAG并没有命中,因此位向量第2个元素为0。依此类推,片段TACTTGCCAAG的位向量为100。然后对于这个DNA段的第2个长度为W的片段ACTTGCCAAGT,因为它在第3个DNA段有命中,因此这个单词位向量第3个元素置1。可得出这个片段的位向量为101。依此类推,遍历整个数据库之后,可以得出一张“单词-位向量”表。
为了更快地进行查找。可以为这张单词-位向量表进行进一步的索引。例如以“单词”为键,建立B+树,树的叶子节点指向这个单词所对应的位向量。假如建立的B+树的一个块有255个指针,那么3层255阶的B+树能够有2553≈1.66×107个指向记录的指针。当BLAST数据库建立的索引表单词长度W=11时,上限单词数目为411≈4.2×106,小于上述B+树可提供指针数目,因此只需要建立一个三层B+树就能够进行索引了。

表1 DNA序列数据库
当BLAST根据查询序列生成单词表时,给出任意一个长度为W的单词,可以根据B+树找到单词对应的位向量,然后利用位向量知道数据库第几条DNA段有命中。从而快速地找到命中的序列。

图2 三阶B+树
4 算法分析
假定B+树每一个块都是充满的(事实上,因为当W=11时单词数目的上限小于叶节点指针数,因此这个B+树是不可能充满的),那么给出一个单词要找到其在B+树对应的键时,程序需要从根节点开始逐层往下比较,每层都顺序比对所有的键,平均比对次数为255/2次,一直到叶节点所在的第三层,共需比对255/2×3=382.5次。
找到对应位向量之后,程序只需读入位向量一次,然后根据位向量查找命中的序列段。每在位向量中发现一个1,则说明有一条序列内部有命中。然后遍历这一段序列,找到所有的命中。设数据库有N个序列段,每个序列段长度最大值为L,假定数据库每个序列段的长度都达到最大,从而对于每一个有命中的序列段,程序都需要比对(L-W+1)次以找出命中的具体位置。
又设查询序列能够生成v个单词,每个单词在数据库中有h个序列段是有命中的,那么平均每个单词查找命中需要比对的次数为:
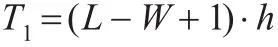
这里的h可以用任意长度为W的单词在数据库有命中的序列段个数的期望近似代替,即:h≅H=N·P。其中P是任意长度为W的单词在数据库的任意一个序列段有命中的概率近似估计(因为不同单词在一个随机序列出现的概率不同,因此这里计算概率时不考虑重复情况只给出概率上限):

因此平均每个单词需进行比对的次数可估计为:

而查询序列生成v个单词,两步共需比对次数为:
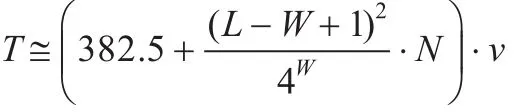
因为数据库的序列条数通常十分巨大,因此可以省略382.5,同时对于较大的L值,W值通常比较小。因此T可以近似估计为:

这里N与L的乘积实际上就是数据库的规模,即C=N×L,则上式可化简为:

把式(3)与式(1)比较,可以得出T与T0的关系:

取W=11,L=250,则 β与v有关,可以绘出 β(v)的图像如图3。
由图3可知随着查询序列长度的增长原算法与改进算法耗时比在降低,即对于较长的查询序列,改进算法的效果将会变差,不过对于DNA序列,查询序列的长度一般小于1 000,根据图像可得 v<1 000时 β(v)>100,即原算法耗时T至少为改进算法耗时T0的100倍,换句话说,执行任何一次搜索的时候,算法查询命中所耗费时间将变为原来的1/100。
除此之外,只读入有命中的序列段而不是全部读入整个序列数据库将会节省大量的时间。另外,为了更快地进行检索,还可以采用把整个B+树索引载入内存,这样只需要一次的磁盘I/O就能找到单词所对应的位向量。
5 速度提升的代价
事实上,为序列数据库建立位图索引将会需要大量空间用于存储单词-位向量表,即使采用压缩的算法[8]存储位向量,根据对几个生物数据库的建表实现的结果,建立单词-位向量表时最好预留30倍原文件大小的空间。也即是说,对于一个大小为1 GB的序列数据库,至少需要30 GB的硬盘空间用于存放改进的数据库。而这正是速度提升的代价所在。
不过鉴于现今PB级的存储设备已相当普及,相对于为了加快搜索速度而增添计算资源,采购大容量存储设备反而更加划算,因此这个代价是完全可以承受的。
另一个速度提升的代价在于对序列数据库的预处理时间,易知如果需要为一个序列数据库建立位图索引,将会需要大量的时间用于建立单词-位向量表。不过在实际应用中,对于一个序列数据库来说,建表将会是一次性的。因此一旦建表完成,之后的任何一次序列搜索将都会获得速度的提升。而B+树的特性又能够使得对数据库的日常维护变得十分容易,例如,当要修改某条序列时,只需要考虑受到影响的单词的位向量即可。
6 结束语
本文分析了BLAST算法其在播种阶段的计算步骤,并提出采用位图索引和B+树重新组织数据库的方法提高计算速度。采用位图索引的思想对序列数据库进行处理,生成单词-位向量表,在使用B+树进行二次索引,从而改变BLAST在播种阶段的处理方式。理论证明,新的算法能够使BLAST查找命中耗费的时间至少缩小为原算法的百分之一。
但还有一些问题尚待进一步研究,例如改进序列数据库的存储效率以及改进算法实现两个问题,这将是下一步研究的重心。
[1]Altschul S,Gish W.Basic local alignment search tool[J].Journal of Molecular Biology,1990,215:403-410.
[2]Altschul S,Madden T,Schäffer A.Gapped BLAST and PSIBLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[3]Korf I,Yandell M,Bedell J.BLAST[M].America:O’Reilly Media,2003.
[4]Bayer R,McCreight E M.Organization and maintenance of large ordered indexes[J].Acta Informatica,1972,1:173-189.
[5]Comar D.The ubiquitous B-tree[J].Computing Surveys,1979,11(2):121-137.
[6]O’Neil P.Model 204 architecture and performance[C]//Proceedings of the 2nd International Workshop on High Performance Transaction Systems,Asilomar,CA,USA,1987:40-59.
[7]O’Neil P,Quass D.Improved query performance with variant indexes[C]//SIGMOD’97.New York:ACM Press,1997:38-49.
[8]Wu K,Otoo E,Shoshani A.Optimizing bitmap indices with efficient compression[J].ACM Transactions on Database Systems,2006,31(1):1-38.
基于位图索引和B+树的BLAST改进算法
黄志洪1,吕 威2,黄 俊1
HUANG Zhihong1,LV Wei2,HUANG Jun1
1.School of Mathematics&Computational Science,Sun Yat-sen University,Guangzhou 510275,China
2.School of Information Technology,Beijing Normal University Zhuhai Campus,Zhuhai,Guangdong 519085,China
It concentrates on the time consuming procedure that goes through the database in the first step of BLAST.In order to speed up the program,it introduces a new approach which using bit map index and B+tree.The developed method builds up a word-bit_vector table according to the database,and reorganizes it with B+tree.It proves theoretically that it decreases the word searching time of BLAST substantially.
sequence alignment;BLAST algorithm;bitmap index;B+tree
针对BLAST算法在查找命中的过程中需要遍历数据库造成计算资源消耗的问题,提出了基于位图索引和B+树的数据存储方式以加快数据的检索。改进算法利用位图索引的原理建立数据库的单词-位向量表,并对这个表使用B+树再次进行索引,最终达到加快BLAST程序的运算速度。对于DNA序列这个方法能够使BLAST查找命中耗费的时间得到极大的减少。
序列比对;BLAST算法;位图索引;B+树
A
TP391
10.3778/j.issn.1002-8331.1110-0392
HUANG Zhihong,LV Wei,HUANG Jun.Improved BLAST algorithm based on bitmap indexes and B+tree.Computer Engineering and Applications,2013,49(11):118-120.
国家自然科学基金(No.11126039)。
黄志洪(1967—),男,讲师,主要研究领域为数据库、数据分析。E-mail:luwei00@126.com
2011-10-20
2012-02-06
1002-8331(2013)11-0118-03
CNKI出版日期:2012-07-19 http://www.cnki.net/kcms/detail/11.2127.TP.20120719.1511.006.html

