黄鲫线粒体DNA控制区结构及长度多态性分析
蔡珊珊徐胜勇宋 娜高天翔张朝晖
(1. 中国海洋大学海洋生物多样性与进化研究所, 青岛 266003; 2. 国家海洋局第一海洋研究所, 青岛 266061)
黄鲫线粒体DNA控制区结构及长度多态性分析
蔡珊珊1徐胜勇1宋 娜1高天翔1张朝晖2
(1. 中国海洋大学海洋生物多样性与进化研究所, 青岛 266003; 2. 国家海洋局第一海洋研究所, 青岛 266061)
鱼类线粒体DNA(Mitochondrial DNA, mtDNA)因为具有结构简单、严格的母系遗传、缺少重组等特点而成为重要的分子标记[1—4]。线粒体DNA控制区(Control region, CR, 又称D-loop区)是一段非编码区, 进化速率快, 容易积累较多的变异(如碱基的替换、插入和缺失等), 被广泛应用于种内分子遗传学研究[5—7]。控制区一般分为终止序列区、中央保守区和保守序列区3部分。控制区变异多发生于终止序列区, 中央保守区和保守序列区相对保守,且存在多个保守片段(CSB-F、CSB-E、CSB-D、CSB-1、CSB-2、CSB-3等)[7]。目前已分析研究了多种鱼类的线粒体DNA控制区结构[7—15], 并总结归纳出控制区各保守片段的普遍形式[7]。在部分鱼类线粒体DNA控制区中亦发现长度多态性现象[7,14,15], 控制区长度多态性可能应用于种内或种间群体学研究[16]。
黄鲫(Setipinna tenuifilis)隶属鲱形目(Clupeiformes)、鳀科(Engraulididae)、黄鲫属, 广泛分布于印度洋西部和西北太平洋海域, 中国沿海各海域均有分布。黄鲫为近海中下层鱼类, 喜栖息于泥沙底、水流较缓的海区[17,18]。黄鲫是一种小型经济鱼类。20世纪80年代以来, 随着小黄鱼、蓝点马鲛、带鱼等传统经济鱼类资源量下降, 黄鲫的捕获量逐年增加[18]。由于捕捞压力过大, 黄鲫资源量已呈现下降趋势[19—21]。目前对黄鲫的研究主要集中在时空分布、种群结构和生物学等方面[18—22]。Li等[23]和张博[24]对黄鲫控制区序列进行分析研究, 识别了CSB-F、CSB-E、CSB-D、CSB-1、CSB-2、CSB-3, 并在终止序列区内发现重复序列现象。本研究在分析黄鲫线粒体DNA控制区结构的基础上, 对控制区长度多态性进行统计分析, 以期为黄鲫群体遗传学研究奠定基础, 同时探讨重复序列应用于黄鲫群体遗传学分析的有效性。
1 材料与方法
1.1 实验材料
本研究所用黄鲫样品于2005—2013年采自中国沿海7个地点(东营、烟台、青岛、南通、温州、厦门、北部湾), 共202尾个体(表1)。在实验样品经形态学鉴定后, 取小块肌肉组织于 95%酒精溶液中固定, 置于–20℃冰箱中保存待用。
1.2 DNA提取及PCR扩增
取肌肉组织约100 mg经蛋白酶K消化后, 使用标准酚-氯仿法抽提获得线粒体基因组DNA。使用100 mL去离子水溶解线粒体基因组 DNA, 置于–4℃冰箱中保存待用。每个群体取5尾个体扩增控制区全序列用于控制区结构分析, 其他个体仅扩增控制区高变区(Hypervariable region I)用于长度多态性分析。

表1 黄鲫样品信息Tab. 1 Sample information of Setipinna tenuifilis
黄鲫控制区全序列扩增分两段测序, 其中控制区高变区引物[25]为: HJF: 5′-CACCCYTRRCTCCCAAAGCYA-3′; HJR: 5′-GGAACACCGAGTAATGCTGAG-3′; 控制区后半段引物为: HJHF: 5′-CATTTTCTATGCGTTCCTCAG-3′; HJHR: 5′-GTGCGGATACTTGCATGTGT-3′。
PCR反应体系为25 μL, 其中Taq酶0.25 μL, DNA模板1 μL, 正、反向引物各1 μL, dNTP 2 μL, 10×PCR buffer 2.5 μL, 去离子水17.25 μL。PCR反应条件为94℃预变性5min; 94℃变性45s, 52℃退火45s, 72℃延伸45s, 共35个循环; 72℃延伸10min。取2 μL PCR产物进行1.5%琼脂糖凝胶电泳(U = 300 V), 使用回收试剂盒(上海沃森生物科技公司)对目的条带进行回收纯化, 使用 ABI Prism 3730型DNA序列分析仪对回收纯化的PCR产物进行正反链测序。
1.3 数据分析
使用 DNASTAR软件包(DNASTAR, Inc., Madison, USA)对序列进行比对、编辑和分析, 并对结果辅以人工校正; 使用Microsoft Excel 2010软件对重复序列频率进行统计分析; 使用RNAstructure 4.3软件[26]分析重复序列二级结构并计算其释放的自由能; 使用SPSS 18.0软件对重复序列频率进行卡方检验(Chi-square test), 以计算两两群体间重复序列频率的差异显著性。
按照Birky等[27]的理论, 计算不同层次遗传多样性。由于黄鲫控制区DNA不存在个体内异质性现象, 个体内遗传多样性 Kb= 0; 群体内个体间遗传多样性 Kc= 1–ΣXi2, Xi指某一DNA类型(长度差异)在群体内的频率; 总体遗传多样性Kt= 1–ΣYi2, Yi指某一DNA类型(长度差异)在所有个体内的频率。根据公式Cip= (mean Kc–Kb)/Kt和Cpt= (Kt–mean Kc)/Kt计算遗传差异在群体内和群体间所占比例[28]。
2 结果
2.1 控制区序列长度和碱基组成
对黄鲫控制区序列进行编辑和人工校正后, 得到黄鲫全序列长度为1193—1271 bp。35条序列共定义了6个单倍型, 检测到9个变异位点, 其中9个位点均为转换。以CSB-F起始位置和CSB-1起始位置分别为终止序列区和中央保守区、中央保守区和保守序列区的界线, 8个变异位点位于终止序列区内, 1个变异位点位于中央保守区,在保守序列区内未发现变异位点。同时, 在终止序列区内(165—437 bp)检测到串联重复序列(Tandem repeat sequence)结构, 重复序列单元长度为 39 bp, 重复次数为 5—7次。重复序列现象仅存在于个体间, 并没有发现个体内异质性(Heterogeneous)现象。
以长度为1271 bp的控制区序列为例分析序列中碱基A、T、G、C的含量(表2)。控制区全序列A+T含量(64.9%)高于 C+G含量, 这一结果与其他鱼类控制区碱基组成相似。重复序列单元 A+T含量(79.5%)均高于全序列含量(64.9%)、终止序列区含量(72.4%)、中央保守区含量(57.1%)和保守序列区(55.9%)。

表2 黄鲫控制区序列碱基组成及长度Tab. 2 Nucleotide compositions and length of mitochondrial DNA control region of S. tenuifilis
2.2 终止序列区
终止序列区是控制区内变异最多的区域。黄鲫控制区终止序列区长度为592—670 bp, 在终止序列区前端发现串联重复序列。参考鲚属鱼类线粒体DNA控制区扩展终止相关序列(Extended termination associated sequence, ETAS)特征序列[9]和 鲿科鱼类ETAS特征序列[13], 确定黄鲫控制区ETAS特征序列为TACAT ACTATGCATTATATT ACAT, 与刘焕章[7]对ETAS的描述略有不同, 与Li等[23]的研究结果相似(图 1), 且 ETAS包含在串联重复单元中,黄鲫控制区终止序列区内存在5—7个ETAS。

图1 黄鲫控制区终止序列区序列Fig. 1 Termination associated sequence of S. tenuifilis
2.3 中央保守区
中央保守区内含有多个保守序列。参照刘焕章[7]给出的 鱼类中央保守区D、E、F 3个保守片段的特征序列及鲚属鱼类中央保守区D、E、F 3个保守片段的特征序列[9], 在黄鲫控制区中央保守区内共识别出CSB-D、CSB-E、CSB-F 3个保守序列。其中CSB-F序列为ATGTAG TA AGAAACCACC, CSB-E序列为AGGG ACAACTATTGTGGGGGACTG GCATCTG , CSB-D 序列为 TATT
GT, 与Li等[23]的研究结果相似(图2)。CSB-E序列中发现 GTGGG-box, 且与鲚属鱼类 CSB-E序列相同, 其他2个序列与鲚属鱼类特征序列分别存在1个碱基差异[9]。
2.4 保守序列区
参照鲚属鱼类的CSB-1、CSB-2和CSB-3的特征序列, 我们识别出黄鲫的3个保守序列区片段。CSB-1的序列为TTGATAGAAGAATTGCATAA; CSB-2和CSB-3的序列分别为AAACCCCCTTACCCCC和TGTCAAACCCC GAAA, 与Li等[23]的研究结果相似(图2)。

图2 黄鲫控制区中央保守区和保守序列区序列Fig. 2 Central conserved domain and conserved sequence block sequence of S. tenuifilis
2.5 重复序列
对202尾黄鲫个体的控制区高变区序列进行3%琼脂糖凝胶电泳(U = 100V), 发现个体间存在长度多态性现象(图 3)。对黄鲫控制区高变区序列进行正反链测序, 测序结果显示黄鲫控制区高变区存在长度为39 bp的重复序列单元, 重复次数为 5—7次。结合琼脂糖凝胶电泳图及测序结果, 统计黄鲫控制区重复序列单元频率, 结果显示 6次重复的个体最多, 其次为7次重复个体, 5次重复个体最少, 仅出现在东营、烟台、南通和温州群体内; 北部湾群体内7次重复个体较多, 其他6个群体均为6次重复个体比例较大(图4)。在重复序列频率上, 卡方检验结果(表3)显示北部湾群体与其他 6个群体存在极显著的差异(χ2>16.655, P=0.000), 其他各群体间差异不显著(χ2<2.945, P > 0.229)。
使用RNAstructure软件分析、构建重复序列二级结构, 发现重复序列能够形成稳定的茎环结构并释放一定的自由能(图5), 5次重复序列释放的自由能为–24.2 kcal/M; 6次重复序列释放的自由能为–28.7 kcal/M; 7次重复序列释放的自由能为–34.2 kcal/M。随着重复次数的增加, 重复序列形成的二级结构越来越稳定, 释放的自由能也越来越高。
对黄鲫控制区重复序列遗传多样性和群体遗传差异进行分析比较, 结果显示 7个群体遗传多样性(Kc)为0.255—0.423, 平均为 0.347; 所有个体遗传多样性为0.435。79.77%的遗传差异来自群体内个体间, 而20.23%的遗传差异来自群体间(表4)。

图3 黄鲫控制区高变区长度多态性(以东营群体为例)Fig. 3 Length polymorphism in hypervariable region I of S. tenuifilis in population Dongying

表3 两两群体间卡方检验结果(对角线下侧为χ2值, 对角线上侧为P值)Tab. 3 Pairwise χ2(below diagonal) and P values (above diagonal) among S. tenuifilis populaitons

图4 黄鲫7个群体重复序列频率Fig. 4 Frequency of tandem repeat sequence in seven populations of S. tenuifilis
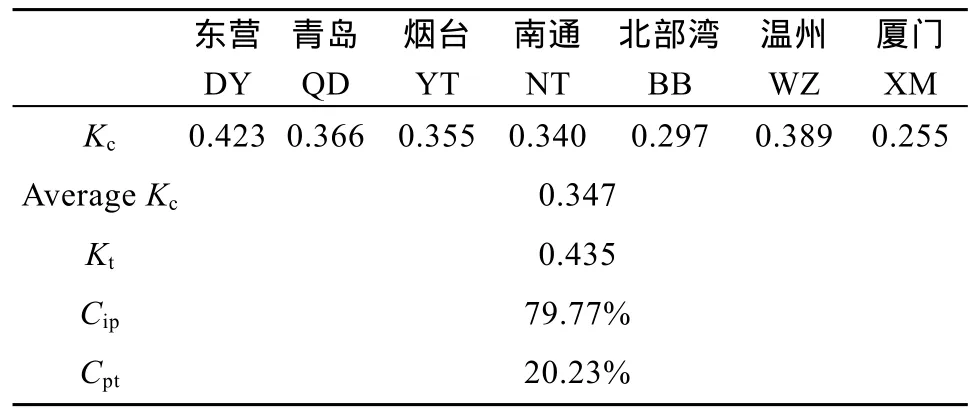
表4 黄鲫群体重复序列遗传多样性和遗传差异分析结果Tab. 4 Results of genetic diversity and genetic differentiation of seven populations of S. tenuifilis
3 讨论
3.1 黄鲫控制区结构
黄鲫控制区结构与其他海洋鱼类相似, 通过比对其他鱼类的控制区结构, 我们发现在黄鲫控制区终止序列区内存在5—7个扩展终止相关序列(ETAS), 这一重复片段也是造成黄鲫控制区长度多态性的主要原因。同时, 在中央保守区和保守序列区内分别识别6个保守序列(CSBD—F和CSB-1—3)。
终止序列区是鱼类控制区内变异积累最多的区域,重复序列也多发生在终止序列区。朱世华等[13]发现在康氏似 鲹(Scomberoides commersonianus)的控制区终止序列区内存在串联重复序列, 在中华鲟[14](Acipenser sinensis)、高首鲟[29](Acipenser transmontanus)控制区终止序列区内也发现重复序列, 且在康氏似 鲹和高首鲟控制区内存在多个 TAS。黄鲫控制区结构分析的研究中, 张博[24]在终止序列区内发现5个ETAS, Li等[23]发现7个ETAS。本研究中, 黄鲫控制区ETAS出现频率为5—7次, 呈现个体间长度多态性。
中央保守区是整个控制区中最为保守的区域[30],Southern等[31]识别出哺乳动物中央保守区B、C、D、E、F 5个保守序列; Randi和Lucchini[32]识别了鸟类中央保守区C、D、E、F 4个保守序列。但在多种鱼类[7—13]中仅识别3个保守片段(CSB-D、E、F)。对黄鲫的中央保守区进行分析, 也仅识别了 CSB-D、E、F等保守片段, 并没有发现CSB-B和CSB-C序列。

图5 重复序列二级结构及自由能Fig. 5 Predicted secondary structure and free energy of tandem repeat sequences
保守序列区可能是控制区中最为重要的区域, 其包含有H链复制起始区(OH)、H链启动子(HSP)、L链启动子(LSP)和 3个保守片段(CSB-1—3)等多个功能区域[30],其中 CSB-1是区分中央保守区和保守序列区的标志, 其变异较大, 而 CSB-2和 CSB-3相对保守[7]。现已识别出多种鱼类保守序列区的保守片段并给出各保守片段的一般形式[7—15]。在黄鲫保守序列区内并没有发现CSB-1的特征序列(GACATA), 参照鲚属鱼类等的CSB-1特征序列,我们发现黄鲫的CSB-1序列为TTGATAGAAGAATTGC ATAA。CSB-2和CSB-3序列相对保守。
3.2 黄鲫控制区重复序列
黄鲫控制区终止序列区出现多个串联重复序列, 且个体间重复次数不同。串联重复序列形成机制较多, 如重组(Recombination)和转座(Transposition)[16]、不等交换(Unequal crossing over)或基因转换(Gene conversion)[33]、滑链(Strand slippage)[34]等。由于脊椎动物线粒体DNA中一般不发生重组现象[35], 滑链错配(Slipped-strand mispairing)是最有可能形成串联重复序列的机制。这一机制在核基因中也同样存在[36,37]。目前仍需要深入研究以确定决定重复次数频率的机制。
重复序列频率差异可能应用于群体比较和种类鉴别。Stepien等[38]对梅花鲈(Gymnocephalus cernua)重复序列多态性进行分析研究, 发现梅花鲈各地理群体间重复序列频率差异显著; Broughton和 Dowling[39]对斑鳍真小鲤(Cyprinella spiloptera)重复序列进行统计分析, 发现其地理群体间重复序列频率存在显著差异; Cesaroni等[40]对2个群体的欧洲鲈鱼(Dicentrarchus labrax)的线粒体重复序列进行分析, 发现串联重复序列频率能够较好的将 2个群体区分开; 但Miracle和Campton[41]对尖吻鲟(Acipenser oxyrhynchus desotoi)的线粒体重复序列进行分析, 没有发现重复序列频率与地理分布存在相关性。Li等[23]利用线粒体DNA控制区序列对中国近海5个黄鲫群体进行遗传学分析, 结果显示东海海域与黄海海域黄鲫群体间不存在显著的遗传差异; 张博[24]利用微卫星标记方法对中国近海5个黄鲫群体进行分析, 发现东海海域与黄海海域黄鲫群体间存在显著的遗传分化; Xu等[25]利用线粒体 DNA控制区序列对中国近海 7个黄鲫群体进行遗传学分析,发现中国沿海黄鲫群体可分为黄渤海组群、东海组群和南海组群, 各组群间存在较大的遗传差异。在本研究中, 对黄鲫控制区重复序列频率进行统计分析, 卡方检验结果显示北部湾群体与其他 6个群体间差异极显著, 这一结果与Xu等[25]的研究结果相似, 佐证了南海组群与其他组群存在遗传差异, 但并未发现东海海域群体与黄渤海海域群体间的遗传差异。仅通过重复序列频率不能将北部湾群体与其他群体完全区分, 重复序列频率可能作为一种补充和辅助手段用于群体遗传学研究。
致谢:
感谢孙典荣研究员、李龙、李渊为本研究采集黄鲫样品!
[1] Buonnacorsi V P, McDowell J R, Graves J E. Reconciling patterns of inter-ocean molecular variance from four classes of molecular markers in blue marlin (Makaira nigricans) [J]. Molecular Ecology, 2001, 10(5): 1179—1196
[2] Domingues V S, Faria C, Stefanni S, et al. Genetic divergence in the Atlantic-Mediterranean Montagu’s blenny, Coryphoblennius galerita (Linnaeus 1758) revealed by molecular and morphological characters [J]. Molecular Ecology, 2007, 16(17): 3592—3605
[3] Engelbrecht C C, Freyhof J, Nolte A, et al. Phylogeography of the bullhead Cottus gobio (Pisces: Teleostei: Cottidae) suggests a pre-Pleistocene origin of the major central European populations [J]. Molecular Ecology, 2000, 9(6): 709—722
[4] Whitehead A, Anderson S L, Kuivila K M, et al. Genetic variation among interconnected populations of Catostomus occidentalis: implications for distinguishing impacts of contaminants from biogeographical structuring [J]. Molecular Ecology, 2003, 12(10): 2817—2833
[5] Birt T P, Green J M, Davidson W S. Mitochondrial DNA variation reveals genetically distinct sympatric populations of anadromous and nonanadromous Atlantic salmon, Salmo salar [J]. Canadian Journal of Fisheries and Aquatic Sciences, 1991, 48(4): 577—582
[6] Xiao W H, Zhang Y P. Genetics and evolution of mitochondrial DNA in fish [J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384—391 [肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化. 水生生物学报, 2000, 24(4): 384—391]
[7] Liu H Z. The structure and evolution of mitochondrial DNA control region of fish: focus on Acheilognathinae fish [J]. Progress in Natural Science, 2002, 12(3): 266—270 [刘焕章.鱼类线粒体DNA控制区的结构和进化: 以鱼类为例.自然科学进展, 2002, 12(3): 266—270]
[8] Zhu S H, Zheng W J, Zou J X, et al. Mitochondrial DNA control region structure and molecular phylogenetic relationship of Carangidae [J]. Zoological Research, 2007, 28(6): 606—614 [朱世华, 郑文娟, 邹记兴, 等. 鲹科鱼类线粒体DNA控制区结构及系统发育关系. 动物学研究, 2007, 28(6): 606—614]
[9] Zhu T J, Yang J Q, Tang W Q. MtDNA control region sequence structure of the genus Coilia in Yangtze River estuary [J]. Journal of Shanghai Fisheries University, 2008, 17(2): 152—157 [诸廷钧, 杨金权, 唐文乔. 长江口鲚属鱼类线粒体 DNA控制区结构分析. 上海水产大学学报, 2008, 17(2): 152—157]
[10] Xie Z Y, Du J Z, Chen X Q, et al. The significance of mitochondrial control region (D-loop) in intraspecific genetic differentiation of fish [J]. Hereditas, 2006, 28(3): 362—368 [谢振宇, 杜继增, 陈学群, 等. 线粒体控制区在鱼类种内遗传分化中的意义. 遗传, 2006, 28(3): 362—368]
[11] Liu H L, Zhang Q, Tang Y L, et al. Structure and genetic diversity of mtDNA D-Loop sequences among Trachidermus fasciatus stocks in Yellow Sea and Bohai Sea of China [J]. Marine Science Bulletin, 2010, 29(3): 283—288 [刘海林,章群, 唐优良, 等. 黄渤海松江鲈鱼线粒体控制区结构与序列多态性分析. 海洋通报, 2010, 29(3): 283—288]
[12] Tan W, Guo Y S, Wang Z D, et al. Structure of the mitochondrial DNA control region of snapper species and their phylogenetic relationship [J]. Acta Oceanologica Sinica, 2010, 32(1): 139—145 [谭围, 郭昱嵩, 王中铎, 等. 笛鲷鱼类的线粒体DNA控制区结构及其系统发育分析. 海洋学报, 2010, 32(1): 139—145]
[13] Zhang Y, Zhang E, He S P. Studies on the structure of the control region of the bagridae in China and its phylogenetic significance [J]. Acta Hydrobiologica Sinica, 2003, 27(5): 463—467 [张燕, 张鹗, 何舜平. 鲿中国 科鱼类线粒体DNA控制区结构及其系统发育分析. 水生生物学报, 2003, 27(5): 463—467]
[14] Zhang S M, Deng H, Wang D Q, et al. Mitochondrial DNA length variation and heteroplasmy in Chinese sturgeon (Acipenser sinensis) [J]. Acta Genetica Sinica, 1999, 26(5): 489—496 [张四明, 邓怀, 汪登强, 等. 中华鲟(Acipenser sinensis)mtDNA个体间的长度变异与个体内的长度异质性. 遗传学报, 1999, 26(5): 489—496]
[15] He C B, Cao J, Liu W D, et al. Structure analysis of mtDNA control region of spotted halibut (Verasper variegatus) and its related species [J]. Hereditas, 2007, 29(7): 829—836 [赫崇波, 曹洁, 刘卫东, 等. 圆斑星鲽及相关种类线粒体DNA控制区结构分析. 遗传, 2007, 29(7): 829—836]
[16] Rand D M, Harrison R G. Molecular population genetics of mtDNA size variation in crickets [J]. Genetics, 1989, 121(3): 551—569
[17] Cheng Q T, Zhou C W. The Fishes of Shandong Province [M]. Jinan: Shandong Science and Technology Press 1997, 81 [成庆泰, 周才武. 山东鱼类志. 济南: 山东科学技术出版社. 1997, 81]
[18] Gu H. Feeding habits and food composition of scaly half-fin anchovy, Setipinna taty (C et V) in the Bohai Sea [J]. Chinese Journal of Oceanology and Limnology, 1990, 8(3): 280—289
[19] Guo B, Zhang B, Dai F Q, et al. Diet composition and ontogenetic variation in feeding habits of juvenile Setipinna taty in the Haizhou Bay [J]. Journal of Fisheries of China, 2010, 34(6): 921—927 [郭斌, 张波, 戴芳群, 等. 海洲湾黄鲫幼鱼的食性及其随叉长的变化. 水产学报, 2010, 34(6): 921—927]
[20] Liu Y, Cheng J H, Li S F. A study on the distribution of Setipinna taty in the East China Sea [J]. Marine Fisheries, 2004, 26(4): 255—260 [刘勇, 程家骅, 李圣法. 东海区黄鲫数量分布特征的分析研究. 海洋渔业, 2004, 26(4): 255—260]
[21] Xiong Y, Tang J H, Liu P T, et al. Resource estimate on Setipinna taty in the southern Yellow Sea [J]. Oceanologia et Limnologia Sinica, 2009, 40(4): 500—505 [熊瑛, 汤建华,刘培廷, 等. 黄海南部黄鲫(Setipinna taty)资源利用分析.海洋与湖沼, 2009, 40(4): 500—505]
[22] Guo A, Zhou Y D, Jin H W, et al. Seasonal changes on food composition and feeding habitat of Colilia mystus (Linnaeus)in the east China sea [J]. Modern Fisheries Information, 2010, 25(8): 10—13 [郭爱, 周永东, 金海卫, 等. 东海黄鲫的食物组成和食性的季节变化. 现代渔业信息, 2010, 25(8): 10—13]
[23] Li H, Xu T J, Cheng Y, et al. Genetic diversity of Setipinna taty (Engraulidae) populations from the China Sea based on mitochondrial DNA control region sequences [J]. Genetics and Molecular Research, 2012, 11(2): 1230—1237
[24] Zhang B. Analysis of genetic diversity in Setipinna taty germplasms [D]. Thesis for Master of Science. Zhejiang Ocean University, Zhoushan. 2013 [张博. 黄鲫分子标记开发及遗传多样性分析. 硕士学位论文, 浙江海洋学院, 舟山. 2013]
[25] Xu S Y, Song N, Lu Z C, et al. Genetic variation in scaly hair-fin anchovy Setipinna tenuifilis (Engraulididae) based on the mitochondrial DNA control region [J]. Mitochondrial DNA, doi: 10.3109/19401736.2013.845754
[26] Mathews D H, Disney M D, Childs J L, et al. Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure [J]. Proceedings of National Academy of Scicences of the United States of America, 2004, 101(19): 7287—7292
[27] Birky C W, Fuerst P, Maruyama T. Organelle gene diversity under migration, mutation and drift: equilibrium expectations, approach to equilibrium, effects of heteroplasmic cells, and comparisons to nuclear genes [J]. Genetics, 1989, 121(3): 613—627
[28] Lewontin R C. The apportionment of human diversity [J]. Evolutionary Biology, 1972, 6: 381—398
[29] Buroker N E, Brown J R, Gilbert T A, et al. Length heteroplasmy of sturgeon mitochondrial DNA: An illegitimate elongation model [J]. Genetics, 1990, 124(1): 157—163
[30] Saccone C, Pesole G, Sbisa E. The main regulatory region of mammalian mitochondrial DNA: structure-function model and evolutionary pattern [J]. Journal of Molecular Evolution, 1991, 33(1): 83—91
[31] Southern S O, Southern P J, Dizon A E. Molecular characterization of a clone dolphin mitochondrial genome [J]. Journal of Molecular Evolution, 1988, 28(1—2): 32—42
[32] Randi E, Lucchini V. Organization and evolution of the mitochondrial DNA control region in the avian genus alectoris [J]. Journal of Molecular Evolution, 1998, 47(4): 449—462
[33] Hoelzel A R, Hancock J M, Dover G A. Generation of VNTRs and heteroplasmy by sequence turnover in the mitochondrial control region of two elephant seal species [J]. Journal of Molecular Evolution, 1993, 37(2): 190—197
[34] Wilkinson G S, Chapman A M. Length and sequence variation in evening bat d-loop mtDNA [J]. Genetics, 1991, 128(3): 607—617
[35] Hayashi J I, Tagashira I, Yoshida M C. Absence of extensive recombination between inter and intra-species mitochondrial DNA in mammalian cells [J]. Experimental Cell Research, 1985, 160(2): 387—395
[36] Broughton R E, Dowling T E. Evolutionary dynamics of tandem repeats in the mitochondrial DNA control region of the minnow Cyprinella spiloptera [J]. Molecular Biology and Evolution, 1997, 14(12): 1187—1196
[37] Levinson G, Gutman G A. Slipped-strand mispairing: A major mechanism for DNA sequence evolution [J]. Molecular Biology and Evolution, 1987, 4(3): 203—221
[38] Stepien C A, Dillon A K, Chandler M D. Evolutionary relationships, phylogeography, and genetic identity of the ruffe Gymnocephalus in the North American Great Lakes and Eurasia from mtDNA control region sequences [J]. Journal of Great Lakes Research, 1998, 24: 361—378
[39] Broughton R E, Dowling T E. Evolutionary dynamics of tandem repeats in the mitochondrial DNA control region of the minnow Cyprinella spiloptera [J]. Molecular Biology and Evolution, 1997, 14(12): 1187—1196
[40] Cesaroni D, Venazetti F, Allegrucci G, et al. Mitochondrial DNA length variation and heteroplasmy in natural populations of the European sea bass, Dicentrarchus labrax [J]. Molecular Biology and Evolution, 1997, 14(5): 560—568
[41] Miracle A L, Campton D E. Tandem repeat sequence variation and length heteroplasmy in the mitochondrial DNA D-loop of the threatened Gulf of Mexico sturgeon, Acipenser oxyrhynchus desotoi [J]. Journal of Heredity, 1995, 86(1): 22—27
MITOCHONDRIAL DNA CONTROL REGION STRUCTURE AND LENGTH POLYMORPHISM ANALYSIS OF SETIPINNA TENUIFILIS
CAI Shan-Shan1, XU Sheng-Yong1, SONG Na1, GAO Tian-Xiang1and ZHANG Zhao-Hui2
(1. Institute of Evolution & Marine Biodiversity, Ocean University of China, Qingdao 266003, China; 2. The First Institute of Oceanography, State Oceanic Administration, Qingdao 266061, China)
黄鲫; 控制区; 结构; 串联重复序列; 长度多态性
Setipinna tenuifilis; control region; Structure; Tandem repeat sequence; Length polymorphism
Q346+.5
A
1000-3207(2014)05-0980-07
2014-04-09;
2014-06-13
国家海洋公益性行业科研专项经费项目(201305030, 201405010); 中央高校基本科研业务费专项资金项目(201262022)资助
蔡珊珊(1990—), 女, 山东潍坊人, 硕士研究生, 主要从事渔业生态学研究。E-mail: cssrsjzdw110@163.com
张朝晖(1970—), 男, 副研究员; E-mail: zhang@fio.org.cn

