基于分子描述符和机器学习方法预测和虚拟筛选乳腺癌靶向蛋白HEC1抑制剂
何 冰 罗 勇 李秉轲 薛 英,3 余洛汀,*邱小龙 杨登贵
(1四川大学华西医院生物治疗国家重点实验室, 肿瘤中心及生物治疗协同创新中心, 成都 610041;2成都师范学院化学与生命科学学院, 成都 611130; 3四川大学化学学院, 成都 610064;4江苏兆邦生物医药研究院有限公司, 江苏 南通 226000; 5江苏海门慧聚药业有限公司, 江苏 海门 226123)
基于分子描述符和机器学习方法预测和虚拟筛选乳腺癌靶向蛋白HEC1抑制剂
何 冰1,2罗 勇1李秉轲2薛 英1,3余洛汀1,*邱小龙4,5杨登贵4
(1四川大学华西医院生物治疗国家重点实验室, 肿瘤中心及生物治疗协同创新中心, 成都 610041;2成都师范学院化学与生命科学学院, 成都 611130;3四川大学化学学院, 成都 610064;4江苏兆邦生物医药研究院有限公司, 江苏 南通 226000;5江苏海门慧聚药业有限公司, 江苏 海门 226123)
HEC1(癌症高表达蛋白)是纺锤体检查点控制、着丝粒功能、细胞存活的关键的有丝分裂调节器, 与原发性乳腺癌的不良预后有关. 筛选具有高亲和力的HEC1新型抑制剂对探索乳腺癌的靶向治疗具有重要意义.本文从结构多样性的化合物库中筛选HEC1抑制剂. 通过对分子描述符的特征筛选, 采用支持向量机(SVM)和随机森林(RF)方法分别对HEC1抑制剂和非抑制剂建立了分类模型. 经对比, RF模型显示了更好的预测精度.我们采用RF模型对HEC1抑制剂进行了虚拟筛选, 从“in-house”实体库筛选得到2个潜在的HEC1抑制剂分子.随后对筛出的化合物进行了体外活性实验, 发现对乳腺癌细胞株MDA-MB-468和MDA-MB-231均有一定程度的抗肿瘤活性. 研究结果表明, 机器学习方法对于设计和虚拟筛选HEC1抑制剂有良好的效果.
HEC1; 选择性抑制剂; 机器学习方法; 支持向量机; 随机森林; 虚拟筛选
1 引 言
乳腺癌是女性癌症高发性恶性肿瘤, 全球范围内位于女性恶性肿瘤首位, 严重威胁女性身心健康.乳腺癌靶向治疗是在分子水平对其通路靶点设计药物, 通过药物与受体或调节分子结合, 下调受体表达或者活化下游基因, 使得肿瘤细胞凋亡或者抑制其生长. 目前乳腺癌常见靶向药物有人表皮生长因子受体(HER)靶向药物曲妥珠单抗(rastuzumab)、帕妥珠单抗(pertuzumab)、西妥昔单抗(cetuximab);还有小分子酪氨酸激酶抑制剂吉非替尼(gefitinib)、厄洛替尼(erlotinib)、拉帕替尼(lapatinib); 哺乳动物雷帕霉素靶蛋白靶向药物依维莫司(everolimus); 血管内皮生长因子(VEGF)靶向药物贝伐单抗(bevacizumab); 多聚二磷酸腺苷核糖聚合酶靶向药物抑制剂Olaparib已进入临床II期.1
HEC1是癌症高表达蛋白,2相对于正常细胞, 它在癌症细胞系中的表达量更高. HEC1也是一类周期蛋白, 主要在G2/M期表达, 参与有丝分裂、动粒组装、有丝分裂检验点以及染色体稳定性的维持等, 在肿瘤的发生发展中占有重要作用. 利用小分子抑制剂来研究HEC1在癌症中高表达的作用对于临床具有极大的意义.3
HEC1抑制剂相关机理已有不少研究.4–6Wu等7通过酵母双杂交筛选得到靶向Hec1/Nek2复合物的小分子抑制剂INH1, 该抑制剂通过抑制Nek2对Hec1的磷酸化从而破坏Nek2-Hec1通路, 另一方面在降低Nek2表达量同时并不降低Hec1表达量的前提下, 导致中期染色体排列错误, 细胞不能进行正常的有丝分裂, 最终导致细胞的死亡. 这在肿瘤治疗中可能具有重要意义. Qiu等8运用反向酵母双杂交系统筛选得到特异性阻断Hec1与Nek2蛋白–蛋白相互作用的小分子化合物, 该小分子能够特异性降低Hec1的磷酸水平, 能够导致染色体不稳定现象的出现. 目前报道的Hec1/Nek2的抑制剂主要是加州大学欧文分校研究的INH系列化合物.9–12
筛选得到对HEC1蛋白高亲和力、高选择性的小分子抑制剂, 特异性阻断Nek2对HEC1的磷酸化作用, 对于乳腺癌靶向治疗有重要意义. 机器学习方法在预测化合物的药效动力学、药代动力学和毒性等方面有非常好的效果.13–15本研究采用支持向量机(SVM)和随机森林(RF)两种机器学习方法, 通过内部五重交叉验证和袋外数据(OOB)估计对训练模型进行优化, 以期获得良好的预测结果. 随后, 我们从这两种模型中选取出与HEC1抑制剂相关的物理化学特征, 用于HEC1潜在抑制剂的虚拟筛选, 并对筛选出的已有化合物进行了体外活性测试.
2 材料与方法
2.1 分子描述符
分子描述符是对分子的结构和物理化学属性的定量描述符号, 广泛应用于定量结构活性关系(QSAR)和其他机器学习模型中, 用于预测各种药物及小分子的毒理学与药理学特征.16–22我们采用课题组编写的分子描述符计算程序, 采用AM1半经验方法对化合物三维(3D)结构进行优化, 然后分别计算了化合物189个描述符相对应的取值. 随后, 我们对SVM模型采用递归特征消除(RFE)方法, 对RF模型使用程序自带的随机选择方法, 剔除掉这189个描述符中与体系不相干的或多余的描述符, 仅保留和体系最相关的关键描述符, 用于进一步虚拟筛选或其它研究. 这两种特征选择方法将在后文中详细介绍. 本研究涉及的189个分子描述符组参见表S1(Supporting Information), 这189个描述符的详细说明请参见我们之前的文章.16–19,23
2.2 数据集
本研究所用的数据集为122个HEC1抑制剂和122个HEC1非抑制剂. HEC1抑制剂均从文献收集得到.8,24为保证所选出的HEC1抑制剂具备一定程度的高效性, 我们去除了HEC1抑制剂中的IC50值大于10 μmolL–1的分子, 确定HEC1抑制剂的IC50值范围为7.5 到9431 nmolL–1. 通过对以上范围抑制剂分子描述符的计算, 我们将具有重复结构的分子舍弃,最终确定了122个HEC1抑制剂加入到数据集中.
为充分表示HEC1非抑制剂的广泛性并作为对比, 我们根据k均值聚类25的方法, 从MDDR (MDL Drug Data Report)数据库中, 挑选出假定存在且对HEC1没有抑制作用的非抑制剂. 我们把MDDR中的未被报道具有任何HEC1抑制活性的分子(去除掉有无效的结构或分子描述符的分子, 剩下超过15万个分子), 根据计算得到它们的189个分子描述符的取值, 并分成122个聚类, 然后挑选出每个聚类中距离相应聚类的质心最近的分子, 即得到122个HEC1非抑制剂化合物.
对选定的244个分子, 均通过ChemBioDraw软件绘制得到其二维(2D)结构, 并用Corina软件将2D结构转换为3D结构, 再用ChemBio3D软件对3D结构进行优化.26,27根据这些分子在化学空间中的相似性和分布的广泛性, 我们将122个抑制剂和122个非抑制剂其分成HEC1的训练集和测试集. 训练集和测试集中化合物的结构、类别和来源参见表S2和S3 (Supporting Information).
2.3 机器学习方法
本研究采用了两种最新的机器学习方法——SVM和RF, 通过SVM和RF方法执行的预测可以归结为一个二元分类问题(即分为HEC1抑制剂或非抑制剂). 对于SVM和RF理论的深入描述可以很容易从一些经典著作和优秀论文中获得, 因此这里仅对SVM和RF分类的主要思想做一个简述.
SVM是一种基于统计学习理论中的结构风险最小化(SRM)原则的方法, 而统计学习理论是一种著名的与核函数相关的机器学习方法.28,29SVM方法通过使用核函数(本研究使用高斯径向基函数(RBF), 把输入变量投射到高维特征空间中, 然后从输入向量中选择一个所谓支持向量的小的子集, 在变换之后的空间中, 通过最大间隔的原则构建一个最优化的分类超平面, 从而把这些输入向量分成了两种不同的类别.30
RF方法则是一种决策树自然生长且很多个决策树预测器组合在一起的分类方法. 每棵决策树依赖于对输入向量进行随机独立抽样所获得的数值,且森林中的所有决策树都具有相同的分布.31,32每棵树都不受干涉地自然生长到最大的规模, 然后对于一个新的数据点给出自己的预测. 也就是说, 这颗树投票决定这一新数据点的类别. 当大量的决策树生成以后, 整个森林就选择最多数的投票结果作为对这个数据点类别的判定. 在对训练集的每次引导过程中, 大约三分之一的实例在训练过程遗漏而组成袋外数据估计(OOB)样本. 作为评估总预测精度指标的一部分, OOB在内部评估了RF的性能, 显示了使用相同大小的测试集具有和训练集同样的精确度, 是一种和经典的交叉验证相类似的新的评估方法.33,34此外, RF可以根据对变量值随机排序所引起的系统性能降低的程度, 选择出对模型预测能力相对重要的描述符.18尽管分子的某种特定描述符与其活性之间的关系被隐藏在一个“黑箱”中, RF模型仍然可以在训练过程中衡量每个描述符对于预测精度的贡献程度.35
2.4 特征选择方法和模型建立
引入RFE方法可以提高机器学习方法的分类性能, 并能筛选出一些最相关的描述符, 这些描述符可以把数据集区分为阴性和阳性两类. RFE在生物化学很多领域, 例如药代动力学相关的药物活性以及毒理学属性等,14,15,16,20显示出了高效性分类和预测能力, 并且发现很多有价值的特征, 获得了令人满意的推广. 因此, RFE和SVM相结合的方法(SVM + RFE)一开始就被应用于本研究中. 我们通过五重交叉验证方法, 从训练集的预测结果中分别选择了与HEC1抑制剂密切相关的一组描述符, 并使用RFE方法, 从整个训练集中产生一个有RFE特征的SVM模型.
对RF模型的每棵树, 均由随机选择的mtry个描述符和与训练集不同的自引导样本共同建立. RF模型的性能通过OOB估计从内部进行评估, OOB测试所选用的数据与构建森林所用到的数据互相独立.在RF的训练过程中, 描述符的相对重要性亦可通过其自带的特征选择程序计算得到.
2.5 性能评估
机器学习方法的预测效果可以通过真阳性(TP), 真阴性(TN), 假阳性(FP)和假阴性(FN)这四个指标来进行衡量. TP是预测正确的HEC1抑制剂的数量, TN是预测正确的HEC1非抑制剂的数量, FP是把HEC1的非抑制剂错误地预测成抑制剂的数量, FN是把HEC1的抑制剂错误地预测成非抑制剂的数量.
一些精确性函数可以用来衡量模型的预测效果, 例如灵敏度(SE, 即HEC1抑制剂的预测精度), 特异性(SP, 即HEC1非抑制剂的预测精度), 错误率(ER), 总预测精度Q, 马氏相关系数C等. 以上这些变量和函数之间的相互作用关系如下所示:

3 结果与讨论
3.1 模型建立与RFE方法的效果
SVM模型根据我们课题组发展的程序13–15建立,而RF模型则由 Breiman和Cutler36提出的Fortran代码生成. 在SVM模型中, 联合五重交叉验证方法, RFE方法的参与显著提高了训练集的预测精度, 详细结果参见表1.
在预测HEC1抑制剂和非抑制剂的过程中, 当SVM的参数σ被确定为5的时候, 模型表现出了最好的预测效果. 对于HEC1抑制剂和非抑制剂而言, 未使用RFE方法的SVM模型(记为SVM)的平均预测精度是95.83%和98.67%; 与此相比, 使用了RFE方法的SVM模型(记为SVM + RFE)的以上数值分别为100%和100%. "SVM"模型的Q和C的平均预测值分别为97.41%和0.9484, 而"SVM + RFE"模型的Q和C值则分别是100%和1. 从以上结果可以看出, 对于HEC1抑制剂和非抑制剂, Q和C这四项指标的平均预测精度而言, 结合了RFE方法的SVM模型比起单纯的SVM模型显示出了较明显的改善. 此外, 使用RFE方法还能有效选出一组与化合物的HEC1抑制活性最相关的分子描述符.
特征选择是通过随机地减少森林中树与树之间的相关度来提高RF模型的预测性能, 因此RF模型的每个节点在构建决策树的过程中使用了特征选择. 树的数量ntree经优化取值为500, 用于生成稳定的袋外数据估计(OBB)预测率.31参数mtry的取值范围是从1到描述符的总个数, 对于本研究1 ≤ mtry≤ 189, mtry的默认值一般设置为描述符总个数的平方根, 即13. 本研究对每个mtry值对应的模型都进行了预测,通过对相应的OOB预测错误率的比较, 选出使OOB预测错误率最小时对应的mtry值为最优值, 即mtry= 9的时候.
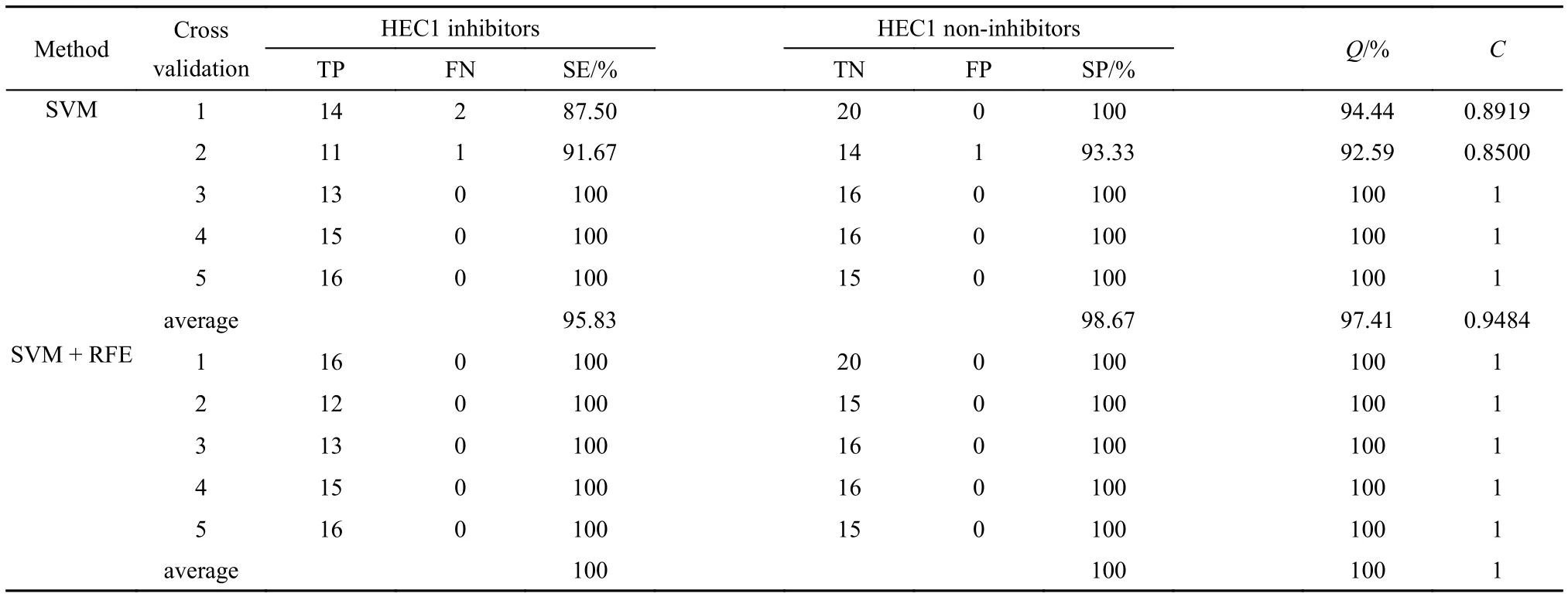
表1 SVM五重交叉验证预测结果Table1 Prediction performance of SVM by 5-fold cross-validation
我们通过测试集进一步评估所生成模型的效能和可靠性. 表2给出了HEC1体系中用RFE选出的描述符所生成的SVM模型对测试集的抑制剂和非抑制剂的预测精度. 作为对比, 使用全部189个描述符所生成的SVM模型相应的预测精度也列在表中.
在该体系中, “SVM”模型和“SVM + RFE”模型对非抑制剂的预测精度相同, 均为100%, 但是对于抑制剂而言, 使用RFE之后预测精度略微有所降低,对“SVM + RFE”模型为98%, 对“SVM”模型为100%.
根据表2数据显示, 这些模型中所采用的分子描述符数量减少, 模型的多余度和分子描述符的不相干性得到了极大程度的降低, 体系中的描述符从189个降到了12个. 表明RFE方法在选择最相关的特征和剔除掉多余分子描述符方面是高度有效的, 大大提高了HEC1体系的抑制剂和非抑制剂分类研究的效率.
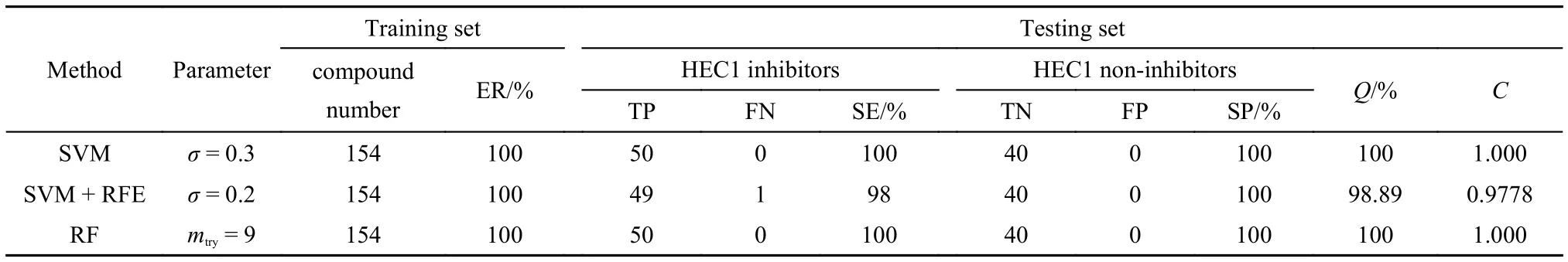
表2 预测精度的比较Table2 Comparison of the prediction accuracies
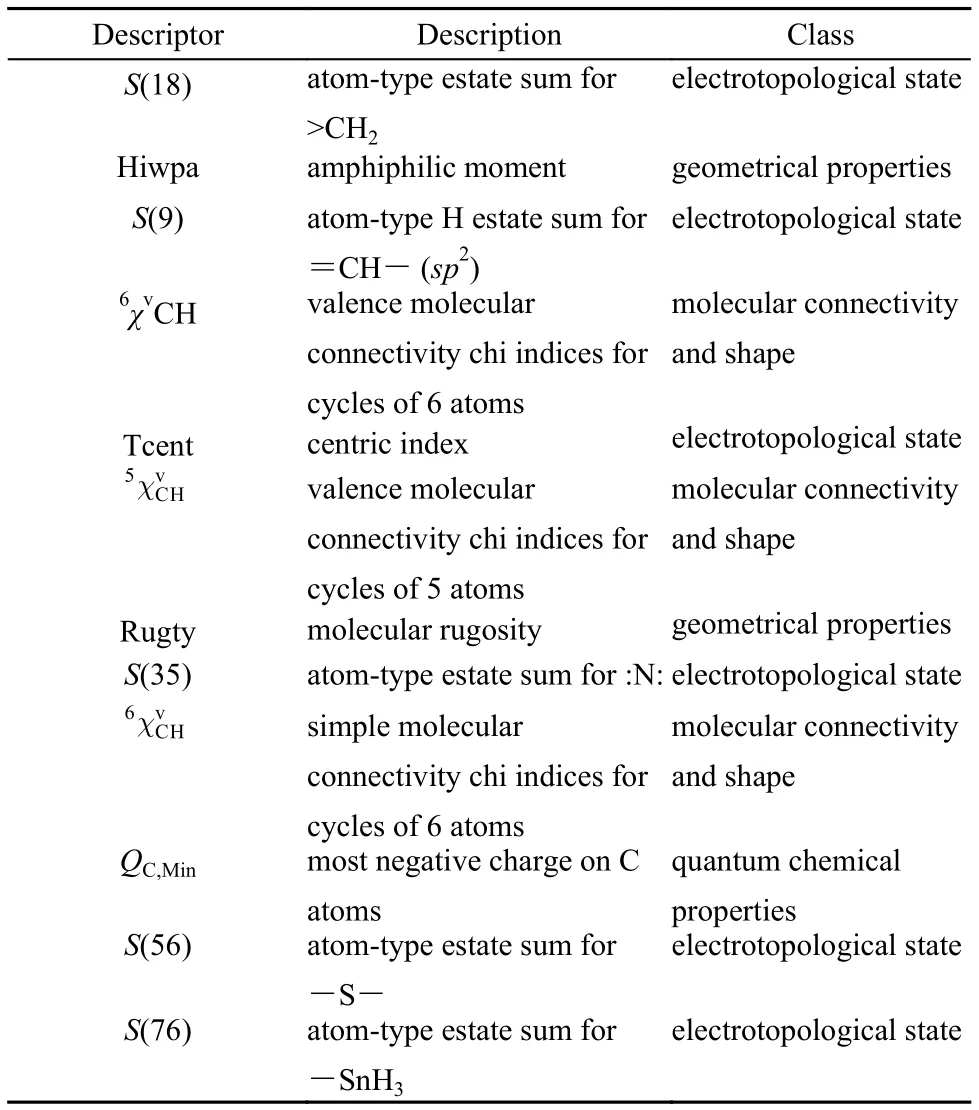
表3 SVM中SVM + RFE模型选择出的12个最相关的描述符Table3 12 descriptors selected by (SVM + RFE) model in the SVM
3.2 模型验证与机器学习方法的性能
表2给出了HEC1体系中SVM和RF方法对抑制剂和非抑制剂预测精度的详细比较, 特别是采用SVM方法所建立模型的参数值也在表中得到体现.对于“SVM + RFE”模型的参数σ = 0.2, 而对于“SVM”模型则为0.3. 相应的模型在这两个参数下都给出了最好的预测结果.
如表3所示, 对于HEC1体系, SVM中“SVM + RFE”模型对抑制剂的预测精度为98%, 对非抑制剂的则为100%, 而“SVM”模型和RF方法对抑制剂和非抑制剂的预测精度均为100%. 相应的, “SVM + RFE”模型的Q和C值分别为98.89%和0.9778, 而“SVM”模型的Q和C值和RF方法的相同, 分别是100%和1.
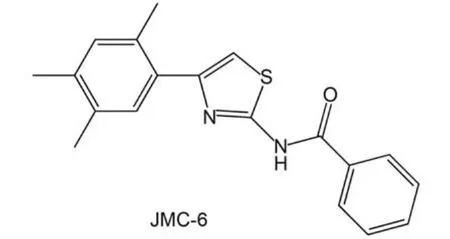
图1 SVM模型对测试集预测错误的HEC1抑制剂的结构Fig.1 Structures of the misclassified HEC1 inhibitors by using testing set in SVM model
由于完全正确的预测准确率, 使得RF模型和“SVM”模型中没有预测错误的抑制剂和非抑制剂分子. 而对于“SVM + RFE”模型, SVM的测试集中有1个抑制剂被预测错误, 该抑制剂的结构信息可以参见图1. 预测错误的原因可能和RFE方法的描述符较少(12个)造成的预测误差有关.
基于以上数据来看, 对于HEC1体系, 尽管“SVM”模型与RF模型具有同样优越的预测性能, 但是“SVM”模型拥有全部189个描述符, 相较于RF模型用自带的特征选择方法挑出的25个最相关描述符而言过于复杂和冗余. 此外, RF方法的计算速度要比SVM方法快的多, 抛开性能不论, 仅从时间和计算成本上考虑, 显然RF模型更利于后续的虚拟筛选工作.
3.3 最相关描述符与对模型的解释
在HEC1体系中, 通过特征选择, SVM和RF预测模型提取了与抑制剂和非抑制剂活性最相关的分子描述符. 部分描述符可为特定类别化合物相关的结构和物理化学属性提供更深层次的了解. 通过RFE方法的SVM模型, 体系分子描述符从189个选出12个(表3).
RF模型选定了25个(表4)与预测性能高度相关的分子描述符, 并按照这些描述符对预测性能的贡献值的高低而对它们进行排序(图2). 体系的RF模型所排出的描述符序列的前三位分别为S(56)(-S-原子类型电拓扑态之和),5χCH(五元环的简单分子连通性Chi指数)和(五元环的价分子连通性Chi指数), 这三个描述符中的两个S(56)和也同时被体系的SVM模型的RFE方法所选出.

表4 腺苷受体模型PROCHECK评价结果Table4 Evaluation results of PROCHECK of modeled adenosine receptor
在HEC1的母核结构中(图3),24存在含有S原子的五元环, 和所选出的贡献最大的三个描述符表述一致. RF选出的nsulph描述符反映了五元环中的S原子; S(35)、S(34)反映了母核结构中的N; S(9)、S(20)、S(25)反映了母核结构的碳碳双键; nhyd反映了母核结构中的氢键. 表明所选出的描述符能够很好地代表HEC1抑制剂的结构特征. 除此之外, SVM模型和RF模型中还有以下重合的描述符: S(18)(>CH2原子类型电拓扑态之和), S(9)(=CH-[sp2]原子类型H电拓扑态之和), Tcent(中心指数), Rugty(分子粗糙度), S(35)(:N:原子类型电拓扑态之和), QC,Min(C原子上最大的负电荷). 以上结果显示两种方法提取出的描述符有所交叉, 但各有自己独特的描述符, 说明学习过程是不同的.
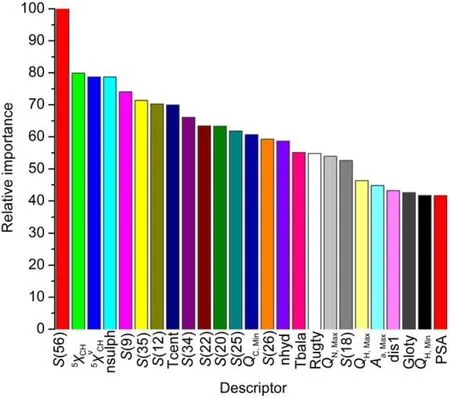
图2 RF模型中得到的25个对HEC1抑制剂和非抑制剂预测最相关的描述符的相对重要性排名Fig.2 Relative importance of the 25 highest ranked descriptors in the RF model for the prediction of HEC1 inhibitors and non-inhibitors
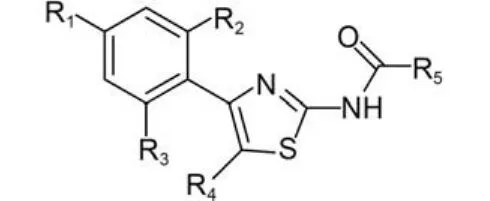
图3 HEC1抑制剂母核结构Fig.3 Mother nuclear structure of HEC1 inhibitors
3.4 虚拟筛选in-house数据库
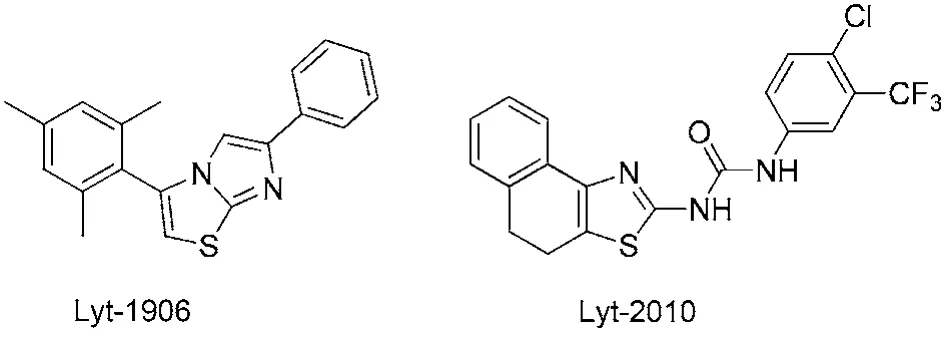
测试集用来验证所建立模型的性能, 预测精度越高, 说明模型的性能就越好. 根据这一原则, 我们采用了效果更优的RF模型来对HEC1的抑制剂进行虚拟筛选. 我们对实验室“in-house”数据库(2100个实体化合物和片段)进行筛选, 最终得到了2个HEC1的潜在选择性抑制剂(图4).
我们虚拟筛选得到的两个分子, 在结构上和数据集的分子相比, 均有噻唑环结构、苯环结构, 以及和噻唑2位相连接的N原子; 且和表4中贡献值最大的前六位描述符S(35)相吻合. 表明筛选结果极大可能具有生物活性.

表5 化合物Lyt-1906和Lyt-2010体外增殖抑制活性Table5 Anti-proliferative activity of compounds Lyt-1906 and Lyt-2010 in vitro
3.5 活性化合物体外抗肿瘤测试
为验证筛选结果, 我们对筛选出的化合物Lyt-1906和Lyt-2010, 进行了体外抗肿瘤活性测试(实验方法见Supporting Information实验1), 选用乳腺癌细胞株为MDA-MB-468和MDA-MB-231. 如表5所示,化合物Lyt-1906和Lyt-2010展示了较好的体外增殖抑制活性, IC50值在5.6–58.3 μmolL–1, 结果验证了虚拟筛选方法的可靠性, 证实RF模型对于筛选HEC1抑制剂的可靠性.
4 结 论
机器学习方法(RF和SVM)对于HEC1抑制剂与非抑制剂分类模型的建立和虚拟筛选, 具有很好的效果. 尤其对于像HEC1这样还没有X射线晶体结构的靶点蛋白, 不能通过小分子和靶点蛋白对接的方法来进行抑制剂的快速筛选, 采用机器学习方法(RF和SVM)进行高通量虚拟筛选, 就具有很重要的现实意义. 我们通过对分子描述符的特征筛选, 采用SVM和RF方法分别对HEC1抑制剂和非抑制剂建立了分类模型. 在RF模型中使用RF方法得到25个分子描述符, 而SVM模型中使用RFE方法选择出12个最相关描述符, 大幅降低了分子描述符的冗余程度.我们对两种模型的预测效果做了对比, 采用具有更好预测性能的RF模型对HEC1抑制剂做了虚拟筛选, 得到了2个潜在的HEC1抑制化合物. 对筛选得到的化合物所进行的体外抗肿瘤活性测试, 均显示出一定活性. 在此基础上, 后期可以通过进一步修饰改构和生物学实验, 以发现HEC1抑制剂, 并对乳腺癌的治疗发挥良好作用.
Supporting Information: The 189 molecular descriptors of this study are listed in Table S1, and the structure, category and source of compounds in the training set and test set are provided in Tables S2 and S3. Test method of antitumor activity in vitro was in experiment 1. This information isavailable free of charge via the internet at http://www.whxb.pk u.edu.cn.
(1)Gan, S. J.; Wang, Q.; Zhu, L. M.; Xie, H.; Ding, X. F. Basic & Clin. Med. 2015, 35 (1), 134. [甘绍举, 王 青, 朱丽敏,谢 浩, 丁先锋. 基础医学与临床, 2015, 35 (1), 134.]
(2)Chen, Y.; Riley, D. J.; Chen, P. L.; Lee, W. H. Mol. Cell Biol. 1997, 17 (10), 6049.
(3)Du, X. L.; Wang, M. R. Acta Acad. Med. Sin. 2007, 29 (1), 137. [杜小莉, 王明荣. 中国医学科学院学报, 2007, 29 (1), 137.]
(4)Hu, C. M.; Zhu, J.; Guo, X. E.; Chen, W.; Qiu, X. L.; Ngo, B.; Chien, R.; Wang, Y. V.; Tsai, C. Y.; Wu, G.; Kim, Y.; Lopez, R.; Chamberlin, A. R.; Lee, E. H.; Lee, W. H. Oncogene 2015, 34, 1220. doi: 10.1038/onc.2014.67
(5)Huang, L. Y.; Chang, C. C.; Lee, Y. S.; Chang, J. M.; Huang, J. J.; Chuang, S. H.; Kao, K. J.; Lau, G. M.; Tsai, P. Y.; Liu, C. W.; Lin, H. S.; Lau, J. Y. Mol. Cancer Ther. 2014, 13 (6), 1419.
(6)Lee, Y. S.; Chuang, S. H.; Huang, L. Y.; Lai, C. L.; Lin, Y. H.; Yang, J. Y.; Liu, C. W.; Yang, S. C.; Lin, H. S.; Chang, C. C.; Lai, J. Y.; Jian, P. S.; Lam, K.; Chang, J. M.; Lau, J. Y.; Huang, J. J. J. Med. Chem. 2014, 57 (10), 4098. doi: 10.1021/jm401990s
(7)Wu, G.; Qiu, X. L.; Zhou, L.; Zhu, J.; Chamberlin, R.; Lau, J.; Chen, P. L.; Lee, W. H. Cancer Res. 2008, 68 (20), 8393. doi: 10.1158/0008-5472.CAN-08-1915
(8)Qiu, X. L.; Li, G.; Wu, G.; Zhu, J.; Zhou, L.; Chen, P. L.; Chamberlin, A. R.; Lee, W. H. J. Med. Chem. 2009, 52 (6), 1757. doi: 10.1021/jm8015969
(9)Chen, Y.; Riley, D. J.; Zheng, L.; Chen, P. L.; Lee, W. H. J. Biol. Chem. 2002, 277 (51), 49408. doi: 10.1074/jbc.M207069200
(10)Diaz-Rodríguez, E.; Sotillo, R.; Schvartzman, J. M.; Benezra, R. Proc. Natl. Acad. Sci. U. S. A. 2008, 105 (43), 16719. doi: 10.1073/pnas.0803504105
(11)Ferretti, C.; Totta, P.; Fiore, M.; Mattiuzzo, M.; Schillaci, T.; Ricordye, R.; Di Leonardo, A.; Degrassi, F. Cell Cycle 2010, 9 (20), 4174. doi: 10.4161/cc.9.20.13457
(12)Wei, R.; Ngo, B.; Wu, G.; Lee, W. H. Mol. Biol. Cell 2011, 22 (19), 3584. doi: 10.1091/mbc.E11-01-0012
(13)Xue, Y.; Li, H.; Ung, C.; Yap, C.; Chen, Y. Chem. Res. Toxicol. 2006, 19, 1030. doi: 10.1021/tx0600550
(14)Xue, Y.; Yap, C. W.; Sun, L. Z.; Cao, Z. W.; Wang, J.; Chen, Y. Z. J. Chem. Inf. Comput. Sci. 2004, 44, 1497. doi: 10.1021/ci049971e
(15)Xue, Y.; Li, Z.; Yap, C. W.; Sun, L.; Chen, X.; Chen, Y. Z. J. Chem. Inf. Comput. Sci. 2004, 44, 1630. doi: 10.1021/ci049869h
(16)Yang, X. G.; Chen, D.; Wang, M.; Xue, Y.; Chen, Y. Z. J. Comput. Chem. 2009, 30, 1202. doi: 10.1002/jcc.v30:8
(17)Yang, X. G.; Lv, W.; Chen, Y. Z.; Xue, Y. J. Comput. Chem. 2010, 31, 1249.
(18)Lv, W.; Xue, Y. Eur. J. Med. Chem. 2010, 45, 1167. doi: 10.1016/j.ejmech.2009.12.038
(19)Cong, Y.; Yang, X.; Lv, W.; Xue, Y. J. Mol. Graph. Model. 2009, 28, 236. doi: 10.1016/j.jmgm.2009.08.001
(20)Luan, F.; Liu, H.; Ma, W.; Fan, B. Eur. Med. Chem. 2008, 43, 43. doi: 10.1016/j.ejmech.2007.03.002
(21)Ung, C. Y.; Li, H.; Yap, C. W.; Chen, Y. Z. Mol. Pharmacol. 2007, 71, 158.
(22)Li, H.; Ung, C.; Yap, C.; Xue, Y.; Li, Z.; Cao, Z.; Chen, Y. Chem. Res. Toxicol. 2005, 18, 1071. doi: 10.1021/tx049652h
(23)Li, B. K.; Cong, Y.; Tian, Z. Y.; Xue, Y. Acta Phys. -Chim. Sin. 2014, 30 (1), 171. [李秉轲, 丛 湧, 田之悦, 薛 英. 物理化学学报, 2014, 30 (1), 171.] doi: 10.3866/PKU.WHXB201311041
(24)Huang, J. J.; Lau, J. Improved Modulators of HEC1 Activity and Methods. CN Patent 103038231.A, 2013-04-10. [Huang, J. J., Lau, J. HEC1活性调节剂及其方法: 中国, CN103038231.A[P]. 2013-04-10.]
(25)Duda, R. O.; Hart, P. E. Pattern Classification and Scene Analysis; John Wiley & Sons: Hoboken, New Jersey, USA, 1973.
(26)ChemDraw 7.0.1 ed.; CambridgeSoft Corporation, Cambridge: Massachusetts, USA, 2007.
(27)Corina 3.4 edn.; Molecular Networks GmbH Computerchemie: Erlangen, Germany, 2006.
(28)Burges, C. J. Data Min. Knowl. Disc. 1998, 2, 121.
(29)Vapnik, V. N. The Nature of Statistical Learning Theory; Springer: Berlin & Heidelberg, Germany, 1995.
(30)Doucet, J. P.; Barbault, F.; Xia, H.; Panaye, A.; Fan, B. Curr. Comput-Aid. Drug. 2007, 3, 263. doi: 10.2174/157340907782799372
(31)Svetnik, V.; Liaw, A.; Tong, C.; Culberson, J. C.; Sheridan, R. P.; Feuston, B. P. J. Chem. Inf. Comput. Sci. 2003, 43, 1947. doi: 10.1021/ci034160g
(32)Breiman, L. Mach. Learn. 2001, 45, 5. doi: 10.1023/A: 1010933404324
(33)Khandelwal, A.; Krasowski, M. D.; Reschly, E. J.; Sinz, M. W.; Swaan, P. W.; Ekins, S. Chem. Res. Toxicol. 2008, 21, 1457. doi: 10.1021/tx800102e
(34)Breiman, L. Out-of-bag Estimation, 1996, http://citeseerx.ist.psu.edu.sci-hub.org/viewdoc/download? doi=10.1.1.45.3712&rep=rep1&type=pdf (accessed Mar 15, 2015).
(35)Breiman, L. Wald Lecture II, Looking inside the Black Box, 2005. http://www.stat.berkeley.edu/users/breiman (accessed Mar 15, 2015).
(36)Breiman, L.; Cutler, A. Random Forests, Version 5.1, 2004. http://www.stat.berkeley.edu/~breiman/RandomForests/ cc_home.htm (accessed Mar 15, 2015).
Predicting and Virtually Screening Breast Cancer Targeting Protein HEC1 Inhibitors by Molecular Descriptors and Machine Learning Methods
HE Bing1,2LUO Yong1LI Bing-Ke2XUE Ying1,3YU Luo-Ting1,*QIU Xiao-Long4,5YANG Teng-Kuei4
(1State Key Laboratory of Biotherapy and Cancer Center, West China Hospital, Sichuan University, and Collaborative Innovation Center for Biotherapy, Chengdu 610041, P. R. China;2College of Chemistry and Life Science, Chengdu Normal University, Chengdu 611130, P. R. China;3College of Chemistry, Sichuan University, Chengdu 610064, P. R. China;4Zhaobang Bio-Med. Institute Co., Ltd., Nantong 226000, Jiangsu Province, P. R. China;5Wisdom Pharmaceutical Co., Ltd., Haimen 226123, Jiangsu Province, P. R. China)
Highly expressed in cancer 1 (HEC1) is a conserved mitotic regulator that is critical for spindle checkpoint control, kinetochore functionality, and cell survival. Overexpression of HEC1 has been detected in a variety of human cancers, and it is linked to poor prognosis of primary breast cancers. Thus, it is important to screen novel inhibitors with high affinity for HEC1. Machine learning (ML) methods were exhibiting good predicting capability in several aspects of the diverse compounds, such as pharmacokinetics,pharmacodynamics, and toxicity. In this work, two ML methods, support vector machines (SVMs) and random forests (RFs), were used to develop a classification method for searching inhibitors and non-inhibitors of HEC1 from the chemical library of structural diversity by screening characteristics of molecular descriptors. Both ML methods achieved promising prediction accuracies, and the RF model showed better performance. We performed virtual screening of HEC1 inhibitors by the RF model from an in-house database to screen potential HEC1 inhibitors. Two novel potential candidates were found. In vitro experiments of the two compounds showed that both had a certain degree of antitumor activity for the MDA-MB-468 and MDA-MB-231 breast cancer cell lines. Our study shows that ML methods are promising to design and virtually screen inhibitors of HEC1.
HEC1; Selective inhibitor; Machine learning method; Support vector machine; Random forest; Virtual screening
O641
10.3866/PKU.WHXB201507301
Received: April 2, 2015; Revised: July 30, 2015; Published on Web: July 30, 2015.
*Corresponding author. Email: luodyu@163.com.
The project was supported by “the Category 1.1 New Drug SKLB1312 of Antitumor (Breast Cancer), which is the Cooperation Project between West China Hospital of Sichuan University and Jiangsu Zhaobang Biological and Medical Research Institute Co., Ltd.”.
四川大学华西医院与江苏兆邦生物医药研究院有限公司合作项目“抗肿瘤(乳腺癌)一类新药SKLB1312”资助
© Editorial office of Acta Physico-Chimica Sinica

