秋、冬季刺参养殖池塘菌群的多样性分析
窦妍,丁君, 曲凌云,刘志敏,王轶南,穆晓虎,常亚青
(1.大连海洋大学 农业部北方海水增养殖重点实验室,辽宁 大连 116023;2.国家海洋局 第一海洋研究所,山东 青岛 266061)
秋、冬季刺参养殖池塘菌群的多样性分析
窦妍1,丁君1, 曲凌云2,刘志敏1,王轶南1,穆晓虎1,常亚青1
(1.大连海洋大学 农业部北方海水增养殖重点实验室,辽宁 大连 116023;2.国家海洋局 第一海洋研究所,山东 青岛 266061)
摘要:以黄海和渤海代表性刺参养殖池塘秋、冬季海水和沉积物基因组DNA为模板,以细菌16S rDNA通用引物进行PCR扩增,构建16S rDNA文库并进行测序分析,研究了秋、冬季刺参养殖池塘菌群的多样性。结果表明:海水和沉积物中主要包括11个门类的细菌,即变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、厚壁菌门(Firmicutes)、梭杆菌门(Fusobacteria)、蓝细菌门(Cyanobacteria)、浮霉菌门(Planctomycetes)、脱铁杆菌门(Deferribacteres)、绿弯菌门(Chloroflexi);黄海和渤海刺参养殖池塘海水和沉积物中优势类群均为变形菌(变形菌比例>48%);用Shannon指数及Simpson优势度指数分析细菌的多样性,黄、渤海刺参养殖池塘中,冬季沉积物细菌Simpson优势度指数均最低,分别为0.014 89和0.016 50,Shannon指数均最高,分别为6.312和5.695。研究表明,黄海和渤海刺参养殖池塘中,冬季沉积物细菌多样性均最高。
关键词:刺参;养殖池塘;16S rDNA;变形菌门
自20世纪80年代中国刺参人工育苗技术取得成功后,刺参的增养殖在北方沿海逐渐实现规模化[1-3]。然而,随着养殖规模的不断扩大,刺参病害也日益增加。良好的环境是刺参健康生长的必备条件,而养殖池塘生物群落的重要组成部分——水体及沉积物中的微生物,可以作为衡量环境优劣的指标[4]。因此,考察养殖环境中细菌的多样性,可以为微生物生态变化与刺参病害发生的关系提供一定的科学依据[5]。
环境中可培养细菌的多样性占微生物总数的量极低,基于培养的研究方面很难全面了解环境微生物的多样性[6]。随着DNA测序技术的发展,以环境总DNA为模板进行微生物群落组成和多样性的研究,已成为分析微生物多样性的一种有效手段[7-9]。目前,有关刺参及养殖环境微生物方面的研究已经逐渐开展。李彬等[10]运用传统细菌分离培养与分子生物学技术相结合的方法研究了冬季刺参养殖环境及其肠道内的细菌菌群。关晓燕等[5]采用16S rDNA的PCR-DGGE基因指纹技术研究了不同盐度的仿刺参养殖水环境中的菌群多样性。张文姬等[11]利用16S rDNA-RFLP法分析了大连地区仿刺参肠道可培养微生物的多样性。
本研究中,采用16S rDNA文库法,通过系统发育分析和多样性分析,对黄、渤海刺参养殖池塘海水和沉积物的菌群结构及多样性进行了研究,并分析了其中潜在的条件致病菌,旨在了解黄、渤海刺参养殖池塘细菌群落结构,为构建中国北方刺参养殖池塘菌库奠定基础。
1材料与方法
1.1材料
黄海秋、冬季刺参养殖池塘沉积物和海水样品于2011年11月和2012年2月采集自大连力源水产有限公司养殖池塘,渤海秋、冬季刺参养殖池塘沉积物和海水样品自2011年11月和2012年2月采集自兴城菊花岛海产品有限公司养殖池塘。2个池塘均为泥沙底,用无菌瓶分别取刺参养殖池塘水面下40 cm左右处海水500 mL和池底5 cm左右处沉积物100 g。将采集的样品置于冰盒中运回至大连海洋大学农业部北方海水增养殖重点实验室。黄海秋、冬季海水和沉积物样品,分别标记为YAW、YAS、YWW、YWS,渤海秋、冬季海水和沉积物样品,分别标记为BAW、BAS、BWW、BWS。
1.2方法
1.2.1DNA提取海水和沉积物样品的DNA提取参照王轶南等[12]的方法。
1.2.216S rDNA基因片段克隆文库的构建采用16S rDNA通用引物27F(5′ AGAGTTTGATCCTGGCTCAG3′)和1492R(5′ GGTTACCTTGTTACGA-CTT 3′)扩增16S rDNA全长序列。PCR反应体系(共25 μL):MasterMix 12.5 μL、上、下游引物(10 μmol/L)各1 μL,ddH2O 9.5 μL,模板 1 μL。PCR反应程序:95 ℃下预变性2 min;94 ℃下变性30 s,52 ℃下退火30 s,72 ℃下延伸90 s,共进行30个循环;最后在72 ℃下延伸10 min,用10 g/L琼脂糖电泳检测PCR产物。PCR产物经割胶纯化连接到pMD19-T载体,于16 ℃下连接30 min后将连接产物注入感受态细胞中,在LB培养基中培养1 h后,涂布于含有X-gal、IPTG和Amp的LB培养基上。从各文库中随机挑选100个左右的阳性克隆子送上海英俊生物技术有限公司进行测序。
1.2.3细菌16S rDNA 系统发育分析用VecScreen程序去除载体序列,用Mallard 1.02软件进行嵌合体检验。应用Mothur 1.31.2软件[13]对所得有效序列进行分析,以97%相似性为标准,划分操作分类单元(operational taxonomic unit,OTU),将每个OUT中的代表序列利用GenBank数据库中的Blast程序进行相似性比对,并挑选相近序列。采用Neighbor-joining法[14],应用Mega 5.22软件构建系统发育树,自展值设为1000[15]。
1.2.4多样性分析基于OTU丰度,用SPADE[16]软件计算8个克隆文库的覆盖率(coverage)、香农指数(Shannon index)和辛普森指数(Simpson index)。
2结果与分析
2.116S rDNA文库的多样性分析
根据OTU丰度,采用SPADE 软件计算得到8个克隆文库的覆盖率、香农指数和辛普森指数分别为10.9%~92.0%、1.508~6.312、0.014 89~0.406 22(表1)。利用Shannon指数及Simpson优势度指数分析细菌的多样性,结果表明:黄海刺参养殖池塘中,冬季沉积物细菌Simpson优势度指数(0.014 89)最低,且Shannon指数(6.312)最高,表明黄海刺参养殖池塘冬季沉积物细菌多样性最高;渤海刺参养殖池塘中,冬季沉积物细菌Simpson优势度指数(0.016 50)最低,且Shannon指数(5.695)最高,表明渤海养殖池塘也是冬季沉积物细菌多样性最高。
表1黄、渤海刺参养殖池塘秋、冬季海水和沉积物细菌的多样性
Tab.1 Bacterial diversity in the seawater and sediments of the sea cucumberApostichopusjaponicusculture ponds from Yellow Sea and Bohai Sea in autumn and winter
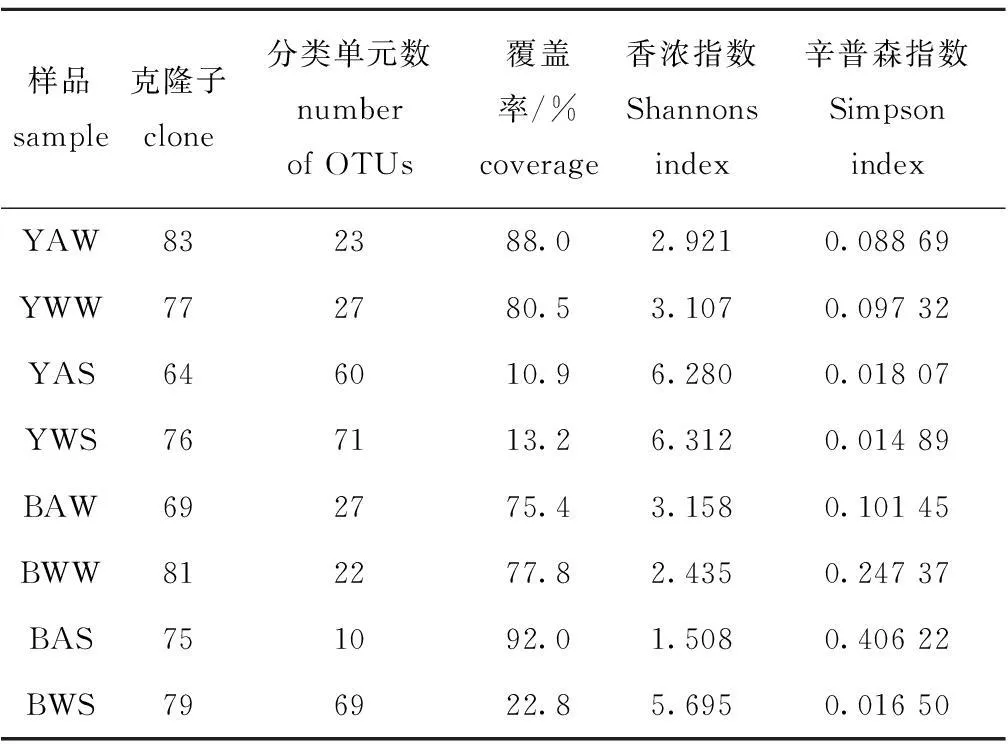
样品sample克隆子clone分类单元数numberofOTUs覆盖率/%coverage香浓指数Shannonsindex辛普森指数SimpsonindexYAW832388.02.9210.08869YWW772780.53.1070.09732YAS646010.96.2800.01807YWS767113.26.3120.01489BAW692775.43.1580.10145BWW812277.82.4350.24737BAS751092.01.5080.40622BWS796922.85.6950.01650
2.2刺参养殖池塘的菌群结构分析
对测序片段去除嵌合体序列后,所有8个文库共得到604个有效序列,其中黄海刺参养殖池塘秋、冬季海水和沉积物样品有效克隆序列分别为83、64、77、76,渤海刺参养殖池塘秋、冬季海水和沉积物样品有效克隆序列分别为69、75、81、79(表1)。将相似性大于97%的序列归为一个OUT后,将每个OUT中代表序列利用GenBank数据库中的Blast程序进行相似性比对,并挑选与之相近序列(相似性为84%~100%)构建系统发育树。由于数据量较大,本研究中随机挑选每个门类的一条克隆序列及其与数据库中相似性较高的序列进行发育树构建,结果如图1所示。
黄、渤海刺参养殖池塘菌群结构分析结果见表2,黄、渤海刺参养殖池塘海水和沉积物样品中16S rDNA序列共分11个门类,即变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、厚壁菌门(Firmicutes)、梭杆菌门(Fusobacteria)、蓝细菌门(Cyanobacteria)、浮霉菌门(Planctomycetes)、脱铁杆菌门黄海养殖池塘秋季海水样品中16S rDNA序列分为4个门类,变形菌门为优势类群(占48.1%),其次为蓝细菌门(38.6%)、拟杆菌门(9.6%)、放线菌门(3.6%);在变形菌所含亚门(α-、β-、γ-)中,α-变形菌为优势亚门。冬季海水样品中16S rDNA序列分为3个门类,变形菌门为优势类群(92.2%),其次为放线菌门(3.9%)、拟杆菌门(2.6%);在变形菌门所含亚门(α-、β-、γ-、δ-)中,α-变形菌为优势亚门(58.4%);部分序列(1.3%)未能确定其分类地位。结果显示,黄海养殖池塘秋、冬季海水菌群结构差别不大,优势菌群均为变形菌门,在变形菌门中优势亚门均为α-变形菌。
表2黄海和渤海秋、冬季刺参养殖池塘海水和沉积物的菌群结构
Tab.2Community structure of bacteria in the seawater and sediments of the sea cucumberApostichopusjaponicusculture ponds from Yellow Sea and Bohai Sea in autumn and winter

%
注:“—”代表未发现
Note:“—” represents not found(Deferribacteres)、绿弯菌门(Chloroflexi)。其中,变形菌门占优势(>48%),在变形菌门所含亚门(α-、β-、γ-、δ-、ε-、ζ-)中,γ-变形菌为优势类群;另有部分序列分类地位尚未明确。
黄海养殖池塘秋季沉积物样品中16S rDNA序列除部分(3.1%)未能确定其分类地位外,其余序列分为6个门类,变形菌门为优势类群(76.6%),其次为拟杆菌门(10.9%)、酸杆菌门(3.1%)、蓝细菌门(3.1%)、疣微菌门(1.6%)、浮霉菌门(1.6%);在变形菌门所含亚门(β-、γ-、δ-、ε-、ζ-)中,δ-变形菌为优势亚门。冬季沉积物样品中除部分序列(2.6%)未能确定其分类地位外,其余序列分为7个门类,优势类群为变形菌门(72.4%),其次为拟杆菌门(6.6%)、蓝细菌门(6.6%)、浮霉菌门(5.3%)、酸杆菌门(2.6%)、疣微菌门(2.6%)、梭杆菌门(1.3%);在变形菌门所含亚门(α-、β-、γ-、δ-、ε-)中,γ-变形菌(23.7%)、δ-变形菌(23.7%)为优势类群。结果显示,黄海养殖池塘秋、冬季沉积物菌群结构相差不大,优势菌群门均为变形菌,在变形菌亚门中γ-变形菌、δ-变形菌较为优势。
渤海养殖池塘秋季海水16S rDNA序列可分为4个门类,变形菌门为优势类群(84.1%),其次为蓝细菌门(13.0%)、放线菌门(1.4%)、拟杆菌门(1.4%);在变形菌门所含亚门(α-、β-、γ-)中,γ-变形菌为优势亚门。渤海养殖池塘冬季海水16S rDNA序列可分为5个门类,变形菌门(90.0%)为优势类群,其次为放线菌门(4.9%)、拟杆菌门(2.5%)、厚壁菌门(Firmicutes)(1.2%)、蓝细菌门(1.2%);在变形菌所含亚门(α-、β-、γ-)中,α-变形菌为优势亚门;其中,单克隆子BWW13与GenBank中的灿烂弧菌Vibriosplendidus(EU091328.1)相似性达99%。结果显示,渤海养殖池塘秋、冬季海水菌群结构差别不大,均以变形菌门为优势类群,但在秋季海水中的优势亚门为γ-变形菌,而冬季海水中的优势亚门为α-变形菌。

图1 黄、渤海刺参养殖池塘秋、冬季海水和沉积物细菌16S rDNA序列的系统发育树Fig.1 Phylogenetic tree of bacterial 16S rDNA sequences from the seawater and sediments of the sea cucumber Apostichopus japonicus culture ponds from Yellow Sea and Bohai Sea in autumn and winter
渤海养殖池塘秋季沉积物16S rDNA序列均为变形菌门;在变形菌门所含的4个亚门(α-、β-、γ-、δ-)中,γ-变形菌为优势亚门;其中,单克隆子BAS12与GenBank中的哈维氏弧菌Vibrioharveyi(HM008702.1)相似性达99%。渤海养殖池塘冬季沉积物16S rDNA序列除部分(1.3%)未能确定其分类地位外,其余可分为9门类,变形菌门占优势(75.9%),其次为酸杆菌门(8.9%)、拟杆菌门(3.8%)、疣微菌门(2.5%)、浮霉菌门(2.5%)、厚壁菌门(1.3%)、梭杆菌门(1.3%)、脱铁杆菌门(1.3%)、绿弯菌门(1.3%);在变形菌门所含亚门(α-、γ-、δ-、ε-、ζ-)中,γ-变形菌(43.0%)为优势亚门。结果显示,渤海养殖池塘秋、冬季沉积物菌群结构变化较大,优势菌均为变形菌门,优势亚门均为γ-变形菌。
3讨论
3.1刺参养殖池塘秋、冬季海水和沉积物细菌的多样性
本研中究发现,黄、渤海刺参养殖池塘海水和沉积物样品中16S rDNA序列的8个克隆文库覆盖率为10.9%~92.0%。利用Shannon指数及Simpson优势度指数分析细菌的多样性,黄、渤海刺参养殖池塘秋、冬季海水和沉积物中,皆以冬季沉积物细菌多样性最高。
关晓燕等[5]在研究不同盐度仿刺参养殖水环境中的菌群多样性时发现,其中所含的优势菌有变形菌门;白洁等[17]在研究黄海北部不同站位海洋细菌群落分布特征时发现,各站位中优势菌为变形菌门。黄海和渤海刺参养殖池塘海水中的优势菌群均为变形菌门,本研究结果与上述研究结果一致。在变形菌门的4个亚门(α-、β-、γ-、δ-)中,黄海秋、冬季海水及渤海冬季海水均以α-变形菌亚门较为优势,这与González等[18]的研究结果一致;渤海刺参养殖池塘秋季和冬季海水中略有不同,秋季海水中γ-变形菌为优势亚门,α-变形菌为次优势亚门,而冬季海水中α-变形菌为优势亚门,γ-变形菌为次优势亚门。总体而言,黄海和渤海刺参养殖池塘海水中菌群结构在秋、冬两季变化不大。
变形菌门是海洋沉积物中的主要类群[19],该类群的代谢是深海沉积物中最主要的微生物活动。本研究表明,黄海和渤海刺参养殖池塘沉积物样品中的主要优势类群为变形菌,这与肖慧等[20]和白洁等[21]对海洋沉积物的研究结果一致。在研究海洋沉积物厌氧条件下氨的代谢情况时,Freitag等[22]发现,γ-变形菌亚门中的细菌在厌氧氨氧化过程中发挥着重要的作用。Urakawa等[23]发现,δ-变形菌中的细菌在海洋沉积物中主要以硫还原细菌(SRB)为主。本研究中,黄海养殖池塘沉积物样品中γ-变形菌和δ-变形菌为优势类群,渤海养殖池塘沉积物样品中γ-变形菌较为优势,因此,黄、渤海养殖池塘沉积物中存在大量的γ-变形菌和δ-变形菌,可能与沉积物中的碳、氮、硫循环密切相关。总体而言,黄海刺参养殖池塘沉积物秋、冬季菌群结构变化不显著,但是渤海刺参养殖池塘秋、冬季菌群结构差异较大。Nelson等[24]对Steel Lake沉积物中细菌群落的分布进行研究时也发现,不同时期形成的沉积物其细菌群落结构不同,主要原因可能是有机质含量不同造成的。本研究中,也推测渤海刺参养殖池塘秋季和冬季沉积物中菌群结构差异较大是由于不同季节沉积物中有机质的含量不同造成的。
3.2刺参养殖环境中的条件致病菌
Stackebrandt等[25]研究发现,同菌种的菌株其16S rDNA序列相似性不低于97%,但是16S rDNA序列相似性达到97%或是更高的菌株却不一定属于同一菌种。目前,将具有97%~99%16S rDNA全序列相似性的菌株判定为一个属,具有99%~100%的16S rDNA全序列相似性的菌株判定为一个种[26]。本研究中发现,渤海冬季海水中克隆子BWW13与灿烂弧菌(EU091328.1)相似性达99%,渤海养殖池塘秋季沉积物克隆子BAS12与哈维氏弧菌(HM008702.1)相似性达99%,可以确定,渤海秋季海水和秋季沉积物中分别含有灿烂弧菌和哈维氏弧菌。 张春云等[27]报道,2003年春季,山东省青岛地区刺参养殖场暴发的较为严重的腐皮综合征的致病菌为灿烂弧菌;Deng等[28]从刺参皮肤溃烂处分离得到6株主要菌株,其中04101和04103菌株为哈维氏弧菌;Becker等[29]在研究糙海参幼体培育过程中暴发的流行性皮肤溃烂病时推测,弧菌属Vibriosp.的哈维氏弧菌或溶藻弧菌V.alginolyticus可能是引发此病的病原菌。虽然本研究中的刺参养殖池塘没有病害发生,但是刺参养殖池塘中有致病菌的存在,应引起关注。
参考文献:
[1]牟绍敦,李远山,刘岗,等.山东增养殖技术发展现状[J].海洋湖沼通报,2000(2):63-65.
[2]隋锡林,邓欢.刺参池塘养殖的病害及预防对策[J].水产养殖,2004,23(6):22-23.
[3]张春云,王印庚,荣小军,等.国内外海参自然资源、养殖状况及存在问题[J].海洋水产研究,2004,25(3):89-97.
[4]Parel H P,Dyble J,Moisander P H,et al.Microbia indicators of aquatic ecosystem change:current applications to eutrophication studies[J].FEMS Microbiology Ecology,2003,46:233-246.
[5]关晓燕,周遵春,陈仲,等.应用PCR-DGGE指纹技术分析高温季节仿刺参养殖水环境中菌群多样性[J].海洋湖沼通报,2010(1):82-88.
[6]Amann R I,Sludig W,Schileifer K H.Phylogenetic identification an in suit detection of individual microbial cells without cultivation[J].Microbiology Review,1995,59:143-169.
[7]郑艳玲,侯立军,陆敏,等.崇明东滩冬夏季表层沉积物细菌多样性研究[J].中国环境科学,2012,32(2):300-310.
[8]Gomez-Balderas C D C,Cochet N,Bert V,et al.16S rDNA analysis of bacterial communities associated with the hyper accumulatorArabidopsishallerigrown on a Zn and Cd polluted soil[J].European Journal of Soil Biology,2014,60:16-23.
[9]Breidt F,Medina E,Wafa D,et al.Characterization of cucumber fermentation spoilage bacteria by enrichment culture and 16S rDNA cloning[J].Journal of Food Science,2013,78(3):470-476.
[10]李彬,荣小军,廖梅杰,等.冬季刺参养殖环境与肠道内细菌菌群的研究[J].海洋科学,2010,34(4):64-69.
[11]张文姬,侯红漫,张公亮,等.仿刺参肠道可培养微生物多样性研究[J].食品工业科技,2011,32(9):149-155.
[12]王轶南,朱世伟,常亚青.刺参肠道及养殖池塘菌群组成的PCR-DGGE指纹图谱分析[J].渔业科学进展,2010,31(3):119-122.
[13]Schloss P D,Westcott S L,Ryabin T,et al.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Applied and Environmental Microbiology,2009,75(23):7537-7541.
[14]Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution,1987,4(4):406-425.
[15]Felsenstein J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985,39(4):783-791.
[16]National Tsing Hua University,TAIWAN.Program SPADE(Species Prediction And Diversity Estimation)[EB/OL].[2014-01-01].http://chao.stat.nthu.edu.tw,2010.
[17]白洁,李海艳,赵阳国.黄海北部不同站位海洋细菌群落分布特征[J].微生物学报,2009,49(3):343-350.
[18]González J M,Moran M A.Numerical dominance of a group of marine bacteria in the alpha-subclass of the class Proteobacteria in coastal seawater[J].Applied and Environmental Microbiology,1997,63(11):4237-4242.
[19]Li L,Kato C,Horikoshi K.Microbial diversity in sediments collected from the deepest cold-seep area,the Japan Trench[J].Marine Biotechnology,1999,1:391-400.
[20]肖慧,张艳,张喆,等.青岛、威海水域夏冬季表层沉积物细菌多样性的初步研究[J].中国海洋大学学报:自然科学版,2009,39(4):641-646.
[21]白洁,张海艳,张健,等.黄海西北部沉积物中细菌群落16S rDNA多样性解析[J].中国环境科学,2009,29(12):1277-1284.
[22]Freitag T E,Prosser J I.Community structure of ammonia-oxidizing bacteria within anoxic marine sedments[J].Applied and Environmental Microbiology,2003,69(3):1359-1371.
[23]Urakawa H,Kita-Tsukamoto K,Ohwada K.Microbial diversity in marine sediments from Sagami Bay and Tokyo Bay,Japan,as determined by 16S rRNA gene analysis[J].Microbiology,1999,145:3305-3315.
[24]Nelson D M,Ohene-Adiei S,Hu F S,et al.Bacterial diversity and distribution in the holocence sediments of a northern temperate lake[J].Microbial Ecology,2007,54:252-263.
[25]Stackebrandt E,Goebel B M.Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic Bacteriology,1994,44:846-849.
[26]Drabcourt M,Bouet C,Caiioz A,et al.16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates[J].Journal of Clinical Microbiology,2000,38(10):3623-3630.
[27]张春云,王印庚,荣小军.养殖刺参腐皮综合征病原菌的分离与鉴定[J].水产学报,2006,30(1):118-123.
[28]Deng H,He C B,Zhou Z C,et al.Isolation and pathogenicity of pathogens from skin ulceration disease and viscera ejection syndrome of the sea cucumberApostichopusjaponicas[J].Aquaculture,2009,287(1/2):18-27.
[29]Becker P,Gillan D,Lanterbecq D,et al.The skin ulceration disease in cultivated juveniles ofHolothuriascabra[J].Aquaculture,2004,242:13-30.
Flora diversity in sea cucumberApostichopusjaponicusculture
ponds in autumn and winter
DOU Yan1, DING Jun1, QU Ling-yun2, LIU Zhi-min1,
WANG Yi-nan1, MU Xiao-hu1, CHANG Ya-qing1
(1.Key Laboratory of Mariculture & Stock Enhancement in North China’s Sea, Ministry of Agriculture, Dalian Ocean University, Dalian 116023, China; 2.First Institute of Oceanography, State Oceanic Administration, Qingdao 266061, China)
Abstract:Flora diversity was studied in seawater and sediments in sea cucumber Apostichopus japonicus culture ponds from Yellow Sea and Bohai Sea in autumn and winter based on total DNA extract. The 16S rDNA genes of bacteria were amplified and 16S rDNA gene libraries were constructed. The results showed that 11 known Phyla of bacteria were found in seawater and sediments: Proteobacteria, Acidobacteria, Actinobacteria, Bacteroidetes, Verrucomicrobia, Firmicutes, Fusobacteria, Cyanobacteria, Planctomycetes, Deferribacteres, and Chloroflexi, with dominant Proteobacteria(>48%). The bacterial diversity in seawater and sediments in sea cucumber culture ponds was evaluated by Simpson index and Shannon index, with the minimum (0.014 89 in Yellow Sea and 0.016 50 in Bohai Sea) in winter by Simpson index, and the maximum (6.312 in Yellow Sea and 5.695 in Bohai Sea) by Shannon index. The findings indicated that there was the maximal bacterial diversity in sediments of sea cucumber culture ponds from Yellow Sea libraries and Bohai Sea libraries in winter.
Key words:Apostichopus japonicus; culture pond; 16S rDNA; Proteobacteria
通信作者:丁君(1973—), 女, 研究员。E-mail:dingjun1119@dlou.edu.cn
作者简介:窦妍(1989 —), 女, 硕士研究生。E-mail:yandou1989@126.com
基金项目:国家海洋局科研项目(201105007-2);辽宁省教育厅优秀人才支持计划项目(LJQ2011073)
收稿日期:2014-06-19
中图分类号:Q954.4
文献标志码:A
文章编号:2095-1388(2015)02-0143-06
DOI:10.3969/J.ISSN.2095-1388.2015.02.006

