PCR产物的高通量测序方法及优化
李桂澜, 胥传来
(1.北京大学 生命科学学院,北京100871;2.食品科学与技术国家重点实验室,江南大学,江苏 无锡214122)
PCR产物的高通量测序方法及优化
李桂澜1, 胥传来2
(1.北京大学 生命科学学院,北京100871;2.食品科学与技术国家重点实验室,江南大学,江苏 无锡214122)
PCR产物的高通量测序被广泛应用于功能基因筛选、肿瘤相关基因的突变和甲基化检测等。在高通量测序技术中,建库和上机测序实验一直是决定最终DNA数据质量的关键。作者优化了PCR产物样品的DNA文库制备条件和体系,设计了适合PCR产物特点上机测序方法,将已建库的PCR样品中混入一定量的基因组标准品后再上机测序,由此保证了测序数据的高质量和低冗余度。为低多样性DNA样品高通量测序技术方法运用提供方法上的参考。
PCR产物;高通量测序技术;样品文库制备;上机测序
DNA测序是常用的分子生物学研究技术,通过测序分析能提供最真实可靠的基因序列信息。从1977年第一代传统 DNA双脱氧链末端终止的sanger测序法问世以来,DNA测序技术经历了快速发展[1-2]。现在基于边合成边测序(sequencing by synthesis)为主的第二代高通量测序技术逐渐被广泛运用于基础研究、临床医学诊断和肠道微生物群与营养及代谢研究领域[2-5]。二代测序以单个读长较短、通量大为特点,实现了对单个物种的转录组和基因组细致全貌分析,也被称为深度测序或高通量测序[6]。二代测序研究平台从最初Roche公司的454焦磷酸测序和Illumina公司早先的Solexa测序技术发展到目前广泛使用的Hiseq、Miseq系列,和Life Technoloiges的Ion PGM和Ion Proton的测序,测序技术无论从速度和通量上得到了前所未有的发展[7]。目前以Illumina为技术平台的Hiseq X 5系统、Hiseq3000/4000最新的测序仪器也相继发布,使二代测序样品建库更加高效,读长(reads)更长,通量更高。
PCR产物测序通过设计恰当引物探针,运用各种PCR技术将待测序的目的基因片段扩增产物进行测序[8-10]。PCR产物的高通量测序技术被辅助运用于细胞文库中功能基因的筛选,肿瘤相关基因突变及甲基化检测和微生物菌群的营养及代谢相关探索研究[11-12]。以Illumina为平台的高通量测序,对片段大小在150~350 bp的PCR混合物样品,与基因组 DNA和微量的 ChIP-seq(Chromatin Immuno Precipitation,ChIP)样品的文库制备方法不同[13-14]。虽然目前建库技术整体一直在被优化,测序通量和测序长度也在不断的进行技术革新使之达到更高的测序要求,但Illumina的边合成边测序技术核心仍无法改变,也决定了Illumina二代测序的技术局限性。所以根据PCR产物样品自身的特点,文库制备和上机测序实验都需要进行特别的设计和处理。作者主要以Illumina系列的二代测序平台,在普通的ChIP建库方法,基于片段已知的DNA样品通过两步末端修饰反应后加DNA通用接头的实验原理,设计建立了一种适合PCR混合物样品性质特点的文库制备及上机操作实验,以确保高效低成本的建库技术及上机测序中得到最佳的测序数据质量。作者以Illumina为代表的高通量测序技术特点及具体实验方法,为不同的研究领域中选择不同测序方法提供参考。
1 材料与方法
1.1 试剂与材料
1.1.1 试剂 ChIP-Seq Sample Prep Master Mix试剂盒:美国 New England Biolabs公司;NEBNext Multiplex Oligos;Agencourt AMPure XP的 DNA纯化磁珠:美国BECKMAN COULTER公司;Brilliant SYBR Green QPCR试剂盒:美国Agilent公司;上机双端测序试剂 Miseq Reagent Kit V3 (2×150 cycles): 美国 Illumina公司;100 bp plus DNA Marker相对分子质量标准品:北京全式金生物技术公司;100%乙醇、Tris、Tween20和 NaOH:美国Sigma公司。1.5 mL低DNA吸附(LoBind)微量离心管:美国Eppendorf。
1.1.2 DNA分子 New England Biolabs公司提供:接头分子(Adaptor):5’-pGATCGGAAGAGCACACG TCTGAACTCCAGTC/ideoxyU/ACACTCTTTCCCTAC ACGACGCTCTTCCGATCT-3’;上游引物:5’-AATG ATACGGCGACCACCGAGATCTACACTCTTTCCCTA CACGACGCTCTTCCGATCT-3’;下游引物:5’-CAA GCAGAAGACGGCATACGAGATAAGCTAGTGACTG GAGTTCAGACGTGTGCTCTTCCGATCT-3’;下游引物中的下划线加粗的6个碱基为第10号标签(index)DNA分子。Takara合成荧光定量PCR引物:上游引物P1:5’-AATGATACGGCGACCACCGA-3’;下游引物P2:5’-CAAGCAGAAGACGGCATACG A-3’。
1.2 仪器
Miseq第二代高通量测序仪:美国Illumina公司;美国Bio-Rad核酸电泳仪、凝胶成像仪和PCR核酸扩增仪;电子天平和pH计:瑞士METTLER TOLEDO公司;NanoDrop2000超微量分光光度计:美国Thermo Scientific公司;Qubit 2.0荧光定量仪:美国Life Technologies公司;Mx300P荧光定量PCR仪:美国安捷伦公司;Fragment Analyzer全自动毛细管电泳仪及其配套试剂:美国Advanced Analytical。ThermoMixer恒温混匀仪:美国Eppendorf。
1.3 实验方法
1.3.1 测序样品文库制备 切胶纯化回收后的PCR产物样品T1、T2和T3,琼脂糖凝胶电泳图中条带是分布在200~300 bp宽峰,见图1。用NanoDrop2000测定质量浓度分别为24.2、25.2、26.2 ng/μL;A260/A280的OD值为1.68、1.59和1.76。
建库的初始样品用 Qubit 2.0荧光定量仪对PCR产物样品准确定量后各取2 μL(DNA总量在10~50 ng)于1.5 mL的Lobind离心管,准备第一步的平末端修复反应:1 μL末端修复反应聚合酶和5μL的10×末端修复反应缓冲液,用RNase free H2O补足至反应终体积50 μL,恒温混匀仪中30℃反应20 min。 结束后加入80 uL的AMPure XP磁珠纯化反应后样品,用44 uL RNase free H2O洗脱。第二步直接向样品溶液加入1 μL Klenow片段酶和5 μL的10×反应缓冲液(含0.2 mmol/L dATP),在37℃反应30 min,完成末端加“A”修饰反应。同样用80 μL磁珠纯化,19 μL的RNase free H2O洗脱。

图1 二代测序的PCR产物样品T1-T3Fig.1 PCR products T1-T3 for next-generation sequencing
将DNA接头分子稀释到5 μmol/L后取1 μL到样品溶液中准备连接反应,加6 μL的5×快速连接缓冲液和4 μL的快速DNA连接酶,小心混匀,室温25℃静置孵育15 min。结束后加入试剂盒的USER酶2 μL,在37℃反应15 min完成接头开环剪切。最后用与样品等倍体积30 μL的磁珠纯化两遍,检测浓度。取10 μL(总量在5~10 ng)样品进行PCR反应,循环数减少至15次。最后用与样品等体积的磁珠纯化两遍,30 μL的ddH2O洗脱,检测浓度。
1.3.2 建库后样品质量检控 将建库后样品稀释至0.5~2 ng/μL,分别用Fragment Analyzer全自动毛细管电泳仪进行片段分析;用Mx300Ps荧光定量PCR仪对样品进行荧光定量检测。选用的标准品为已准确定量并上机测序后的建库样品。用10 mmol/L Tris-HCl pH 8.0,0.05%Tween 20缓冲液按10倍梯度配制成0.002~20 pmol/L五个不同浓度以获得定量标准曲线。建库样品再次稀释 104倍到 0.1~ 2 pmol/L后取2 μL,2×Brilliant SYBR Green Master Mix 5 μL,10 μmol/L引物P1/P2 Mix 0.4 μL,最后ddH2O补足为10 μL的反应体系,阴性对照组NTC(No Template Control)和所有样品及标准品做三组重复。
[Final Conc(nmol/L)]=[QPCR]×340×104×10-6/Fragm
式中,[QPCR]代表仪器根据标准曲线检测出来的平均样品浓度 (fM);340 bp代表选用的DNA标准品的片段大小;104代表样品稀释倍数;Fragm代表建库后样品的平均片段大小。
1.3.3 Illumina Miseq上机测序 对已建库样品T2准备上机测序,将T2样品与Phix基因组标准样品等摩尔混合,用10 mmol/L Tris-HCl pH 8.0缓冲液稀释至终浓度2 nmol/L,取10 μL的上机混合样品,加入10 μL 2 mol/L的NaOH充分混匀后室温变性5 min。用双端测序试剂盒里的杂交缓冲液将样品稀释到18 pmol/L,取600 μL上机测序。Illumina数据收集和分析软件:Miseq Control Software;Real Time Analysis(RTA);Offline Basecaller(OLB);CASAVA软件用于进一步数据分析。
2 结果与分析
2.1 建库后样品检测
PCR产物样品T1,T2和T3为同一样本的3个平行生物组重复。建库结束后检测终质量浓度为:22.2、15.3、16.1 ng/μL各30 μL体系。随机选取样品T2的建库及上机测序结果分析。用 Fragment Analyzer分析初始样品和建库后样品片段分布,见图2-3。样品建库后片段的出峰位置出现移动,增加约120 bp,满足接头序列成功连接后的片段分布情况。根据标准品所得的荧光定量标准曲线其相关系数为Rsq:99.9%,计算得到稀释后的3个建库样品浓度12.46、6.78、7.81 nmol/L,满足上机测序要求。T2样品准备上机测序。

图2 高通量测序样品T2的DNA片段分析Fig.2 DNA fragment analysis of sample T2
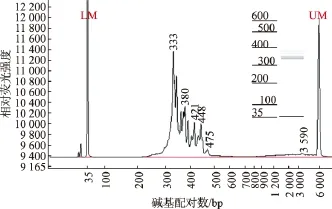
图3 测序样品T2建库后的DNA片段分析Fig.3 DNA fragment analysis of final library sample T2
2.2 上机测序结果
Illumina Miseq测序中最主要指标参数是数据质量Q30值和测序数据量统计结果见图4-5。其中百分比≥Q30代表每轮测序碱基的质量不低于99.9%所占的百分比。统计T2样品测序最终的Q值分布(横坐标)和得到的数据量(纵坐标,单位是million)。统计得到全部测序总数据量为5.8G,数据质量≥Q30的占94.3%。最终统计得到的T2样品原始测序数据量(Raw Data)为3.725 G。进一步生物信息过滤处理得有效可用数据(clean data)为3.705 G,数据有效率为99.46%,Q20值为94.09%,Q30值为92.85%;GC含量比例为48.19%。结果满足后续对T2测序样品的生物学研究分析的基本要求。
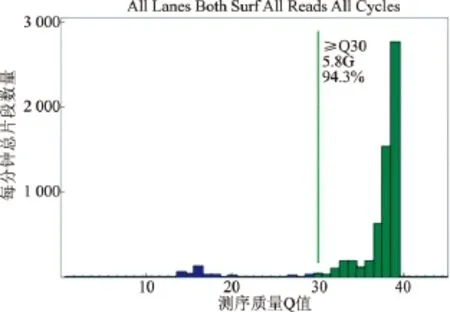
图4 样品T2在Miseq上机测序得到的全部数据Q值(Quality Score)分布统计Fig.4 Statistic of the QScore distribution on illumina Miseq
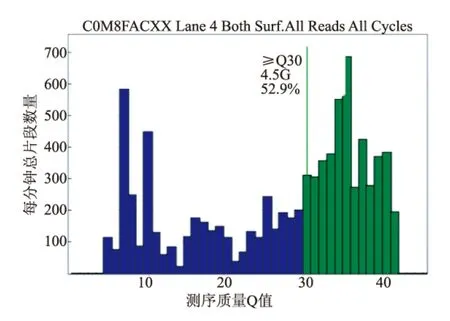
图5 Hiseq2000的测序芯片 (flowcell)中第4条测序通道(lane 4)里所有样品都只是PCR产物样品测序时的数据Q30分布统计Fig.5 Statistic of the Qscore distribution without the phix standard mixture on lane 4 at Hiseq 2000
2.3 上机测序实验优化分析
当前二代高通量测序正突飞猛进地不断进行技术革新,但以Illumina为平台的边合成边测序的核心技术原理保持不变[2]。在上机测序过程中前25个碱基测序循环(cycle)结果质量的统计方法无法改变,依旧决定了整个测序的数据质量。测序中监测参数Cluster PF(Pass Filter)仍是重要的上机质检标准。当测序的前25个碱基低质量的数据有两个以上(即PF<60%),测序仪器将判定这条读长质检不通过。而PCR产物样品DNA多样性差,在测序中极易引起两个碱基的识别可靠性低于60%,从而减损最终测序的Q30值和判定合格测序数据总量,见图6。图4为Illumina Hiseq2000测序中的第5测序通道(Lane 5)标准基因组样品测序监测的%Base正常分布情况,Cluster PF(%)为95.23±1.15,可获得的数据量约34 G;图7为同时测序中第4通道(Lane 4)的样品当全为PCR产物时%Base分布情况,Cluster PF(%)为21.24±14.83。使最终得到的合格数据量和Q30的统计值降低,测序通道Lane 4里全部测序数据质量≥Q30仅为52.9%,最终获得的测序数据量降低到4.5 G。
作者尝试将待测的PCR产物样品T2中混合了等摩尔的Phix基因组标准品后进行Miseq测序,监测得到的Cluster PF(%)为89.81±0.72。最终目的样品T2占整个有效测序数据量的61.77%,大大改善了PCR混合物测序样品的数据质量和有效数据总量。对于多样性差的其它待测序DNA样品,比如RRBS 样 品 (Reduced Representation Bisulfite Sequencing,简化的表观亚硫酸氢盐测序),由于含有固定的酶切位点序列降低了样品的多样性,但通过混入一定量比例的多样性好的基因组标准品,实验结果显示得到的测序数据质量可提高约50%。
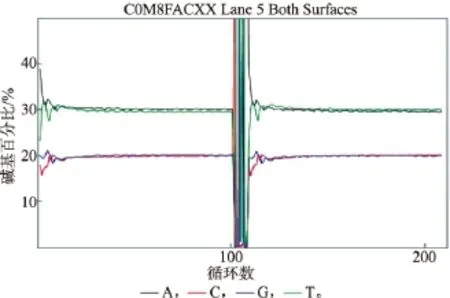
图6 在Illumina Hiseq2000测序仪器中监测的第5条测序通道标准基因组样品的碱基分布图Fig.6 Base distribution of genomic DNA sequencing on lane 5 of Illumina Hiseq 2000
目前还可以尝试降低待测序样品上样量的方法改善低多样性样品引起的低数据质量的情况。但研究表明至少要降低一半的PCR样品量才可能达到略微的效果,也大大折损了测序数据总量,并非经济有效的方案。而选择将多样性好的基因组样品掺入到多样性差的样品中上机测序的方法,实验操作简单,能改善整个测序通道的样品多样性表现,从而提高测序质量,同时也能保证得到的最终有效的测序数据总量损失最少。

图7 第4条测序通道为全部PCR产物样品测序时的碱基分布图Fig.7 Base distribution of all PCR samples sequencing on lane 4 of Illumina hiseq 2000
3 结语
作者研究探索了一套对普通PCR产物样品的高通量测序建库和上机实验方法,对低多样性样品的高通量测序方法做了探索研究,通过对样品的上机测序数据监测,证实所建立的方法保证样品制备的高效经济和测序质量的稳定可靠。
[1]MARDIS E R.A decade’s perspective on DNA sequencing technology[J].Nature,2011,470(7333):198-203.
[2]METZKER M L.Sequencing technologies-the next generation[J].Nat Rev Genet,2010,11(1):31-46.
[3]NIEDRINGHAUS T P,Milanova D,Kerby M B,et al.Landscape of next-generation sequencing technologies[J].Anal Chem,2011,83(12):4327-4341.
[4]王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114. WANG Xingchun,YANG Zhirong,WANG Min,et al.High-throughput sequencing technology and its application[J].China Biotechnology,2012,32(1):109-114.(in Chinese)
[5]MCCANN J C,WICKERSHAM T A,Loor J J,et al.High-throughput methods redefine the rumen microbiome and its relationship with nutrition and metabolism[J].Bioinformatics and Biology Insights,2014,8:109-125.
[6]MOROZOVA O,MARRA M A.Applications of next-generation sequencing technologies in functional genomics[J].Genomics,2008,92(5):255-264.
[7]MARDIS E R.Next-generation sequencing platforms[J].Annu Rev Anal Chem(Palo Alto Calif),2013(6):287-303.
[8]YU C,ZHANG Y,YAO S,et al.A PCR based protocol for detecting indel mutations induced by TALENs and CRISPR/Cas9 in Zebrafish[J].PLoS One,2014,9(6):e98282.
[9]NELSON M C,MORRISON H G,BENJAMINO J,et al.Analysis,optimization and verification of Illumina-generated 16S rRNAgene amplicon surveys[J].PLoS One,2014,9(4):e94249.
[10]BOUTIN S,SEVELLEC M,Pavey S A,et al.A fast,highly sensitive double-nested PCR-based method to screen fish immunobiomes[J].Mol Ecol Resour,2012,12(6):1027-1039.
[11]ZHOU Y,ZHU S,CAI C,et al.High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J]. Nature,2014,509(7501):487-491.
[12]CAPORASO J G,LAUBER C L,WALTERS W A,et al.Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J].ISME J,2012,6(8):1621-1624.
[13]AMIR A,ZEISEL A,ZUK O,et al.High-resolution microbial community reconstruction by integrating short reads from multiple 16S rRNA regions[J].Nucleic Acids Res,2013,41(22):e205.
[14]BOWMAN S K,SIMON M D,DEATON A M,et al.Multiplexed Illumina sequencing libraries from picogram quantities of DNA [J].BMC Genomics,2013,14:466.
Methods and Optimization of High Throughput Sequencing Technologies on PCR Products
LI Guilan1, XU Chuanlai2
(1.School of Life Sciences,Peking University,Beijing 100871,China;2.State Key Laboratory of Food Science &Technology,Jiangnan University,Wuxi 214122,China)
The development of the new generation sequencing technology extends the application field of gene sequencing.High throughput sequencing of PCR products has been widely used in functional gene screening,mutation and methylation detection of tumor related genes,etc.In the high throughput sequencing technology,database and computer sequencing experiments are the key factor to decide the quality of DNA data.Based on Illumina sequencing instruments,the sample preparation method and sequencing running for PCR products were optimized,the results make us clearly understand the primary features of high-throughput sequencing techniques,and provide us important references of sequencing methods for low diversity DNA samples to address biological questions of interest.
PCR products,high-throughput sequencing,sample library preparation,sequencing runs
Q 523.8
A
1673—1689(2016)12—1317—06
2015-02-03
国家“十二五”科技支撑计划项目(2012BAC01B071)。
李桂澜(1980—),女,重庆人,理学博士,工程师,主要从事DNA测序技术的应用及生物分子相互作用方面的研究。E-mail:liglan@pku.edu.cn

