水稻籼粳亚种分化关联性状的QTL分析
郑秀娟, 解慧芳, 曹红瑞, 杨桂英, 谢长荣, 梁康迳, 孙新立
(1.福建农林大学作物遗传育种与综合利用教育部重点实验室;2.福建农林大学作物遗传改良研究所作物种质资源创新与利用研究室,福建 福州 350002)
水稻籼粳亚种分化关联性状的QTL分析
郑秀娟1, 解慧芳1, 曹红瑞1, 杨桂英2, 谢长荣2, 梁康迳2, 孙新立1
(1.福建农林大学作物遗传育种与综合利用教育部重点实验室;2.福建农林大学作物遗传改良研究所作物种质资源创新与利用研究室,福建 福州 350002)
摘要:亚洲栽培稻分为籼粳两个亚种,程氏形态指数法已经广泛应用于籼粳分类的研究和生产实践中,但籼粳分化相关性状的遗传基础还多不清楚.本研究以典型粳稻牡丹江8号和典型籼稻明恢63的杂交F2群体为材料,构建遗传连锁图,检测到了11个籼粳分化相关性状(稃毛长、粒长、粒宽、倒一节间长和抽穗期)的QTL.同时定位了13个与这些性状高度相关的株高、穗长和护颖长的QTL.结果表明,检测到5个控制稃毛长的QTL,最大贡献率为25.57%;4个控制穗长的QTL,总贡献率为47.13%;4个控制抽穗期的QTL,分别位于第3、第7染色体上;控制粒长和控制粒长的QTL各检测到1个,贡献率分别为38.03%、20.56%.本研究为相关性状基因的克隆打下了基础,并加深了人们对籼粳分化的认知.
关键词:水稻; 籼粳分化; 遗传连锁图; QTL
亚洲栽培稻(OryzasativaL.)是最重要的粮食作物之一,约8000-9000年前从普通野生稻驯化而来[1,2].亚洲栽培稻分为籼、粳两个亚种,籼稻进一步分为籼型和aus型;粳稻分为温带粳稻和热带粳稻[3].籼粳两亚种在长期的自然和人工选择下,表现出丰富的多态性.在籼、粳两亚种的鉴别上,众多学者依据二者之间的差异,建立了不同分类标准.程侃声等[4]提出了“程氏形态指数法”,通过对水稻稃毛、酚反应、谷粒长宽比、抽穗时颖壳色、倒一节间长、叶毛有无等6个性状区分籼粳亚种,其中前4个性状可以区分95%以上的品种[4].此方法简单快速,但需要丰富的经验.Oka[5]提出采用判别式进行籼粳分类,判别因子包括酚反应、氯酸钾抗性、稃毛长度和低温敏感性.这一判别式可以将误分率降低到0.4%,但操作变得复杂.同工酶技术及电泳技术的发展使得籼粳分化的研究深入到蛋白质水平.Glaszmann et al[6]利用15个同工酶位点,将亚洲栽培稻分为6组,其中Ⅰ,Ⅱ和Ⅲ属于籼稻,Ⅳ,Ⅴ和Ⅵ属于粳稻.孙新立等选择6个籼粳分类表现好的位点,建立了同工酶分类的判别式[7].
随着RFLP、SSR和SNP等分子标记技术的飞速发展和应用,为籼粳的分类、分化提供了更深一步的证据[3,8,9].首个克隆、涉及籼粳分类性状的基因是控制酚反应的基因—Phr1[10].该基因在粳稻中编码一个失活的氧化酶,因而酚反应不能使粳稻谷粒着色[10].水稻稃毛长度是划分籼粳稻的主要性状之一,籼稻短而整齐、粳稻长而密,且多集中在谷粒顶部[4].Sato et al[11,12]认为稃毛长度受一个隐性主效基因控制,并将该基因位于第6染色体,命名为aph与Est-2和Pgi-2连锁.钱等也在第6染色体定位了一个数量性状座位(quantitative trait locus, QTL),但位置与Sato et al[13]研究结果不同.而Cai et al[14]发现稃毛长度受多个基因控制并检测到11个QTL.乔宝健等[15]利用籼粳杂交的BIL群体在2个不同地点,分别检测到5个和7个控制水稻倒一节间长度的QTL,其中有4个相同.He et al[16]利用单染色体片段代换系,检测出4个控制水稻倒一节间长QTL.但仅有第1染色体上的QTL可能与乔等的相同.目前,尚未见到克隆控制该性状QTL的报道.但基于对株高或倒一节间长的突变体的分析,克隆了3个(OsLIS1、EUI1和SUI1)调控该性状的基因[17-19].但根据这3个基因突变体的性状分析,似乎与涉及籼粳分类无关.籽粒长宽比是另一个重要的涉及籼粳分类的性状,分别由粒长和粒宽基因控制,这类基因已有多个被克隆,但不清楚哪几个基因涉及籼粳分类性状[20-27].故籼粳分化相关性状的遗传基础,除酚反应外,还所知甚少.
本研究以典型粳稻牡丹江8号和典型籼稻明恢63杂交的F2群体为材料,选取122个SSR标记构建分子连锁图,定位了多个籼粳分化相关性状(稃毛、粒长、粒宽、生育期和倒一节间长等)的主效QTL,为这些性状的基因克隆奠定了基础.
1材料与方法
1.1种植方法及性状考察
本研究以典型籼稻品种明恢 63(MH63)与典型粳稻品种牡丹江8号(MDJ8)为亲本,构建含有186个株系的F2群体.供试材料于2012年4月10日浸种,催芽露白后随机点播于25 cm×20 cm塑料育秧盘中,5月5日移栽至福建农林大学作物遗传改良研究所作物种质资源创新与利用研究室网室内,株行距18 cm×20 cm,采用常规的田间栽培管理,力求均匀一致,并及时进行病虫害防治.
田间考察MDJ8/MH63的F2群体的抽穗期、株高、穗长、倒一节间长;收获种子后,加标尺,用带MP-E 65 mm镜头的CANON相机放大3-5倍拍照,Image J软件辅助测量稃毛、粒长、粒宽、外护颖和内护颖的长度,记录数据并进行统计分析.
1.2性状表型值及相关性分析
采用SPSS 13.0软件的频率分布对F2群体各性状表型值进行分析(P<0.05),得出性状分析表和频率分布图,同时通过该软件计算两两性状的相关系数(P<0.05),分析各个性状的相关性.
1.3PCR检测、SSR遗传连锁图和QTL定位
选取群体中约20天的幼嫩叶片,采用Dellaporta et al[28]提出的SDS抽提法提取水稻基因组DNA.并将每份DNA稀释至20 ng·μL-1,进行PCR检测.
选取均匀分布于水稻12条染色体上的SSR分子标记612对,分别对MDJ 8号、MH 63及F1代进行多态性筛选.利用多态性分子标记分别对F2群体进行检测,并统计带型.运用JoinMap 4.0作图软件,以LOD=2.5为阈值,采用Kosambi函数将重组率转化成遗传图距(cM),构建遗传连锁图谱.QTL分析采用QTL IciMapping version 3.2(www.isbreeding.net)软件中ICIM-ADD和Windows QTL Cartographer 2.5[29](简写WinQTLCart 2.5)软件中CIM,各个性状的LOD阈值采用Permutation test(1000次,Type I error小于0.05)获得.
2结果与分析
2.1F2群体及亲本表型值分析
MH 63和MDJ 8分别是典型的籼稻和粳稻,其F2群体变化范围广,株高、穗长、倒一节间长、稃毛长等各性状都存在一定程度的超双亲分离(图1).F2群体中,穗长、稃毛长、粒长、粒宽的峰度和偏度的绝对值接近于0,最接近正态分布.群体株高、倒一节间长均值超越了高值亲本MH 63.所研究的性状表现明显的数量性状遗传特征.
2.2所考察性状间的相关性分析
为证实抽穗期、株高、倒一节间长、穗长、粒长和内外护颖间可能存在的相关性.本研究采用SPSS 13.0,计算了所研究性状间的相关系数(表1).结果表明,抽穗期、株高、倒一节间长和穗长4个性状间存在极显著的正相关,穗长和株高间相关系数高达0.7.内外护颖长度之间相关系数高达0.81,可能受相同的基因控制.护颖长度与粒长存在极显著的正相关(表1).

表1 F2群体各个性状相关分析1)
1)**表示P<0.01,*表示P<0.05.
2.3分子遗传连锁图谱的构建和QTL分析
采用122个SSR标记构建水稻全基因组遗传连锁图,总图距为1660.0 cM,相邻标记平均遗传距离为13.5 cM,每个连锁群上的标记数为8~14个,遗传距离为89.4~202.9 cM(图2).
依据构建的遗传连锁图,本研究采用2种方法对所考察的9个性状进行QTL分析.采用ICIM-ADD共检测到24个QTL(图2,表2),分布在第1~7染色体上,单个表型贡献率从6.12%到38.03%.第3染色体上检测到9个QTL.本研究亦用WinQTLCart 2.5-CIM检测相关性状,共检测到26个QTL(资料未显示),其中有21个QTL为2种方法同时检测到.而仅有1种方法检测到的QTL,贡献率均较低.
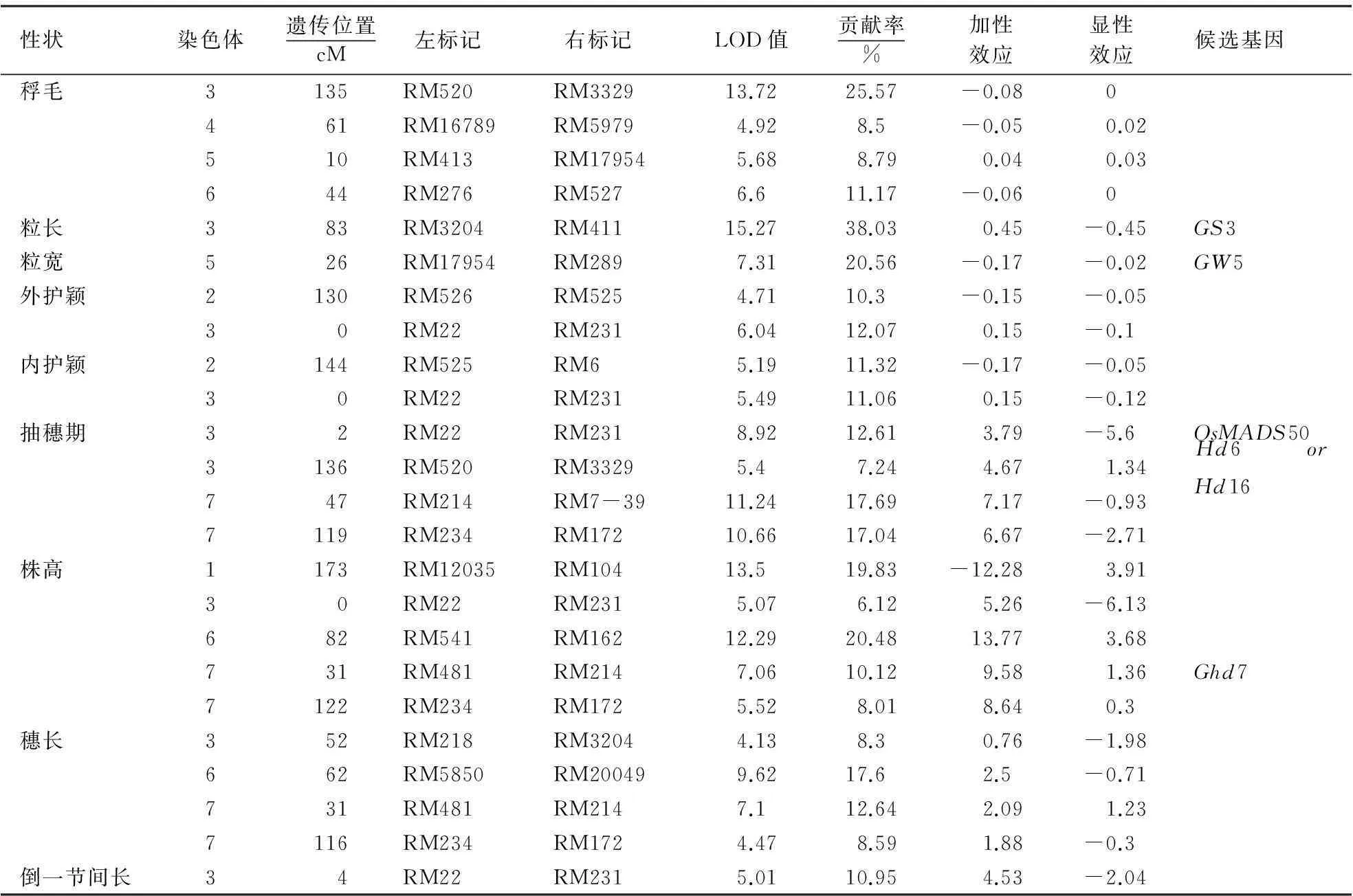
表2 QTL定位和效应分析
结合文献分析和搜索NCBI等数据库,发现本研究定位的QTL区间包含已经克隆的相关基因.MH63含有突变的GS3[20],而MDJ8含有突变的Ghd7[25].在二者所在的位置,都检测到相关的QTL.因控制粒宽的QTL区间包含GW5/qSW5基因,因此,本研究测序分析了MDJ8的GW5/qSW5基因,发现它带有粒宽基因gw5/qsw5[25,30].此外,第3染色体RM22-RM231区间,包含抽穗期基因Hd6或Hd16[31,32].RM520-RM3329区间包含抽穗期基因OsMADS50(表2)[33]. 上述研究表明,QTL的分析结果和主效QTL的定位是非常可靠的.
第3染色体前端和第7染色体的两个区段,存在多个QTL聚集的密集区(图2,表2).第3染色体前端检测到控制株高、倒一节间长、抽穗期和内外护颖的QTL在同一区间;第7染色体上,控制穗长,株高和抽穗期的基因在同一区间(图2).然而,第3染色体的QTL密集区,包含OsMADS50,该基因的突变,改变了水稻的抽穗期、株高[33].Ghd7基因包含在第7染色体的RM214-RM7-39区段内,该区段抽穗期QTL波峰覆盖RM481-RM7-39,包含株高和穗长QTL.此外,这些QTL聚集在一起,较好的解释了株高、穗长、倒一节间长和抽穗期间的相关性(表2).
3讨论
本研究采用两种方法检测了MDJ8/MH63 F2群体中的籼粳分化相关的QTL,两种检测方法检测到的QTL多数重叠.另外,一些MDJ8和MH63中已可隆的基因,如GS3、GW5和Ghd7,在定位结果中均检测出来,这些说明本研究的QTL分析结果,特别是主效QTL的定位是非常可靠的.
稃毛长度是籼粳程氏指数分类法中的重要指标之一,只采用稃毛长度对籼粳分类,86.5%的品种可以正确的分到相应的亚种中[5].对稃毛长度QTL的定位可以为籼粳分类指标提供遗传依据.本研究采用带有高分辨率、高放大倍数镜头的相机拍照,Image J软件辅助,精确测量稃毛长度,共检测到4个控制稃毛长度的QTL,能解释54.0% 的群体总变异.其中,贡献率最大的两个QTL分别位于第3和6染色体,与Cai et al[14]检测到的QTL重叠.位于第4、第5染色体上的QTL可能是新的控制稃毛长的QTL.
籽粒长宽比是籼粳分类的另一指标,若单采用该指标的误分率高达39.0%[5].本研究分别定位了控制籽粒长度和宽度的QTL,各检测到1个贡献率较大的QTL(表2).这两个QTL很可能是gs3和gw5/qsw5[20,25,30].本研究也尝试直接采用复合性状谷粒长宽比定位QTL,分别在粒长和粒宽QTL区检测到控制长宽比的2个QTL(结果未显示).这些结果表明,用于籼粳分类的长宽比指标可分解为粒长和粒宽两个因素.MH63中的GS3突变类型广泛存在于栽培稻中,Takano-Kai等[23]对235份栽培稻和284份野生稻的分析发现,34%的栽培稻和4%普通野生稻带有该基因的突变体.其中,这一突变体主要分布在热带粳稻和籼稻中.GW5/qSW5的突变体主要分布在粳稻和宽粒籼稻中[25,30].这些结果暗示,这两个基因可能是控制籼粳分类的主要基因.钱前等分别在第1、2和3染色体上检测到控制籽粒长宽比的QTL,分析其定位结果,在其QTL所覆盖的物理区间,与本研究检测到的不同,同时也没有发现已克隆的基因处在这些区间[13].这些表明可能还有其它粒形基因涉及籼粳分类.
水稻光周期基因不仅影响水稻的抽穗期,而且影响水稻的株高、倒一节间长和穗长[25,34].分布于不同纬度的品种,因其涉及光周期基因的序列及表达差异,表现出不同的光温反应[35,36].本研究分别在第3染色体的RM22-RM231区间,第7染色体的两个区间检测到涉及光温反应的QTL.其中第3染色体的该QTL区间包含OsMADS50[33],而第7染色体的一个区间包含Ghd7基因[25].这两个基因都属于多效基因,同时影响抽穗期、株高和穗长[30,33,37].然而,本研究仅检测到一个涉及倒一节间长的QTL,在第3染色体的RM22和RM231区,但贡献率只有10.9%.这一结果是否意味着籼粳稻间倒一节间长的差异,是由于其对光温反应的不同造成的呢?这些,尚待深入研究.
用于籼粳分类的形态及生理性状,除酚反应外,都是由多基因控制.Cai et al[14]研究表明,可以区分97.1%的籼粳品种的氯酸钾抗性,亦由多基因控制.这为相关基因的克隆增加了难度.本研究的QTL定位,尤其是稃毛QTL的定位,为其进一步的克隆奠定了基础;同时抽穗期、倒一节间长、株高和穗长间的相互关系的揭示,加深了人们对籼粳分化性状的认知.
参考文献
[1] HIGHAM C, LU T L D. The origins and dispersal of rice cultivation[J]. Antiquity, 1998(72):867-877.
[2] FULLER D Q, SATO Y-I, CASTILLO C, et al. Consilience of genetics and archaeobotany in the entangled history of rice[J]. Archaeol Anthropol Sci, 2010(2):115-131.
[3] HUANG X, KURATA N, WEI X, et al. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature, 2012,490(7421):497-501.
[4] 程侃声,周季维,卢义宣,等.云南稻种资源的综合研究与利用Ⅱ. 亚洲栽培稻分类的再认识[J].作物学报,1984(10):271-280.
[5] OKA H I. Origin of Cultivated Rice[M]. Tokyo; Japan Scientific Societies Press, 1988.
[6] GLASZMANN J C. Isozymes and classification of Asian rice varieties[J]. Theor Appl Genet, 1987(74):21-30.
[7] 孙新立,才宏伟,王象坤.同工酶基因数量化方法对亚洲栽培稻的分类研究[J].作物学报,1996,22(6):693-639.
[8] GARRIS A J, TAI T H, COBURN J, et al. Genetic structure and diversity inOryzasativaL[J]. Genetics, 2005,169(3):1631-1638.
[9] RESURRECCION A P, VILLAREAL C P, PARCO A, et al. Classification of cultivated rices intoindicaandjaponicatypes by the isozyme, RFLP and two milled-rice methods[J]. Theor Appl Genet, 1994,89(1):14-18.
[10] YU Y, TANG T, QIAN Q, et al. Independent losses of function in a polyphenol oxidase in rice: differentiation in grain discoloration between subspecies and the role of positive selection under domestication[J]. Plant Cell, 2008,20(11):2946-2959.
[11] SATO Y E. Genetic control of apiculus hair length[J]. Rice Genetics Newsletter, 1985(2):72-74.
[12] SATO T I, ISHIKAWA R, MORISHIMA H. Linkage analysis of geneAphfor apiculus hair length[J]. Rice Genetics Newsletter, 1987(4):74-5.
[13] 钱前,何平,郑先武,等.籼粳分类的形态指数及其相关鉴定性状的遗传分析[J].中国科学(C辑),2000,30(3):305-310.
[14] CAI W, MORISHIMA H. QTL clusters reflect character associations in wild and cultivated rice[J]. Theor Appl Genet, 2002,104(8):1217-1228.
[15] 乔保建,王盈盈,朱晓彪,等.不同生长环境下水稻最上节间长度QTL定位研究[J].遗传,2007,29(8):1001-1007.
[16] HE F, XI Z, ZENG R, et al. Identification of QTLs for plant height and its components by using single segment substitution lines in rice[J]. Rice Science, 2005,12(3):151-156.
[17] GAO X, CHEN Z, ZHANG J, et al.OsLIS-L1 encoding a lissencephaly type-1-like protein with WD40 repeats is required for plant height and male gametophyte formation in rice[J]. Planta, 2012,235(4):713-727.
[18] LUO A, QIAN Q, YIN H, et al.EUI1, encoding a putative cytochrome P450 monooxygenase, regulates internode elongation by modulating gibberellin responses in rice[J]. Plant Cell Physiol, 2006,47(2):181-191.
[19] ZHU L, HU J, ZHU K, et al. Identification and characterization ofSHORTENEDUPPERMOSTINTERNODE1, a gene negatively regulating uppermost internode elongation in rice[J]. Plant Mol Biol, 2011,77(4-5):475-487.
[20] FAN C, XING Y, MAO H, et al.GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]. Theor Appl Genet, 2006,112(6):1164-1171.
[21] MAO H, SUN S, YAO J, et al. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proc Natl Acad Sci U S A, 2010,107(45):19579-19584.
[22] LI Y, FAN C, XING Y, et al. Natural variation inGS5 plays an important role in regulating grain size and yield in rice[J]. Nat Genet, 2011,43(12):1266-1269.
[23] TAKANO-KAI N, JIANG H, KUBO T, et al. Evolutionary history ofGS3, a gene conferring grain length in rice[J]. Genetics, 2009,182(4):1323-1334.
[24] SONG X J, HUANG W, SHI M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nat Genet, 2007,39(5):623-630.
[25] XUE W, XING Y, WENG X, et al. Natural variation inGhd7 is an important regulator of heading date and yield potential in rice[J]. Nat Genet, 2008,40(6):761-767.
[26] ZHANG X, WANG J, HUANG J, et al. Rare allele ofOsPPKL1 associated with grain length causes extra-large grain and a significant yield increase in rice[J]. Proc Natl Acad Sci U S A, 2012,109(52):21534-21539.
[27] WANG S, WU K, YUAN Q, et al. Control of grain size, shape and quality byOsSPL16 in rice[J]. Nat Genet, 2012,44(8):950-954.
[28] DELLAPORTA S, WOOD L H, HICKS J B. A plant DNA minipreparation[J]. Plant Mol Biol Rep, 1983(1):19-21.
[29] WANG S, BASTEN C J, ZENG Z B. Win QTL Cartographer 2.5. [EB/OL]. Raleigh:North Carolina State University, 2007.http://statgen.ncsu.edu/qtlcart/WQTLCart.htm.
[30] SHOMURA A, IZAWA T, EBANA K, et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nat Genet, 2008,40(8):1023-1028.
[31] TAKAHASHI Y, SHOMURA A, SASAKI T, et al.Hd6, a rice quantitative trait locus involved in photoperiod sensitivity, encodes the alpha subunit of protein kinase CK2[J]. Proc Natl Acad Sci U S A, 2001,98(14):7922-7927.
[32] HORI K, OGISO-TANAKA E, MATSUBARA K, et al.Hd16, a gene for casein kinase I, is involved in the control of rice flowering time by modulating the day-length response[J]. Plant J, 2013,76(1):36-46.
[33] LEE S, KIM J, HAN J J, et al. Functional analyses of the flowering time geneOsMADS50, the putativeSUPPRESSOROFOVEREXPRESSIONOFCO1/AGAMOUS-LIKE20 (SOC1/AGL20) ortholog in rice[J]. Plant J, 2004,38(5):754-764.
[34] YAN W H, WANG P, CHEN H X, et al. A major QTL,Ghd8, plays pleiotropic roles in regulating grain productivity, plant height, and heading date in rice[J]. Mol Plant, 2011,4(2):319-330.
[35] HUANG C L, HUNG C Y, CHIANG Y C, et al. Footprints of natural and artificial selection for photoperiod pathway genes inOryza[J]. Plant J, 2012,70(5):769-782.
[36] ITOH H, IZAWA T. The coincidence of critical day length recognition for florigen gene expression and floral transition under long-day conditions in rice[J]. Mol Plant, 2013,6(3):635-649.
[37] RYU C H, LEE S, CHO L H, et al.OsMADS50 andOsMADS56 function antagonistically in regulating long day (LD)-dependent flowering in rice[J]. Plant Cell Environ, 2009,32(10):1412-1427.
(责任编辑:吴显达)
QTL analysis of the differentiation characteristics ofindicaandjaponicarice and associated traits
ZHENG Xiujuan1, XIE Huifang1, CAO Hongrui1, YANG Guiying2,
XIE Changrong2, LIANG Kangjing2, SUN Xinli1
(1.Fujian Agriculture and Forestry University, Provincial and Ministerial Key Laboratory of Crop Genetics Breeding and Comprehensive Utilization; 2.Fujian Agriculture and Forestry University, Institute of Plant Genetics and Breeding, Fuzhou, Fujian, 350002)
Abstract:Morphological index method has been widely applied in distinguishing two subspecies of Asian cultivated rice, indica and japonica, and in production. But genetic bases of characteristics that related to differentiation of indica and japonica is still unclear. Therefore, we constructed a genetic linkage map of the F2 population that crossed between a typical japonica Mudanjiang 8 and a typical indica Minghui 63 and identified 11 QTLs of indica-japonica differentiation traits, including apiculus length, first internode length, grain length, grain width and heading date. We also detected 13 QTLs that associated with plant height, panicle length, and empty glume length, which highly correlated with one or two upper traits. To summarize, among the 13 QTLs, 5 QTLs related to apiculus length and the highest contribution rate is 25.57%; 4 QTLs controlled panicle length with a total contribution rate of 47.13%; 4 QTLs controlled heading date which located in chromosome 3 and 7; 1 QTL related to grain length and 1 to grain width, whose contribution rate was 38.03% and 20.56%, respectively. The study may facilitate the map-based cloning, and offer deeper insight to the indica-japonica differentiation.
Key words:rice; indica-japonica differentiation; linkage-map; QTL
DOI:10.13323/j.cnki.j.fafu(nat.sci.).2016.01.001
中图分类号:S511
文献标识码:A
文章编号:1671-5470(2016)01-0001-07
作者简介:郑秀娟(1987-),女,研究实习员,硕士.研究方向:分子生物学.Email:chenxizxj@126.com.通讯作者孙新立(1965-),男,教授,博士.研究方向:植物分子生物学.Email:xinlisun@hotmail.com.
基金项目:国家自然科学基金(31371557).
收稿日期:2015-04-07修回日期:2015-05-20

