HIV-1逆转录酶抑制剂的3D-QSAR和分子对接研究
仝建波, 吴英纪, 白 敏
(陕西科技大学 化学与化工学院, 陕西 西安 710021)
HIV-1逆转录酶抑制剂的3D-QSAR和分子对接研究
仝建波, 吴英纪, 白敏
(陕西科技大学 化学与化工学院, 陕西 西安710021)
摘要:采用分子对接和三维定量构效关系研究了二芳基苯胺衍生物与HIV-1非核苷类逆转录酶的相互作用,并运用经典的三维全息原子场作用矢量的方法(3D-HoVAIF)和多元线性回归方法(MLR)研究了药物分子的化学结构与生物活性之间的关系.MLR建模得出的复相关系数(R(cum))为0.949、 留一法交互校验复相关系数(Q(CV))为0.799,从该结果可以看出,三维定量构效关系对化合物的抗艾滋病活性具有比较好的预测能力.最后,运用分子对接研究了小分子药物和大分子HIV-1逆转录酶的氨基酸活性残基之间的结合模式,对今后设计合成新的抗艾滋病药物具有很好的指导作用.
关键词:HIV-1逆转录酶; 三维定量构效关系; 多元线性回归; 分子对接
0引言
人类免疫缺陷病毒1型(HIV-1)是致病性的逆转录病毒,是艾滋病(获得性免疫缺陷综合征)的病原体[1],严重地威胁着人类的健康,至今为止依然没有可以完全治愈的药物.根据统计数据显示,2011 年约有170万人死于与艾滋病相关的疾病[2].
HIV-1是一种逆转录病毒,对于病毒复制,它会编码一个逆转录酶(RT)[3].对于HIV-1感染患者来说,由于药物具有合规性、负反应性和交叉耐药性,所以需要发展一种新的、强效的抗突变非核苷类逆转录酶抑制剂,这是一个具有高挑战性的任务[4].目前,二芳基苯胺衍生物已经引起了人们的高度重视,因为其与野生型的HIV-1逆转录酶菌株不同,具有较好的活性并且能够高度识别有效化合物[5,6].
本实验先采用三维全息原子场作用矢量法和多元线性回归法研究了药物的三维定量构效关系,然后运用分子对接方法对二芳基苯胺衍生物与HIV-1非核苷类逆转录酶残基之间的相互作用进行了分析研究,这为今后抗艾滋病药物的研究和发展提供了有利的基础.
1材料与方法
1.1软件与硬件
三维定量构效关系及分子对接均在Window.8系统下完成.其中,三维全息法涉及的软件有Chemdraw 12.0(用于画图)、Spss 12.0(用于逐步线性回归筛选)、应用二程序集(用于多元线性回归建模),以及AutoDock 4.2[7](用于分子对接).
1.2数据集
本实验采用24种二苯胺衍生物作为HIV-1逆转录酶抑制剂,实验活性值pEC50取自文献[8]中,其化合物的公共结构如图1所示、结构和活性数据如表1所示.
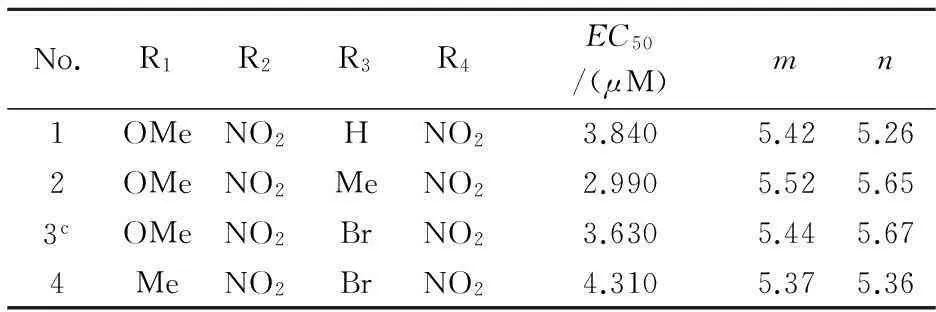
表1 二芳基苯胺衍生物的结构及EC50活性值
续表1
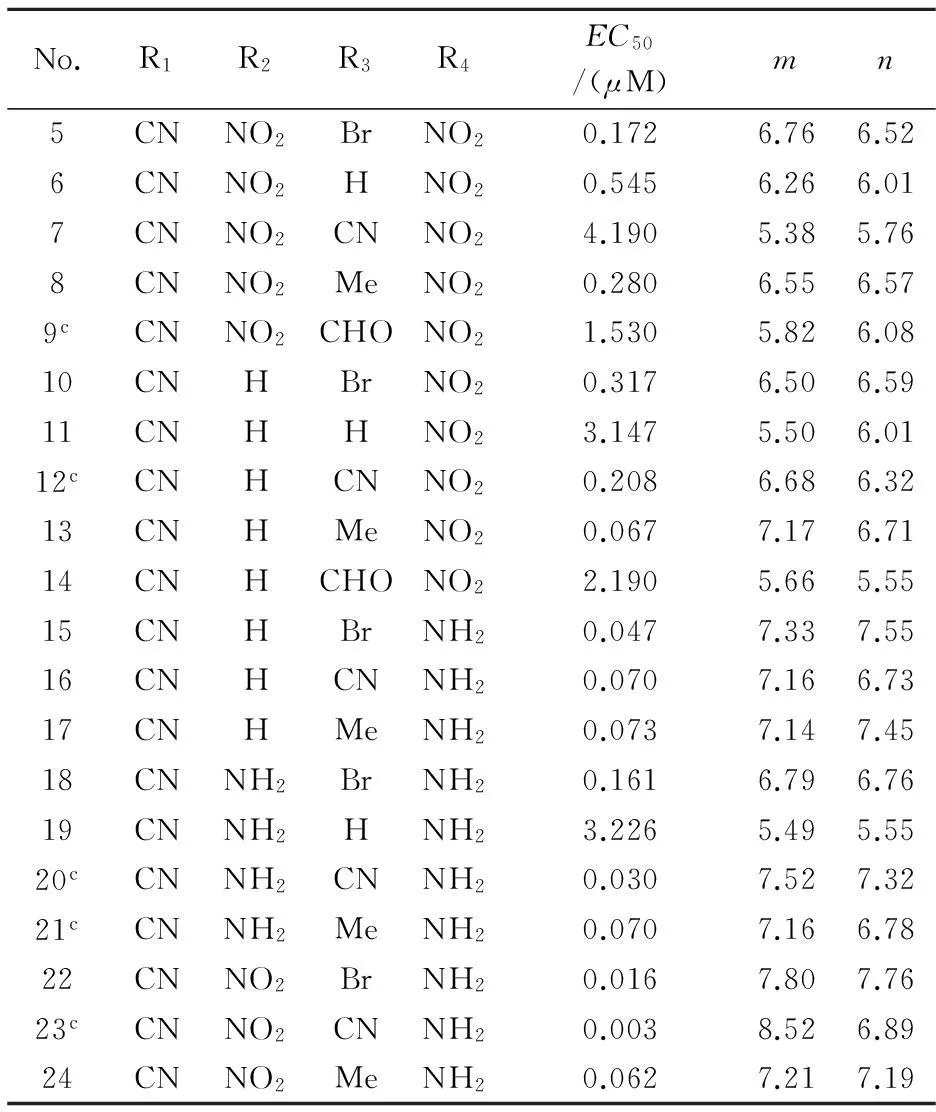
No.R1R2R3R4EC50/(μM)mn5CNNO2BrNO20.1726.766.526CNNO2HNO20.5456.266.017CNNO2CNNO24.1905.385.768CNNO2MeNO20.2806.556.579cCNNO2CHONO21.5305.826.0810CNHBrNO20.3176.506.5911CNHHNO23.1475.506.0112cCNHCNNO20.2086.686.3213CNHMeNO20.0677.176.7114CNHCHONO22.1905.665.5515CNHBrNH20.0477.337.5516CNHCNNH20.0707.166.7317CNHMeNH20.0737.147.4518CNNH2BrNH20.1616.796.7619CNNH2HNH23.2265.495.5520cCNNH2CNNH20.0307.527.3221cCNNH2MeNH20.0707.166.7822CNNO2BrNH20.0167.807.7623cCNNO2CNNH20.0038.526.8924CNNO2MeNH20.0627.217.19
注:m为实验活性值( Exp.(pEC50) );n为预测活性值((Pred.pEC50);c为测试集.
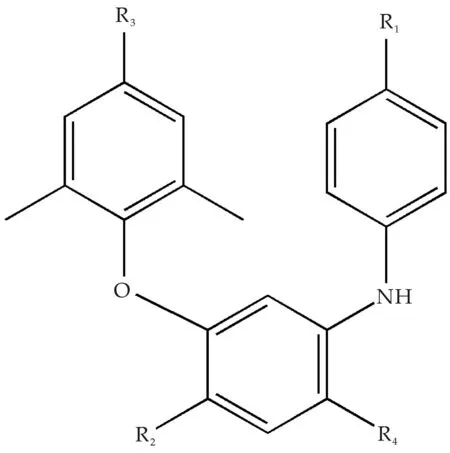
图1 二芳基苯胺衍生物的公共结构
1.3三维定量构效关系
三维全息原子场作用矢量(3D Holographic Vector of Atomic Interaction Field, 3D-HoVAIF)是一种描述三维分子结构的方法,已用于多种药物体系中[9].它将有机分子中常见的原子(氢、碳、氮、磷、氧、硫、氟、氯、溴和碘等)按其所处元素周期表的位置分为五类.并根据原子的杂化状态,这些原子又被分为十类.因此,在一个分子中就有55种相互作用.再基于以前的静电、立体和疏水性作用,一个有机化合物就会产生3×55=165种相互作用.
采用经典的库仑定理( Coulomb )(1)表示其静电相互作用(Electro Static Interaction);采用 Lennard-Jones方程(2)来表征立体作用 (Steric Interaction);采用Kellogg等提出的hint 方法(3)来定义计算两原子间的疏水作用(Hydrophobic Interraction).
(1≤m≤10, m≤n≤10)
(1)
在方程(1)中:rij是原子间Euclid 距离,单位nm;e是单位电荷电量1.602 189×10-19C;ε0为真空中的介电常数8.854 187 82×10-12C2/J·m;Z为原子净电荷数, 以电子为单位;m和n为原子所属种类.
(1≤m≤10, m≤n≤10)
(2)
在方程(2)中:εij为原子对势能,取文献值[10,11];D为经验推导的原子间作用能校正常数, 取0.01;Rij*=(Ch·Rii*+Ch·Rjj*)/2为校正后的原子对Vander Waals半径;校正因子Ch当sp3杂化时取1.00;sp2杂化时取0.95;sp杂化取0.90[12].
(1≤m≤10, m≤n≤10)
(3)
在方程(3)中:T是作用形式的二值判别函数,以表明不同类型原子疏水作用的熵效应变化方向;S为原子溶剂可及表面积(Solvent Accessible Surface Area of Atom,SASA),是以水分子(范德瓦尔斯半径为0.14 nm)为探针在原子表面滚动其球心形成的表面面积[13];a为原子疏水性常数,取文献值[14].
在3D-QSAR研究中,24个二芳基苯胺衍生物根据活性范围的高低被随机地分为18个训练集样本和6个测试样本.其中,训练集用于多元线性回归建模,测试集用于外部模型的有效验证.化合物的实验活性值和预测活性值如表1所示.
1.4分子对接方法
分子对接法是基于生物受体三维结构的一种药物设计方法,将小分子药物放置于受体的活性位点处,并寻找合理的取向和构象使得配体与受体的形状和相互作用达到最佳匹配构象.配体和受体的结合程度由结合过程中自由能的变化来决定.
分子对接采用AutoDock4.2软件,通过AutoGrid程序来测定活性位点,并运用拉马克遗传算法(LGA)进行优化.在这个算法中,先利用遗传算法进行全局搜索,然后采用局部搜索法进行能量优化.
利用半经验自由能的计算方法来评价配体与受体间的能量匹配,每一个化合物会产生10个对接构象,根据各构象的结合模式和结合自由能选择合适的构象来进行研究.其余计算参数采用应用程序的默认值.采用式(4)的函数形式计算[15].
(4)
在方程(4)中:ΔGvdw、ΔGH-bond、ΔGele、ΔGtor和ΔGsol都是半经验参数,可用拟合得到;Ntor指配体在对接后被约束的可旋转键的数目.
本研究选用24个二芳基苯胺衍生物中实验活性值最高的22号药物分子进行分子对接.HIV-1非核苷类逆转录酶受体的3D共晶结构取自生物信息学研究合作实验室(Research Collaboratory for Structural Bioinformatics,RCSB)的蛋白质数据库[16],它的PDB ID 为1S6Q,分辨度为3.0 Å.
在进行分子对接之前,先对配体小分子和HIV-1非核苷类逆转录酶受体分别进行优化处理(这些优化操作均在Chemdraw 12.0 中完成),包括删除受体中原有的配体、合并其中非极性氢原子、加电荷(Gasteiger )、去水,再对小分子进行加氢、去水、加电荷(Gasteiger)操作.实验中盒子的格点设为40×40×40,格点距离为0.037 5 nm,中心坐标为(147.889,-24.383,73.213),其它参数均采用程序默认数值.
2结果与讨论
2.1模型分析
运用三维全息得到165个变量,再使用逐步线性回归(Stepwise Multiple Regression,SMR)筛选得到17个非零变量.根据变量不超过样本数1/3的经验规则,从变量数为6开始计算.逐步回归按Fisher显著性检验依次引入变量,随变量的不同,每一步建模都会得到相应的复相关系数(Rcum)、标准偏差(SD)、交互校验复相关系数(QCV)和标准偏差(SDCV).当模型的Rcum≥0.6、QCV≥0.5时,认为此模型可行.最后,得出自变量数为6时结果最佳,其结果如下:
pEC50=6.660-130.531×S1-9+0.010×H5-8-75.182×E7-10+0.000×H2-2-33.024×E2-2-93.059×E5-10
N=18,Rcum=0.949,SD=0.322,F=16.726,
QCV=0.799,SDCV=0.460,FCV=7.285
其中,N为训练集样本数,Rcum为复相关系数,SD为标准偏差,F为F检验值,QCV、SDCV、FCV为交互校验的复相关系数、标准偏差、F检验值.
在模型中,S1-9指H原子与SP2杂化的O原子之间的立体作用,H5-8指SP3杂化的N原子与SP3杂化的O原子之间的疏水作用,E7-10指SP杂化的N原子与Br原子之间的静电作用,H2-2和E2-2分别指SP3杂化的C原子之间的疏水作用和静电作用,E5-10指SP3杂化的N原子与Br原子之间的静电作用.
运用多元线性回归模型来预测样本的活性值,并将得出的预测值与实验值做图,如图2所示.从图2可以看出,此药物模型的实验活性值与预测活性值为线性趋势,所有模型都在45 °线周围.模型中内部训练集的Rcum为0.949、QCV为0.799,外部测试集的Qext为0.736,从而可以总结出实验所构建的3D-QSAR模型具有良好的稳定性,而且此模型对同系列化合物的活性值均有好的预测能力.因为所参考文献[8]中的22号分子结构与其它分子相比,差异较大、不饱和键较多,所以明显偏离了45 °线,而且有些药物在实验测试条件下难以得出较理想的结果.但从图的基本趋势来看,此模型的预测能力还是比较可靠.
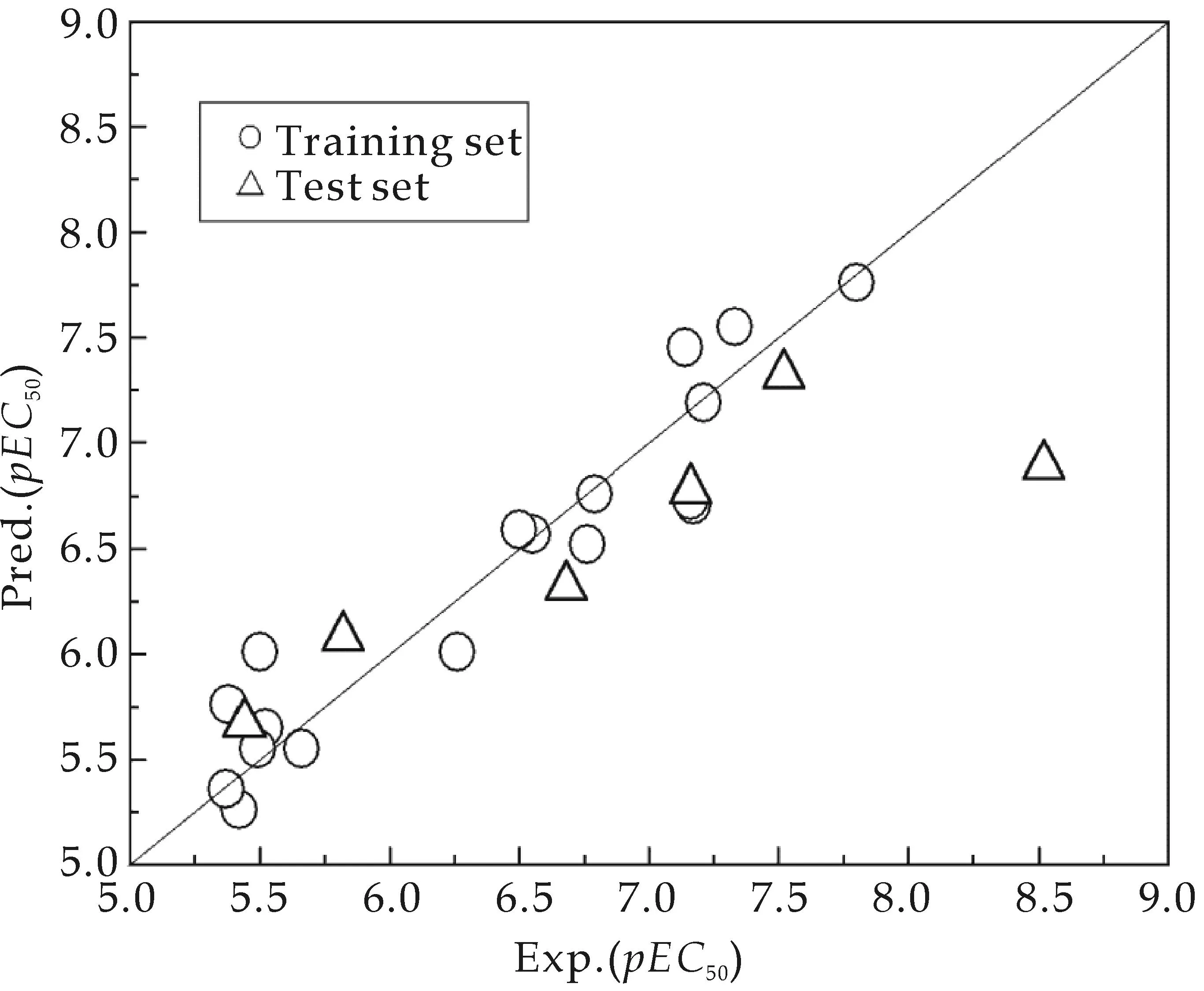
图2 二芳基苯胺衍生物的实验值与预测值散点图
2.2分子对接研究
实验随机选取了结构差异较大并且活性值相对较高的六个化合物(2、5、15、20、22、23)分别进行了分子对接,并将对接结果列于表2中,其预测的结合自由能(ΔG)与实验活性值的关系如图3所示.
从图3可以观察到,这几个点均在拟合线附近,这些化合物结合能呈逐渐降低的趋势,而其实验活性值呈逐渐增大的趋势,这说明化合物的预测结合自由能(ΔG)与实验活性值(pEC50)呈现较好的单调性,这与理论上这些化合物的活性值应该相对比较高的观点刚好吻合,表明了分子对接计算结果具有一定的可靠性.
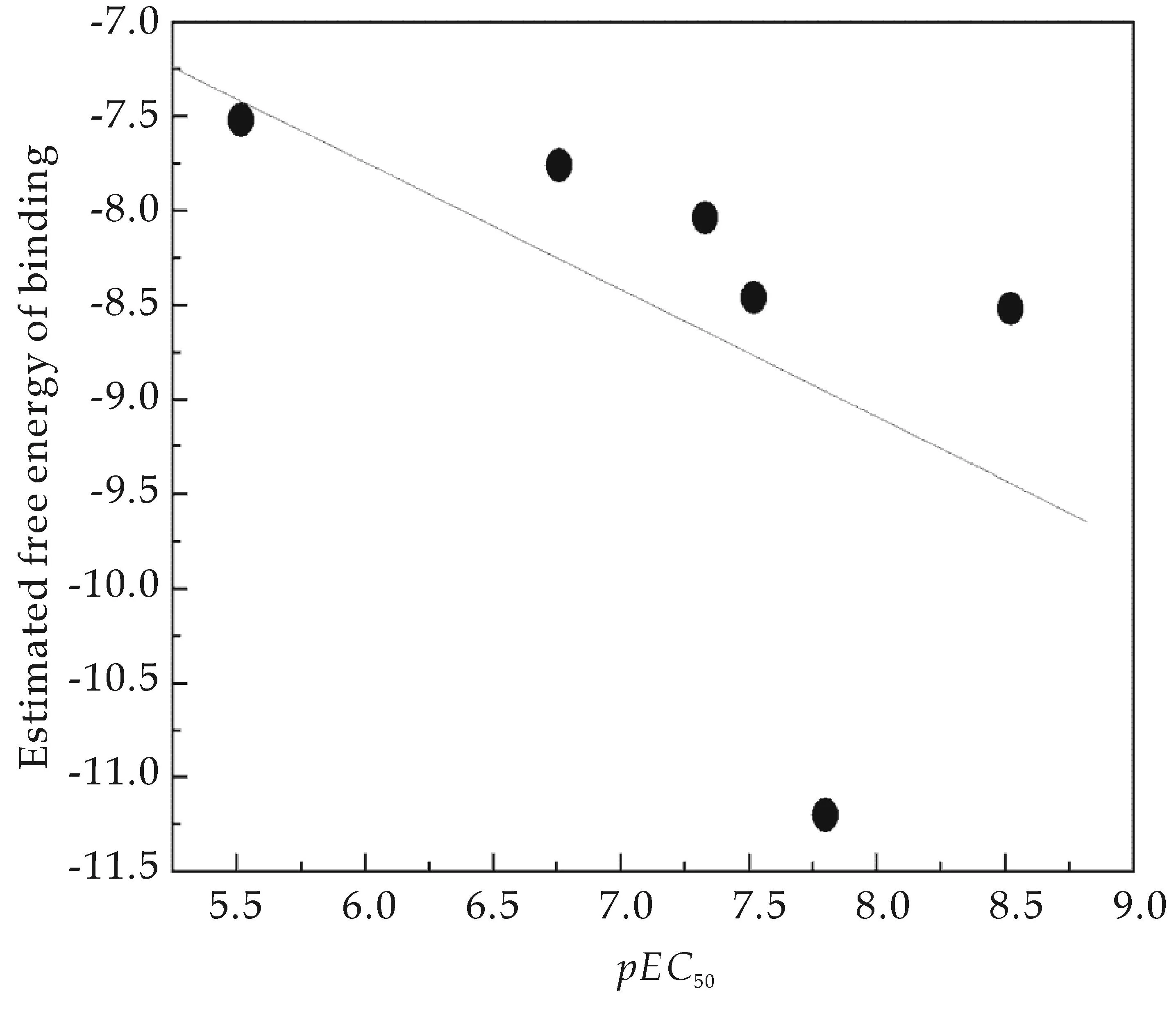
图3 部分化合物实验值与对接预测结合能的关系图
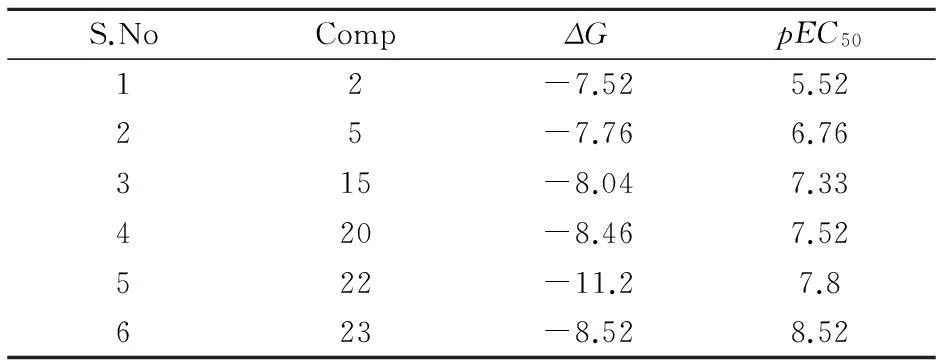
S.NoCompΔGpEC5012-7.525.5225-7.766.76315-8.047.33420-8.467.52522-11.27.8623-8.528.52
化合物中活性最高的22号二芳基苯胺衍生物与HIV-1逆转录酶残基的对接模型见图4所示.此构象是化合物分子与 HIV-1逆转录酶对接后10个构象中结合自由能最低的构象,其结合能是-11.4 kcal/mol,故也是结合比较稳定的模式.从图4可见,22号化合物与HIV-1逆转录酶活性部位氨基酸残基形成了两个氢键,其一是-NH中的SP3杂化H原子与LYS101的-OH基原子之间形成一个-NH…OH氢键,距离为2.0 nm;其二是-NH2中的SP3杂化H原子与LYS101的-OH基原子之间形成一个-NH2…OH氢键,距离为1.8 nm.
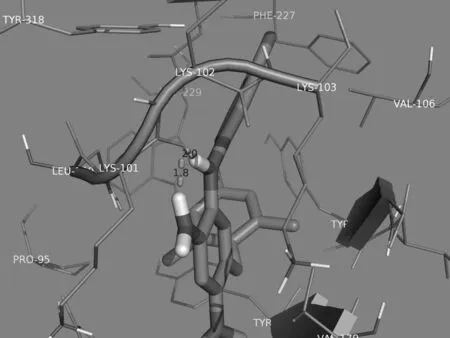
图4 22号二芳基苯胺衍生物与HIV-1逆转录酶的对接图
从对接后的结果可以看出,在配体与受体结合的过程中,氢键可以决定和影响配体在活性空腔中的位置[17].当小分子在大分子的活性位点上与大分子结合时,小分子的活性部位与大分子活性部位中极性氨基酸残基形成氢键,而小分子的疏水部分能够与大分子活性部位中非极性氨基酸残基产生疏水作用[18]. 药物分子和生物大分子间比较常见的一种分子间作用力就是氢键,它能够增加化合物的活性.二芳基苯胺衍生物苯基上的推电子基能提供孤对电子,它与LYS101氨基酸上的羟基分别形成-NH…OH和-H…OH 两个氢键.
3结论
二芳基苯胺衍生物是一种具有良好外部预测能力和稳定性的强效抗HIV药物.本实验通过三维定量构效关系研究了药物结构和活性之间的关系,并采用分子对接分析了二芳基苯胺衍生物与HIV-1逆转录酶之间的相互作用.对接结果表明小分子与大分子的氨基酸残基LYS101可以形成氢键.这些研究结果可以引导人们从多种角度设计合成有效的抗艾滋病药物.
参考文献
[1] Fatemi M H,Heidari A,Gharaghani S.QSAR prediction of HIV-1 protease inhibitory activities using docking derive d molecular descriptors[J].J Theor Biol,2015,369:13-22.
[2] International C.Global report:UNAIDS report on the global AIDS epidemic 2013[R].Geneva:UNAIDS,2013.
[3] Corbett J W,Ko S S,Rodgers J D,et al.Inhibition of clinically relevant mutant variants of HIV-1 by quinazolinone non-nucleoside reverse transcriptase inhibitors[J].J Med Chem,2000,43(10):193-195.
[4] Liang Y H,He Q Q,Zeng Z S,et al.Hybrid diarylbenzopyrimidine non-nucleoside reverse transcriptase inhibitors as promising new leads for improved anti-HIV-1 chemotherapy[J].Bioorg Med Chem,2010,18(13):4 601-4 605.
[5] Zeng Z S,He Q Q,Liang Y H,et al.Hybrid diarylbenzopyrimidine non-nucleoside reverse transcriptase inhibitors as promising new leads for improved anti-HIV-1 chemotherapy[J].Bioorg Med Chem,2010,18(14):5 039-5 047.
[6] Qin X,Jiang H,Lu X,et al.Diarylaniline derivatives as a distinct class of HIV-1 non-nucleoside reverse transcriptase inhibitors[J].J Med Chem,2010,53(13):4 906-4 916.
[7] Morris G M,Goodsell D S,Halliday R S,et al.Automated docking using a lamarckian genetic algorithm and an empirical binding free energy function[J].Comput. Chem,1998,19(14):1 639-1 662.
[8] Bhadoriya K S,Sharma M C,Jain S V,et al.2,4-dihydropyrano[2,3-c] pyrazole:Discovery of new lead as through pharmacophore modeling,atom-based 3D-QSAR,virtual screening and docking strategies for improved anti-HIV-1 chemotherapy[J].Journal of Taibah University for Science,2015,44(4):521-530.
[9] Damme S V,Bultinck P.3D-QSAR based on conceptual DFT molecular fields:Antituberculotic activity[J].Journal of Molecular Structure:Theochem,2010,943(1):83-89.
[10] Levitt M.Protein folding by restrained energy minimization and molecular dynamics[J].J.Mol.Biol,1983,170(3):723-764.
[11] Levitt M,Perutz M F.Aromatic rings act as hydrogen bond acceptors[J].J.Mol.Biol,1988,201(4):751-754.
[12] Hahn M.Receptor surface models.1.definition and construction[J].J.Med.Chem,1995,38(12):2 080-2 090.
[13] Hasel W,Hendrikson T F,Still W C.A rapid approximation to the solvent accessible surface areas of atoms[J].Tetrahed.Comp.Method,1988,1(2):103-116.
[14] Pei J,Wang Q,Zhou J,et al.Estimating protein-ligand binding free energy:Atomic solvation parameters for partition coefficient and solvation free energy calculation[J].J.Proteins:Struct.Funct.Genet,2004,57(40):651-664.
[15] Huey R,Morris G M,Olson A J.Software news and update a semiempirical free energy force field with charge-based desolvation[J].J Comput Chem,2007,28(6):1 145-1 152.
[16] Das K.,Clark A D.,Lewi P J.,et al.Roles of conformational and positional adaptability in structure-based design of TMC125-R165335 (Etravirine) and related non-nucleoside reverse transcriptase Inhibitors that are highly potent and effective against wild-type and drug-resistant HIV-1 variants[J].J.Med.Chem,2004,47:2 550-2 560.
[17] 李月婷,刘永澜,史博智,等.分子对接技术用于马来酰亚胺类GSK-3α抑制剂的作用特征分析[J].分子科学学报,2013,29(4):265-275.
[18] 孙苗,李泽生,郑清川,等.细胞色素P450 2f1(CYP2f1)的三维结构模建和与维甲酸的对接研究[J].分子科学学报,2005,21(2):6-10.
【责任编辑:晏如松】
Study on 3D-QSAR and molecular docking of HIV-1 reverse transcriptase inhibitors
TONG Jian-Bo, WU Ying-ji, BAI Min
(College of Chemistry and Chemical Engineering, Shaanxi University of Science & Technology, Xi′an 710021, China)
Abstract:In this study, using molecular docking and three-dimensional quantitative structure-activity relationship as a method to study the interaction between a series of diaryl aniline analogues and HIV-1 non nucleoside reverse transcriptase,through the establishment of three-dimensional quantitative structure-activity relationship model and multiple linear regression (MLR) model study the relationship between structure and biological activity of the drug moleculesl.The correlation coefficient (R(cum)) of MLR is 0.949,the correlation coefficient (Q(CV)) is 0.799,the results show that the three-dimensional quantitative structure-activity relationship has good prediction ability for the anti HIV activity of the compound.The binding model of small molecule drugs and macromolecular HIV-1 reverse transcriptase active amino acid residues was studied by molecular docking.It is a good guide for the design and synthesis of new anti AIDS drugs.
Key words:HIV-1 reverse transcriptase; three-dimensional quantitative structure-activity relationship; multiple linear regression; molecular docking
中图分类号:O6-04
文献标志码:A
文章编号:1000-5811(2016)02-0134-05
作者简介:仝建波(1975-),男 ,山西怀仁人,副教授,博士,研究方向:计算机辅助药物设计、相关化学信息与计量学
基金项目:国家自然科学基金项目 (21475081)
收稿日期:2015-11-14

