伞裙追寄蝇不同地理种群遗传多样性的ISSR分析
王梦圆, 韩海斌, 王惠萍, 岳方正, 丛靖宇,刘爱萍*
(1. 内蒙古农业大学,呼和浩特 010010;2. 中国农业科学院草原研究所,呼和浩特 010010;3. 锡林郭勒盟太仆寺旗草原站,内蒙古宝昌 027000)

伞裙追寄蝇不同地理种群遗传多样性的ISSR分析
王梦圆1,2, 韩海斌2, 王惠萍3, 岳方正2, 丛靖宇1*,刘爱萍2*
(1. 内蒙古农业大学,呼和浩特 010010;2. 中国农业科学院草原研究所,呼和浩特 010010;3. 锡林郭勒盟太仆寺旗草原站,内蒙古宝昌 027000)
为从分子水平探索不同地区伞裙追寄蝇种群间的内在联系,本文利用ISSR分子标记技术对8个不同地区伞裙追寄蝇自然种群进行遗传多样性、种群间分化程度以及聚类分析等研究。结果表明:筛选出11对多态性稳定的ISSR引物,对8个地区伞裙追寄蝇群体的80个个体进行PCR扩增,共获得166个重复性好且清晰可辨的ISSR条带,平均每条引物扩增出15.0909个片段,且均为多态性条带,多态信息含量(PIC)为0.8441-0.8653;香农信息指数(I)为0.1240-0.3455;Nei’s遗传多样性指数(H)为0.0841-0.2285;基因分化率(Gst)为28.78%,基因流(Nm)的均值为1.5702,即遗传变异主要存在于个体之间,不同种群间基因交流处于中等水平;8个地区伞裙追寄蝇种群被划分为4个类群,种群间的遗传分化与地理距离呈正相关关系。
伞裙追寄蝇;地理种群;ISSR;遗传多样性;遗传分化
伞裙追寄蝇Exoristacivilis属双翅目Diptera寄蝇科 Tachinidae,是多主寄生性天敌昆虫(相红燕等,2012)。近年来,因其对多种草地害虫,如小地老虎Agrotisypsilon、棉铃虫Helicoverpaarmigera、粘虫Mythimnaseparata和银纹夜蛾Argyrogrammaagnata等有控制作用而逐渐引起重视,尤其是对草地螟LoxostegesticticalisL.等间歇性大发生的害虫有很好的防控作用(王建梅等,2013)。由于伞裙追寄蝇的控害作用近几年才被人们发现,故对伞裙追寄蝇的研究主要集中在其生物学特性等方面(相红燕等,2013),对不同地理种群伞裙追寄蝇基因水平的研究国内外尚无报道。
简单重复序列间区(inter-simple sequence repeat, ISSR),被认为是目前最简便有效的分子标记技术之一,是基于SSR标记开发的一种分子标记方法(Ziekienlczetal.,1994)。因其操作简单、多态性丰富、重复性好等优点(Guietal.,2008),被广泛的应用到遗传多样性(梁红蕾等,2012;宋忠魁等,2012;朱勋等,2012)、亲缘关系(贺学勤等,2005)、种质资源鉴定(Ammirajuetal.,2001;侯永翠等,2005;卢家仕等,2013)等研究当中。近年来ISSR技术主要应用于植物领域(Bornetetal.,2004;Li Hetal.,2009;Christopoulosetal.,2010;李娜,2014;周兰英,2014),在动物、昆虫遗传结构研究方面也已有一些报道(余德亿等,2011;林杰君等,2012)。本文首次采用ISSR分子标记技术对不同地区伞裙追寄蝇种群遗传多样性进行了研究,以期从分子水平探索8个不同地区自然种群伞裙追寄蝇的基因交流程度、遗传分化及内在联系,为今后利用伞裙追寄蝇防控害虫提供理论依据。
1 材料与方法
1.1供试虫源
本实验所用伞裙追寄蝇成虫个体采自锡林郭勒盟太仆寺旗(TQ)、新疆哈巴河县(XJ)、河北康保县(KB)、呼和浩特第七牧场(HH)、四子王旗(SQ)、土默特左旗(TZ)、兴安盟代钦塔拉苏木(DS)、兴安盟义和道卜苏木(YS)8个地区的自然种群(表1),采集后的样本经100%乙醇浸泡后放置于-80℃冰箱中保存备用。每个种群分别取10-15头个体进行DNA的提取。
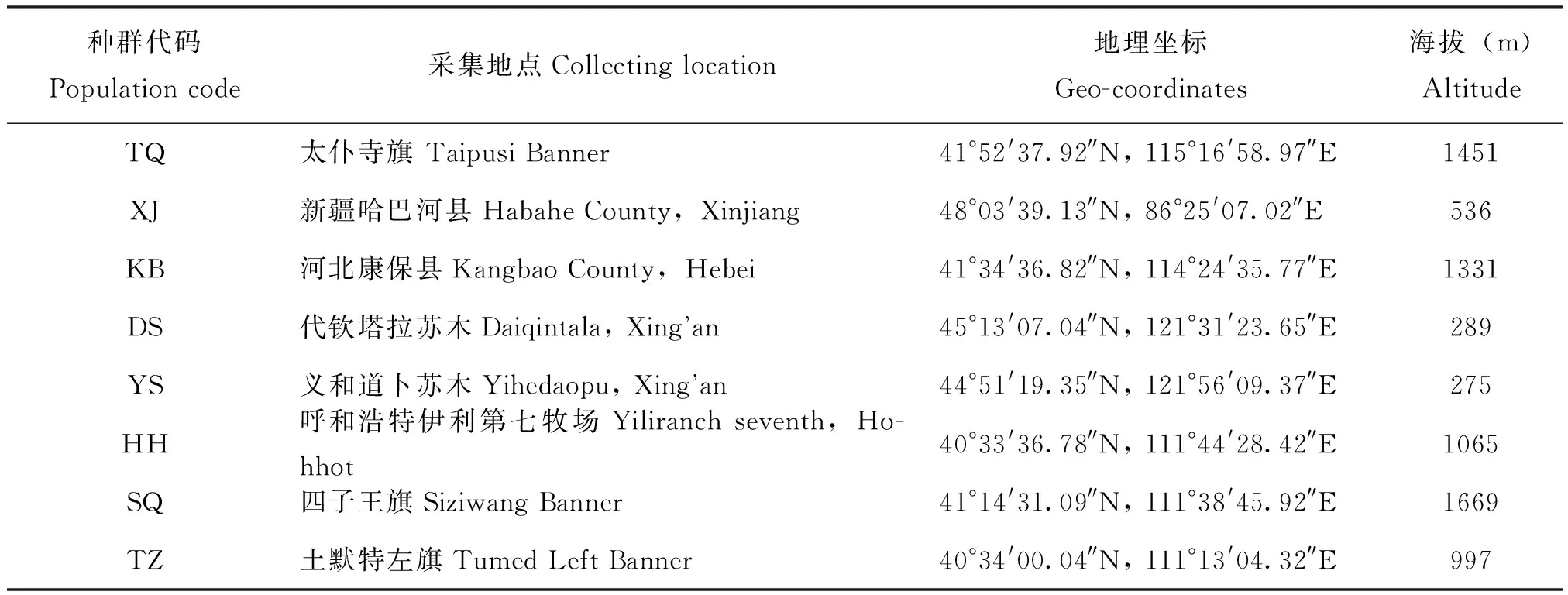
表1 供试伞裙追寄蝇采集信息
注:种群代码全文同。 Note:Population code all same in this paper.
1.2基因组DNA的提取与检测
伞裙追寄蝇虫体用无菌双蒸水冲洗2-3次,晾干。单头伞裙追寄蝇,去头、足、翅于1.5 mL离心管中,加液氮研磨成粉(每个个体单独研磨,不要混用研磨棒)。使用天根dp304动物基因组DNA提取试剂盒对样本进行DNA提取。提取出的基因组DNA用1%的琼脂糖凝胶电泳进行检测,然后置于-20℃冰箱中保存备用。每个种群选取10个DNA样品进行ISSR遗传多样性分析。
1.3ISSR引物的合成与筛选
从加拿大哥伦比亚大学设计并公布的100条通用引物中筛选出扩增条带明亮、清晰、重复性好的引物,用于伞裙追寄蝇遗传多样性的ISSR-PCR扩增反应,引物由上海生工生物工程技术服务有限公司进行合成。
1.4ISSR-PCR扩增体系的优化及产物检测
经预实验,对DNA模板、Mg2+、dNTPs、引物和Taq聚合酶用量进行筛选,得到最适反应体系(20 μL)为:10×PCR Buffer 2.0 μL,DNA模板1.0 μL,Mg2+1.8 μL,dNTPs 1.8 μL,引物1.0 μL,Taq聚合酶0.4 μL,ddH2O补足。经预实验得到最佳的扩增程序为:94℃预变性10 min;94℃变性45 s,54℃退火45 s,72℃延伸2 min,40个循环;72℃补充延伸10 min;最后4℃保存备用。
扩增出的ISSR-PCR产物用1.5%的琼脂糖凝胶在100 V恒压条件下电泳1 h,对扩增产物进行检测。电泳完成后,在紫外凝胶成像仪上对结果进行检测并拍照保存。
1.5数据统计与分析
对电泳图谱中清晰可见的扩增条带进行统计,一些亮度较低、较模糊的条带视为无效的杂带。在同一迁移位置上有清晰可辨的条带时,被认为是同一DNA片段产物,计为“1”,无带的则计为“0”,建立“01”二元数据矩阵,对数据进行分析。
采用群体遗传学分析软件Popgene 32 (Version 1.31)、Excel软件对多态位点百分率(P)、Nei’s遗传多样性指数(H)、香农信息指数(I)、基因分化系数(Gst)、基因流(Nm)、Nei’s遗传相似度(S)、遗传距离(D)和多态信息含量(PIC)进行计算(孙洁茹等,2011)。应用MEGA 4.0软件,通过UPGMA(unweighted pair-group method with arithmetic averages)聚类法构建聚类图。选用TFPGA软件对遗传距离和地理聚类进行mantel测定,其中地理距离由Distance 3.2软件根据经纬度坐标计算得出。
2 结果与分析
2.18个地区伞裙追寄蝇的ISSR-PCR扩增结果
8个地区自然种群伞裙追寄蝇的ISSR-PCR产物部分电泳结果如图1和图2。11对ISSR引物共扩增出166个色彩明亮、清晰可辨的条带,11对引物扩增出的条带数在11-18条,平均为15.0909,其中引物841和809扩增出的条带数最多为18条,引物848扩增出的条带数最少为11条。本实验中11对引物扩增出的166个条带均为多态性条带,平均多态信息含量(PIC)为0.8591,大于0.5,均呈现高度多态性(表2)。
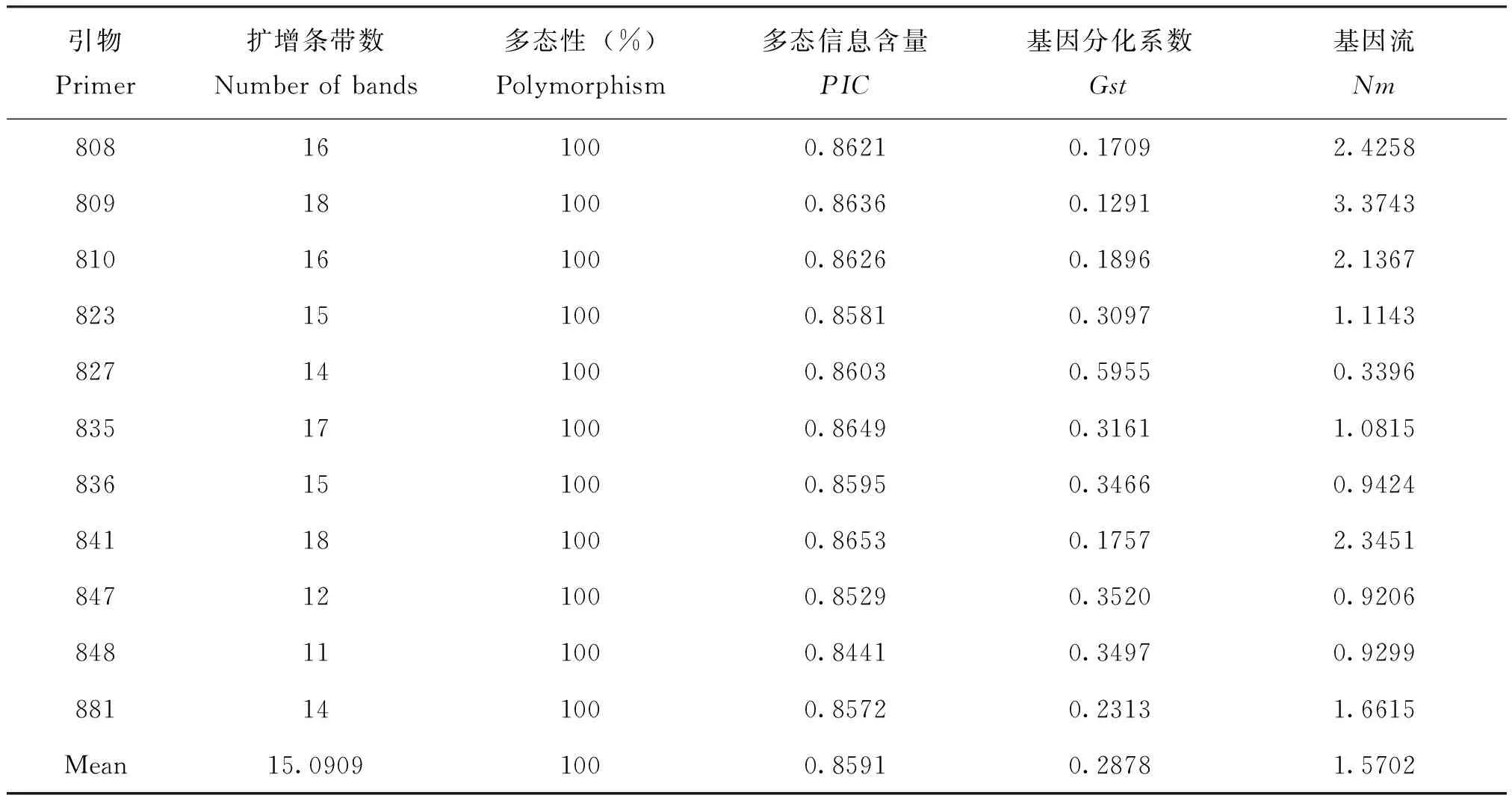
表2 11对ISSR引物对8个伞裙追寄蝇种群扩增信息
图1 引物827对伞裙追寄蝇的ISSR扩增结果
注:M, DNA分子量标准物。图2同。Note:M,DNA molecular weight marker.Some to Pig.2.

图2 引物823对伞裙追寄蝇的ISSR扩增结果
2.2伞裙追寄蝇种群的遗传多样性分析
由表3可知,伞裙追寄蝇8个不同地理种群扩增出的条带范围为75-127条,其中土默特左旗种群(TZ)最多,新疆种群(XJ)最少,均值为107.1250。8个地区伞裙追寄蝇种群扩增条带的多态比例(P)平均为79.85%,其中新疆种群最低为49.33%,多态性条带37条,锡林郭勒盟太仆寺旗种群(TQ)最高为88.39%,多态性条带有99条。8个地区伞裙追寄蝇的香农信息指数(I)为0.1240-0.3455,平均值0.2736。新疆种群最低,土默特左旗种群最高。Nei’s遗传多样性指数(H)在0.0841-0.2285,平均值为0.1814。新疆种群最低,土默特左旗种群最高。以上结果表明:锡林郭勒盟太仆寺旗种群和土默特左旗种群的遗传多样性程度最丰富,而新疆种群的遗传多样性较差。
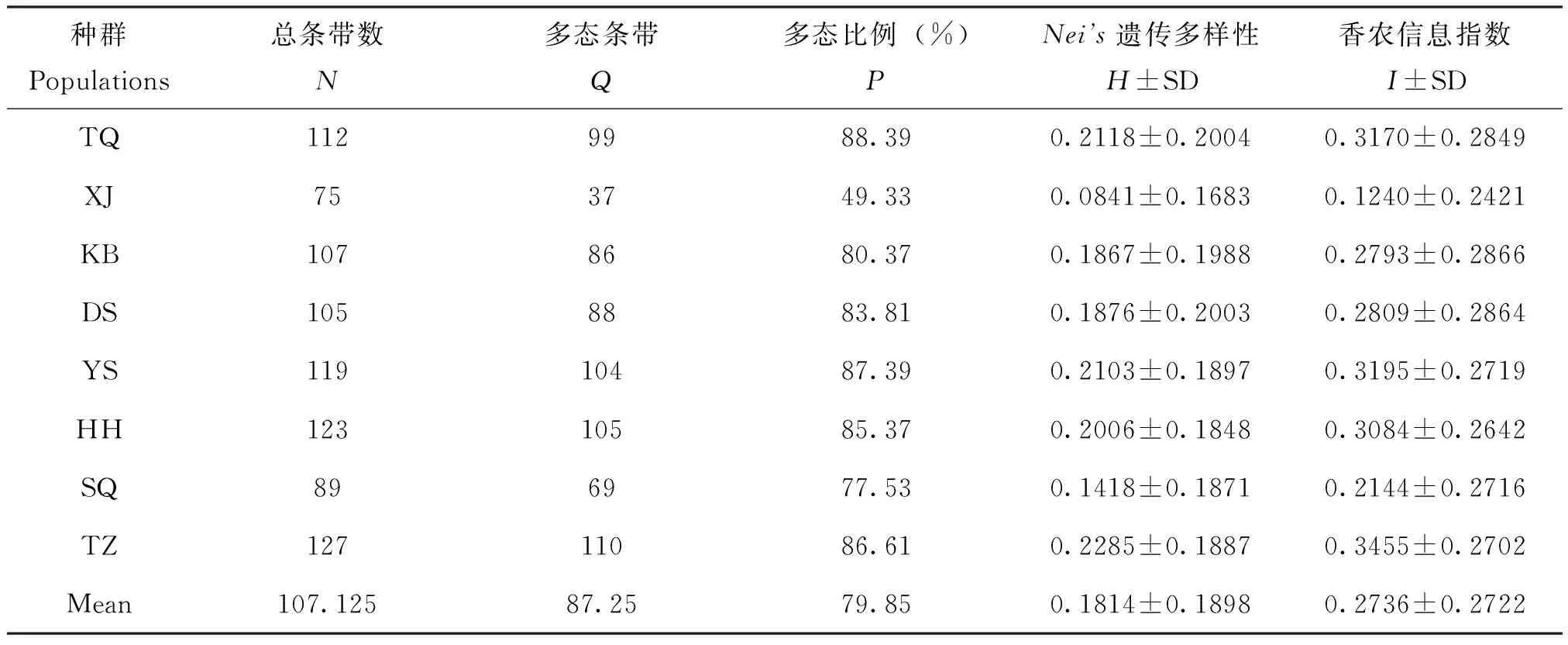
表3 伞裙追寄蝇8个种群的种群内遗传变异统计
注:N,扩增总条带数;Q,多态性条带数;P,多态比例;H,Nei’s遗传多样性;I,香农信息指数;SD,标准差。Note:N, Number of bands;P, Percentage of polymorphism;H,Nei’sgene diversity;I, Shannon’s information index; SD, Standard deviation.
2.3伞裙追寄蝇种群间的遗传分化分析
11对ISSR引物对8个地区伞裙追寄蝇自然种群的扩增中(表2),基因分化系数(Gst)在0.1291-0.5955,均值为0.2878,其中引物827最大,引物809最小。基因流(Nm)平均值为1.5702,引物809最大,为3.3743,引物827最小,为0.3396。对于整个群体而言,平均遗传分化率为28.78%,说明有71.22%的变异来源于个体之间,群体内的变异远高于群体间的变异;根据基因流的大小,种群间基因交流较大(Nm<1)的位点有827、836、847、848,种群间基因交流较小(Nm>4)的位点没有,其余位点在各种群间存在中等程度的分化。
根据11对引物扩增片段大小的统计分析,计算出伞裙追寄蝇8个种群间遗传距离(D)和Nei’s遗传相似度(S),进一步分析了种群间的遗传分化程度(表4)。四子王旗种群(SQ)与土默特左旗种群(TZ)之间的遗传距离最小,为0.0734,新疆种群(XJ)与四子王旗种群之间的遗传距离最大,为0.3556;Nei’s遗传相似度的范围在0.7008-0.9292,其中新疆种群与四子王旗种群之间最小,S为0.7008,四子王旗种群与土默特左旗种群之间最大,S为0.9292。
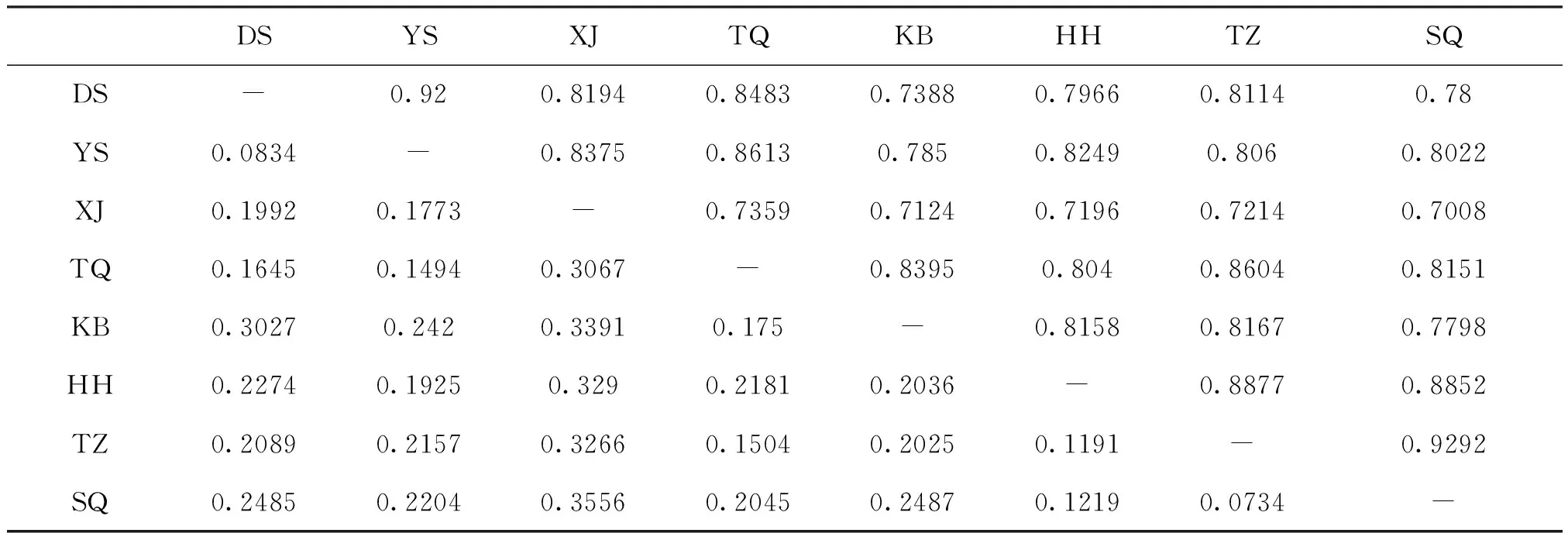
表4 伞裙追寄蝇8个地理种群间的遗传距离和遗传相似度系数
注:对角线上方为遗传相似度系数,对角线下方为遗传距离。Note:Genetic similarity index is above the diagonal, while the genetic distance is below the diagonal.
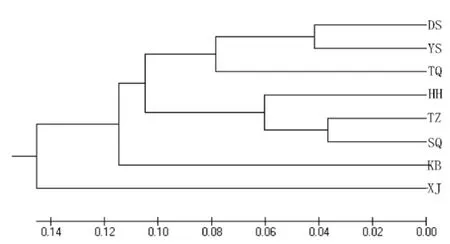
图3 伞裙追寄蝇8个种群间基于Nei’s遗传距离的UPGMA聚类图Fig.3 UPGMA cluster analysis of the 8 Exorista civilis populations generated from the Nei’s genetic distance
2.4伞裙追寄蝇种群间的聚类分析
采用UPGMA方法聚类,对供试的8个不同地理种群伞裙追寄蝇进行聚类分析,得到聚类图(图3)。11对引物对8个地区伞裙追寄蝇种群的聚类分析结果如下:兴安盟代钦塔拉苏木种群、兴安盟义和道卜苏木种群以及锡林郭勒盟太仆寺旗种群聚为第1支;呼和浩特第七牧场种群、四子王旗种群及土默特左旗种群聚为第2支;河北康保县种群单独聚为第3支;新疆哈巴河县种群单独聚为第4支。伞裙追寄蝇8个种群的聚类结果与其自然地理分布呈现出一定的规律性,说明地理距离较近的种群聚为同一组或相邻组,伞裙追寄蝇种群间的遗传距离与其地理距离呈正相关关系。
2.5遗传距离与地理距离的相关性分析
利用TFPGA软件对伞裙追寄蝇8个种群间的遗传距离与相应的地理距离进行Mantel测定(图4),回归方程为y=7410.8x-625.60,相关系数r=0.6292(P=0.0130)。由此可见,伞裙追寄蝇种群间的遗传距离与其地理距离呈显著正相关关系。
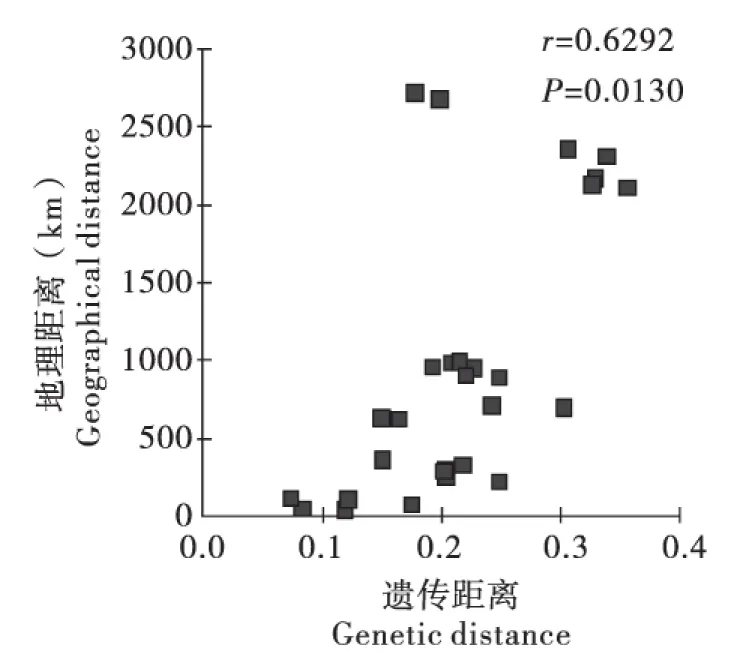
图4 伞裙追寄蝇8个种群的遗传距离与地理距离间的回归分析Fig.4 Regression analysis between genetic distance and geographical distance of the 8 Exorista civilis populations
3 结论与讨论
ISSR分子标记技术在遗传学研究领域发挥着重要作用,在昆虫上的应用多数以农业、蔬菜、瓜果类害虫为主(朱振华等,2005;张长禹,2007;王帅宇,2010;张敏哲等,2013;韩海斌等,2013;陈英才,2014;高立志,2014),但应用ISSR分子标记技术对天敌昆虫遗传多样性研究却少有报道。本研究首次选用ISSR标记的方法对伞裙追寄蝇种群的遗传多样性进行研究,从分子水平上探讨了伞裙追寄蝇不同地理区域自然种群的内在联系,为进一步研究天敌昆虫伞裙追寄蝇提供了理论依据。
多态信息含量(PIC)是衡量遗传多样性较好的指标,其值大小反映基因丰富程度(杨建宝等,2012)。Botstein等(1980)提出,当PIC>0.5时,该基因位点为高度多态性位点。本文检测到11对ISSR引物的PIC值均大于0.5,呈现高度多态性,能为本研究的遗传多样性分析提供充足信息。其中引物841的多态信息含量最高,为0.8653,引物848的多态信息含量最低,为0.8441。说明不同引物对伞裙追寄蝇种群的多态性表现不同,故对不同种群的伞裙追寄蝇群体,选择合适的引物十分重要。
多态位点比例(P)、香农信息指数(I)和Nei’s遗传多样性指数(H)是衡量种群多样性最常用的指标。综合以上3个反映群体遗传多样性的重要参数指标表明,锡林郭勒盟太仆寺旗种群和土默特左旗种群的遗传多样性程度最丰富,而新疆种群的遗传多样性较差。这可能是由于太仆寺旗、土默特左旗两地地势平坦,无高山河流的阻隔,自然种群基因交流频繁,从而导致遗传多样性比较丰富,而新疆哈巴河县的伞裙追寄蝇自然种群由于与其他采集地区相隔较远,基因交流受阻,其遗传多样性较差。
基因分化系数(Gst)是反映群体间遗传分化的重要指标。根据Wright(1978)提出的标准,本研究中Gst平均值为0.2878(Gst>0.15),说明伞裙追寄蝇8个种群的遗传分化较大,群体中有28.78%的变异来自种群间,71.22%的变异来源于群体内,即群体间的变异远低于群体内的变异。基因流(Nm)的基本作用是消弱种群间的遗传差异,故其存在是影响种群间遗传分化的重要因素。本研究中,11对ISSR引物检测到基因流的均值为1.5702(1 群体间的遗传距离揭示了群体遗传分化的程度。本研究中8个种群伞裙追寄蝇的UPGMA聚类分析结果表明,8个地理种群共聚为4支。兴安盟的代钦塔拉苏木种群、义和道卜苏木种群以及锡林郭勒盟太仆寺旗种群聚为第1支;呼和浩特第七牧场种群、四子王旗种群、土默特左旗种群聚为第2支;河北康保县种群、新疆哈巴河县种群分别单独聚为第3支和第4支。其中兴安盟代钦塔拉苏木与义和道卜苏木相邻,为伞裙追寄蝇种群之间的交流提供了便利条件,且相似的生活习惯也使得基因交流变得容易。锡林郭勒盟太仆寺旗种群与以上两地区种群共同聚为一支,可能是农作物在收割过程中夹带被伞裙追寄蝇寄生过的害虫,由于交通贸易与当地的伞裙追寄蝇种群进行基因交流,造成锡林郭勒盟太仆寺旗与兴安盟两地伞裙追寄蝇种群遗传距离较近而共同聚为一类;呼和浩特第七牧场、四子王旗、土默特左旗三地地理距离较近,故三地伞裙追寄蝇种群在基因交流上受到的阻碍较小,基因交流频繁;而新疆地区种群单独聚为一支可能是因为地理距离较远,与其他种群形成较大的地理隔离造成的,并且新疆哈巴河县种群样本采集于贺兰山以西,由于贺兰山的阻隔,在某种程度上起着天然屏障的作用,地理隔离较大,在生物资源方面有一定的差异,总体与其它种群差距较大,故而新疆哈巴河县种群单独聚为一支;河北康保县种群单独聚为一支可能是由于康保县位于阴山山脉以东,内蒙古高原南缘的坝上草原地区,同其他种群的基因交流较少引起的。地形的差异可能是伞裙追寄蝇种群遗传变异的重要因素。Mantel检测结果发现,种群间遗传分化与地理距离呈正相关。地理距离和地形差异是限制其种群间基因交流,导致遗传分化较高的主要原因。说明伞裙追寄蝇不同群体间的地理距离通过影响基因流而形成了现有的遗传结构。综上所述,伞裙追寄蝇8个种群的遗传分化较大,不同种群间基因交流处于中等水平。 由于ISSR分子标记技术不能区分显性纯合基因型和杂合基因型(张民照和康乐,2002),为伞裙追寄蝇种群遗传结构分析所提供的信息量比较有限,加之本文所涉及伞裙追寄蝇的个体数量和种群还不足以覆盖全国,故本文虽为伞裙追寄蝇不同地理种群的遗传多样性提供了有价值的分子生物学依据,但由于技术和采集数量的局限,很难全面反映全国各地伞裙追寄蝇的整体情况,尚需进一步研究。 References) Ammiraju JSS, Dholakia BB, Santra DK. Identification of inter simple sequence repeat (ISSR) markers saaociated with seed size in wheat[J].TheoreticalandAppliedGentics, 2001, 102(5): 726-732. Botstein D, White RL, Skolnick M,etal. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].AmericanJournalofHumanGenetics, 1980, 32(3): 314-331. Bornet B, Antoine E, Bardouil M,etal. ISSR as new markers for genetic characterization and evaluation of relationships among phytoplankton[J].JournalofAppliedPhycology, 2004, 16(4): 285-290. Chen YC. Population Genetic Structure ofBactroceracorrecta(Diptera: Tephritidae) in Main Distributions Areas[D]. China Agricultural University, 2014. [陈英才. 世界主要分布区番石榴果实蝇种群遗传结构研究[D]. 中国农业大学, 2014] Christopoulos MV, Rouskas D, Tsantili E,etal. Germplasm diversity and genetic relationships among walnut (JuglansregiaL.) cultivars and Greek local selections revealed by Inter-Simple Sequence Repeat (ISSR) markers[J].ScientiaHorticulturae, 2010, 125(4): 584-592. Gui FR, Wan FH, Guo JY. Population genetics ofAgeratinaadenophorausing inter-simple sequence repeat (ISSR) molecular markers in China[J].PlantBiosystems, 2008, 142(2): 255-263. Gao LZ. Population Genetic Structure Analysis ofBactroceraminax(Diptera: Tephritidae) in China Inferred from ND4 and Microsatellite Markers[D]. Southwest University, 2014. [高立志. 基于ND4和微卫星标记的中国柑橘大实蝇种群遗传结构分析[D]. 西南大学, 2014] He XQ, Liu QC, Zhai H,etal. The use of RAPD, ISSR and AFLP markers for analyzing genetic relationships amongSweetpotatoCultivarswith known origin[J].ActaAgronomicasinica, 2005, 31(10): 1300-1304. [贺学勤, 刘庆昌, 翟红, 等. 用RAPD、ISSR、和AFLP标记分析系谱关系明确的甘薯品种的亲缘关系[J]. 作物学报, 2005, 31(10): 1300-1304] Hou YC, Yan ZH, Lan XJ,etal. Genetic diversity among barley germplasm with known origins based on the RAMP and ISSR markers[J].ScientiaAgriculturaSinica, 2005, 38(12): 2555-2565. [侯永翠, 颜泽洪, 兰秀锦, 等. 利用RAMP和ISSR标记分析大麦种质资源的遗传多样性[J]. 中国农业科学, 2005, 38(12): 2555-2565] Han HB, Zhou XR, Pang BP,etal. Microsatellite marker analysis of the genetic diversity ofOedaleusasiaticus(Orthoptera: Acrididae) populations in Inner Mongolia, northern China[J].ActaEntomologicaSinica, 2013, 56(1): 79-87. [韩海斌, 周晓榕, 庞保平, 等. 内蒙古亚洲小车蝗种群遗传多样性的微卫星分析[J]. 昆虫学报, 2013, 56(1): 79-87] Liang HL, Bao CH, Jiang YL,etal. Genetic diversities of two geographical populations ofPelodiscussinensisby ISSR[J].JournalofAnhuiAgriculturalUniversity, 2012, 39(1): 31-35. [梁红蕾, 鲍传和, 蒋业林, 等. 中华鳖两个地理种群遗传多样性的ISSR分析[J]. 安徽农业大学学报, 2012, 39(1): 31-35] Lu JS, Bo ZY, Lv WL,etal. ISSR analysis on genetic diversity of germplasms resources inDendrobiumSW. from different habitats[J].ChineseTraditionalandHerbalDrugs, 2013, 44(1): 96-100. [卢家仕, 卜朝阳, 吕维莉, 等. 不同产地石斛属种质资源的ISSR遗传多样性分析[J]. 中草药, 2013, 44(1): 96-100] Li H, Ruan CJ, Silva JAT.Identification and genetic relationship based on ISSR analysis in a germplasm collection of sea buckthorn (Hippophae L.) from China and other countries[J].ScientiaHorticulturae, 2009, 123(2): 263-271. Li N. Using ISSR Marker to Analyze Genetic Resources ofQuercusvariabilis[D]. North West Agriculture and Forestry University, 2014. [李娜. 栓皮栎遗传资源的ISSR分析[D]. 西北农林科技大学, 2014] Lin JJ, Bao YX, Liu J,etal. ISSR marker and its applications in analyzing animal genetic structure[J].ChineseJournalofEcology, 2012, 31(5): 1319-1326. [林杰君, 鲍毅新, 刘军, 等. ISSR分子标记及其在动物遗传结构研究中的应用[J]. 生态学杂志, 2012, 31(5): 1319-1326] Song ZK, Sun FY, Li MY,etal. Genetic diversity of six mud crab (Scyllaparamamosain) populations in Beibu Gulf of South China based on ISSR analysis[J].ChineseJournalofEcology, 2012, 31(10): 2585-2590. [宋忠魁, 孙奉玉, 李梦芸, 等. 北部湾6个拟穴青蟹群体遗传多样性的ISSR分析[J]. 生态学杂志, 2012, 31(10): 2585-2590] Sun JR, Li Y, Yan S,etal. Microsatellite marker analysis of genetic diversity ofCacopsyllachinensis(YangetLi)(Hemiptera: Psyllidae) populations in China[J].ActaEntomologicaSinica, 2011, 54(7): 820-827. [孙洁茹, 李燕, 闫硕, 等. 微卫星标记分析中国梨木虱种群的遗传多样性[J]. 昆虫学报, 2011, 54(7): 820-827] Wang JM, Liu AP, Gao SJ,etal. Parasitic functional response ofExoristacivilisRond. toLoxostegesticticalisL. larvae[J].ChineseJournalofGrassland, 2013, 35(5): 169-172. [王建梅, 刘爱萍, 高书晶, 等. 伞裙追寄蝇对草地螟幼虫的寄生功能反应[J]. 中国草地学报, 2013, 35(5): 169-172] Wang SY. Analysis of Mitochondrial Genome Sequences and Population Differentiation of Several Invasion Leafminers[D]. Chinese Academy of Agricultural Sciences, 2010. [王帅宇. 几种入侵斑潜蝇线粒体全基因组序列分析及种群分化研究[D]. 中国农业科学院, 2010] Wright S. Evolution and the Genetics of Population Variability Within and Among Natural Population[M]. Chicago: University of Chicago Press, 1978. Xiang HY, Liu AP, Gao SJ,etal. Selectivities ofExoristacivilisRond. to different hosts[J].JournalofEnvironmentalEntomology, 2012, 34(3): 333-338. [相红燕, 刘爱萍, 高书晶, 等. 伞裙追寄蝇对不同寄主的选择性[J]. 环境昆虫学报, 2012, 34(3): 333-338] Xiang HY, Liu AP, Gao SJ,etal. A biological study onExoristacivilis, a tachinid parasitoid ofLoxostegesticticalis[J].ActaPrataculturaeSinica, 2013, 22(3): 92-98. [相红燕, 刘爱萍, 高书晶, 等. 草地螟优势寄生性天敌——伞裙追寄蝇生物学特性研究[J]. 草业学报, 2013, 22(3): 92-98] Yu DY, Yao JA, Hu JF,etal. Genomic DNA extraction of three kinds of fruit flies and their ISSR-PCR reaction system establishment[J].JournalofAgriculture, 2011, 1(5): 22-28. [余德亿, 姚锦爱, 胡进锋, 等. 3种实蝇基因组DNA提取及ISSR-PCR反应体系的建立[J]. 农学学报, 2011, 1(5): 22-28] Yang JB, Zhang YP, Su JH,etal. Genetic analysis of culturedSalvelinusfontinalisin China inferred form microsatellites[J].SichuanJournalofZoology, 2012, 31(3): 348-352. [杨建宝, 张艳萍, 苏军虎, 等. 引进美洲红点鲑群体遗传多样性微卫星的分析[J]. 四川动物, 2012, 31(3): 348-352] Ziekienlcz E, Rafashl A, Labuda D. Genome fingerprinting by simple sequence repeats (SSR) anchored polymerase chain reaction amplification[J].Genomics, 1994, 20(2): 176-183. Zhu X, Yang JQ, Wu QJ,etal. Genetic diversity of different geographical populations ofPlutellaxylostella(Lepidoptera: Plutellidae) from China based on ISSR analysis[J].ActaEntomologicaSinica, 2012, 55(8): 981-987. [朱勋, 杨家强, 吴青君, 等. 小菜蛾不同地理种群遗传多样性的ISSR标记研究[J]. 昆虫学报, 2012, 55(8): 981-987] Zhou LY. ISSR Analysis on the Diversity of Oil-teaCamelliaGermplasms[D]. Hunan Normal University, 2014. [周兰英. 油茶种质资源多样性的ISSR分析[D]. 湖南师范大学, 2014] Zhang MZ, Pang BP, Zhou XR,etal. Optimization of SSR-PCR reaction system forPararcypleramicropterameridionalis[J].JournalofInnerMongoliaAgriculturalUniversity(Natural Science Edition), 2013, 34(3): 26-31. [张敏哲, 庞保平, 周晓榕, 等. 宽翅曲背蝗SSR反应体系的优化[J]. 内蒙古农业大学学报 (自然科学版), 2013, 34(3): 26-31] Zhu ZH, Ye H, Zhang ZY. Genetic relationships among fourBactroceracucurbitaegeographic populations in Yunnan Province[J].ChineseJournalofAppliedEcology, 2005, 16(10): 1889-1892. [朱振华, 叶辉, 张智英. 云南四个瓜实蝇地理种群的遗传关系分析[J]. 应用生态学报, 2005, 16(10): 1889-1892] Zhang CY. Molecular Phylogenetic Study and Molecular Identification in Five Species offruitflies(Diptera: Tephritidae)[D]. Huazhong Agricultural University, 2007. [张长禹. 五种实蝇的分子系统发育分析与分子快速鉴定研究[D]. 华中农业大学, 2007] Zhang MZ, Kang L. Amplified fragment length polymorphism (AFLP) and its applications in entomological research[J].ActaEntomologicaSinica, 2002, 45(4): 538-543. [张民照, 康乐. AFLP标记的特点及其在昆虫学研究中的应用[J]. 昆虫学报, 2002, 45(4): 538-543] Genetic diversity ofExoristacivilisfrom different geographical populations based on ISSR marker WANG Meng-Yuan1,2, HAN Hai-Bin2, WANG Hui-Ping3,YUE Fang-Zheng2,CONG Jing-Yu1*, LIU Ai-Ping2* (1.Inner Mongolia Agricultural University, Hohhot 010010, China; 2. Grassland Research Institute of Chinese Academy of Agricultural Sciences, Hohhot 010010, China; 3. Taipusi Banner Grassland Station, Baochang 027000, Inner Mongolian Autonomous Region, China) In order to explore the genetic link of the different geographic populations ofExoristacivilisat the molecular level, the molecular marker technique based on inter-simple sequence repeat (ISSR) was used to assess the genetic diversity, population differentiation and cluster analysis in natural populations ofE.civilisfrom eight different regions. The results showed that eleven primers with stable polymorphic were sieved from primers of ISSR and a total of 166 clear ISSR bands with high repeatable were amplified from the 80 individuals, on average, each primer amplified segments were 15.0909, and these were all the polymorphic bands, Polymorphism Information Content(PIC)of per locus was 0.8441-0.8653; the Shannon’s Information Index(I)and theNei’sGene Diversities Index(H)were 0.1240-0.3455 and 0.0841-0.2285, respectively; Coefficient of Gene Differentiation (Gst) was 28.78%, the index of Gene Flow (Nm) an average of 1.5702, that is, genetic variability mainly exist in individual within population, and moderate gene flow among the different popilations;E.civilisindividuals from eight different habitats natural populations were divided into four groups and between the genetic differentiation of interspecific and geographical distance shows a positive correlation. Exoristacivilis; geographical population; ISSR; genetic diversity; genetic differentiation 公益性行业项目(201103002);“十二五”国家科技支撑计划项目(2012BAD13B07);农业部“948”项目(2011-G4) 王梦圆, 女,1989年生,内蒙古锡林郭勒盟人,硕士,研究方向为生物化学与分子生物学,E-mail:wmy19891012@126.com Author for corresponding,E-mail:congjingyuwyh@163.com;E-mail:liuaiping806@sohu.com 2015-10-15; 接受日期 Accepted: 2015-11-12 Q968.1;S433.5 A 1674-0858(2016)04-0805-08

